惩罚广义线性模型在遗传关联研究中的应用及R软件实现*
广东药学院公共卫生学院流行病与卫生统计学系(510310)
张俊国 刘 丽 李丽霞 张 敏 郜艳晖△
惩罚广义线性模型在遗传关联研究中的应用及R软件实现*
广东药学院公共卫生学院流行病与卫生统计学系(510310)
张俊国刘丽李丽霞张敏郜艳晖△
【提要】目的遗传关联研究中高维数据与日俱增。本文探讨基于岭估计、LASSO和弹性网的广义线性模型在遗传关联研究的应用及软件实现,为高维关联分析提供方法学参考。方法介绍惩罚广义线性模型原理及软件实现方法,并采用模拟的连锁平衡和连锁不平衡的SNPs关联研究数据,以惩罚logistic模型例证R软件glmnet包对广义线性模型的拟合。结果对连锁平衡和连锁不平衡SNPs模拟数据,LASSO与弹性网均给出稀疏解,较好地选择有关联SNPs而剔除无关联变量;而岭估计把所有变量都保留在模型中,模型复杂度高但相应的解释度未增加。结论LASSO和弹性网可对高维遗传关联数据进行有效降维,筛选变量的同时提供参数估计,从而降低模型的复杂度。R软件的glmnet包灵活拟合各类惩罚广义线性模型,可在高通量遗传关联分析中推广应用。
惩罚广义线性模型遗传关联研究LASSO弹性网glmnet包
近年来随着高通量测序技术在遗传流行病学研究中的应用,大量的高维遗传关联数据不断涌现,如动辄成百上千甚至数以万计的SNPs(single nucleotide polymorphisms)数据呈现高维度的特征,给遗传关联统计分析带来极大挑战[1]。在许多情况下,研究中响应变量的分布非正态,且与高维自变量的关系也非线性,因此统计分析可在广义线性模型(generalized linear model,GLM)框架下实现[2]。广义线性模型是经典线性模型的自然推广,包含有一些极具实用价值的模型,如被学者广为熟知的logistic回归、计数资料中发挥重要作用的Poisson回归等。在高维数据中运用广义线性模型,其首要任务是降维,去除数据中的冗余信息或综合数据中相关信息以减少自变量的个数[3];而如何从这些高维数据中攫取有价值的信息,即变量选择显得尤为重要。
传统广义线性模型通过对数似然函数进行参数估计,系数估计值一般非零,自变量多时模型复杂度大大提高,而存在多重共线性时参数估计更易于失效。从模型简洁度和解释力的角度看,研究者希望从大量的预测变量中挑选尽量小的子集,用尽可能简单的模型获得最好的解释效果[4]。针对传统参数估计的缺陷,1970年Hoerl和Kennard提出岭估计,在似然函数基础上对系数平方进行惩罚(称L2惩罚),可在一定程度上解决共线性问题来提高模型的精确度[5];但是它将系数压缩趋于0却不为0,因此不能给出简单且解释性强的模型。1996年Tibshirani提出称之为LASSO(least absolute shrinkage and selection operator)的变量选择方法,用模型系数的绝对值函数作为惩罚来压缩模型(称L1惩罚),使绝对值较小的系数自动压缩为0,同时实现变量选择和参数估计[6]。然而在变量数p远远大于观测值n的情形下,LASSO最多只能选择n个变量,导致模型过于稀疏。因此Zou等学者在2005年提出将岭估计和LASSO凸结合的惩罚方法,称为“弹性网(elastic net)”,可有效解决该问题[7]。目前惩罚广义线性模型可在R软件中实现,运用Glmnet包可有效、灵活地拟合各类惩罚模型,为高维数据提供了有力的分析工具。本文采用模拟的SNPs关联研究数据,以惩罚logistic模型例证glmnet包对广义线性模型的拟合。
模型原理和方法
假定观测值xi∈Rp,因变量yi∈R,i=1,…,N,并假定因变量取值为1和0,构建logistic模型为:
(1)
式(1)通常采用最大似然估计。而惩罚logistic回归是在对数似然函数的基础上,加入惩罚项[8],通过最小化惩罚目标函数得到参数估计值,即:

(2)
研究表明,如果自变量间存在相关,岭估计倾向于同时压缩相关自变量的系数,而LASSO倾向于仅选择相关自变量中的一个,弹性网则是岭估计和LASSO的有效结合。设置α=0.5将倾向于将相关变量同时纳入或同时排除,当α=1-ε(ε为虽小却大于0的数值)时,会让弹性网模型执行起来更倾向于LASSO,可以排除许多存在高度相关的变量。
惩罚logistic回归的算法多采用坐标下降法(coordinate descent),这是目前关于LASSO的最快计算方法[9]。算法原理为对每一个参数在保持其他参数固定的情况下进行优化循环,直到系数稳定为止。计算在λ的格点值上进行,而λ的确定可以通过k折交叉验证(k-fold cross-validation)得出。
R软件glmnet包的基本语法
R软件中可使用glmnet包实现惩罚广义线性模型[8]。glmnet能够处理各种数据类型,拟合线性、多项式、logistic和Poisson回归等广义线性模型。它的正则化路径是在λ的格点值上计算LASSO或者弹性网的惩罚项。主要函数为:
glmnet(x,y,family=c(“gaussian”,“binomial”,“Poisson”,“multinomial”,“mgaussian”),alpha=1,…)
上述语句可拟合广义线性模型,得到正则化的路径;其中,x为自变量矩阵,y为因变量,family中指定因变量的分布类型,默认各类分布的典型链接函数,如上述选择项分别对应正态分布(恒等链接),二项分布(logit链接),Poisson分布(log链接),多项分布(累积logit链接)和多元正态分布(恒等链接);alpha是弹性网的混合参数,取值范围为[0,1]。当设置alpha=1时拟合LASSO(缺省值),alpha=0时拟合岭估计;在(0,1)区间内拟合弹性网。实际应用中,alpha取值的选择也可借助cv.glmnet函数,通过设定不同的alpha值进行交叉验证:
cv.glmnet(x,y,nfolds,alpha,…)
上述函数可完成模型的K折交叉验证,除选择alpha值外,还主要用于确定模型的调整参数λ。其中x和y意义同前,nfolds指定交叉验证的K值,表示将所有观测随机分为K份,循环采用K-1份作为训练样本,剩余1份作为验证样本,默认值为10,最小可设定为3。
模型拟合后,可用coef和predict函数显示惩罚模型的参数估计值及据此模型进行预测:
coef(object,s=c(“lambda.1se”,“lambda.min”)or null,…)
predict(object,newx,s=c(“lambda.1se”,“lambda.min”)or null,…)
上述函数中,如对参数s进行设定,object定义为cv.glmnet交叉验证选择的模型,否则为glmnet估计的所有模型;s中lambda.min为交叉验证结果中平均误差最小的模型对应的值,lambda.1se则为最小平均误差1倍标准误内对应λ中的最大值;一般而言,定义Lambda.min输出最优模型(平均误差最小),但可能存在模型复杂及过拟合的问题;定义Lambda.1se可输出更简洁的模型,但较最优模型存在相对误差。此外,predict函数中的newx为参与预测的自变量矩阵。
实例分析
为说明惩罚广义线性模型及R软件glmnet包在遗传关联研究中的应用,本文采用病例对照设计的遗传关联研究模拟数据,探讨不同的遗传情景下各类惩罚模型的参数估计效果。数据模拟过程中,设置了连锁不平衡(linkage disequilibrium,LD)参数,以反映群体中不同基因座上等位基因间的关联。在模拟数据中LD定义为高维SNPs间的相关系数,分两种情况:(1)SNPs变异间独立(LD=0);(2)SNPs间高度相关(LD=0.9)。病例组和对照组样本量各为500,为便于说明glmnet包的输出结果,模拟数据集中只包含20个SNPs,并随机抽取其中5个(X1,X4,X5,X12,X15)设置为与因变量有关联的SNPs,剩余15个设置为与因变量无关联的噪声变异;与疾病有关联的变异效应值ORs均设为2。模拟数据在R软件中完成,具体模拟方法参见文献[10]。数据集形式为1000个观测行,22个变量列,如表1所示。
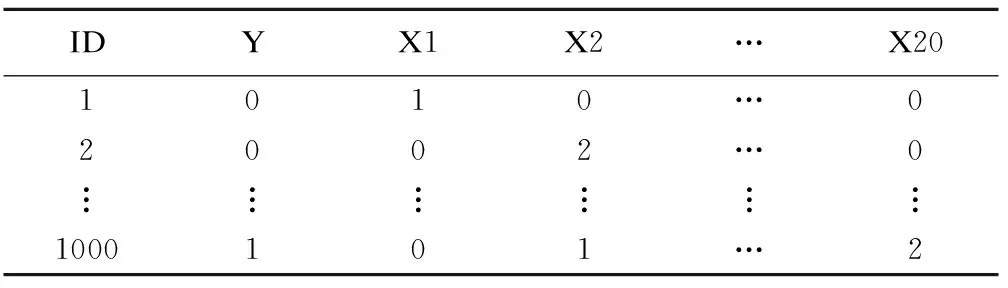
表1 数据集形式
实例分析时,先在R软件中载入glmnet包,读取R软件工作目录的数据,建立自变量和因变量矩阵;设定随机数种子,可以保证结果的唯一性。例如对连锁平衡数据集SAMPLE_LD0.csv拟合惩罚logistic回归模型,具体程序为:
library(glmnet)#载入glmnet包
linkage<-read.csv(“SAMPLE_LD0.csv”)#读入数据
linkage.x<-as.matrix(linkage[,3:22])#生成自变量矩阵
linkage.y<-as.matrix(linkage[,2])#生成因变量矩阵
set.seed(2015)#设定随机数种子
link_glm<-glmnet(linkage.x,linkage.y,family=“binomial”)#拟合LASSO模型
plot(link_glm)#绘制系数正则化的路径图
由于因变量为二分类,故在glmnet函数的family选项中选择binomial,alpha缺省时为1,拟合基于LASSO的惩罚logistic回归;如定义为0或(0,1)之间的数值,则拟合基于岭估计和弹性网的惩罚logistic回归。Link_glm中包含不同取值时的所有模型。图1为alpha=1,0和0.5时分别拟合LASSO,岭估计和弹性网系数惩罚的收缩情况;其中横坐标为L1正则化,即系数的绝对值之和,又称曼哈顿距离;纵坐标为系数,上方数字是模型中保留的变量个数,图中可看到系数在正则化路径上的选择。此例中LASSO和弹性网压缩过程相似,可将部分系数连续压缩为0,从而实现变量选择的目的,而岭估计压缩系数的过程中仍保留所有变量在模型中。
为选择适宜的λ值,使用cv.glmnet函数,通过交叉验证观察不同λ取值时的模型误差,具体程序为:
link_cv <-cv.glmnet(linkage.x,linkage.y,nfolds=10,family=“binomial”)#10折交叉验证
plot(link_cv)#绘制交叉验证曲线图
link_cv$lambda.min#最小误差模型的lambda
link_cv$lambda.1se#1倍SE内的最大lambda
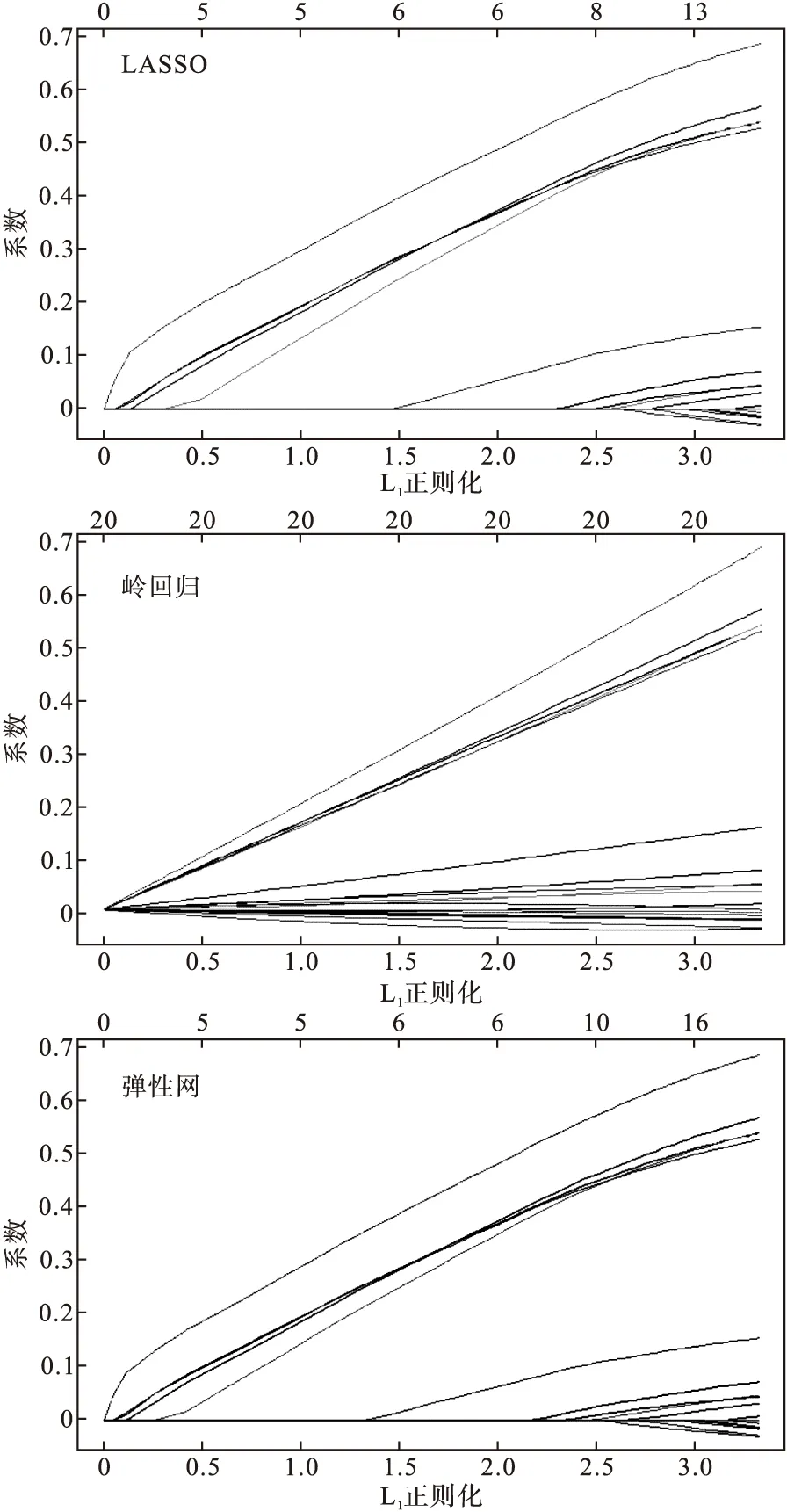
图1 参数估计变化趋势图
图2为分别拟合LASSO,岭估计和弹性网时取不同log(λ)时模型误差的变化趋势。其中左侧虚线对应平均误差最小时(最优模型)的log(λ)(lambda.min),右侧另一条虚线是其1倍SE时对应的log(λ)(lambda.1se),对应更简洁的模型;图形上方数字为模型对应的变量个数。本例中LASSO和弹性网各模型平均误差接近,而岭估计较高。如基于LASSO惩罚回归,由于lambda.min和lambda.1se 对应的模型误差变化不大,更偏好简洁模型时,选择lambda.1se对应的模型,此时λ值为0.0255,交叉验证模型的平均误差为1.3558。
选择适宜的λ后,可提取最终模型参数估计的结果,包括回归系数(beta),自由度(df),λ和决定系数R2(dev.ratio)等;其中决定系数是评判自变量对因变量解释程度的重要指标,可进行模型之间的比较,具体程序为:
link_cv.best <-link_cv$glmnet.fit#提取最佳模型
link_cv.coef <-coef(link_cv$glmnet.fit,s=link_cv$lambda.1se)#提取模型系数
link_cv.coef #输出模型系数
R<-link_cv.best$dev.ratio[which(link_cv.best$lambda==link_cv$lambda.1se)]#输出R2

图2 交叉验证λ和模型误差的关系
根据以上的语句,可输出模型最终参数估计结果及R2。本例中,调整glmnet函数和cv.glmnet函数内的alpha值依次为1,0.5,0,分别对连锁平衡数据和连锁不平衡数据拟合LASSO、弹性网和岭估计,各模型参数估计值及R2值如表2所示。

表2 连锁平衡与连锁不平衡数据拟合模型的参数估计值及R2
由表2可得,本例中,对连锁平衡和连锁不平衡数据,LASSO均给出稀疏解,较好地选择有关联SNPs而剔除无关联变量;除关联SNPs外,弹性网多保留一个无关联SNP;而岭估计因无法把参数压缩为0,所有变量都保留在模型中,模型的复杂度高,但解释度未增加。
讨 论
由于生物学测序技术的迅猛发展,遗传关联数据趋于高维,需要处理的信息量越来越大且存在大量的冗余信息和噪音,选择显著变量、拟合较简单却解释程度好的模型是研究者面临的共同问题,相应的统计分析方法也越来越被关注。基于惩罚的广义线性模型在处理共线性,降维以及变量选择方面有着独特的优势,在各类科研领域尤其是遗传流行病学研究中的应用也随之逐步兴盛起来[3]。本文以遗传关联分析中常用的病例对照设计模拟数据为例,拟合LASSO,岭估计和弹性网三种惩罚模型,运用glmnet包进行关联性SNPs位点选择,说明惩罚模型在遗传关联分析中应用的可行性及科学性。
实例分析可见,岭估计能够有效克服自变量间的高度相关,得到稳定的模型,但是它没有稀疏性,不能有效地筛选自变量;但有研究表明,在岭估计基础上引入“Boosting”后可得到与LASSO同样的估计,实现筛选变量的目的[11]。LASSO即L1惩罚可很好地降低维度,轻微解决共线性问题,但也有研究表明,LASSO有时可能过度压缩参数[6]。弹性网对系数的惩罚介于岭估计与LASSO之间,在本例结果中的表现更接近LASSO,且和LASSO一样能同时进行模型选择和参数估计。以往研究显示,弹性网一般不会过度压缩参数,能有效处理组群效应(group effect),特别是能直接处理变量数远远大于样本数的问题,这些是LASSO所不能达到的[12]。因此在实际分析中,还需根据具体情况选择适宜的方法,如自变量弱共线时可考虑LASSO,强共线、高维小样本数据时倾向于弹性网。本研究为便于说明R软件结果,只采用了单个模拟数据集,无法体现惩罚模型更进一步的性质,如不同样本量和连锁不平衡参数时参数估计的准确度、混杂无关联变异比例对变量选择的影响等;更深入研究和比较各种惩罚方法,还需设置更多遗传情境的模拟研究和实例应用。
glmnet包作为R数据挖掘中的三驾马车之一,过程灵活,功能性强,可以拟合各类基于惩罚的广义线性模型,通过最小化惩罚目标函数得到参数估计值。但是与一般的广义线性模型不同,glmnet包无法直接计算Fisher信息阵进而估计参数的方差。有研究者提出可采用bootstrap方法计算估计值的标准误[6],或采用Bayesian LASSO获得有效的β及标准误[13]。还有学者提出可在似然比中设定“三明治”(sandwich formula)用于协方差估计[14],这些方法及应用程序将进一步补充和完善glmnet包的功能。此外,在研究SNPs与疾病预后常用的生存时间资料时,glmnet包还可拟合基于三种惩罚的Cox模型。除此之外,R中还有许多其他拟合LASSO的相关软件包:如较早时基于最小角回归拟合传统线性模型的lars;运用同伦算法拟合广义线性模型的LASSO 2;还有和glmnet类似的penalized,也可选择L1,L2或弹性网惩罚拟合各种广义线性模型;而基于LARS-EN算法的elasticnet可以进行弹性网惩罚,能够估计稀疏主成分。实际应用中,研究者可结合数据特征和实际需求加以选择。
总之,运用R对高维数据建模时,惩罚广义线性模型克服了传统线性模型存在的预测精度和模型解释度较差的缺陷,基于惩罚的变量选择方法能够在选择变量的同时得到参数估计,加之计算量小,因此在处理高维数据时,有着传统变量选择方法无可比拟的优越性[15]。相信随着惩罚广义线性模型及软件的推广使用,它将在高通量遗传关联分析中发挥极其重要的作用。
[1]Ayers KL,Cordell HJ.Analysis of Genetic Analysis Workshop 18 data with gene-based penalized regression.BioMed Central Ltd,2014,S43.
[2]Zhang Z,Ersoz E,Lai C,et al.Mixed linear model approach adapted for genome-wide association studies.Nature genetics,2010,42(4):355-360.
[3]龚建朝.Lasso及其相关方法在广义线性模型模型选择中的应用.中南大学,2008.
[4]马文浩.各种L_q惩罚在变量选择中的应用及其比较.山东大学,2012.
[5]Hoerl AE,Kennard RW.Ridge Regression:Biased Estimation for Nonorthogonal Problems.Technometrics,1970(12):55-67.
[6]Tibshirani R.Regression shrinkage and selection via the lasso.Journal of the Royal Statistical Society.Series B(Methodological),1996:267-288.
[7]Zou H,Hastie T.Regularization and variable selection via the elastic net.J R Statist,2005(67):301-320.
[8]Friedman J,Hastie T,Simon N,et al.glmnet:Lasso and elastic-net regularized generalized linear models.CRAN,2009.
[9]Friedman J,Hastie T,Tibshirani R.Regularization paths for generalized linear models via coordinate descent.Journal of Statistical Software,2009,33(1):1-22.
[10]梁融.稀有变异关联性分析中折叠与非折叠法的模拟比较研究.广东药学院,2014.
[11]Tutz G,Binder H.Boosting ridge regression.Computational Statistics & Data Analysis,2007,51(12):6044-6059.
[12]Bhlmann P,Sara VDG.Statistics for high-dimensional data:methods,theory and applications.Springer Science & Business Media,2011.
[13]Casella TPG.The Bayesian Lasso.Journal of the American Statistical Association,2008,103(482):681-686.
[14]Fan J,Li R.Variable Selection via Nonconcave Penalized Likelihood and its Oracle Properties.Journal of the American Statistical Association,2001,96(456):1348-1360.
[15]张秀秀,王慧,田双双,等.高维数据回归分析中基于LASSO的自变量选择.中国卫生统计,2013,30(6):922-926.
(责任编辑:邓妍)
Application of Penalized Generalized Linear Model in Genetic Association Study and its Software Implementation in R
Zhang Junguo,Liu Li,Li Lixia,et al
(Department of Epidemiology and Biostatistics,School of Public Health,Guangdong Pharmaceutical University(510310),Guangzhou)
ObjectiveWith the development of high-dimensional data in genetic association studies,this study was aimed to depict the application of penalized generalized linear model based on ridge,LASSO and elastic net in genetic association study and its software implementation.MethodsWe introduced the penalized generalized linear model in detail,and performed the analyses in the simulated SNPs data in linkage equilibrium and linkage disequilibrium,respectively.At last,we used the ‘glmnet’ package in R software to fit the penalized generalized linear model with the example of logistic model.ResultsFor both of the SNPs data in linkage equilibrium and linkage disequilibrium,LASSO and elastic net could worked out sparse solution and effectively detected out the associated SNPs,as well as fairly excluded the non-associated variables.By contrast,the ridge model included all variables,and thus promoted the complexity of model,but meanwhile its explanation was not added.ConclusionFor the high-dimensional association data,LASSO and Elastic net can achieve effective dimensionality reduction,variables selection,and parameters estimation simultaneously,and thus reduce the complexity of the model.R glmnet package can flexibly fit different types of generalized linear model.Therefore,it can be wildly used in high-throughput genetic association studies.
Penalized generalized linear model;Genetic association studies;LASSO;Elastic net;Glmnet package
国家自然科学基金(81302493);广东省科技厅社会发展基金(2014A020212307);广东省自然科学基金(S2013040013590)
郜艳晖,E-mail:gao_yanhui@163.com

