野生玫瑰基因组DNA提取及CDDP引物筛选
姜丽媛+臧德奎+李文清
摘要:野生玫瑰中多糖多酚含量较高,DNA提取较为困难,本试验在植物基因组DNA CTAB提取法的基础上,通过延长水浴时间、调整氯仿-异戊醇(24∶1)抽提次数为2~3次等一系列方法提高野生玫瑰基因组DNA的提取纯度。以2×Es Taq MasterMix(含染料)10 μL,40 ng/μL DNA模板1 μL,10 pmol/μL引物 1 μL, ddH2O 8 μL为CDDP-PCR反应体系,对21条CDDP引物进行多态性筛选。结果表明,用改良的CTAB法提取的野生玫瑰基因组DNA纯度较高;21条CDDP引物中有16条对野生玫瑰品种的鉴别能力较高。本研究为野生玫瑰CDDP分子标记及后续各种分子生物学研究奠定基础。
关键词:野生玫瑰;改良CTAB法;DNA提取; CDDP分子标记
中图分类号:S685.120.1 文献标识号:A 文章编号:1001-4942(2016)11-0013-05
Abstract DNA extraction of Rosa rugosa is difficult for their high contents of polyphenol and polysaccharide. Here a modified CTAB method for Rosa rugosa DNA extraction was presented through extending water-bath time, appropriately adjusting chloroform isoamyl alcohol (24∶1) extraction time to 2~3 times and etc. Twenty-one CDDP polymorphic primers were screened with reaction system containing 10 μL of 2 ×Es Taq MasterMix (including dye), 1 μL of 40 ng/μL DNA template, 1 μL of 10 pmol/μL primer and 8 μL of ddH2O. The results showed that the genomic DNA of Rosa rugosa extracted with modified CTAB method owned high purity,and 16 CDDP primers had higher discriminating ability on Rosa rugosa. This research provided bases for CDDP molecular markers and subsequent molecular biology researches of Rosa rugosa.
Keywords Rosa rugosa; Modified CTAB method; DNA extraction; CDDP molecular marker
野生玫瑰(Rosa rugosa)是国家二级濒危保护植物,分布在我国吉林图们江河口、辽宁南部海岸以及山东东部沿海海岸[1]。野生玫瑰内含有芬香、抗寒、抗旱等性状基因,是栽培玫瑰育种资源的依托,因此深入了解野生玫瑰的遗传变异、保护野生玫瑰资源具有重要意义。
基因组DNA是文库构建、分子标记、基因克隆等分子生物学研究的基本前提,其提取纯度直接影响整个试验的质量。由于不同植物体细胞内各种化学成分含量存在很大的差异,其DNA提取方法可能不同,甚至相同植物不同部位的 DNA 提取策略也不同[2]。
随着以PCR为基础的分子标记技术的迅速发展,分子标记辅助育种成为当前国际植物遗传育种研究的热点[3]。来源保守的DNA序列多态性(conserved DNA-derived polymorphism, CDDP)标记是由Collard和Mackill开发的基于DNA保守序列的新型分子标记[4],它是一种单引物扩增反应,最先应用于水稻,后来迅速在其他植物中得到应用[5]。CDDP分子标记的开发利用受益于植物基因组学和功能基因组学的快速发展,其引物序列的设计是根据植物基因组DNA中起重要作用的保守序列。不同植物间DNA保守序列均相当保守,所以CDDP分子标记技术能在不同物种间通用。在水稻上的研究表明,CDDP分子标记具有操作便捷、应用成本低、多态性丰富、可以有效产生与目标性状有关的标记等优点,是对RAPD、ISSR、TRAP、CoRAP、SCoT等标记方法的有效补充,具有较好的应用前景[6]。目前,CDDP分子标记已经应用于菊花[7]、牡丹[8,9]等多种植物,但尚未见野生玫瑰种质资源上CDDP分子标记应用的报道。
本研究主要着力于优化野生玫瑰基因组DNA的提取方法和在CDDP引物中筛选出对野生玫瑰品种鉴别力较高、多态性丰富的引物,为后续进行野生玫瑰的CDDP分子标记研究奠定基础。
1 材料与方法
1.1 材料来源
以吉林珲春、辽宁庄河以及山东牟平、威海等地的野生玫瑰为研究材料,在2015年5月和7月采集新鲜幼叶,共120个样品,用硅胶保存于-20℃。
1.2 试剂
液氮;PVP;提取介质(4℃保存);2×CTAB提取缓冲液;β-巯基乙醇;Tris-酚;氯仿-异戊醇(24∶1);3 mol/L NaAc;无水乙醇;70%乙醇;1×TE;双蒸水(ddH2O)。
1.3 仪器
电子天平;研钵;研棒;药匙;冰盒;水浴锅;低温离心机;移液枪。
1.4 改良CTAB法提取野生玫瑰基因组DNA
1.4.1 准备工作 ①将水浴锅升温至65℃;②将无水乙醇放入-20℃的冰箱内预冷;③配制提取介质(表1)和2×CTAB提取缓冲液(表2);④将枪头、CTAB提取缓冲液、提取介质灭菌;⑤将药匙放在液氮中预冷。
1.4.2 提取方法 ①用移液枪取1 mL提取介质于2 mL离心管中,置于冰盒内,标好标号。
②用电子天平称取0.12~0.20 g幼嫩叶片于干燥研钵中,在研钵内加入少许PVP,倒入约35 mL液氮,将叶片捣碎,待液氮即将挥发完时,快速研磨叶片,重复加入液氮2~3次,反复研磨直至叶片成细小的粉末状(研磨的越细越好)。用泡在液氮中提前预冷的小匙快速将研钵中的叶片粉末转移至已加好提取介质的离心管内,混匀,冰盒内放置10 min, 7 000 r/min、4℃离心10 min,弃上清液。离心结束时把CTAB放入65℃水浴锅内预热。
③加提取介质,重复上述步骤,离心后弃上清液,向沉淀中加入40 μL β-巯基乙醇、800 μL CTAB,摇离心管至沉淀散开,放入65℃水浴锅内1 h,15 min摇匀一次。
④将离心管从水浴锅中取出,冷却5~6 min至室温,摇匀加入400 μL Tris-酚、400 μL氯仿-异戊醇(24∶1),缓慢摇动10 min,放入低温离心机内,12 000 r/min、4℃离心10 min。
⑤准备新的2 mL离心管,写好编号。从离心机内取出离心管,取上清液800 μL(每次取200 μL ,最多取800 μL,若上清液较少也可以取600 μL)于新的2 mL离心管内,加入等体积的氯仿-异戊醇(24∶1),轻轻缓慢摇动10 min,使其充分混匀至上下不分层。
⑥将离心管放入离心机内,12 000 r/min、4℃离心10 min,离心后观察离心管内中间的蛋白层,若中间蛋白质较多,重复上述步骤(取上清液600 μL左右)。
⑦准备新的1.5 mL离心管,写好编号。用黄色平底枪头取上清液400 μL(每次取100 μL)于1.5 mL离心管中,先加入上清液1/10体积的3 mol/L NaAc试剂,然后加入上清液2倍体积的-20℃无水乙醇,放入冰箱-20℃保存20 min。取出离心管,12 000 r/min、4℃离心3 min。
⑧弃上清液,用1 000 μL的70%乙醇洗涤5 min,重复2~3次,倒出乙醇,放置3~4 h晾干。
⑨用50 μL的1×TE溶解沉淀, 4℃冰箱内保存(-20℃长期保存)。
1.4.3 DNA质量检测 向提取的DNA样品中加入1 μL RNAase,37℃水浴锅内放置1 h,去除DNA内的RNA。
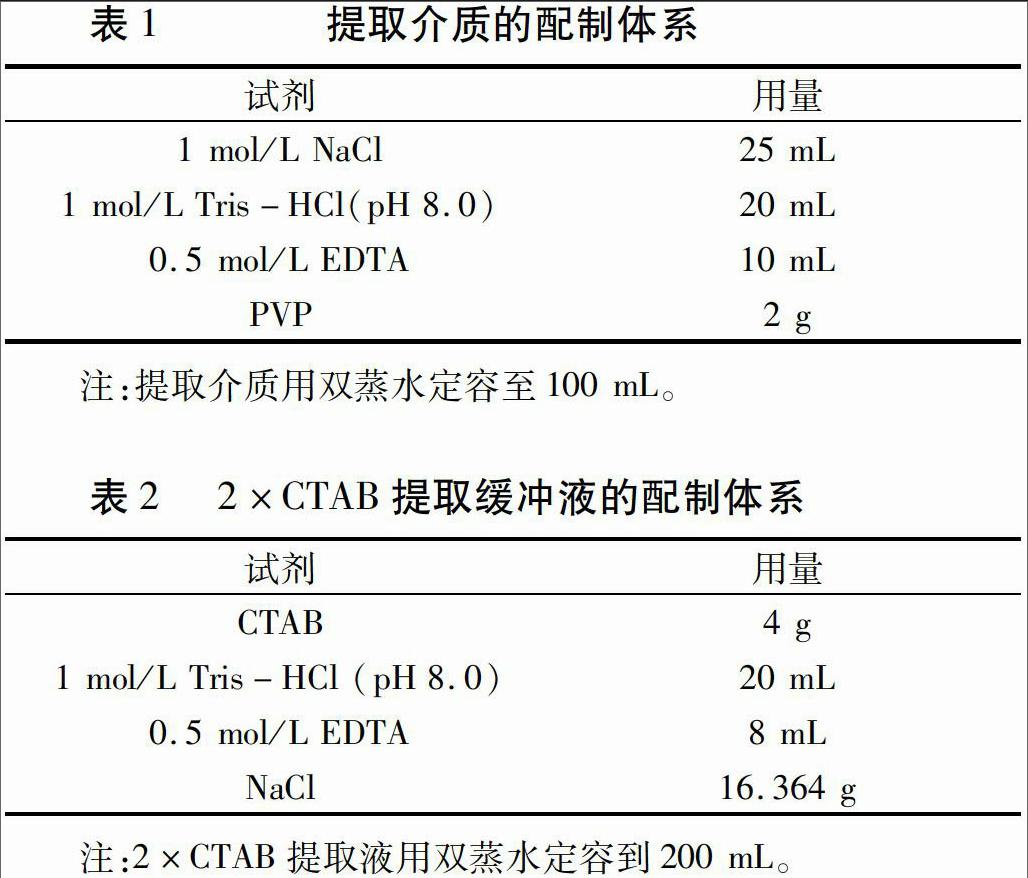
琼脂糖凝胶电泳检测:配制0.8%琼脂糖凝胶,取5 μL提取的DNA样品加入2 μL loading buffer,混匀,点入琼脂糖凝胶内,在120 V电压下电泳30 min,电泳结束后将琼脂糖凝胶放于EB中染色30 min,最后在凝胶成像系统中观察、拍照[10]。
分光光度计检测DNA纯度,高纯度DNA的A260/280值应在1.8~2.0之间,当A260/280小于1.8时,DNA样品中可能存在蛋白质污染,当A260/280大于2.0时,DNA样品中RNA的含量较高[11]。
1.5 CDDP分子标记引物筛选
从吉林珲春、辽宁庄河、山东威海、山东牟平四个地区的高质量野生玫瑰基因组DNA中各挑选一个样品,利用21条CDDP引物对其进行扩增。经多次反应体系试验,确定稳定性良好的PCR扩增体系:2×Es Taq MasterMix(含染料)10 μL,40 ng/μL DNA模板1 μL,10 pmol/μL引物1 μL,ddH2O 8 μL。PCR反应程序设为94℃预变性3 min;94℃变性1 min,50℃退火1 min,72℃延伸2 min,35个循环;72℃后延伸5 min,4℃保存。
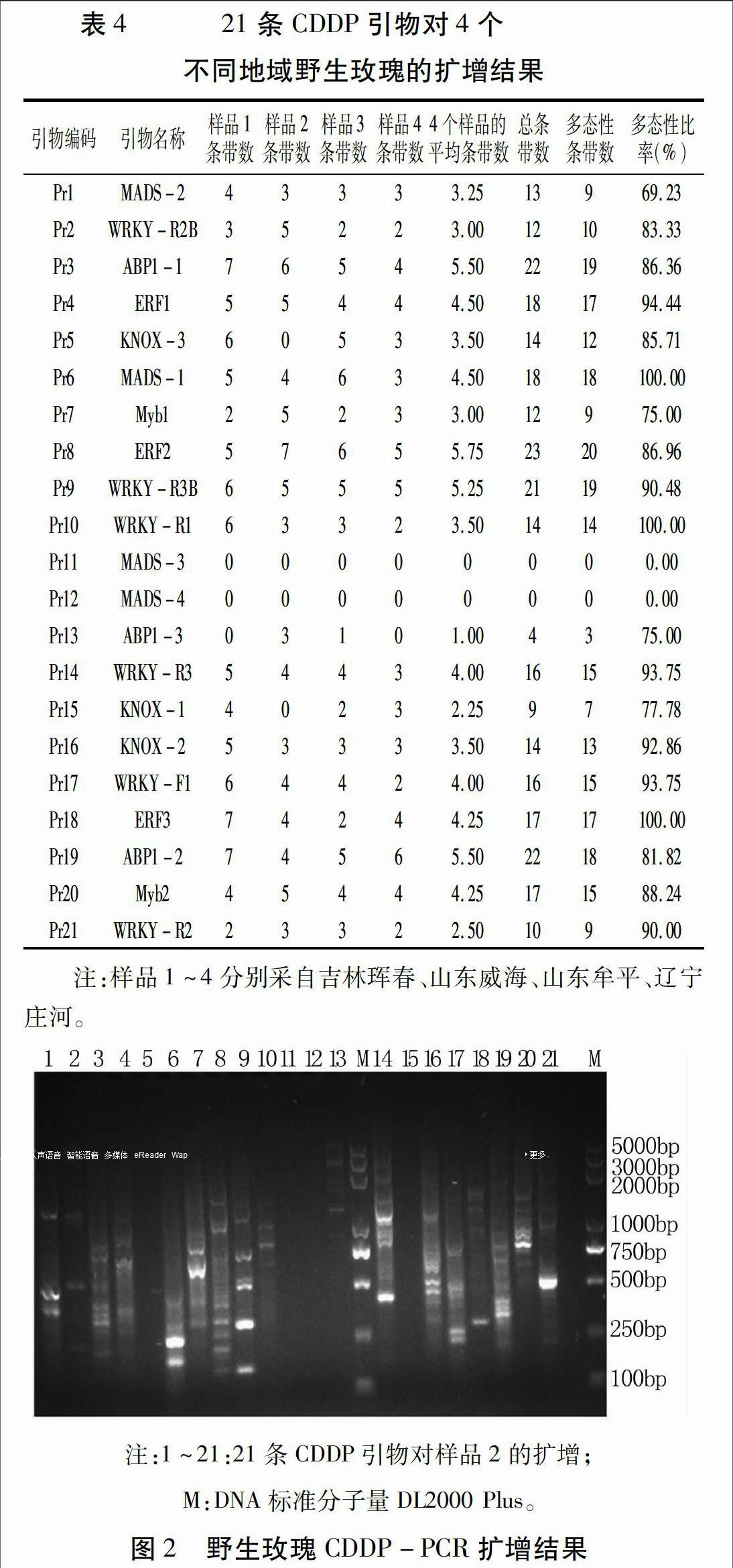
CDDP-PCR扩增结果检测:取6 μL扩增产物于2%琼脂糖凝胶上进行电泳分离,电压5 V/cm,电泳1.5~2 h,EB染色10 min后,在凝胶成像分析仪上进行观察并采集图像。
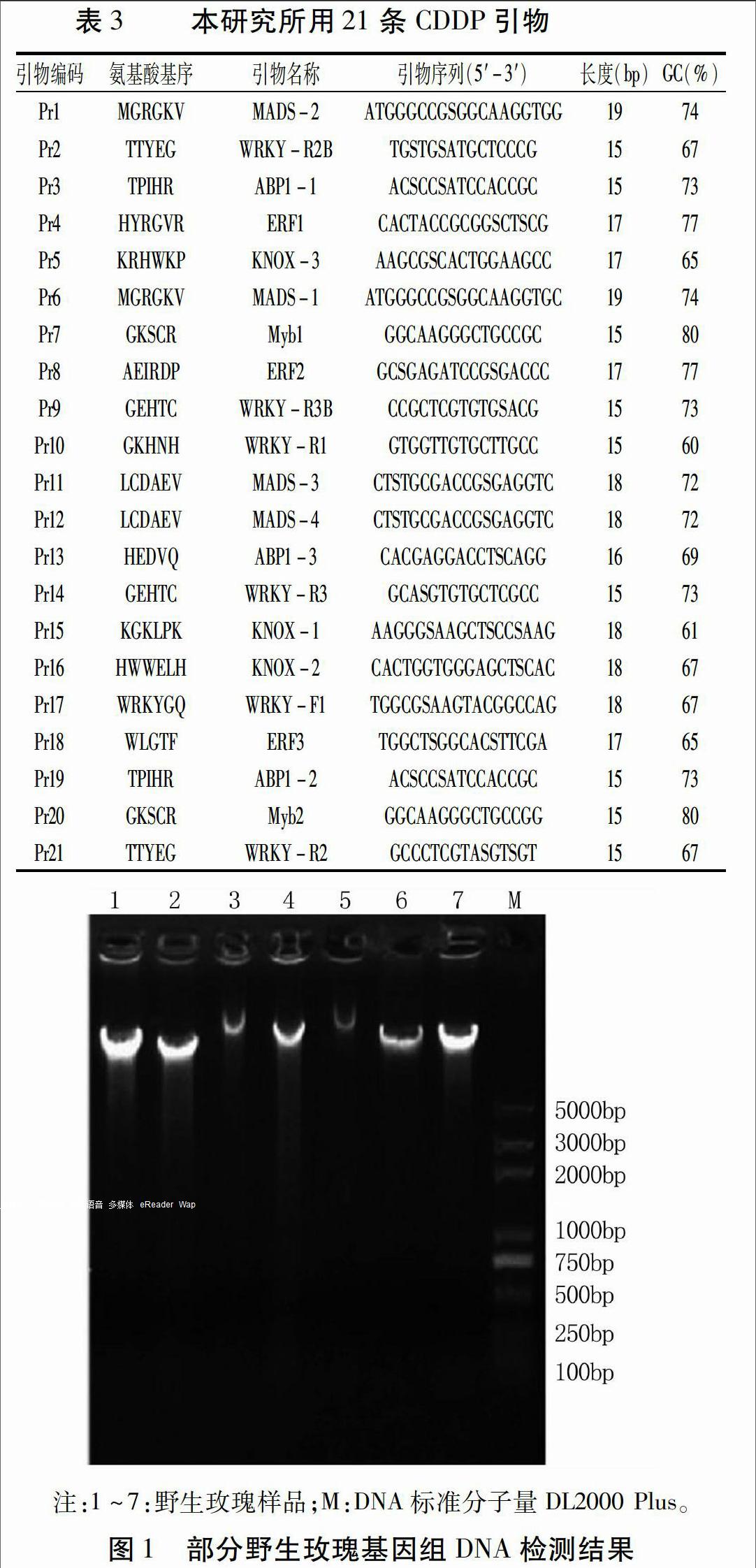
本试验所用的21条CDDP引物都是由上海生工生物工程有限公司合成,引物编号及序列见表3。
2 结果与分析
2.1 野生玫瑰基因组DNA提取
本研究在植物基因组DNA CTAB提取法的基础上,延长水浴时间至1 h,调整氯仿-异戊醇(24∶1)的抽提次数为2~3次,用此方法提取野生玫瑰基因组DNA。琼脂糖凝胶电泳检测结果(图1)表明,DNA条带明显清晰、拖尾较轻,无明显降解现象、完整性较好,说明改良CTAB法可以高效、便捷地提取野生玫瑰基因组DNA。
提取的野生玫瑰基因组DNA的A260/280值在1.60~1.85之间,提取的DNA浓度在1 200~2 500 ng/μL之间,DNA得率较高,因此改良CTAB法能有效提取出较高质量的野生玫瑰基因组DNA。
2.2 CDDP分子标记引物筛选
用21条CDDP引物对4个不同地区的野生玫瑰基因组DNA进行扩增,结果(图2,表4)表明,Pr11和Pr12没有扩增出任何条带,其他引物均能扩增出有效条带;Pr5和Pr15可以对其中三个样品进行有效扩增;引物Pr13的扩增结果中仅有威海和牟平两个地区出现条带,且条带数量较少;其余16条引物对四个不同地域的野生玫瑰基因组DNA均有理想的扩增效果,其中引物Pr3、Pr8、Pr9和Pr19的扩增总条带数多且条带清晰易辨。不同引物对不同地域的野生玫瑰基因组DNA的扩增情况不同,体现了不同地域间的野生玫瑰资源存在遗传差异。从表4可知,多态性比率达到100%的有Pr6、Pr10和Pr18 三条引物,最低的是Pr1为69.23%,其他引物的多态性条带比例均较高。综合考虑每个引物的鉴别能力和扩增效果等情况,筛选出16条(Pr1、Pr2、Pr3、Pr4、Pr6、Pr7、Pr8、Pr9、Pr10、Pr14、Pr16、Pr17、Pr18、Pr19、Pr20、Pr21)CDDP引物,可作为核心引物用于野生玫瑰的鉴定与识别及遗传多样性分析等研究。
3 讨论与结论
基因组DNA的提取是分子标记研究的重要前提,DNA的质量影响着试验的成败。不同的植CDDP分子标记是一种新型的标记技术,引物设计的锚定位点在功能蛋白质或功能基因序列中的保守区域,可以有效地产生其他性状的连锁标记。本研究利用21条CDDP引物对吉林珲春、辽宁庄河以及山东牟平、山东威海四个地区的野生玫瑰基因组DNA进行扩增,以2×Es Taq MasterMix(含染料)10 μL,40 ng/μL DNA模板1 μL,10 pmol/μL引物1 μL,ddH2O 8 μL为反应体系,多态性比率达到100%的有Pr6、Pr10和Pr18三条引物,最低的是Pr1为69.23%,其他引物的多态性条带比例均较高,有16条引物对野生玫瑰具有较高的鉴别能力。由于不同植物间的种质资源遗传多样性较大,相同CDDP引物对不同植物的鉴别能力有所差异,如李莹莹[8]用同样的21条引物进行牡丹的CDDP分子标记研究,最终有20条引物得到扩增条带;李田等[7]将此21条CDDP引物用于菊花,得到19条有效引物。但另一方面也证明了CDDP引物具有通用性、操作简单、成本较低、易得到遗传信息等优势[14],更有利于反映物种遗传变异的多样性。
本研究结果证明了CDDP引物在野生玫瑰种质资源的遗传多样性分析和品种鉴定等方面具有应用的可行性,为以后利用CDDP-PCR这一新型目的分子标记技术进行野生玫瑰遗传变异的深入研究提供一定的应用基础,也为此标记在其它植物上的应用研究提供了经验与借鉴。
参 考 文 献:
[1]傅立国, 金鉴明. 中国植物红皮书——稀有濒危植物(第一册)[M].北京:科学出版社, 1992:558-559.
[2] Matasyoh L G, Wachira F N, Kinyua M G, et al. Leaf storage conditions and genomic DNA isolation efficiency in Ocimum gratissimum from Kenya[J]. African Journal of Biotechnology, 2008, 7(5): 557-564.
[3] 王书珍, 张传进, 程华, 等. 杜鹃花表达序列标签资源中的微卫星信息分析[J]. 湖北林业科技,2014, 43(2):7-10.
[4] 熊发前, 蒋菁, 钟瑞春, 等. 两种新型目标分子标记技术——CDDP与 PAA[J]. 植物生理学通讯,2010(9):871-879.
[5] 熊发前, 蒋菁, 钟瑞春, 等. 分子标记技术的两种新分类思路及目标分子标记技术的提出[J]. 中国农学通报,2010, 26 (10): 60-64.
[6] Collard B C Y, Mackill D J. Conserverd DNA-derived polymorphism (CDDP): a simple and novel method for generating DNA markers in plants[J]. Plant Cell Reports, 2012, 31: 299-310.
[7] 李田, 郭俊娥, 郑成淑, 等. 菊花品种的遗传多样性分析及CDDP指纹图谱构建[J].北京林业大学学报, 2014, 36(4): 95-101.
[8] 李莹莹. 牡丹CDDP分子标记引物筛选及多态性分析[J].核农学报,2013,27(8):1099-1105.
[9] 李莹莹, 郑成淑. 利用CDDP标记的菏泽牡丹品种资源的遗传多样性[J]. 中国农业科学,2013,46(13): 2739-2750.
[10]李强, 高聚林, 苏二虎, 等. 基于单因素试验的大豆CDDP-PCR反应体系优化[J]. 安徽农业科学, 2015, 43(3):32-34,43.
[11]吴艳艳, 代德艳, 蔡春梅, 等. 一种改良的大豆DNA提取方法[J].大豆科学,2015,34(1):113-115.
[12]王书珍, 张传进, 查三省, 等. 杜鹃基因组DNA提取方法的研究及应用[J].林业科技通讯, 2015(2):5-8.
[13]李承哲, 刘占林, 杨红超, 等. 改良CTAB法提取鸢尾属植物DNA[J].现代农业科技,2011(8):31-34.
[14]王小文. 基于CDDP标记的不同花色牡丹的遗传关系分析[D]. 泰安:山东农业大学, 2015.

