转肽酶A在蛋白质和多肽修饰中的应用
马颖,刘忞之,王伟
转肽酶A在蛋白质和多肽修饰中的应用
马颖,刘忞之,王伟
在20世纪90年代末,Navarre 和 Schneewind[1]首次在金黄色葡萄球菌(Staphylococcus aureus)中发现了转肽酶 A(sortase A),它催化了表面蛋白与多种革兰阳性菌的肽聚糖骨架的共价结合,从而开启了转肽酶A对蛋白质或多肽作用的研究[2-10]。
近年来,人们发现从金黄色葡萄球菌中分离得到的转肽酶A在蛋白质或多肽的合成与修饰中表现出了良好的反应活性和特异性[11]。转肽酶A可以特异性结合蛋白质 C末端的 LPXTG 序列(X 是任意氨基酸),切断苏氨酸和甘氨酸之间的肽键,而后与 N-端含有甘氨酸残基的蛋白质或多肽末端连接形成肽键[11-12]。转肽酶A不仅可以用于修饰蛋白质或多肽增强其稳定性和活性,还可以实现蛋白质和蛋白质、蛋白质和多肽以及多肽和多肽之间的特异性位点的连接[13];其催化反应的高特异性使得转肽酶A在蛋白质或多肽合成与修饰中得到了广泛的应用。本文就转肽酶A的作用机制、结构特征及其在蛋白质和多肽修饰中的应用展开综述。
1 转肽酶A的作用机制及结构特征
转肽酶A是一种膜结合巯基转肽酶[12],能够特异性地识别蛋白质或多肽 C端的保守氨基酸序列 LPXTG(X 指任意氨基酸)。首先,转肽酶A的 184 位半胱氨酸上的巯基进攻苏氨酸(Thr)和甘氨酸(Gly)之间的肽键,生成乙酰化的转肽酶A中间产物,然后另一蛋白质或多肽N端的甘氨酸的氨基亲核进攻硫酯中间体,使得亲核基团和苏氨酸之间的肽键形成,而原有的保守序列 LPXTG 中的甘氨酸残基及其相关部分被切除(图 1)[14-17]。
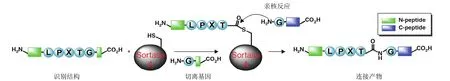
图1 转肽酶A作用机制[17]
对于转肽酶A的三维结构,Bradshaw 等[12]利用 NMR进行了解析(图 2A)。结果显示,转肽酶A包含 2 个短螺旋和 8 个 β 折叠结构。后经其他研究表明,在其第 4 个和第 7 个 β 折叠的筒状结构的一侧具有一个疏水沟,这个结构被 β2-β3、β3-β4、β6-β7 和 β7-β8 四个 loop 结构包围,催化中心 His120、Cys184、Arg197分别位于 β4 和 β7 的尾部以及 β8 起始端的沟中(图 2B)[18-20],该结构可以容纳并识别底物,发生转肽反应。
转肽酶A通过催化活性中心的 Val166、Val168和Leu169识别 LPXTG 序列[19]。这三个氨基酸残基位于β6-β7 的 loop 区,这一 loop 结构在结合底物的过程中由一个无序的开放状态变为有序的闭合状态。而闭合结构β6-β7 loop 区的稳定性是由钙离子与 β6-β7 loop 区的Glu171、β3-β4 loop 区的 Glu105、Glu108和 Asp112结合所决定的(图 2C)。因此,钙离子的结合可增强酶对底物的亲和力,钙离子浓度低时催化效率也低,对钙离子的高度依赖性限制了转肽酶A的使用范围。
2 转肽酶A的蛋白质工程及催化活性的研究
为了解决钙离子浓度依赖而催化效率低的问题,Hirakawa 研究组于 2012 年开展了不同菌株的转肽酶 A催化特性的对比实验研究,结果发现来源于化脓性链球菌(Streptococcus pyogenes)和炭疽芽胞杆菌(Bacillus anthraci)的转肽酶A本身就是非依赖钙离子的,说明对钙离子的依赖是金黄色葡萄球菌转肽酶A的特性。因此,Hirakawa 等提出可以通过突变获得钙离子非依赖的转肽酶A。他们对来源于化脓性链球菌和炭疽芽胞杆菌的转肽酶及其他转肽酶进行序列比对,发现转肽酶家族的酶活性位点区域高度保守,但是侧链的钙离子结合位点区域并不保守,即对应于转肽酶A的第 105 位和第 108 位氨基酸残基在其他两个转肽酶中不保守。于是他们设计了五组突变体,即E105K、E108A、E108Q、E105K/E108A 和 E105K/E108Q。通过对结果进行分析,得到了在钙离子不存在的情况下,仍可发生催化反应的双突变体(E105K/E108A 和E105K/E108Q),只是催化效率较野生型有所降低[21]。
该研究组为解决双突变体催化效率低的问题,继续开展了转肽酶A的突变研究。他们合成(或表达)了引入五个突变位点(P94R/D160N/D165A/K190E/K196T)的转肽酶A,这一突变体在没有钙离子的情况下不具有催化活性,但是将五个突变位点引入双突变体,形成的具有七个突变位点的转肽酶 A,不仅可不依赖于钙离子进行转肽反应,而且活性较野生型的酶提高了 120 倍[22]。由此可见,具有七个突变位点的转肽酶A将成为选择性蛋白连接的强有力工具。

图2 转肽酶A结构特征(A:转肽酶A的三维结构;B:催化中心结构;C:钙离子结合区)[12,21]
3 转肽酶A在蛋白质或多肽修饰中的研究进展
转肽酶A对于底物的特异性,使之成为对蛋白或多肽特异性修饰的一个重要工具。关于转肽酶A的多种应用已经被报道,例如蛋白连接[23]、细胞表面蛋白标记[24-25]、蛋白-脂质连接[26]和蛋白质与多肽的环化[27-28]等。
2004 年,Mao 等[29]通过将天然多肽以及非天然多肽连接到 LPXTG 标记的绿色荧光蛋白上,首次证实了转肽酶A 催化的转肽反应可以作为蛋白质修饰的一个重要工具。2009 年,Antos 等[30]扩大了其在蛋白质修饰中的应用。此后,转肽酶A被用于修饰多种结构不同的多肽序列[23,31-33]。2011 年,Popp 等[34]又发现应用转肽酶A的环化作用可增加细胞因子的稳定性。在已报道的关于转肽酶A 应用的诸多研究中,多数是阐述转肽酶A用于蛋白质修饰,尤其是其在环肽的生物合成中的应用[27,35-40]。
3.1 增加细胞因子的稳定性
重组蛋白疗法因血循环半衰期短或稳定性较差使得临床疗效差。尽管聚乙二醇缀合修饰可延长很多蛋白质的半衰期,但是这种方法存在特异性差的弊端,使得蛋白质的生物活性丧失。Popp 等[34]以具有四个螺旋结构的细胞因子为例,利用转肽酶A介导的转肽反应来促进聚乙二醇和细胞因子的位点特异性结合,实现了在不影响活性的前提下延长细胞因子半衰期的目的。
3.2 标记蛋白或多肽
目前转肽酶A介导的转肽反应已成功地应用于目的蛋白的多肽标记、荧光标记、生物素标记、放射性金属复合物标记、聚乙二醇衍生物标记、多聚蛋白标签标记等[40]。研究表明,在利用转肽酶A对蛋白质或多肽进行标记时,需先将目的蛋白构建成具有转肽酶识别位点(LPXTG)的形式[41],然后通过转肽酶A介导的转肽反应实现目的蛋白的多肽标记。
3.3 蛋白-蛋白融合
2004 年,Pollok 研究组首次阐述了转肽酶A可用作催化多肽和多肽、多肽和蛋白、蛋白和蛋白融合的工具酶[29]。在研究中,他们以一系列含有不同甘氨酸残基数(1/2/3/5)的短肽和含有转肽酶A识别序列的荧光蛋白为底物,通过检测荧光蛋白荧光信号的变化来证实融合反应的完成。从此,转肽酶A被用于各种不同长度的蛋白或多肽链之间的融合[1,33,42-43]。
3.4 蛋白质或多肽的环化
蛋白质或多肽的稳定性差限制了其在临床治疗与实验研究中的应用,而环状结构因具有环化骨架及二硫键结构可大大增强蛋白质或多肽的稳定性。环肽由于含有共价闭合的酰胺骨架而具有独特的特性,使得它作为多肽中一种特殊肽类而存在。自然存在的环肽通常含有 14~78 个氨基酸残基,与线形肽相比,环肽具有更强的蛋白酶稳定性[37]、化学稳定性和热稳定性[30]。除自然存在的环肽外,环状肽也可通过化学方法、其他连接方法或酶介导法获得。在这些方法中,化学方法及其他连接方法存在产量低、纯化困难等问题,而酶法介导的环化反应不仅产量高,而且特异性强,利于纯化。
Ploegh 实验组首次报道了转肽酶A可介导 C-端含有LPXTG 识别序列且 N-端含有甘氨酸残基的蛋白环化[30],这使得转肽酶A的应用不再局限于分子间的反应,分子内的快速且简单的转肽反应同样受到关注(图 3)。从此,转肽酶A介导的环化反应得以更广泛深入的研究。
2009 年,Antos 等[30]利用转肽酶A环化了三种不同结构的蛋白质底物(Cre 融合酶、eGFP 和 UCHL3),通过对这三种不同结构的蛋白质底物及其环化后产物进行研究,发现转肽酶A催化的环化反应是可逆的,并且具有反应快速、特异性强等特点。Antos 等首先将 Cre 和 eGFP 构建成 C 端含有转肽酶识别序列、N 端含有甘氨酸残基的蛋白底物,通过与转肽酶A反应,得到环化产物,经验证确定了 Cre 和 eGFP 发生了分子内转肽反应。进一步研究发现,在已环化的产物中仍然含有转肽酶A识别序列,后经进一步实验证明,发现转肽酶A也可以剪切环肽中的苏氨酸和甘氨酸之间的肽键,使环肽再次转变为线形肽,也就是说转肽酶介导的反应是可逆的,在环肽与线性肽之间存在一个平衡。此外,通过对 eGFP 的结构进行研究,发现经过修饰的 N-端和 C-端位于 β 折叠的同一端,是环化反应的理想的底物结构。但是,将转肽酶A识别序列构建在泛素化 C-端水解酶(UCHL3)内部的一个交叉环上,环化反应仍可发生,且发生反应的方式与 eGFP 相同。由此可见转肽酶A催化的环化反应具有反应特异性强的特点。
转肽酶A介导的环化作用受底物多肽长度的影响,后者至少长达 19 个肽,才可得到单体直接环化的环肽。Wu等[39]分别将含有 17、18、19 个氨基酸残基的肽链与转肽酶A进行反应,发现得到的单体环肽随线性肽氨基酸残基数量的增加而增加,而二聚体及三聚体环肽随线性肽氨基酸数量的增加而减少。另外,该酶也可以催化富含二硫键的多肽环化,Jia 等[27]通过研究发现转肽酶A对于富含二硫键的多肽具有广泛适用性。在研究中,他们利用转肽酶A分别将 kalata B1、α-芋螺霉素 Vc1.1 和向日葵胰蛋白酶抑制剂 1 进行环化,这些多肽含有 14~29 个氨基酸残基,在环化生成的环肽中分别具有 3、2 和 1 个二硫键。
转肽酶A介导的环化反应在提高环肽产量以及多肽活性方面发挥着重要作用。2011 年,Bolscher 等[38]以histatin-1 作为模板,分别通过化学法和转肽酶A介导的转肽反应对模板蛋白进行环化。实验结果表明,化学法仅得到约 3% 的环肽,而转肽酶A介导的环化反应产量则大于90%;另外,在伤口愈合活性方面,10 nmol/L 的环肽即可达到最大伤口愈合活性,而线形肽达到此活性需要 10 μmol/L。由此可见,转肽酶A介导的环化对于环肽产量以及多肽活性的提高具有重要作用。
此外,在本研究组的前期研究中,构建了含有双亲和色谱标签的融合转肽酶 A,并通过酶促动力学实验证明了融合标签对于转肽反应的催化活性没有影响[44],初步建立可固定化转肽酶A介导的多肽环化方法。
综上所述,转肽酶A催化的转肽反应为多肽和蛋白的环化提供了一种较好的方法。与化学法合成环肽的方法相对比,转肽酶A催化的反应具有产量高和特异性强的特点。因此,通过对目的蛋白进行适当修饰,利用转肽酶催化的反应获得环肽无疑更具有潜力。

图3 转肽酶A介导的分子内与分子间转肽反应[30]
4 小结
尽管金黄色葡萄球菌的转肽酶A及其突变体有着快速高效地催化转肽反应的优点,但是反应的可逆性依然是一个不可忽视的缺陷。目标蛋白或多肽与转肽反应产物之间存在平衡,为提高转肽反应产物的产率通常使用过量的含有多聚甘氨酸的多肽底物(衍生物或亲核分子)促进正向的转肽反应;这从一定程度上导致了多聚甘氨酸亲核试剂的浪费和目标产物的纯化困难。为解决逆反应的不足,已有研究报道利用转肽酶A对识别位点 C-端结构的非特异性合成含有缩肽(depsipeptide)类或经转肽反应后形成二酮哌嗪的类多肽标记底物用于目标蛋白的 N-端标记[45-46]。这些实验结果为进一步提高转肽酶A的正向转肽反应效率的深入研究奠定了基础。不仅如此,目前还通过定向进化和噬菌体展示技术获得了识别不同位点(APxTG 或 FPxTG)的突变体和从其他菌株如溶血性葡萄球菌(Staphylococcus simulans)中鉴定出识别位点为 LXPTG 的转肽酶,这将促进转肽酶介导的蛋白或多肽不同标记修饰的应用[47-48]。
自首次发现转肽酶A至今已将近 20 年,对于它的应用研究也已有 10 多年,转肽酶A介导的酶法连接逐渐被认为可以用于替代天然化学连接法。转肽酶A对于识别序列 LPXTG 具有很高的特异性,使其在蛋白质或多肽的修饰方面具有很高的应用价值,其中最重要的应用是利用转肽酶A标记的抗体类药物在临床诊断和治疗中发挥了重要作用[40,49]。在环肽研究方面,由于环化的多肽比线性多肽具有更强的稳定性,使得蛋白质或多肽在疾病治疗方面具有更广泛的潜在价值[37]。此外,随着蛋白或多肽传递(BioPorter)技术的发展,转肽酶A已发展成为活细胞内直接进行蛋白标记的分子工具[50-51]。因此,可以预见随着分子生物学技术的发展,更多的研究将被用于转肽酶A的结构优化,发掘其更多的潜在功能,扩大其应用范围,作为强大的蛋白质或多肽修饰工具,转肽酶A将被广泛地应用于生物医药研究。
参考文献
[1]Navarre WW,Schneewind O.Surface proteins of gram-positive bacteria and mechanisms of their targeting to the cell wall envelope.Microbiol Mol Biol Rev,1999,63(1):174-229.
[2]Spirig T,Weiner EM,Clubb RT.Sortase enzymes in Gram-positive bacteria.Mol Microbiol,2011,82(5):1044-1059.
[3]Mazmanian SK,Liu G,Ton-That H,et al.Staphylococcus aureus sortase,an enzyme that anchors surface proteins to the cell wall.Science,1999,285(5428):760-763.
[4]Scott CP,Abel-Santos E,Jones AD,et al.Structural requirements for the biosynthesis of backbone cyclic peptide libraries.Chem Biol,2001,8(8):801-815.
[5]Lee SF,McGavin MK.Identification of a point mutation resulting in loss of cell wall anchoring activity of SrtA of Streptococcus mutans NG5.Infect Immun,2004,72(7):4314-4317.
[6]Saska I,Gillon AD,Hatsugai N,et al.An asparaginyl endopeptidase mediates in vivo protein backbone cyclization.J Biol Chem,2007,282(40):29721-29728.
[7]Raz A,Fischetti VA.Sortase A localizes to distinct foci on the Streptococcus pyogenes membrane.Proc Natl Acad Sci U S A,2008,105(47):18549-18554.
[8]Cascioferro S,Totsika M,Schillaci D.Sortase A:an ideal target for anti-virulence drug development.Microb Pathog,2014,77:105-112.
[9]Koussa MA,Sotomayor M,Wong WP.Protocol for sortase-mediated construction of DNA-protein hybrids and functional nanostructures.Methods,2014,67(2):134-141.
[10]Zhang J,Yamaguchi S,Nagamune T.Sortase A-mediated synthesis of ligand-grafted cyclized peptides for modulating a model protein-protein interaction.Biotechnol J,2015,10(9):1499-1505.
[11]Tan XL,Xu L,Shi J,et al.Application of transpeptidase sortase A in protein modification.Prog Chem,2014,26(10):1741-1751.(in Chinese)谭祥龙,许玲,石景,等.转肽酶sortase A在蛋白质修饰中的应用.化学进展,2014,26(10):1741-1751.
[12]Bradshaw WJ,Davies AH,Chambers CJ,et al.Molecular features of the sortase enzyme family.FEBS J,2015,282(11):2097-2114.
[13]Proft T.Sortase-mediated protein ligation:an emerging biotechnology tool for protein modification and immobilisation.Biotechnol Lett,2010,32(1):1-10.
[14]Bentley ML,Lamb EC,McCafferty DG.Mutagenesis studies of substrate recognition and catalysis in the sortase A transpeptidase from Staphylococcus aureus.J Biol Chem,2008,283(21):14762-14771.
[15]Frankel BA,Tong Y,Bentley ML,et al.Mutational analysis of active site residues in the Staphylococcus aureus transpeptidase SrtA.Biochemistry,2007,46(24):7269-7278.
[16]Perry AM,Ton-That H,Mazmanian SK,et al.Anchoring of surface proteins to the cell wall of Staphylococcus aureus.III.Lipid II is an in vivo peptidoglycan substrate for sortase-catalyzed surface protein anchoring.J Biol Chem,2002,277(18):16241-16248.
[17]Schmohl L,Schwarzer D.Sortase-mediated ligations for the site-specific modification of proteins.Curr Opin Chem Biol,2014,22:122-128.
[18]Ilangovan U,Ton-That H,Iwahara J,et al.Structure of sortase,the transpeptidase that anchors proteins to the cell wall of Staphylococcus aureus.Proc Natl Acad Sci U S A,2001,98(11):6056-6061.
[19]Suree N,Liew CK,Villareal VA,et al.The structure of the Staphylococcus aureus sortase-substrate complex reveals how the universally conserved LPXTG sorting signal is recognized.J Biol Chem,2009,284(36):24465-24477.
[20]Call EK,Klaenhammer TR.Relevance and application of sortase and sortase-dependent proteins in lactic acid bacteria.Front Microbiol,2013,4:73.
[21]Hirakawa H,Ishikawa S,Nagamune T.Design of Ca2+-independent Staphylococcus aureussortase A mutants.Biotechnol Bioeng,2012,109(12):2955-2961.
[22]Hirakawa H,Ishikawa S,Nagamune T.Ca2+-independent sortase-A exhibits high selective protein ligation activity in the cytoplasm of Escherichia coli.Biotechnol J,2015,10(9):1487-1492.
[23]Policarpo RL,Kang H,Liao X,et al.Flow-based enzymatic ligation by sortase A.Angew Chem Int Ed Engl,2014,53(35):9203-9208.
[24]Shi J,Kundrat L,Pishesha N,et al.Engineered red blood cells as carriers for systemic delivery of a wide array of functional probes.Proc Natl Acad Sci U S A,2014,111(28):10131-10136.
[25]Swee LK,Lourido S,Bell GW,et al.One-step enzymatic modification of the cell surface redirects cellular cytotoxicity and parasite tropism.ACS Chem Biol,2015,10(2):460-465.
[26]Antos JM,Miller GM,Grotenbreg GM,et al.Lipid modification of proteins through sortase-catalyzed transpeptidation.J Am Chem Soc,2008,130(48):16338-16343.
[27]Jia X,Kwon S,Wang CI,et al.Semienzymatic cyclization of disulfide-rich peptides using sortase A.J Biol Chem,2014,289(10):6627-6638.
[28]Hu J,Zhao W,Gao Y,et al.Site-specific in situ growth of a cyclized protein-polymer conjugate with improved stability and tumor retention.Biomaterials,2015,47:13-19.
[29]Mao H,Hart SA,Schink A,et al.Sortase-mediated protein ligation:a new method for protein engineering.J Am Chem Soc,2004,126(9):2670-2671.
[30]Antos JM,Popp MW,Ernst R,et al.A straight path to circular proteins.J Biol Chem,2009,284(23):16028-16036.
[31]Cambria E,Renggli K,Ahrens CC,et al.Covalent modification of synthetic hydrogels with bioactive proteins via sortase-mediated ligation.Biomacromolecules,2015,16(8):2316-2326.
[32]Beerli RR,Hell T,Merkel AS,et al.Sortase enzyme-mediated generation of site-specifically conjugated antibody drug conjugates with high in vitro and in vivo potency.PLoS One,2015,10(7):e0131177.
[33]Wu Z,Guo Z.Sortase-mediated transpeptidation for site-specific modification of peptides,glycopeptides,and proteins.J Carbohydr Chem,2012,31(1):48-66.
[34]Popp MW,Dougan SK,Chuang TY,et al.Sortase-catalyzed transformations that improve the properties of cytokines.Proc Natl Acad Sci U S A,2011,108(8):3169-3174.
[35]Stanger K,Maurer T,Kaluarachchi H,et al.Backbone cyclization of a recombinant cystine-knot peptide by engineered Sortase A.FEBS Lett,2014,588(23):4487-4496.
[36]Strijbis K,Ploegh HL.Secretion of circular proteins using sortase.Methods Mol Biol,2014,1174:73-83.
[37]Van't Hof W,Hansenová Maňásková S,Veerman EC,et al.Sortase-mediated backbone cyclization of proteins and peptides.Biol Chem,2015,396(4):283-293.
[38]Bolscher JG,Oudhoff MJ,Nazmi K,et al.Sortase A as a tool for high-yield histatin cyclization.FASEB J,2011,25(8):2650-2658.
[39]Wu Z,Guo X,Guo Z.Sortase A-catalyzed peptide cyclization for thesynthesis of macrocyclic peptides and glycopeptides.Chem Commun (Camb),2011,47(32):9218-9220.
[40]Voloshchuk N,Liang D,Liang JF.Sortase A mediated protein modifications and peptide conjugations.Curr Drug Discov Technol,2015,12(4):205-213.
[41]Ritzefeld M.Sortagging:a robust and efficient chemoenzymatic ligation strategy.Chemistry,2014,20(28):8516-8529.
[42]Levary DA,Parthasarathy R,Boder ET,et al.Protein-protein fusion catalyzed by sortase A.PLoS One,2011,6(4):e18342.
[43]Lee T,Min J,Hirakawa H,et al.Fusion protein bilayer fabrication composed of recombinant azurin/cytochrome P450 by the sortase-mediated ligation method.Colloids Surf B Biointerfaces,2014,120:215-221.
[44]Zhang SX,Liu MZ,Yang Y,et al.Enzymatic cyclization of peptides using immobilized sortase A.Acta Pharm Sinica,2015,50(5):627-632.(in Chinese)张书香,刘忞之,杨燕,等.固定化分选酶介导的多肽环化.药学学报,2015,50(5):627-632.
[45]Williamson DJ,Fascione MA,Webb ME,et al.Efficient N-terminal labeling of proteins by use of sortase.Angew Chem Int Ed Engl,2012,51(37):9377-9380.
[46]Liu F,Luo EY,Flora DB,et al.Irreversible sortase A-mediated ligation driven by diketopiperazine formation.J Org Chem,2014,79(2):487-492.
[47]Schmohl L,Bierlmeier J,Gerth F,et al.Engineering sortase A by screening a second-generation library using phage display.J Pept Sci,2017.[Epub ahead of print]
[48]Chen J,Dong H,Murfin KE,et al.Active site analysis of sortase A from Staphylococcus simulans indicates function in cleavage of putative cell wall proteins.Biochem Biophys Res Commun,2016,478(4):1653-1659.
[49]Massa S,Vikani N,Betti C,et al.Sortase A-mediated site-specific labeling of camelid single-domain antibody-fragments:a versatile strategy for multiple molecular imaging modalities.Contrast Media Mol Imaging,2016,11(5):328-339.
[50]Glasgow JE,Salit ML,Cochran JR.In vivo site-specific protein tagging with diverse amines using an engineered sortase variant.J Am Chem Soc,2016,38(24):7496-7499.
[51]Zhang J,Yamaguchi S,Hirakawa H,et al.Intracellular protein cyclization catalyzed by exogenously transduced Streptococcus pyogenes sortase A.J Biosci Bioeng,2013,116(3):298-301.
中国医学科学院医学与健康科技创新工程(2016-I2M-3-012);北京协和医学院“协和青年基金”(3332016057)
100050 北京,中国医学科学院北京协和医学院药物研究所天然药物活性物质与功能国家重点实验实验室/国家卫生和计划生育委员会天然药物生物合成重点实验室
王伟,Email:wwang@imm.ac.cn
2017-02-20
10.3969/j.issn.1673-713X.2017.02.010

