新发动物疫病检测方法研究及口岸检疫应用展望
杜方原,冯春燕,刘丹丹,仇松寅,张永宁,林祥梅,吴绍强
(中国检验检疫科学研究院,北京 100176)
新发动物疫病检测方法研究及口岸检疫应用展望
杜方原,冯春燕,刘丹丹,仇松寅,张永宁,林祥梅,吴绍强
(中国检验检疫科学研究院,北京 100176)
目前我国口岸已有的一些疫病检测方法,主要是针对进境动物检疫疫病名录中提及的已知病原,而针对新发病原的检测方法较少。本文介绍了传统病原的检测方法,包括病原分离和培养、血清学方法、蛋白质免疫印迹和电子显微镜观察等,阐述了新型分子生物学检测方法,如随机引物PCR法、序列非依赖性单引物扩增技术、小RNA深度测序技术、病毒宏基因组学技术等,并对上述方法在口岸中的应用前景进行了分析。
新发病;宏基因组;高通量测序;病原分离;口岸检疫
随着人类文明的发展以及自然环境的大幅度改变,病原微生物与宿主的接触频率不断加速。在过去的数十年里,世界范围内不断出现变异和新发病原,如施马伦贝格病毒、埃博拉病毒、新型猪圆环病毒(PCV3)。这些病原引起的疫病不仅严重威胁畜禽养殖产业的经济和生态安全,同时给人类和动物的健康以及社会稳定带来巨大威胁。通常将新的或者有重大变化的病原体引起的动物疫病称为新发动物疫病[1]。由于病毒的复制能力强、突变速率高、并且具有重配和重组的变异方式[2],大多数新发病的病原体为病毒。由于新发疫病的症状一般具有不确定性、缺乏检测方法储备以及难以预测等特点,无法对其作出快速诊断和响应。尤其在当今交通和物流运输发达的情况下,疫病的快速传播可能会造成难以估量的经济损失。因此,如何发现新发动物病毒病的感染并对新发病进行确诊是新发病研究中的首要问题。
早期新发动物疫病检测主要依赖传统的病原检测方法,包括从临床病例中进行病原分离和培养、血清学检测、Western blot和病原形态的电镜观察等。这些方法在早期新病原的发现过程中起了十分重要的作用,但由于耗时长、无法进行确诊或对生物安全资质有较高要求等原因,逐渐被新发展起来的分子生物学诊断方法所替代,如序列非依赖性扩增技术、小RNA深度测序技术和宏基因测序技术等。尤其是基于高通量测序技术的病毒宏基因组学等方法,能在核酸水平从少量病料中迅速发现病原群体,并测序得到全基因组序列,在对变异病原和新发病原的识别中起了十分重要的作用。本文就上述各方法进行了综述,尤其是对近年来发展起来的宏基因组技术及其在口岸检疫的应用前景进行详尽的介绍。
1 传统病原检测
1.1 病原分离和培养
病原分离是鉴定病毒的常规方法。通过敏感细胞系接种病料或者培养组织细胞等原代细胞,来分离病毒,鸡胚接种或动物试验接种病料,观察病变或者动物的发病情况。病毒培养是筛查和鉴定不明病原体最直接有效的方法。2009年,Yu等[3]收集病因不明的急性发热患者的血液样本,利用细胞系分离出新的布尼亚病毒病原体。
1.2 血清学方法和蛋白质免疫印迹(Western blot)
血清学方法包括酶联免疫吸附试验(ELISA)和间接免疫荧光试验(IFA)等。ELISA原理是酶分子与抗体或抗抗体分子共价结合,滴加底物溶液后,底物可在酶作用下出现颜色反应,通过底物的颜色反应来判定有无相应的免疫反应。IFA是利用血清和相应的荧光标记物共同孵育细胞,在荧光显微镜观察荧光。血清学方法通常用来检测宿主体内针对病毒的抗体,来达到检测抗体的目的。Western blot是将蛋白质转移到膜上,利用相应的抗体进行检测的方法。血清学方法和Western blot的方法可用于新发疫病的初步诊断。
1.3 电子显微镜观察
电子显微镜技术能够直观且快速鉴别出临床样品中的病毒形态,初步确定病毒的种类,是病原学研究的必要手段之一。对于混合感染的病例,电子显微镜技术还可以同时检测出病料中的几种病原体。
2 新型分子生物学检测方法
2.1 随机引物PCR法
1992年,Patrick[4]建立了随机引物PCR法。其核心是引物设计,应用随机PCR技术构建cDNA文库,再结合基因芯片或测序等技术发现新型病毒或与某类病毒相关的片段,或者采用此方法对提取的核酸直接进行扩增,检测未知病原。Li等[5-6]利用随机PCR法对从未知原因的脑炎病人肛拭子中和原因未明的肠胃炎病人粪便中提取的核酸进行随机PCR,通过测序、分析,分别得到新型γ-乳头瘤病毒和新型小核糖核酸病毒。
2.2 序列非依赖性单引物扩增技术(Sequenceindepen dent single primer amplification,SISPA)
SISPA最早于1991年由Reyes[7]建立,经过多年来的发展优化,形成了与随机PCR方法联合使用,从而扩增病毒遗传物质的固定模式,尤其适用于样本中病毒颗粒少、缺少核酸序列信息的情况。Kirkland等[8]采用这种方法从不明原因致死的3~4周龄猪的身上发现了2种新型瘟疫病毒Bungowannah毒株。
2.3 小RNA深度测序技术
2009年,Kreuze等[9]在检测被病毒感染的甘薯中首次建立了小RNA深度测序技术。病毒感染宿主后,将病毒基因组释放入宿主体内,宿主细胞内会形成大量的病毒衍生小RNA(Virus-derived small interfering RNAs,vsiRNA),通过序列特异性结合使其同源RNA高效特异性降解,沉默病毒RNA,使宿主获得系统性的病毒免疫。通过第二代高通量测序技术可以获得这些siRNA的序列,应用生物信息学工具将测序数据进行重叠组装,获得宿主体内的病毒序列信息。2012年,Thomas等[10]应用该技术分析巨细胞病毒(Cytomegalovirus,CMV)感染的人包皮成纤维细胞,从中获得了病毒基因组信息以及有CMV编码的sRNA信息。
2.4 病毒宏基因组学技术
1998年,Handelsman等[11]首次提出“宏基因组”的概念。宏基因组学研究是建立在核酸测序基础上的一种研究手段,已广泛应用于环境、动物以及人体的微生物的监测和新病原的发现中。病毒宏基因组学研究的基本路线包括4个主要的步骤:一是样品处理;二是提高目标样品丰度,包括构建宏基因组文库或序列非依赖性扩增等技术;三是测序技术;四是生物信息学分析。近年来随着高通量测序技术的出现,尤其是下一代测序技术(NGS)的广泛应用,可以应用序列非依赖性扩增技术来提高目的基因的丰度,来代替基因组文库的构建及筛选。
2.4.1 样品处理。样品经过离心、过滤、核酸酶处理等步骤,去除掉病毒以外的其他杂质,尽量降低背景核酸的干扰,得到纯度较高的病毒核酸悬液。利用试剂盒提取病毒核酸。Li等[12]对比使用3种试剂盒提取病毒核酸,发现Qiagen的试剂盒能更多的提取到病毒核酸。对于病毒含量较低的样品可通过SISPA或者基于片段多态性扩增构建病毒cDNA技术(VIDISCA)来提高病毒丰度,为后续测序做好准备[13]。
2.4.2 文库的构建及筛选。宏基因组学文库筛选方法和研究目的的不同,构建文库的载体和方法也各有不同。构建的文库可分为载体克隆文库和基于下一代测序技术的加接头的文库。文库筛选的方法包括功能性筛选法、荧光原位杂交技术法以及直接测序法等[14]。这些方法操作繁琐耗时、效率低下,限制了宏基因组学的推广及应用,目前主要应用高通量测序技术对宏基因组学基因文库直接测序并筛选出目的基因。
2.4.3 高通量测序。高通量测序技术即下一代测序技术(NGS),是指能够在短时间内对几十万甚至几百万条DNA分子序列进行测定的技术。2005年底,美国454公司推出了基于焦磷酸测序法的高通量测序系统(Genome Sequencer 20 System),开启了高通量测序新纪元。时至今日,Roche、Illumina、Life Technologies、Pacific Biosciences 等公司相继推出了各自的高通量测序系统。其技术原理主要包括焦磷酸测序、边合成边测序、基于磁珠的大规模并行连接测序、单分子荧光测序、半导体测序、单分子实时测序等。Illumina公司于2014年推出基于边合成边测序的HiSeqX Ten系统,拥有十台测序仪,每台仪器3天内可输出高达1.8 Tb的数据量。2017年1月,Illumina公司宣布推出NovaSeq系列测序仪,该仪器具有可扩展的通量和测序灵活性的特点,在应用方面与HiSeqX Ten互补。技术的不断进步与完善,使得测序成本不断下降。由于该技术对标本类型及标本中所含有的核酸信息不具偏见性,因此被广泛应用于新病原的发现。目前应用广泛的高通量测序平台主要有5种,包括HiSeq/Miseq、454、SOLID 、Ion Torrent、PacBio(表1)。这5种方法在误差率、读取长度、产量、运行成本和预期应用等方面存在差异,每个系统都有其自身的优缺点[15]。研究人员可根据需求选择合适的系统进行测序。
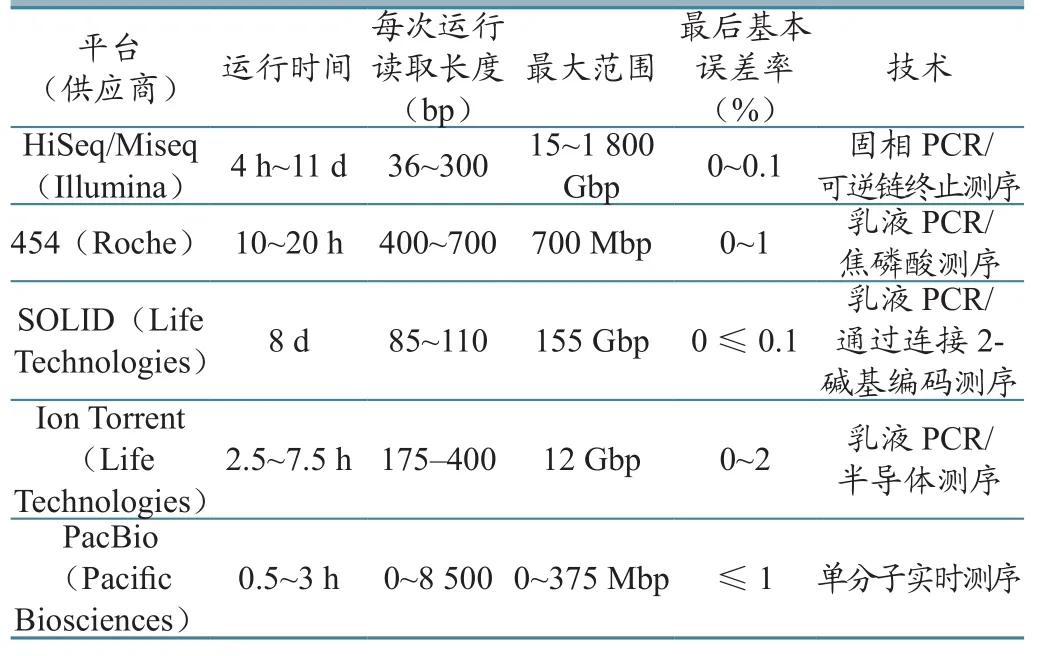
表1 5种NGS系统平台比较
2.4.4 序列的生物信息学分析。病毒宏基因组的分析流程包括原始数据的预处理、基因组装、功能分类和基因预测等[16]。高通量测序获得的结果需要专门的软件来处理,如RAMMCP和MetaStats[17],通过与数据库比对才能找出目标序列,而且得到的数据量十分庞大。运用RAMMCP和MetaStats等软件去除序列中低质量的和重复的序列后,使用SNAP 在 GenBank 的非冗余核苷酸序列数据库(Nonredundant Database),将获得的所有核酸序列进行比对,E value≤0.000 01 的序列作为有意义的序列,按照注释的物种信息去掉人、鼠、噬菌体、真菌等有关序列后,分析余下数据。对于发现的新病毒可以将单个序列和contigs在blastn的nr数据库中比对,如果比对结果无法判断出是何种序列,则在blastx的nr数据库中比对[18]。
2.4.5 病毒宏基因组学的应用。2002年,Breitbart等[19]最早应用宏基因组学理论分析病毒群落,他应用鸟枪测序法首次对美国加州海岸的两处海洋中病毒群体进行研究,发现374~7 114个潜在的新病毒。2003年,Breitbart等[20]应用宏基因组学分析人粪便中不能培养的病毒群落,并且在该研究中首次提到研究病毒群落方面的宏基因组学这一概念,目前,宏基因组学检测技术在新发动物疫病的诊断方面已得到广泛应用。2011年,Hoffmann[21]利用宏基因组测序的方法在患病母牛的血液中发现了新发病病毒——施马伦贝格病毒。2012年,YANG X等[22]用病毒宏基因组学方法系统地研究了中国6个不同地区的食虫蝙蝠粪便样品中的病毒,发现蝙蝠肠道中存在有大量浓核病毒、双顺反子病毒、冠状病毒等,证明蝙蝠肠道有大量新的昆虫病毒、哺乳动物病毒,推测蝙蝠在这些病毒的传播中起着重要的作用。2016年,Wang等[23]利用病毒宏基因组学方法从中国西南部马蹄蝠的直肠拭子和尿液中发现1株新型冠状病毒。2017年,Palinski等[24]利用宏基因组测序的方法在母猪和仔猪中确诊了新型的猪圆环病毒——PCV3。
5 口岸检疫中的应用展望
随着我国经济及贸易的不断发展,我国畜禽动物的贸易量逐年增大,进境动物的种类和数量不断增多,进境畜禽动物携带外来及新发疫病的风险与日俱增,给经济发展、人民健康和社会安定带来严重威胁。国境口岸是国际间动物及动物产品贸易的首站,是新发动物疫病传入的第一个关口。据不完全统计,仅2015年我国进口种牛超过13万头,进口种羊超过1.7万只,种猪约5 000头,马约1 700匹。由于对未知病原缺乏检测手段,导致未知病原随着进口动物及动物产品传入的可能性增大,对我国养殖业的安全及国民经济的稳定是一个巨大的威胁。
口岸检疫的要求是“检得出、检得准、检得快”,用传统的检测方法来检测新发病存在诸多问题。病原的分离涉及到操作活病毒和动物试验,操作繁琐,耗时长,且对口岸检疫的实验室提出了进一步的要求,一些地方性口岸很难达到生物安全资质的要求。血清学检测方法中,ELISA检测只能对病原做初步诊断,无法实现“检得准”的目标;IFA试验也由于操作繁琐且对试验操作人员要求较高,而逐渐将被稳定克隆细胞系的方法所代替[25]。电镜观察能实现“检得准”的要求,但是受活病毒操作资质的限制,对试验及分析人员也提出很高的要求。
相较于传统检测方法,快速、便捷且高通量的新型分子生物学检测方法在口岸检测中具备巨大的应用前景。采用序列非依赖扩增技术与病毒宏基因组学相结合的方式,通过序列非依赖扩增技术提高病毒丰度,利用病毒宏基因组学对样本中可能携带的病毒进行系统性的分析和鉴定,能够实现“检得出、检得准”的要求。随着该技术的不断发展以及新的检测试剂及策略的制定,该方法的检测时间也将大大缩短。将该方法应用于口岸进行新发动物疫病的检测把关,对将疫情挡于国门之外,并进行疫情早期防控,防治方案研究等都具有十分重要的意义。
[1] SMITH R D. Responding to global infectious disease outbreaks:Lessons from SARS on the role of risk perception,communication and management[J]. Social science & medicine,2006,63(12):3113-3123.
[2] SUTTLE C A. Viruses in the sea[J]. Nature,2005,437,7057:356-361.
[3] YU X J,LIANG M F,ZHANG S Y,et al. Fever with Thrombocytopenia Associated with a Novel Bunyavirus in China[J]. New England journal of medicine,2011,364(16):1523-1532.
[4] FROUSSARD P. A random-PCR method(rPCR) to construct whole cDNA library from low amounts of RNA[J]. Nucleic acids research,1992,20(11):2900.
[5] LI L,BARRY P,YEH E,et al. Identification of a novel human gammapapillomavirus species[J]. Journal of general virology,2009,90(Pt 10):2413-2417.
[6] LI L,VICTORIA J,KAPOOR A,et al. A novel picornavirus associated with gastroenteritis[J]. J Virol,2009,83(22):12002-12006.
[7] REYES G R,KIM J P. Sequence-independent,single primer amplification(SISPA) of complex DNA populations[J]. Mol cell probes,1991(5):473-481.
[8] KIRKLAND P D,FROST M J,FINLAISON D S,et al. Identification of a novel virus in pigs-Bungowannah virus:a possible new species of pestivirus[J]. Virus res,2007,129(1):26-34.
[9] KREUZE J F,PEREZ A,UNTIVEROS M,et al. Complete viral genome sequence and discovery of novel viruses by deep sequencing of small RNAs:A generic method for diagnosis,discovery and sequencing of viruses[J]. Virology,2009,388(1):1-7.
[10] STARK T J,ARNOLD J D,SPECTOR D H,et al. High-Resolution Profiling and Analysis of Viral and Host Small RNAs during Human Cytomegalovirus Infection[J]. Journal of virology,2012,86(1):226-235.
[11] HANDELSMAN J,RONDON M R,BRADY S F,et al. Molecular biological access to the chemistry of unknown soil microbes:a new frontier for natural products[J]. Cell chemical biology,1998,5(10):245-249.
[12] LI L,DENG X,MEE E T,et al. Comparing viral metagenomics methods using a highly multiplexed human viral pathogens reagent[J]. Journal of virological methods,2015,213:139-146.
[13] DATTA S,BUDHAULIYA R,DAS B,et al. Nextgeneration sequencing in clinical virology:Discovery of new viruses[J]. World journal of virology,2015,4(3):265-276.
[14] NG T F,MANIRE C,BORROWMAN K,et al. Discovery of a novel single-stranded DNA virus from a sea turtle fibropapilloma by using viral metagenomics[J]. Journal of virology,2009,83(6):2500-2509.
[15] HEAD S R,KOMORI H K,LAMERE S A,et al. Library construction for next-generation sequencing:overviews and challenges[J]. Biotechniques,2014,56(2):61-92.
[16] 浦绍艳,张鑫磊,蒋太交,等. 宏基因组学研究方法及其在病原体检测中的应用[J]. 生物物理学报,2014,30(1):3-14.
[17] WHITE J R,NAGARAJAN N,POP M. Statistical methods for detecting differentially abundant features in clinical metagenomic samples[J]. Plos computational biology,2009,5(4):e1000352.
[18] HOLTZ L R,FINKBEINER S R,KIRKWOOD C D,et al. Identification of a novel picornavirus related to cosaviruses in a child with acute diarrhea[J]. Virology journal,2008,5(1):1-5.
[19] BREITBART M,SALAMON P,ANDRESEN B,et al. Genomic analysis of uncultured marine viral communities[J]. Proceedings of the national academy of sciences of the United States of America,2002,99(22):14250-14255.
[20] BREITBART M,HEWSON I,FELTS B,et al. Metagenomic Analyses of an Uncultured Viral Community from Human Feces[J]. Journal of bacteriology,2003,185(20):6220-6223.
[21] HOFFMANN B,SCHEUCH M,HÖPER D:et al. Novel orthobunyavirus in Cattle,Europe,2011[J]. Emerging infectious diseases,2012,18(3):469-472.
[22] YANG X,ZHANG Y,GE X,et al. A novel totiviruslike virus isolated from bat guan [J]. Archives of virology,2012,157(6):1093-1099.
[23] WANG L,FU S,CAO Y,et al. Discovery and genetic analysis of novel coronaviruses in least horseshoe bats in southwestern China[J]. Emerging microbes & infections,2017,6(3):e14.
[24] PALINSKI R,PIÑEYRO P,SHANG P,et al. A novel porcine circovirus distantly related to known circoviruses is associated with porcine dermatitis and nephropathy syndrome and reproductive failure[J]. Journal of virology,2016,91(1):16.
[25] 张永宁,吴绍强,宋姗姗,等. 稳定表达施马伦贝格病毒核衣壳蛋白的BHK-21细胞系的建立[J]. 中国预防兽医学报,2015,37(6):438-443.
(责任编辑:杜宪)
The Detection Methods of New Animal Diseases and the Application Prospect in Quarantine Area
Du Fangyuan,Feng Chunyan,Liu Dandan,Qiu Songyin,Zhang Yongning,Lin Xiangmei,Wu Shaoqiang
(Chinese Academy of Inspection and Quarantine,Beijing 102600)
At present,some methods of disease detection in China's ports are mainly aimed at the known pathogens mentioned in the list of quarantine list of imported animals,and the detection methods for new pathogens are few. The detection methods of traditional pathogens,including isolation and culture of pathogens,serological methods,Western blotting and electron microscope observation,were introduced. The new methods of molecular biology detection,such as random primer PCR,sequence independent single primer amplification,small RNA deep sequencing technology,virus macro genomics technology,were described. And the application prospect of these methods in port was also analyzed.
new animal diseases:macro genome:high throughput sequencing:pathogen isolation:port quarantine
S862.23
B
1005-944X(2017)09-0071-05
10.3969/j.issn.1005-944X.2017.09.020
十二五科技支撑计划(2013BAD12B01);国家重点研发计划(2016YFD0501105)
并列第一作者:杜方原、冯春燕