蛋白质序列与蛋白质结构关系的研究
管维红
(江苏信息职业技术学院 物联网工程学院,江苏 无锡 214153)
蛋白质序列与蛋白质结构关系的研究
管维红
(江苏信息职业技术学院 物联网工程学院,江苏 无锡 214153)
蛋白质结构与功能的研究一直是分子生物学研究的热点之一.基于混沌理论对蛋白质序列特性进行研究,先将蛋白质序列转为时间序列,再对其进行相空间重构,通过计算确定时间延迟t、嵌入维数m等参数,最后计算得出蛋白质序列的最大Lyapunov指数,通过对蛋白质结构分类数据库SCOP中七类蛋白质结构的蛋白质序列最大Lyapunov指数计算和比较,发现蛋白质整体序列和蛋白质结构没有明显关联.
蛋白质序列;蛋白质结构;混沌;最大Lyapunov指数;关联
0 引言
分子生物学研究中还存在很多未解之谜,蛋白质结构和功能的研究还处在初始阶段,蛋白质结构和功能的预测一直是研究热点,其中蛋白质序列和蛋白质结构及功能之间是否存在某种关系也尚未明确[1].关于蛋白质序列的特性早期也曾有不少学者做过相关研究:有的认为蛋白质序列是随机的[2],有的认为蛋白质序列具有分形特征[3],笔者基于混沌理论并结合前人基础进行研究,给出蛋白质序列具有混沌特性的结论[4-5],并且先后完善了蛋白质序列最大Lyapunov指数的计算方法.关于蛋白质序列与结构的关系,也有学者给出一些结论,有的采用功率谱的方法对不同结构类蛋白质序列进行研究,认为不同结构类蛋白质序列具有与它们的结构类相对应的不同的关联特性[6].有的发现蛋白质序列具有隐含的与结构相同的对称性,序列的对称性可能决定结构的对称性[7].
ANFINSEN曾通过实验证明:一般情况下,蛋白质能够自发折叠形成特定的结构构象,也就是说,蛋白质的结构信息就蕴含于其序列之中[8].本文首先基于混沌理论计算蛋白质结构分类数据库SCOP(Structural Classification of Proteins,蛋白质结构分类)[9]中的七大类结构的蛋白质序列的最大Lyapunov指数,通过对七类结构蛋白质序列的最大Lyapunov指数的对比,分析研究蛋白质序列和蛋白质结构的关系.
1 蛋白质序列的选取
蛋白质分类数据库很多,最为常用的蛋白质结构分类数据库是SCOP和CATH(class, architecture, topology, homology).本研究蛋白质序列来自蛋白质结构分类数据库SCOP,是由MRC(Medical Research Council,英国医学研究委员会)的分子生物学实验室和蛋白质工程研究中心开发和维护[9].SCOP数据库利用计算机程序自动监测和人工验证结合的方法,把PDB(protein data bank,蛋白质结构数据库)中的蛋白质按传统分类方法分成α型、β型、α/β型、α+β型以及多结构域蛋白、膜蛋白和细胞表面蛋白、小蛋白,一共七大类,在此基础上再按折叠类型、超家族、家族三个层次逐级分类[9].蛋白质序列按照七大类结构依次从SCOP数据库中任意选取若干条,选取的原则是长度适合,便于下载和获取.
蛋白质由氨基酸组成,蛋白质序列中常见的氨基酸有20种.对蛋白质序列进行研究,首先要把序列转化为时间序列,即对每种氨基酸进行数值化,这里采用的是EIIP(electron interaction potential,电子—离子相互作用势)匹配法[10],氨基酸的电子—离子相互作用势是一种表示氨基酸价电子平均能量的物理属性,与蛋白质的生物性质有很大关联[10-11].与其他氨基酸对应数值的映射相比,EIIP匹配法对于蛋白质序列分析是最为合适的[12].每个氨基酸对应的EIIP值见表1.
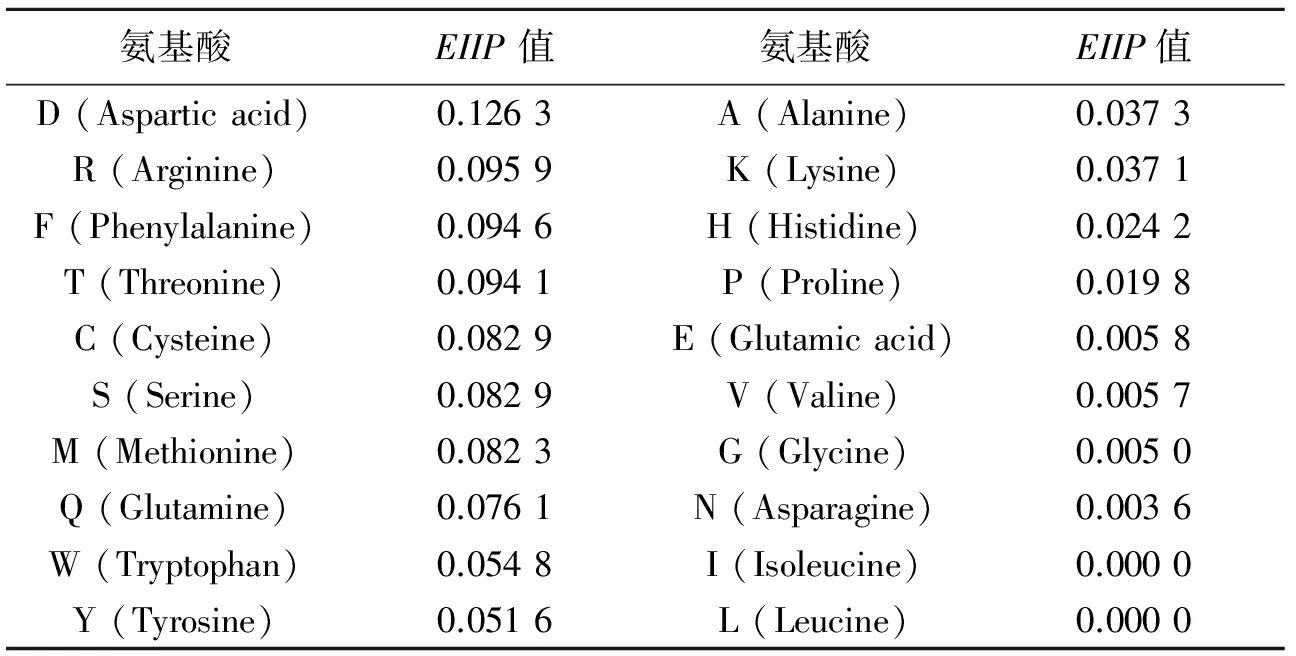
表1 20种氨基酸对应的EIIP值Tab.1 EIIP values for 20 amino acids
2 蛋白质序列混沌特性的计算方法
蛋白质序列混沌特性的计算方法,在笔者以前的文章中曾详细描述计算步骤并举例说明[5],这里只作简单介绍.蛋白质序列转化为时间序列后,要对其进行相空间重构,采用时间坐标延迟法[13].设蛋白质时间序列为{xi},i=1,2,…,N,N为蛋白质时间序列总长,时间延迟t,嵌入维数m,Yj为相空间中的一点,Yj=(xj,xj+t,xj+2t,…,xj+(m-1)t),j=1,2,…,n,n=N-(m-1)t.首先需要确定时间延迟t和嵌入维数m.
2.1 时间延迟t的计算
时间延迟t采用去偏自相关法确定[13].对于一个时间序列{xi},i=1, 2, …,N,去偏自相关函数定义为[13-14]
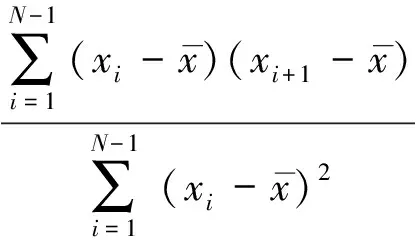
(1)
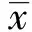
2.2 嵌入维数m的计算
嵌入维数m的选择采用G-P算法[13,15]:对于时间序列{xi},i=1,2,…,N,先尝试取一个较小的嵌入维数m0,则对应的相空间为Yj=(xj,xj+t,xj+2t,…,xj+(m0-1)t),j=1,2,…,n,n=N-(m0-1)t.计算其关联函数
(2)
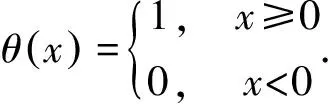
C(r)的值与r的取值有关,在实际应用中,通过给定一些m,r的值进行计算.对于蛋白质时间序列,经前期实验,r依次取1,0.9,0.8,…,0.1,嵌入维数m取1,2,3,4,5,6,7,8,…,可以得到比较好的结果[5].当lnC(r)-lnr的曲线图上通过最佳拟合得到该直线斜率即为关联维数D,即C(r)=rD,则
(3)
一般随着m增大,关联维数D也相应增大,当m增大到一定值M时,D达到峰值,则M即为嵌入维数.如果D随m的增长而增长,不收敛于一个稳定的值,表明该序列具有随机性,它在有限维的相空间中不存在吸引子[13-14].
2.3 最大Lyapunov指数的计算
最大Lyapunov指数的计算采用改进的最大Lyapunov指数的方法[13-14].设有一条蛋白质时间序列{xi},i=1,2,…,N,通过时间延迟t和嵌入维数m重构相空间,相空间中的点为Yj=(xj,xj+t,xj+2t,…,xj+(m-1)t),j=1,2,…,n,n=N-(m-1)t.任意选取两相邻初始位置(通常取初始点与其最邻近点),计算两点间的初始距离d1(0),计算经过时间延迟t演化后两点间的距离为d1(t),则可得
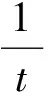
(4)
接着选取第j个点,计算第j个点与其最近邻之间的距离dj(0),再计算经过时间t后两点间的距离变为dj(t),则
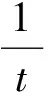
(5)
所以最大Lyapunov指数为
(6)
该方法十分简单,而且充分利用了所有数据,是一种既简便又适用于小数据量的方法[13].当蛋白质时间序列的最大Lyapunov指数λ1大于0时,即说明蛋白质时间序列具有混沌特性[13-14].
3 计算结果
从蛋白质结构分类数据库SCOP中按α型、β型、α/β型、α+β型以及多结构域蛋白、膜蛋白和细胞表面蛋白、小蛋白这七大类分别随机选取多条蛋白质序列,每类随机给出10条计算结果,为了便于比对分析,依次给出时间延迟t、嵌入维数m、关联维数D以及最大Lyapunov指数λ1,并且按最大Lyapunov指数λ1从小到大的顺序进行排序.具体结果见表2~表8.
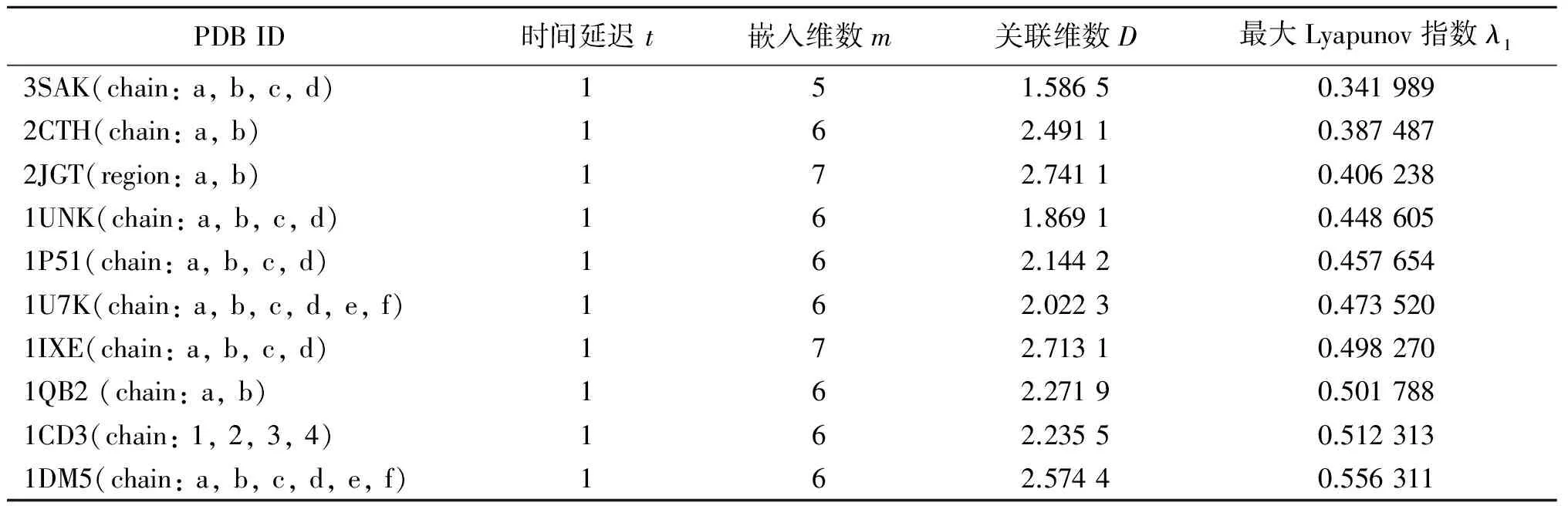
表2 α型结构的蛋白质时间序列的分析结果Tab.2 The analysis results of all alpha protein time sequences
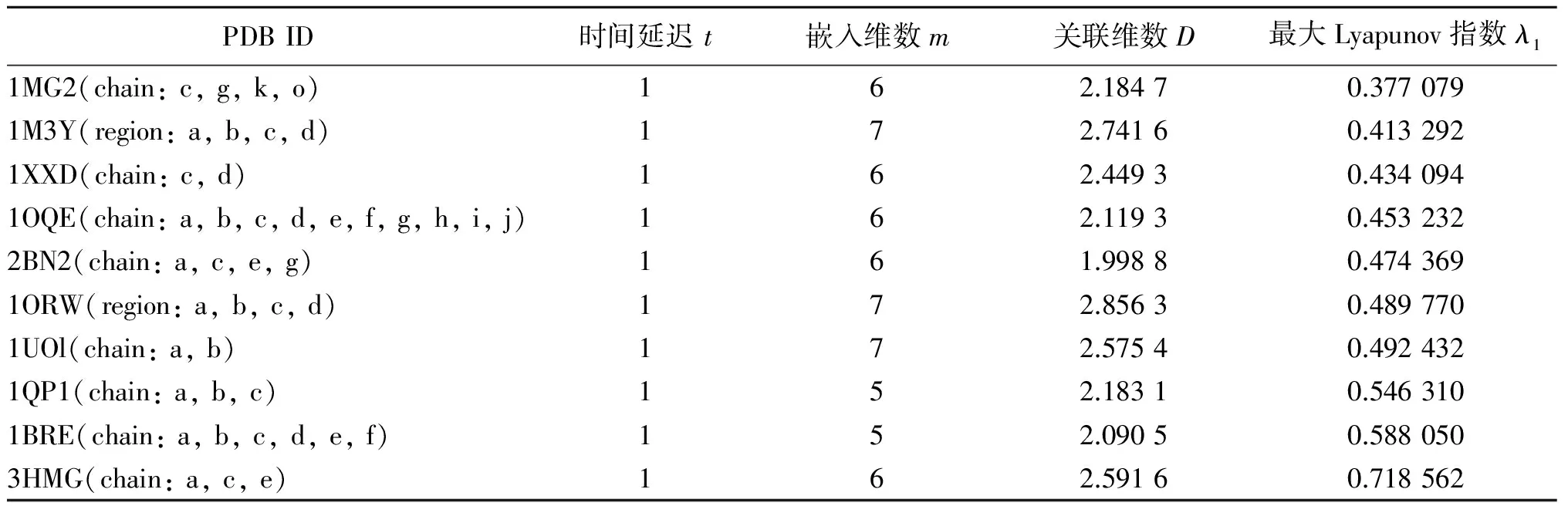
表3 β型结构的蛋白质时间序列的分析结果Tab.3 The analysis results of all beta protein time sequences
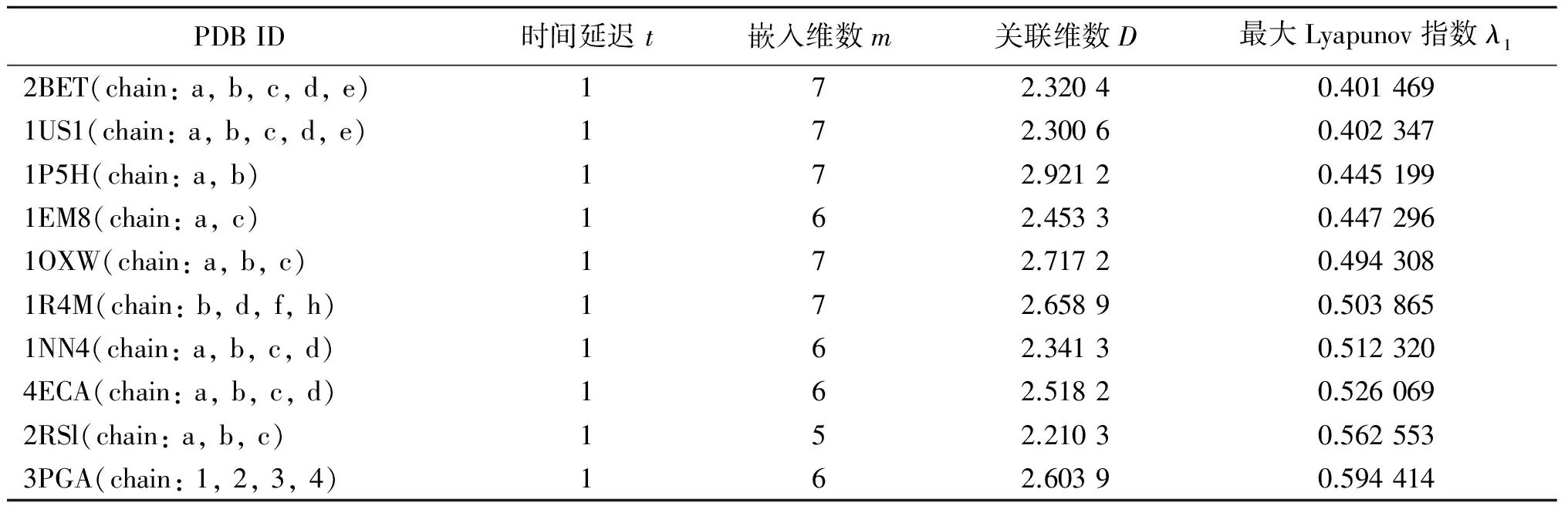
表4 α/β型结构的蛋白质时间序列的分析结果Tab.4 The analysis results of alpha and beta proteins (a/b) protein time sequences
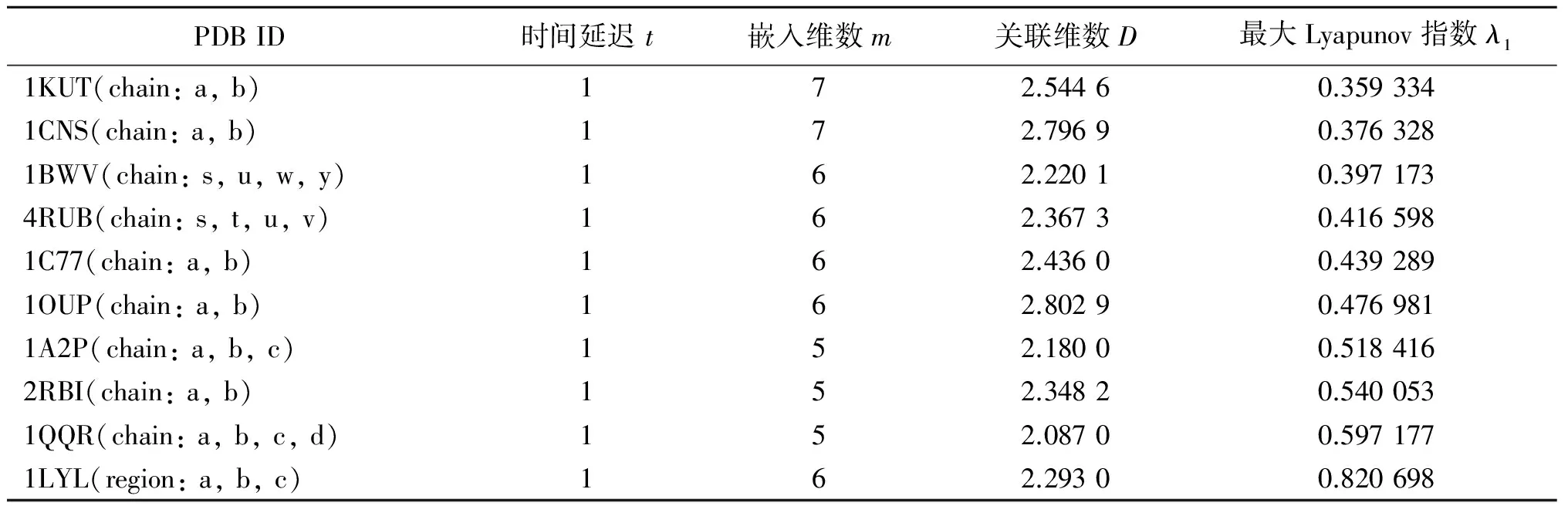
表5 α+β型结构的蛋白质时间序列的分析结果Tab.5 The analysis results of alpha and beta proteins (a+b) protein time sequences
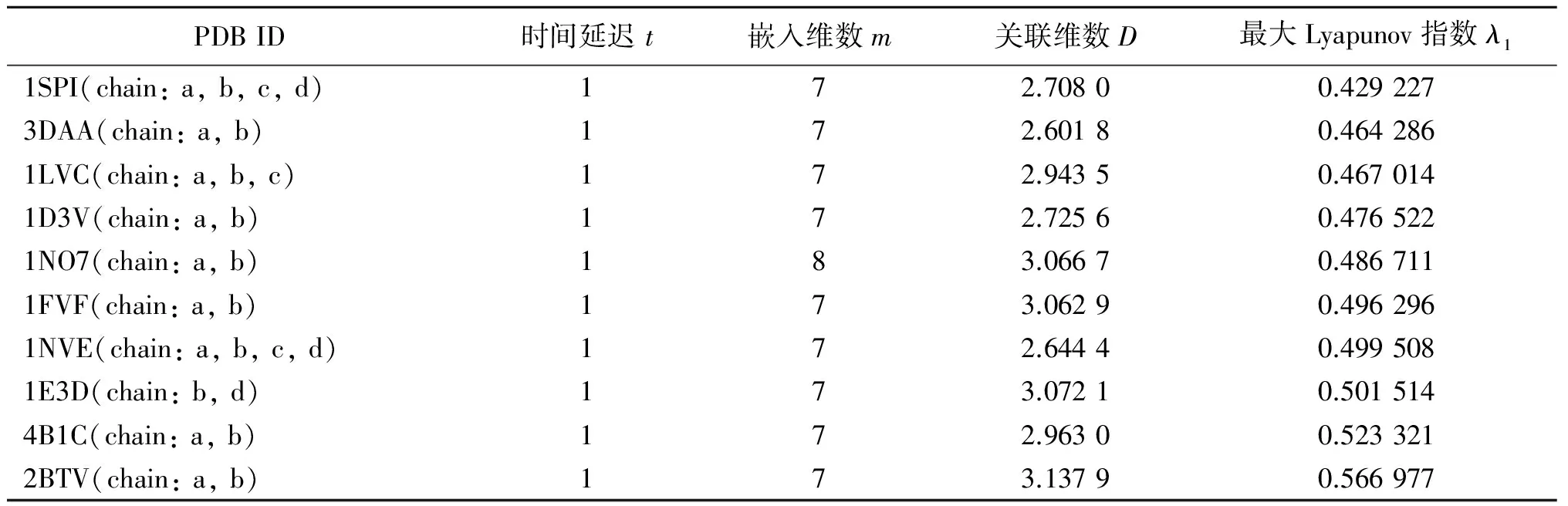
表6 多结构域蛋白结构的蛋白质时间序列的分析结果Tab.6 The analysis results of Multi-domain proteins (alpha and beta) protein time sequences

表7 膜蛋白和细胞表面蛋白结构的蛋白质时间序列的分析结果Tab.7 The analysis results of membrane and cell surface proteins and peptides protein time sequences

续表7 膜蛋白和细胞表面蛋白结构的蛋白质时间序列的分析结果Tab.7(Continued) The analysis results of Membrane and cell surface proteins and peptides protein time sequences
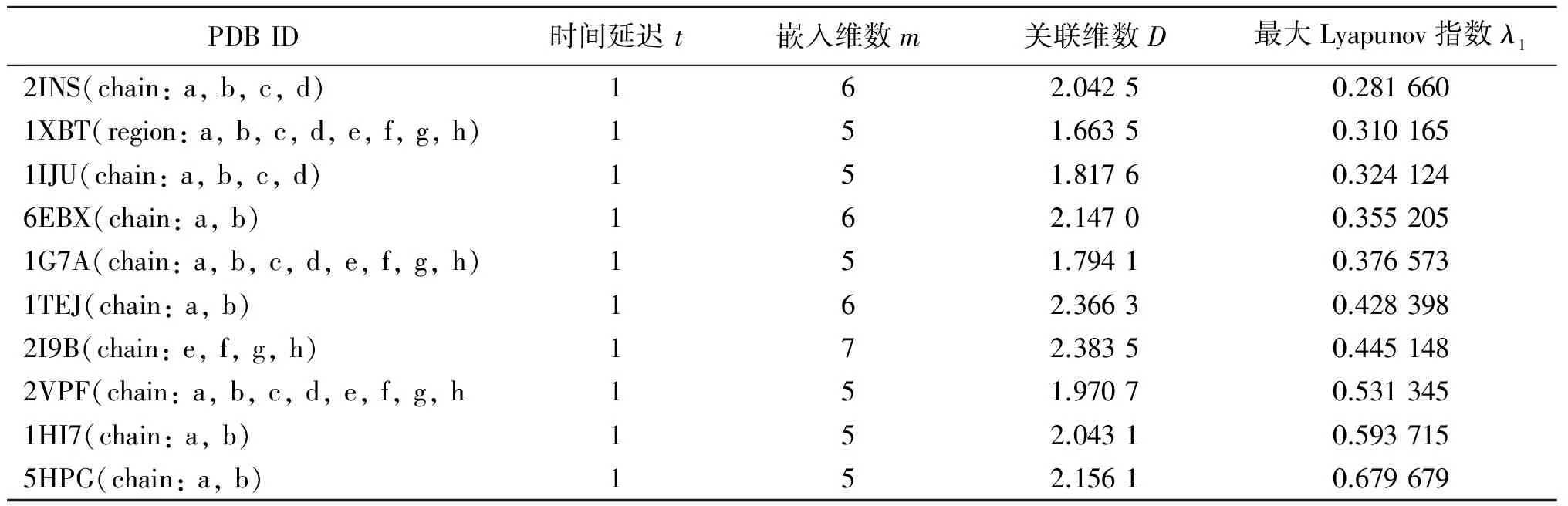
表8 小蛋白结构的蛋白质时间序列的分析结果Tab.8 The analysis results of Small protein time sequences
从表2~表8中可以看出,每条蛋白质序列的延迟时间t都为1,这和之前的研究结果一致,说明每个氨基酸都是独立的个体, 都可能对蛋白质时间序列系统造成影响[5,8].嵌入维数m的值总体在[5,8]这个区间上,每类结构嵌入维数m的值都不同,α型、β型大多为6,α/β型6、7几乎各占一半,α+β型则是集中在5、6,多结构域蛋白几乎都为7,膜蛋白和细胞表面蛋白比较分散,从5到8都有,小蛋白则大多为5.关联维数D最小值为1.586 5,最大值为3.137 9,每类结构的值总体差异不大,最大Lyapunov指数值λ1的数值分布及差异也不大,为了便于观察,现把每类结构的最大Lyapunov指数值λ1集中在一张图上进行对比,见图1.

图1 七种结构的蛋白质时间序列最大Lyapunov指数的对比图Fig.1 The comparison of the largest Lyapunov exponents for seven structures of protein time sequences
从对比图可以看出七种结构的蛋白质时间序列的最大Lyapunov指数值区间非常接近,除了小蛋白的值偏低,以及α+β型有一个最大Lyapunov指数值较为偏高,总的来说蛋白质结构及功能的差异和蛋白质时间序列表现出的混沌特性之间的差异相比要大得多,蛋白质整体序列和蛋白质结构之间没有发现明显关联.
4 结束语
首先从蛋白质结构分类数据库SCOP中按照七大结构分类分别随机选取多条蛋白质序列,并且通过氨基酸的电子—离子相互作用势EIIP值转换为时间序列,再对其进行相空间重构,利用去偏自相关系数和经典G-P算法分别得出时间延迟t和嵌入维数m两个最为关键参数,最后通过改进的最大Lyapunov指数计算方法计算序列的最大Lyapunov指数值.在此说明,在计算中也还是有一些序列不收敛[5],没有吸引子,无法计算最大Lyapunov指数值,文中没有列出这些序列,占比很少,可能和蛋白质序列准确性以及本身特性有关.
通过对七大类蛋白质结构最大Lyapunov指数值的对比发现,蛋白质整体序列和蛋白质结构没有明显关联.对于想从蛋白质整体序列上进行研究推测蛋白质结构的做法不大可行.蛋白质整条序列是混沌的,蛋白质序列里一些短的片段可能蕴含结构信息,以促使蛋白质自发进行折叠形成特定的结构,以行使其生物学功能.蛋白质结构和蛋白质某些序列到底具有怎样的关联性,有很多蛋白质序列很相似,但是结构和功能差异非常大,这个差异可能就在某些关键的氨基酸或者氨基酸组合上.这需要结合蛋白质序列和蛋白质结构在大量的数据基础上再作进一步研究.
[1] 叶子弘.生物信息学[M].浙江: 浙江大学出版社, 2011:2-16.
[2] HUANG Y Z, XIAO Y. Nonlinear deterministic structures and the randomness of protein sequences[J]. Chaos, Solitons and Fractals, 2003, 17(5): 895-900.
[3] GOPAKUMAR G, NAIR A S. Lacunarity analysis of protein sequences reveal fractal like behavior of amino acid distributions[J]. Advances in computing and communications in computer and information science, 2011, 190(4):320-327.
[4] 管维红,张立婷, 徐振源,等. 蛋白质序列混沌特性的研究[J].生物信息学, 2008, 6(4): 148-151.
[5] 管维红.基于混沌理论的蛋白质序列特性的研究[J].生物信息学, 2012,10(3):194-198.
[6] 黄敏,沈晖,肖奕.不同结构类蛋白质序列中的关联特性[J].生物物理学报, 2000,16(4):755-759.
[7] 肖奕, 冯建辉, 黄延昭.对称蛋白质序列与结构关系研究[J].生命科学, 2010, 22 (11) :1129-1137.
[8] ANFINSEN C B. Principles that govern the folding of protein chains[J]. Science,1973,181:223-230.
[9] Medical Research Council. Structural Classification of Proteins[DB/OL]. 2009-2-23[2016-3-1]. http://scop.mrc-lmb.cam.ac.uk/scop/data/scop.b.html.
[10] TRAD C H, FANG Q, COSIC I. Protein sequence comparison based on the wavelet transform approach[J]. Protein Engineering, 2002, 15(3): 193-203.
[11] SAMBUK N, KONJEVODA P, POKRIC B, et al. Resonant recognition model defines the secondary structure of bioactive proteins[J]. Croatica Chemica Acta, 2002, 75(4): 899-908.
[12] 刘宏德, 孙啸. 蛋白质序列的特征周期研究[J].生物物理学报, 2008, 24 (2) :145-154.
[13] 陈士华, 陆君安. 混沌动力学初步[M]. 武汉: 武汉水利大学出版社, 1998:95-106.
[14] 黄延林, 韩晓刚, 卢金锁. 基于Lyapunov指数的混沌预测方法及在水质预测中的作用[J]. 西安建筑科技大学学报 (自然科学版), 2008, 40(6): 846-851.
[15] 王卫宁, 汪秉宏, 史晓平. 股票价格波动的混沌行为分析[J]. 数量经济技术经济研究, 2004, 21(4): 141-147.
ResearchontheRelationshipBetweenProteinSequencesandProteinStructures
GUAN Weihong
(InternetofThingsEngineeringDepartment,JiangsuCollegeofInformationTechnology,Wuxi214153,China)
The research on protein structure and function is one of the hotspots in molecular biology. The characteristics of protein sequences based on chaos theory is studied. Firstly, the protein sequences are transformed into numerical time series, then reconstructed phase space for the series, and calculated the related parameters of the system: delay timetand embedding dimensionm, at last the largest Lyapunov exponent is calculated by improved maximal Lyapunov exponent method. Through the calculation and comparison in the largest Lyapunov exponent of seven structures protein sequences from SCOP (Structural Classification of Proteins) database, it is found that the whole protein sequence and protein structure has no obvious correlation.
protein sequence; protein structure; chaos; the largest Lyapunov exponent; correlation
2017-10-22
江苏省现代教育技术资助项目(2016-R-48107,2016-R-48055)
管维红(1983—),女,江苏连云港人,江苏信息职业技术学院物联网工程学院实验师.
10.3969/j.issn.1007-0834.2017.04.009
Q516
A
1007-0834(2017)04-0037-06
——兼析少数民族地区官方微博综合绩效现状

