基于深度学习的肝脏肿瘤图像分割
黄佳佳+赵曙光+张笑青+杨峰+许方成

摘要:传统肝脏CT 图像肿瘤分割算法常需引进先验信息,分割效率低,无法满足临床实时性要求。针对这些问题,提出基于卷积-反卷积神经网络的肝脏肿瘤图像分割算法,自动提取肝脏肿瘤的特征。实验表明,DSC指标约85.32%,且分割每幅图像只需用时3至5秒,实时性好。
关键词:肝脏CT图像;肿瘤分割;反卷积网络;自动提取特征
中图分类号:TP391 文献标识码:A 文章编号:1007-9416(2017)11-0081-02
近年来,有许多算法实现肝脏及其肿瘤图像的分割,但这些算法存在的共性是在处理的过程中或多或少的需要人工干预。如Seung-Jin Park等[1]提出基于最优阈值的肝脏肿瘤图像分割,该方法的缺点是必须先将肝脏分割出来,然后才能对肿瘤进行分割。本文提出一种基于卷积-反卷积神经网络的肝脏肿瘤图像分割方法,能够自动分割肝脏肿瘤图像。
1 卷积神经网络
卷积神经网络(Convolutional Neural Networks,CNN)[2]是深度学习算法中的一种,CNN由多个层次组成的可训练的架构,每一层的输入和输出包含多个特征映射层,主要由若干个卷积层、池化层、激活函数层以及全连接层组成。
2 肝脏肿瘤图像分割方法
2.1 图像的预处理
如图1所示,(a)是原始的肝脏CT图像,在直方图均衡化之后的(b)中,肝脏肿瘤和其它组织的灰度值有明显的差异,这将有助于提取有效特征。由于图像采集时受到设备噪声的影响,CT图像中含有噪声。本文采用中值滤波器对其进行滤波,结果如(c)所示,滤波效果较好。
2.2 卷积-反卷积神经网络
如前文所述,由于卷积网络在池化过程中缩小了图片的尺寸,而对于图像分割的任务,最终需要得到与原图尺寸相同的结果,为了解决此问题,文献[3]引入反卷积网络,其主要由反卷积层和反池化层组成。
本文在卷积网络的基础上加上对称的反卷积网络,构成卷积-反卷积网络。在网络中,本文增加Batch normalization层[4],使模型尽可能地收敛到全局最优解。
3 实验与结果分析
3.1 實验数据集
本次实验采用MICCAI 2017数据集。本实验中从数据集中筛选出含有肿瘤的样本,得到训练图像4983张,测试图像2141张,尺寸为像素。
3.2 实验流程与评价指标
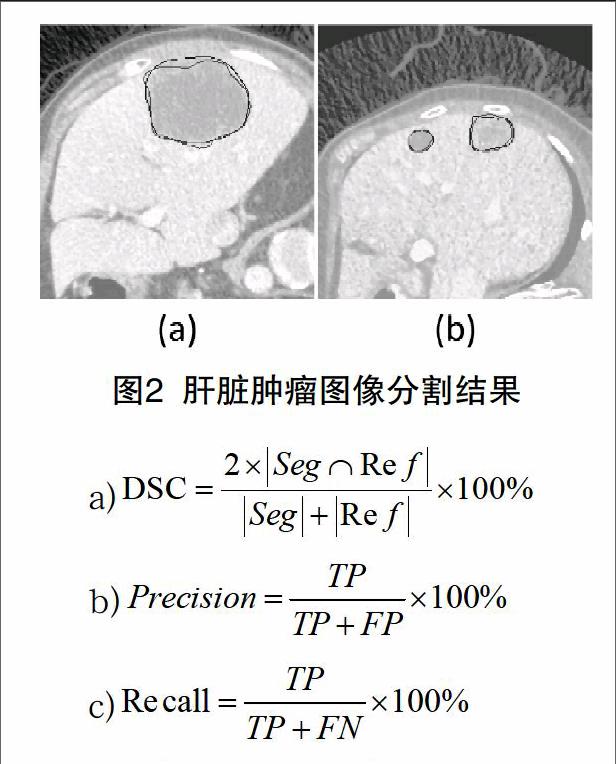
实验流程是先对图像进行预处理,再将预处理过后的图像作为训练集提供给卷积-反卷积网络进行训练。利用训练得到参数模型对测试图像进行分割之后,利用MATLAB对分割图进行后续处理能得到分割最终结果(如图2)。本实验的评价指标分别采用:a)Dice相似系数;b)准确率;c)召回率等医学图像分割的常用指标。Seg表示为算法分割结果,Ref为金标准,TP表示真阳性,FP表示假阳性。FN表示假阴性。
3.3 实验结果及分析
部分实验结果如图2所示。图中的蓝色的实线是标准分割的结果,红色的线条是本文的分割结果。从图中可看出,本文的肝脏肿瘤预测接近真实值,分割的准确度较高。
本文的方法与Adaboost[5]、Random Forests[6]等传统分割方法进行比较,在DSC(%)指标中Adaboost、Random Forests及本文的算法分别是77.22%、78.94%、85.32%。Precision(%)指标中三种算法的指标分别是77.34%、80.99%、86.53%。Recall(%)指标中三种算法的指标分别是82.91%、83.43%、85.37%。本文分割方法的效果好于其它方法。分割一幅图像只需2至3秒,速度明显快于其它算法。
4 结语
本文结合肝脏CT图像的特点,提出了一种基于全卷积-反卷积的肝脏肿瘤图像分割方法。该方法无需手工设计特征,自动提取的深层特征能很好地描述图像变化。经过实验证明,该方法对肝脏肿瘤图像分割具有较好的准确性和实时性。
参考文献
[1]Park Seung-Jin,Seo Kyung-Sik,Park Jong-An.Automatic hepatic tumor segmentation using statistical optimal threshold[J].Lecture Notes in Computer Science,2005,3514:275-283.
[2]Y.Le Cun,L.Bottou,Y.Bengio,P.Haffner.Gradient-based Learning Applied to Document Recognition. Proceedings of the IEEE, 86(11):2278-2324,1998.
[3]NohH,HongS,HanB.Learning deconvolution network for semantic segmentation[C]//The IEEE International Conference on Computer Vision .NewYork:IEEE,2015:1520-1528.
[4]S.Ioffe and C.Szegedy.Batch normalization:Accelerating deep network training by reducing internal covariate shift.arXiv preprint arXiv:1502.03167,2015.5.
[5]Y.Freund,R.E.Schapire. A Decision-theoretic Generalization of On-line Learning and An Application to Boosting.Journal of Computer and System Sciences,55,1997.
[6]L.Breiman.Random forests.Mach.Learning,45(1):5-32,2001.
Abstract:The traditional liver CT image segmentation algorithm often needs to introduce prior information, and the segmentation efficiency is low, which can not meet the real-time clinical requirements. To solve these problems, an algorithm of hepatic tumor segmentation based on convolution-deconvolution neural network is proposed to automatically extract features of liver tumors. Experiments show that the DSC index is about 85.32%, and the segmentation of each image takes only 3 to 5 seconds, good real-time.
Key Words:liver CT image; tumor segmentation; deconvolution network; automatic extraction of featuresendprint

