西藏青稞种质清蛋白和球蛋白遗传多样性分析
侯维海 王建林 胡 单 旦 巴
(西藏农牧学院高原作物分子育种实验室, 林芝 860000)
青稞(Hordeum vulgare Linn.var.nudum Hook.f.)又称裸大麦,是西藏高原最古老的作物之一,也是西藏高原第一大粮食作物,广泛分布于西藏高原多样的生态区,主要分布范围在北纬28°~30°,东经88°~94°,海拔1 900~4 200 m的河谷地带[1-3]。近年来,发现青稞具有丰富的膳食纤维、较高的保健组分和饲用价值,在酿酒、保健食品、再生能源等方面具有很大的开发潜力[4-5],随即青稞育种和利用逐渐受到人们重视。前人通过对西藏高原青稞种质资源表观性状调查、细胞学鉴定、生理生化分析等,提出了西藏高原大麦变异极其丰富,是世界栽培大麦多样性分布中心或初生起源中心之一[6-10]。随后,随着分子标记技术的发展,有学者利用RAPD、SSR、ISSR等分子标记技术开展了青藏高原青稞种质资源遗传多样性分析、亲缘关系预测和种质鉴定工作[11-15],但针对西藏高原青稞种质遗传多样性的评价和研究仍较为薄弱和有限。西藏高原是青稞资源重要的基因库,这里蕴藏着多种现代农业所急需的抗旱、抗寒、耐盐碱、高品质基因[16],加强对西藏本地青稞资源的研究将有利于青稞资源的保护和促进优质高抗青稞品种的选育和利用,有效缓解目前青稞遗传基础狭窄的困境,为西藏青稞育种亲本选择提供参考。
种子贮藏蛋白(Seed Storage Proteins,简称SSP)是指不具备代谢和结构功能的种子蛋白,其组成由基因决定,在品种间存在明显的差异,其电泳后条带的多少及组合方式受基因型控制,几乎不受外界环境因子的影响。因此,SSP在品种间组分上的差异可以反映基因型的不同,故被称为品种的生化“指纹”[17]。SSP已被作为遗传标记广泛应用于四个方面:材料间遗传多样性分析;鉴定基因组关系;遗传资源保存和育种相关的植物驯化;作为植物改良育种的手段[18]。基于SDS-PAGE种子蛋白质电泳已经成为一种有效工具解决植物分类、进化问题、品种和变种的鉴定、种质特征分析和补充评价相关信息等[19-20]。前人通过对小麦、大麦、辣椒、玉米、水稻、大豆,蚕豆、云豆等作物研究表明,不同生态类群、品种和材料间SSP是高度多态的,能够反映材料大量的遗传变异[21-23]。目前,有关青稞蛋白遗传多样性分析主要以醇溶蛋白进行研究,例如曾兴权[24]利用SDS-PAGE对347份西藏青稞农家种、野生近缘种和育成品种进行了醇溶蛋白的遗传多样性分析,发现青稞育成品种的遗传多样性指数最高,地方品种次之,野生近缘种最小。冯宗云等[25]对106份西藏野生二棱大麦、野生瓶形大麦和野生六棱大麦3个醇溶蛋白位点的遗传多样性分析表明,不同棱型大麦蛋白位点有一定差异。闫敏[26]分析了18份西藏青稞育成品种醇溶蛋白的遗传多样性,共分离出22种相对迁移率不同的醇溶蛋白谱带,认为西藏青稞育成品种具有较丰富的醇溶蛋白遗传多态性。迄今为止,仅零星描述西藏高原青稞醇溶蛋白遗传多样性的文章外,很少发现从种子清蛋白和球蛋白水平上研究西藏高原青稞种质变异程度。本研究以54份西藏青稞农家种和野生近缘种为材料,利用SDS-PAGE技术对种子蛋白多态性进行分析,旨在探索西藏高原青稞种质资源中蛋白亚基组成以及变异规律,筛选优质蛋白种质,为青稞资源开发和优良亲本选配提供参考。
1 材料和方法
1.1 材料
参试材料为西藏青稞农家种,野生近缘种共计54份,由西藏农牧学院农学教研室于2015年从西藏各县市偏远农村搜集获得,并就地搜集经度、纬度、海拔高度等地理信息数据(表1)。2016年3月20日在西藏农牧学院农场对54份参试材料进行大田种植(北纬26°52′~30°40′,东经92°09′~98°47′,海拔2 900 m),小区面积2 m×3 m=6 m2,随机区组排列,3次重复,采用常规田间管理措施,于材料成熟后,单独采集5个麦穗,分类保存,自然晾干后脱粒,放通风干燥处保存备用。
1.2 主要仪器设备
台式冷冻离心机:美国Beckman Coulter(贝克曼库尔特)公司;DYCZ-24B型电泳仪:北京六一仪器厂。
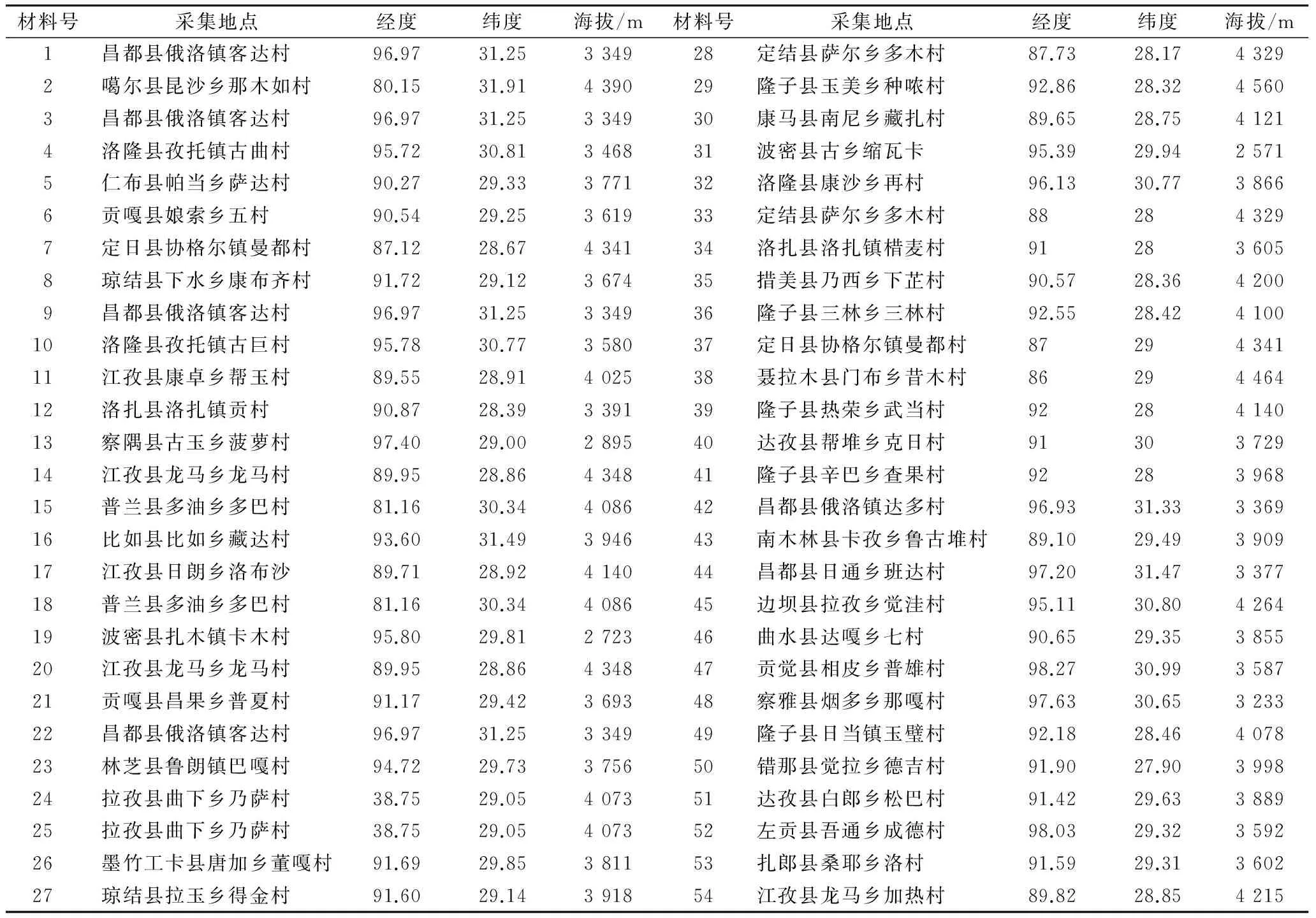
表1 54份西藏青稞种质材料基本信息
1.3 实验方法
1.3.1青稞清蛋白、球蛋白的分离
参考宋晓敏等[27]、刘玉皎等[28]清蛋白、球蛋白的分离提取方法并作适当改动。取4~5粒青稞种子置于预冷的研钵,捣碎并快速充分研磨至粉状,称取0.3 g青稞粉移入2 mL离心管,加入1.5 mL蒸馏水涡旋振荡混匀,置于4 ℃冰箱提取30 min,期间每5 min充分振荡混匀1次。6 500 r/min离心20 min,取上清液移入新离心管,为清蛋白,放4 ℃保存备用。取0.3 g青稞粉移入2 mL离心管,加入1.5 mL 1 mol/L的NaCl溶解,放4 ℃冰箱提取30 min,期间每5 min充分振荡混匀1次。6 500 r/min离心20 min,取上清液置4 ℃冰箱透析24 h得干粉,离心,用40 μL 1 mol/L的NaCl溶解干粉,为盐容性蛋白,放4 ℃保存备用。
1.3.2青稞清蛋白、球蛋白的SDS-PAGE电泳
分别取40 μL清蛋白和球蛋白,各加入10 μL上样缓冲液(250 mmol/L,pH 6.8的Tris-HCl、10%SDS、0.5%溴酚蓝、50%甘油、5% mLβ-巯基乙醇),于100 ℃烘箱煮沸5 min,冷却后,离心20 s,进行点样。电泳采用不连续聚丙烯酰胺凝胶电泳,浓缩胶浓度5%,分离胶浓度10%,以Tris-甘氨酸作为电极缓冲液,每份样品上样量为30 μL,凝胶厚度1.0 mm。在16 mA恒流条件下电泳,待指示剂迁移至凝胶底部后结束电泳。
清蛋白和球蛋白凝胶采用银染色法,固定液(50%无水乙醇+12%冰醋酸+0.1%甲醛+38 mL双蒸水)固定60 min;50%无水乙醇洗涤3次,5 min/次;敏化(0.02% Na2S2O3)1 min;双蒸水漂洗3次,1 min/次;银染(0.5% AgNO3,0.075%甲醇)20 min;双蒸水漂洗2次,1 min/次;显色(6% Na2CO3,0.000 4 Na2S2O3+0.05% HCHO)至带型隐约看见,立即停止染色;终止反应(50%无水乙醇+12%冰醋酸)。
1.3.3数据统计分析
利用Quantity one软件自动读取青稞清蛋白和球蛋白电泳谱带,人工辅助修正,按照同一位置,蛋白电泳条带有,赋值为1,无赋值为0,生成0,1矩阵。通过NTSYS-pc 2.1软件对生成的0,1矩阵进行非加权类平均法进行聚类,计算遗传距离和遗传相似系数。利用popgen 32软件计算参试材料不同群见和不同群体内某一位点的等位变异数、等位变异频率、有效等位基因数、Shannon’s信息指数、基因多样性指数。通过Microsoft Excel计算出现频率。
2 结果与分析
2.1 青稞清蛋白的SDS-PAGE分析
各参试材料籽粒清蛋白SDS-PAGE电泳图见图1所示,蛋白谱带遗传多样性分析见表2和表3所示。参试材料经SDS-PAGE电泳共分离出26条迁移率不同的谱带,其范围在10~21条,平均每份材料清蛋白谱带条数为17.3条。蛋白谱带数因材料而异,其中6、23、24、33和42号青稞材料分离出的清蛋白谱带数最多,为21条,而7和43号材料谱带最少,为10条。各迁移率不同的谱带在参试材料中出现频率为25.93%~100%,其中6、9、21、24号谱带在参数材料中均有出现,其出现频率为100%,为共有带;13号谱带出现频率最低,为25.93%。参试材料有效等位基因数(ne)为1.0~1.99;多样性指数(H)为0~0.5;Shannon’s信息指数为0~0.69。这些数据表明54份来源不同的西藏青稞资源球蛋白谱带存在丰富的变异类型。
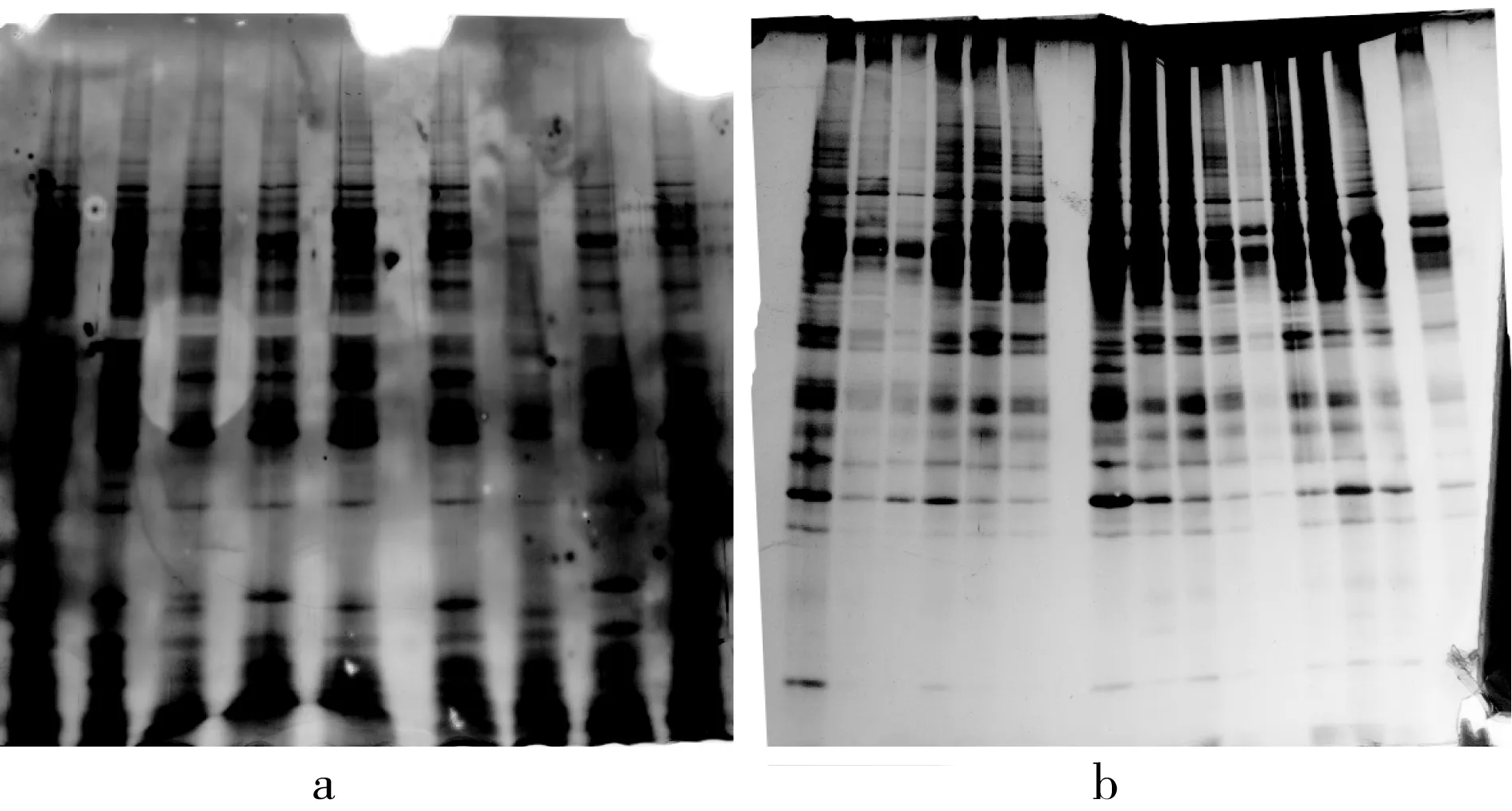
图1 西藏青稞材料籽粒清蛋白和球蛋白SDS-PAGE电泳
2.2 青稞球蛋白的凝胶电泳分析
参试材料球蛋白SDS-PAGE电泳图见图1a所示,球蛋白遗传多样性分析见表2所示。54份西藏青稞共分离出迁移率不同的蛋白谱带31条,变异范围为12~26条,平均每份材料蛋白谱带为21条,其中13、28和44号青稞材料分离出的球蛋白谱带数最多,为26条,而3、11、15和30号青稞材料分离出的蛋白谱带最少,为12或13条(表2、表4)。迁移率不同的31条谱带在54份参试材料中出现的频率为15.63%~100%,其中9、12、13、28、29号谱带在参数材料中均有出现,其出现频率为100%,为共有带;23号谱带出现频率最低,为15.63%。参试材料有效等位基因数(ne)、多样性指数(H)为0~0.5;Shannon’s信息指数与清蛋白数据相似。这些数据表54份来源不同的西藏青稞资源球蛋白谱带存在丰富的变异类型。
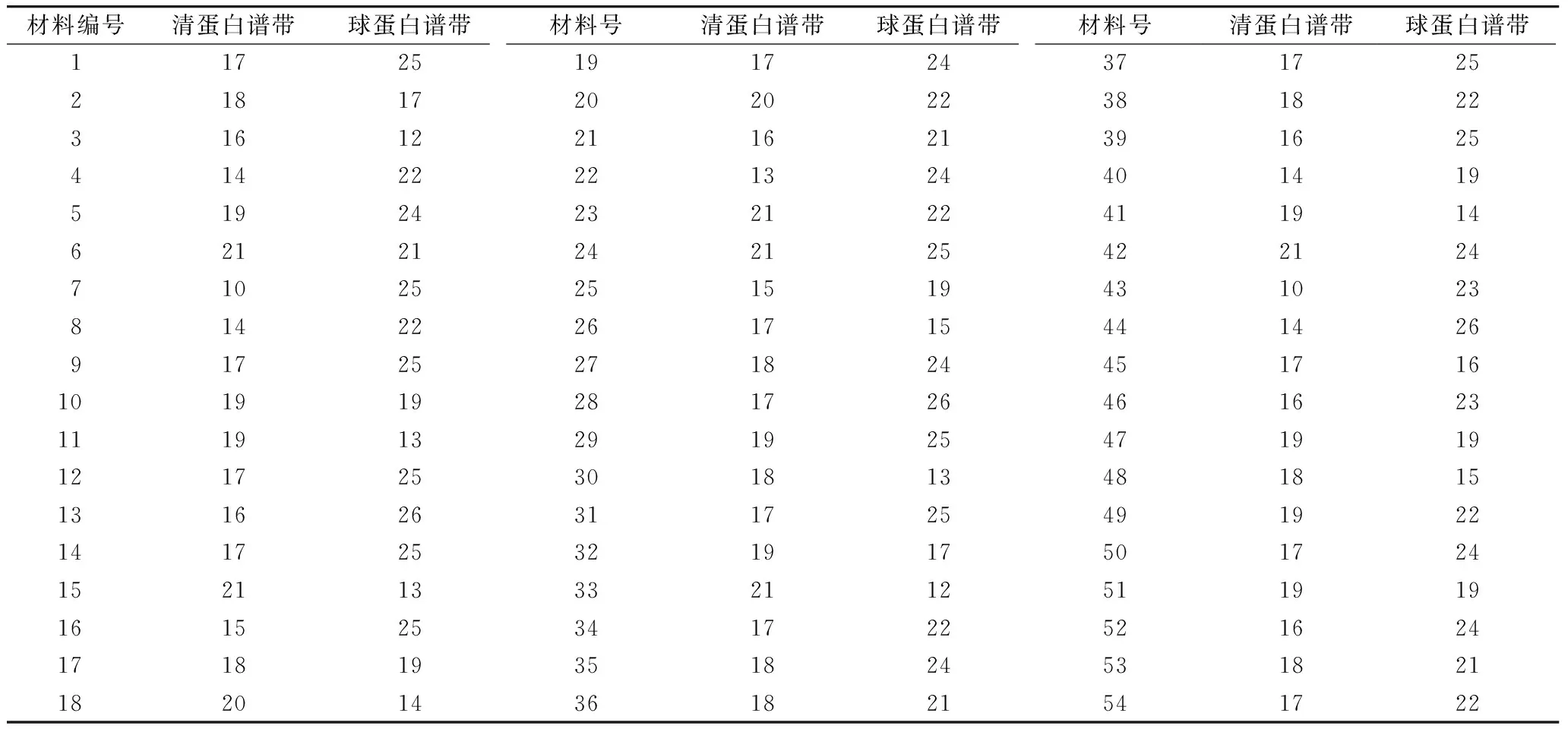
表2 54份西藏青稞材料种子SDS-PAGE电泳谱带统计
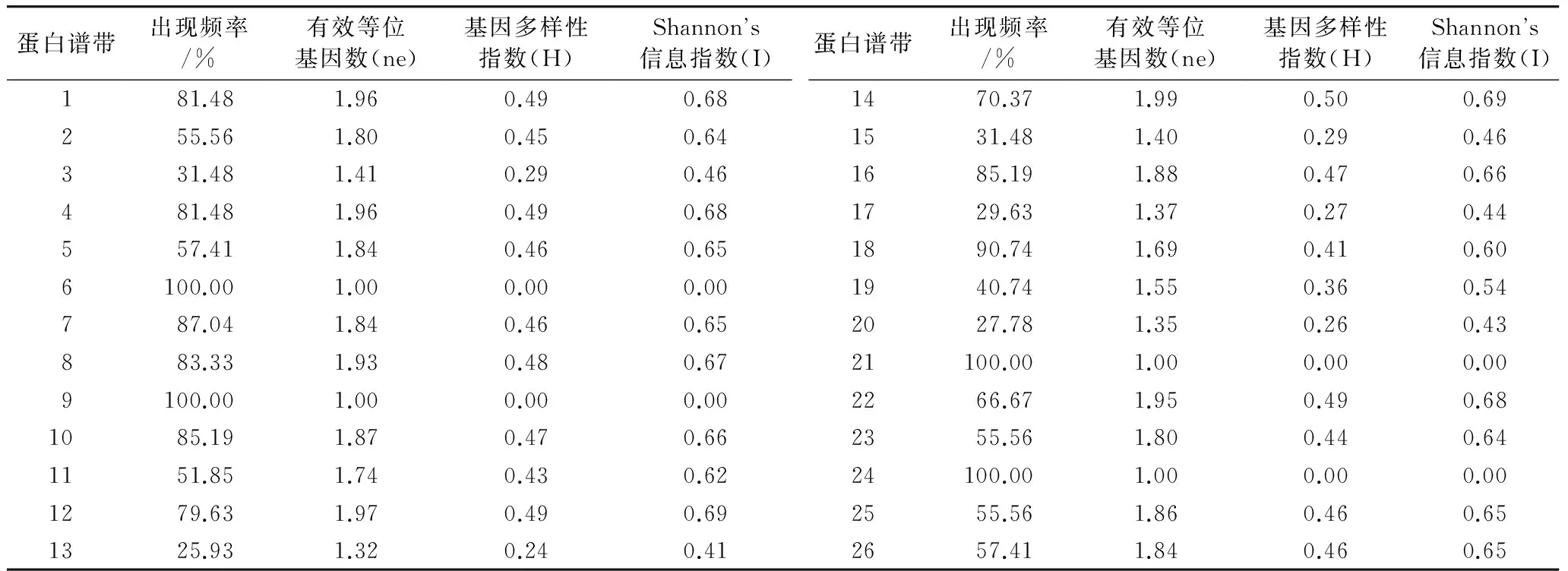
表3 54份青稞材料种子清蛋白谱带遗传多样性分析
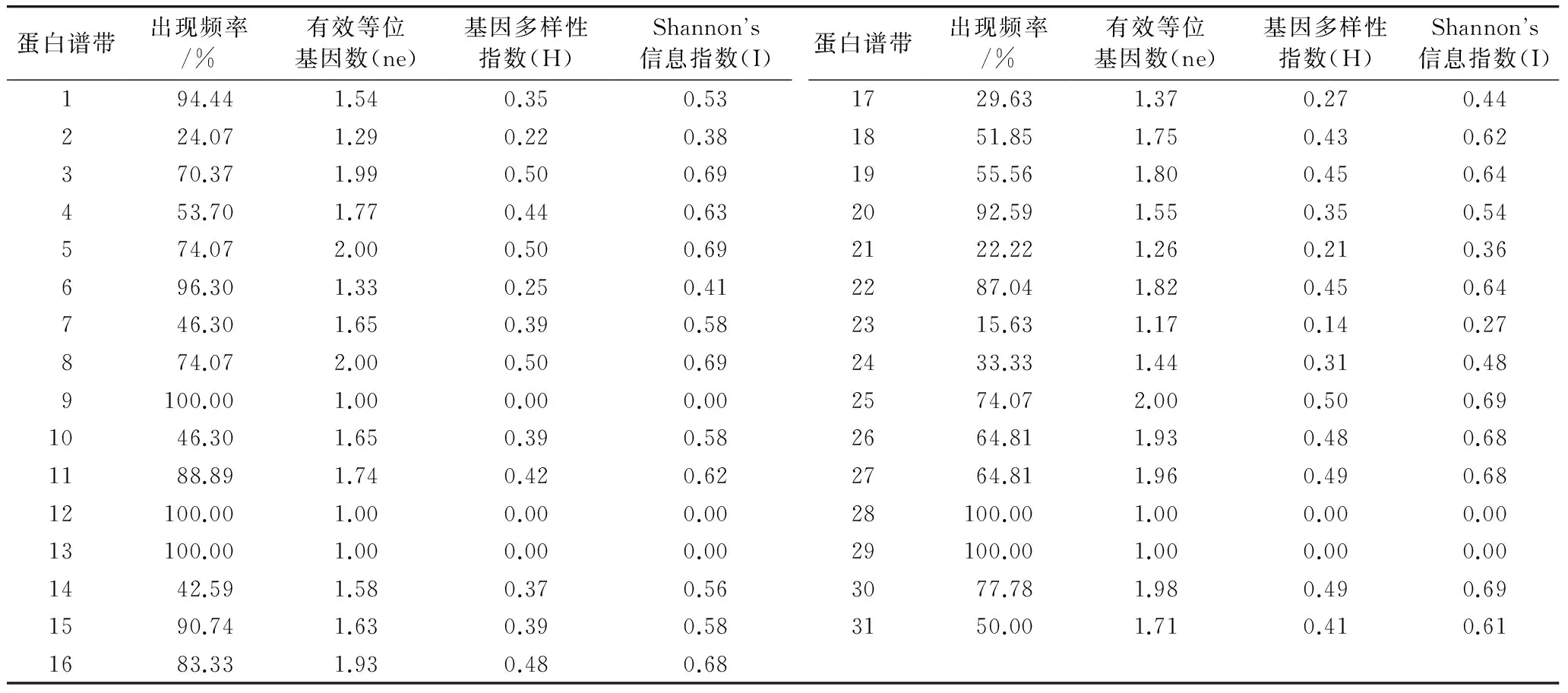
表4 54份青稞材料种子球蛋白谱带遗传多样性分析
2.3 聚类分析
利用NTSYS-PC软件采用非加权类平均聚类法(UP-GMA)对54份青稞材料清蛋白和球蛋白谱带进行聚类分析(图2),发现参试材料遗传相似系数(GS)变异范围在0.71~0.96,其中1号和46号材料间的相似系数最大,为0.96,其次为2号和47号,其相似系数为0.955,表明这两组材料遗传背景差异极小,亲缘关系最近。其他材料间有一定距离的遗传差异。根据GS值为0.71水平上可将参试材料聚为一个类群,GS在0.775水平上将参试材料聚为8个类群,第I类为1份材料;第Ⅱ类群为1份材料;第Ⅲ类群有1份材料,分为两个大的亚群;第Ⅳ类群为3份材料;第Ⅴ类群有1份材料;第Ⅵ类群有8份材料;第Ⅶ类群有3份材料;第Ⅷ类群为最大的类群,有29份材料,GS在0.79水平上,可将第Ⅷ类群分为3个小亚群。聚类分析发现来自西藏相似生态地理区位的青稞地方种、农家种并未聚为一类,材料间互相交叉聚类。表现出对于参试材料蛋白的遗传距离与地理来源之间无明显关系。这可能与西藏多变的地理环境因素有关,表明来源于西藏不同区县的清蛋白农家种、地方种间遗传背景差异较大。
2.4 不同海拔梯度种质群间的遗传多样性差异
据参试材料采集地海拔高度,将54份青稞材料分为4个海拔梯度类群,即海拔低于3 000 m的类群、海拔高度在3 000~3 500 m的类群、3 500~4 000 m的类群和>4 000 m的类群。对各类群谱带进行遗传多样性分析,发现4个类群间各考察参数均表现出一定的差异(表5),表现出随着海拔高度的增加,观测等位基因数(na)、有效等位基因数(ne)、基因多样性指数(H)和Shannon’s信息指数呈有规律增加。这一结果暗示着,在西藏高原地区随着海拔高度的增加,使得青稞遗传变异相应增大。此结果也印证了在海拔3 500~4 000 m的日喀则、山南等西藏高原青稞主产区,青稞种质在长期的栽培驯化和人为选育过程中,遗传资源变得丰富。此外,分布于海拔>4 000 m的极端恶劣环境下,青稞产生了极其丰富变异。
54份青稞材料类群间基因多样性指数(Ht)是0.322 3,类群体内基因多样性指数(Hs)是0.292 9,基因变异的平均系数(Gst)是0.091 3,群体估计的基因流(Nm)是4.797 1(表6)。
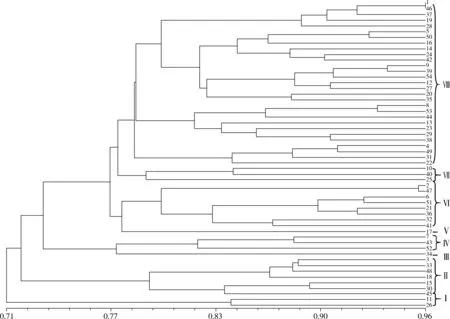
图2 54份西藏青稞种质材料清蛋白和球蛋白亚基带型N-J进化树
表5 不同海拔梯度的清蛋白生态类群遗传多样性分析
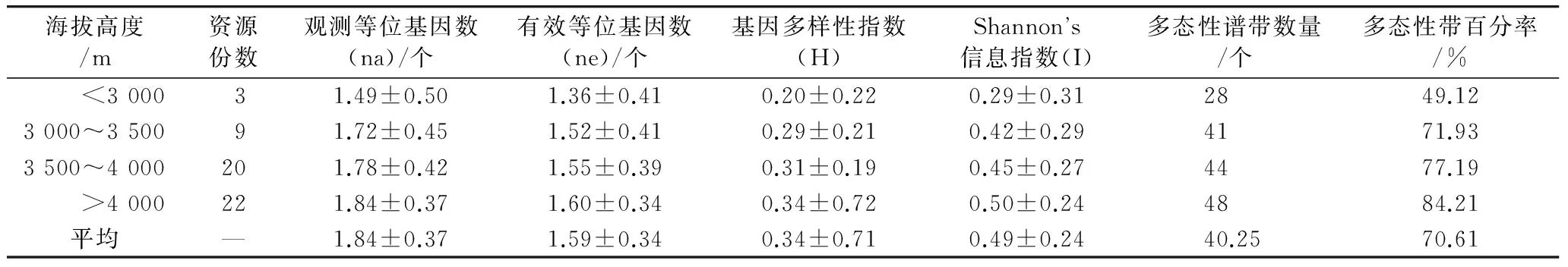
海拔高度/m资源份数观测等位基因数(na)/个有效等位基因数(ne)/个基因多样性指数(H)Shannons信息指数(I)多态性谱带数量/个多态性带百分率/%<300031.49±0.501.36±0.410.20±0.220.29±0.312849.123000~350091.72±0.451.52±0.410.29±0.210.42±0.294171.933500~4000201.78±0.421.55±0.390.31±0.190.45±0.274477.19>4000221.84±0.371.60±0.340.34±0.720.50±0.244884.21平均—1.84±0.371.59±0.340.34±0.710.49±0.2440.2570.61

表6 54份青稞材料基因多样性评价
3 讨论与结论
研究物种的遗传变异有助于解析基因库和遗传差异,鉴定物种的亲缘关系和相似性,并能更全面的比较品种特征,对挖掘优异种质资源和新品种选育具有重要意义[7]。前人利用蛋白质、DNA分子标记、细胞遗传学、生物化学和表型特征等方法对小麦、大豆、玉米等作物的遗传变异和品种进行了鉴定[29-32]。本研究通过SDS-PAGE方法对来自西藏不同生态区的56份青稞材料间SSP的遗传变异鉴定,分析材料间的遗传多样性和亲缘关系,有助于了解西藏青稞材料的遗传背景,拓宽亲本谱,为优选亲本组配提供奠定基础。
前人利用SDS-PAGE方法对青藏高原青稞或大麦种质资源蛋白遗传多样性分析主要集中于醇溶蛋白,认为该地区存在非常丰富的醇溶蛋白等位变异。例如,Yin等[33]对西藏的211份近缘野生大麦种质的醇溶蛋白多态性进行了分析,发现其多态性远远高于来源于以色列、约旦等地的大麦材料。侯永翠等[34]对来自西藏及青海省36个不同行政县的105份近缘野生大麦的醇溶蛋白SDS-PAGE电泳、共分离出36条不同谱带,谱带变幅在5~24条,平均14条,未发现共有带,多态性达到100%,醇溶蛋白谱带聚类分析显示,材料的遗传相似系数(GS)变异范围为0.182~0.966,平均值为0.580。闫敏等[26]对18份西藏青稞育成品种进行醇溶蛋白遗传多样性研究,共分离出22种不同谱带,平均等位变异数为10个,传相似系数(GS)变异范围为0.00~0.61。曾兴权[24]对347份西藏青稞种质材料进行了醇溶蛋白的遗传多样性分析,共分离得到169条不同谱带;谱带变幅在2~17条,平均7.59条,平均遗传相似系数为0.453 8,变化范围为0.25~1.0。本研究利用SDS-PAGE对来自西藏不同生态区的54份青稞资源籽粒清蛋白和球蛋白遗传多样性分析发现,共分离出迁移率不同的清蛋白谱带26种,变幅在10~21条,平均17.3条;球蛋白谱带31条,其变幅在12~26,平均21条;清蛋白和球蛋白谱带的遗传相似系数变异范围在0.71~0.96。此结果与前人利用SDS-PAGE分离出的青稞醇溶蛋白遗传多样性相比,分离出的蛋白谱带略多,但遗传多样性略低,这可能归咎于本实验采用痕量银染法,与常规的考马斯亮蓝颜色相比,其显色敏感型约为后者的10倍,同时推测清蛋白和球蛋白较醇溶蛋白稳定,发生遗传变异的可能性低有关,也支持了前人的研究结果,认为醇溶蛋白具有较多的突变[26]。由此,建议今后全面解析青稞籽粒蛋白的遗传多样性,应全面分析清蛋白、球蛋白和醇溶蛋白的遗传多样性。
本研究根据54份西藏青稞资源种子清蛋白和球蛋白谱带差异,利用聚类分析发现,在相似系数为0.775水平上,将54份青稞种质材料分8个类群,不同类群的材料间并未表现出明显的地域分布,这可能与西藏地区特殊的地理环境条件有关,不同区县间环境条件迥异,且大山阻隔了青稞种质间的基因流。同时在不同海拔高度的生态环境中,青稞种质资源遗传多样性表现出一定的规律,即随着海拔高度增加,评价青稞资源的遗传多样性各指标均呈增加趋势。这表明,随着海拔高度增加,氧浓度逐渐下降,气压下降,辐射强度增加,干旱少雨等,各种诱变因素增加,导致青稞种质资源变异频率提高,加之在海拔3 500~4 000 m西藏青稞主产区,在长期的人工驯化和种质资源的人为流动,导致天然群体混杂度提高,使得青稞遗传多样性增加。
54份青稞种质基因遗传多样性(H)为0.34,暗示着所有参试材料间遗传变异的34%,而剩余的64%的遗传变异发生在群体内。Yeh等[35]认为Gst范围在0.151~0.25代表大的遗传差异,基于此范围,西藏清蛋白4个地理生态群体表现出较弱的群体间分化。本研究估算的基因流Nm为4.797 1,Wright[36]认为Nm低于1暗示着群体因为遗传漂移开始分化,而Mcdermott等[37]认为Nm低于0.5暗示着群体因基因漂移而产生广泛的遗传多样性。本试验Nm远高于1,暗示西藏不同生态类群的青稞资源处于分化进程中。
[1]冯继林,甲干,向明华,等.藏区青稞考察与思考[J].大麦与谷类科学,2007,(3):6-9
FENG J L,JIA G,XIANG M H,et al.Investigation and thinking of hulless barley in tibet[J].Barley and Cereal Sciences,2007,(3):6-9
[2]王建林,胡单.西藏栽培大麦的遗传多样性中心[J].植物生态学报,2004,28(1):133-137
WANG J L,HU D.A study on genetic diversity centers ofbibet cultivated barley[J].Acta Phytoecologica Sinica,2004,28(1):133-137
[3]马得泉,李雁勤,洛桑更堆,等.西藏栽培大麦研究进展[J].西藏科技,1997(1):2-8
MA D Q,LI Y Q,LUO S G D,et al.Research progress of cultivated barley in tibet[J].Tibet’s Science & Technology,1997(1):2-8
[4]党斌,杨希娟,刘海棠.青稞加工利用现状分析[J].粮食加工,2009,34(3):69-71
DANG B,YANG X J,LIU H T.Analysis on status of processing and utilization of highland barley[J].Grain Processing,2009,34(3):69-71
[5]苟安春,李良.青稞产品开发大有可为[J].大麦科学,2004(4):40-44
GOU A C,LIL.The product development of highland barley has bright prospects[J].Barley Science,2004(4):40-44
[6]朱印酒.西藏青稞资源与分布特征[J].西藏大学学报(自然科学版),2011,26(1):42-45
ZHU YJ.Resources and distribution characteristics of tibetanHordeumvulgare[J].Journal of Tibet University(Nature Science Edition),2011,26(1):42-45
[7]孙立军,陆炜,张京,等.中国大麦种质资源鉴定评价及其利用研究[J].中国农业科学,1999,32(2):24-31
SUN L J,LU W,ZHANGJ,et al.Evaluation and utilization of barley germplasm resources of china[J].Scientia Agricultura Sinica,1999,32(2):24-31
[8]FAIRS D G.Physiology and genetics of the kernel color of barley[M].Columbia:University of British Columbia,1995
[9]BOTHMER R V,SATO K,KOMATSUDA T,et al.Diversity in Barley(Hordeum vulgare):the domestication of cultivated barley[M].Amsterdam,Netherlands:Elsevier Science B.V.2003
[10]TAKAHASHI R.Genetic features of east asian barleys[R].Okayama Japan:Barley Genetics V.proc.5th Int.Barley Genet Symp.1987:7-20
[11]吴昆仑.青稞种质资源的SSR标记遗传多样性分析[J].麦类作物学报,2011,31(6):1030-1034
WU K L.Genetic diversity analysis ofhulless barley germplasm by SSR markers[J].Journal of Triticeae Crops,2011,31(6):1030-1034
[12]潘志芬,邹弈星,邓光兵,等.青藏高原栽培青稞SSR标记遗传多样性研究[J].中山大学学报,2007,46(2):82-86
BAN Z F,ZHOU Y X,ZHENG G B,et al.Genetic diversity of SSR markers in cultivated hulless barley from qinghai-tibet plateau in china[J].Acta Scientiarum Naturalium Universitatis Sunyatseni,2007,46(2):82-86
[13]曾兴权,王玉林,徐齐君.等.利用SSR引物分析西藏青稞种质资源的遗传多样性[J].麦类作物学报,2013,33(2):260-267
ZENG X Q,WANG Y L,XU Q J,et al.Assessment of genetic diversity in tibetan hulless barley germplasm(Hordeum vulgare L.var.nudum h k.f.)by SSR primers[J].Journal of Triticeae Crops,2013,33(2):260-267
[14]孟亚雄,孟祎林,汪军成,等.青稞遗传多样性及其农艺性状与SSR标记的关联分析[J].作物学报,2016,42(2):180-189
MENG Y X,MENG W L,WANG J C,et al.Genetic diversity and association analysis of agronomic characteristics with SSR markers in hulless barley[J].Acta Agronomica Sinica,2016,42(2):180-189
[15]汪爱华,丁毅.西藏近缘野生大麦PAPD和ISSR分子标记的遗传多样性[J].武汉大学学报(理学版),2007,53(6):723-730
WANG A H,DING Y.Genetic diversity of wild close relatives of barley(Hordeum vulgare L.)in tibet of china by using RAPD and ISSR markers[J].Journal of Wuhan University(Science Edition),2007,53(6):723-730
[16]王建林,栾运芳,大次卓嘎,等.西藏栽培大麦变种组成和分布规律研究[J].中国农业科学,2006,39(11):2163-2169
WANG J L,LAN Y F,DA C Z G,et al.Distribution and composition study of tibetan cultivated barley varieties[J].Scientia Agricultura Sinica,2006,39(11):2163-2169
[17]KIM W T,OKITA T W.Expression of storage protein multigene families in developing rice endosperm.Plant Cell Physiology,1993,34(4):595-603
[18]KHOSHROO S M,KHAVARINEJAD R,BAGHIZADEH A,et al.Seed storage protein electrophoreticprofiles in some Iranian date palm(Phoenix dactylifera L.)cultivats[J].Affrican Journal of Biotechnology,2013,10(77):17793-17804
[19]VELUSAMY V,LEE K J,HA B K,et al.Evaluation of genetic diversity in Korean soybean landraces by protein banding patterns using high-throughput screening[J].Journal of Crop Science and Biotechnology,2013,16(3):189-207
[20]GHAFOOR A,AHMAD Z,QURESHI A S,et al.Genetic relationship inVignamungo(L.)HepperandV.radiata(L.)R.Wilczekbased on morphological traits and SDS-PAGE[J].Euphytica,123(3):367-378
[21]EMRE I.Electrophoretic analysis of some Lathyrus L.species based on seed storage proteins[J].Genetic Resources and Crop Evolution,2009,56(1):31-38
[22]YUZBASIOGLU E,ACIK L,OZCAN S.Seed protein diversity among lentil cultivars[J].Biologia Plantarum,2008,52(1):126-128
[23]LEE K J,LEE G A,LEE J R,et al.Comparison of eating quality and seed storage protein among Korean rice Landraces[J].Journal of Crop Science and Biotechnology,2016,19(3):241-247
[24]曾兴权.西藏青稞种质资源醇溶蛋白遗传多样性分析[J].西藏科技,2014,(6):5-9
ZENG X Q.Genetic diversity of alcohol soluble protein of hulless barley germplasm in Tibet highland[J].Tibet’s Science & Technology,2014,(6):5-9
[25]冯宗云,李宏,张立立,等.西藏野生大麦醇溶蛋白的遗传多样性[J].四川大学学报,2004,41(2):440-445
FENG Z Y,LI H,ZHANG L L,et al.Genetic diversity of hordein in three groups of wild barley from tibet[J].Journal of Sichuan University(Natural Science Edition),2004,41(2):440-445
[26]闫敏.西藏青稞育成品种醇溶蛋白的遗传多样性分析[J].大麦与谷类科学,2007(3):1-5
YAN M.Genetic diversity of hordein came from hulless barley varieties bred in tibet of china[J].Barley and Cereal Sciences,2007(3):1-5
[27]宋晓敏,李素芬,刘建福,等.220 种蚕豆样品蛋白质含量与其蛋白组分的分子质量[J].中国粮油学报,2013,28(12):42-46
SONG X M,LI S F,LIU J F,et al.Protein content and relative molecular mass of their subunit from twenty broad bean varieties grown in china[J].Journal of the Chinese Cereals and Oils Association,2013,28(12):42-46
[28]刘玉皎,侯万伟,李萍,等.青海不同基因型蚕豆蛋白组成及清蛋白和球蛋白亚基的SDS-PAGE分析[J].甘肃农大学报,2012,47(2):68-71
LIU Y J,HOU W W,LI P.et al.Analysis on protein composition and subunit of albumin and globulin in fava beans of different genotype by SDS-PAGE in Qinghai province[J].Journal of Gansu Agricultural University,2012,47(2):68-71
[29]COLLARD B C,MACKILL D J.Marker-assisted selection:an approach for precision plant breeding in the twenty-first century[J].Philosophical Transactions of the Royal Societ of London,2008,363(1491):557-572
[30]MALIK M F A,QURESHI A S,ASHRAF M,et al.Evaluation of genetic diversity in soybean(Glycinemax)lines using seed protein electrophoresis[J].Australian Journal of Crop Science,2009,3(2):107-112
[31]MIERNYK J A.Seed proteomics[J].Journal of Proteomics,2014,74(4):389-400
[32]TAVAUD P M,SARTRE P,NELSON R,et al.Genetic diversity in a soybean collection[J].Crop Science,2009,49(3):895-902
[33]YIN Y Q,MA D Q,DING Y.Analysis of genetic diversit y of hordein in wild close relatives of barley from Tibet[J].Theor Appl Genet,2003,122:1-17
[34]侯永翠,郑有良,魏育明.青藏高原近缘野生大麦醇溶蛋白遗传多样性分析[J].西南农业学报,2004,17(5):545-551
HOU Y C,ZHENG Y L,WEI Y M.Analysis of genetic diversity of hordein in wild relatives of barley from Qingzang plateau[J].Southwest China Journal of Agricultural Sciences,2004,17(5):545-551
[35]YEH F C,YANG R C,BOYLE T B J,et al.Popgene,A user-friendly shareware for population genetic analysis[M].Edmonton:Molecular and Biotechnology Center,University of Alberta,1997
[36]WRIGHT S.Evolution and the genetics of populations[J].Quarterly Review of Biology,1968(8):1191-1192
[37]MCDERMOTT J M,MCDONALD B A.Gene flow in plant photosystems[J].Annual Review of Phytopathology,1993,31(1):353-357.

