白皮小麦抗穗发芽资源评价及抗性候选位点关联分析
周 勇,李净琼,李 嘉,毛瑞文,谢 娇,刘亚西,江千涛,蒲至恩,谭 志,王际睿,3
(1.四川农业大学小麦研究所, 四川成都 611130; 2.成都海关/食品安全检测四川省重点实验室, 四川成都 610041;3.作物基因资源与遗传改良国际合作联合实验室, 四川成都 611130)
穗发芽(Pre-harvest sprouting, PHS)是指作物成熟未收获前,籽粒在穗上发芽的现象[1]。小麦穗发芽现象与种皮颜色具有特定的相关性[2]。小麦种皮有多种颜色,如白色、红色、棕色、紫色等,生产上以白皮小麦和红皮小麦为主。其中,白皮小麦因其皮薄且胚乳相对含量及出粉率高,较红皮小麦更受农户和企业青睐。但白皮小麦更易发生穗发芽[2],在成熟期遇到阴雨天气往往会造成巨大的经济损失,使其在收获季多雨的地区推广困难。因此,当务之急是筛选并培育具有穗发芽抗性的白皮小麦品种。
对于白皮抗穗发芽种质资源的挖掘始于20世纪90年代。江登阳等[3]从269个普通小麦材料中筛选出涪陵须须白麦等发芽率在10%以下的6个中国白皮小麦材料。Morris等[4]研究发现,RioBlanco是一个高抗穗发芽白皮小麦材料。张海峰等[5]鉴定到9个发芽率在20%以下的白皮小麦材料,包括丰产3号、ID80-115、ID83-196、Vakka、B/V-3、B/V-5、ID83-3、Ford和Peck。蒋国梁等[2]对48个白皮小麦的休眠和穗发芽抗性鉴定发现,涪陵须须麦、永川白麦子、遂宁坨坨麦、梓潼女儿麦和宜宾白麦子休眠期超过6周,穗发芽抗性较好。肖世和等[6]筛选出20个高抗穗发芽的白皮小麦材料,如万县白麦子、遂宁坨坨麦、永川白麦子以及涪陵须须麦等。Graybosch等[7]从美国种质资源库中筛选出一个高抗穗发芽的白皮小麦品种Nuplains。上述研究表明,从白皮小麦资源中选择抗穗发芽品种是可行的,但近年来对白皮小麦抗穗发芽资源进行评价和筛选的研究开展得较少。
小麦穗发芽是受多基因和环境因素共同调节的数量性状[6, 8-9]。目前,己在小麦全基因组的21条染色体上挖掘到上百个抗穗发芽位点(QTLs)[3-4],主要集中在3号染色体同源群[10-16]以及4A染色体上[17-23]。3号染色体同源群定位到的候选基因或者QTL位点中,包括一个与种皮颜色相关的重要候选基因 myb10[24]。4A染色体上的主效QTL位点 QPhs.dpivic-4A.1与种皮颜色无关,其连锁标记Xgwm397和Xbarc170可以在不同的作图群体以及生态环境下同时鉴定到[25]。随着对白皮小麦抗穗发芽工作的关注度不断加强,从白皮材料中也定位到一些与穗发芽抗性相关的QTL区段[11,15,20,26],其连锁的分子标记包括传统的SSR标记及高通量的DArT[20]、GBS[27]和SNP[21]标记。利用已报道抗穗发芽连锁标记进行分子标记辅助选择,培育抗穗发芽品种是防治穗发芽灾害的有效途径[28],如Xiao等[29]利用小麦3A染色体上与穗发芽抗性连锁的SSR分子标记Xbarc57、Xbarc310和Xbarc321,结合传统育种,成功选育出白皮抗穗发芽品种中麦911;Hickey等[25]利用 QPhs.dpivic-4A.1的连锁SSR分子标记Xbarc170从休眠品种AUS1408与无休眠品种后代中选择到抗穗发芽白皮小麦材料。
本研究搜集来自世界主要小麦产区的555个小麦种质资源(502个白皮种质、53个深色种质),在7个不同环境条件下进行穗发芽抗性鉴定和评价,旨在筛选具有穗发芽抗性的白皮小麦材料,为育种家提供抗穗发芽白皮小麦种质资源;同时,对白皮小麦穗发芽抗性进行关联分析,筛选与白皮小麦穗发芽抗性连锁的SSR标记,为白皮小麦抗穗发芽标记辅助选择育种提供理论依据。
1 材料与方法
1.1 材 料
本研究所用材料包括世界主要小麦产区的502个白皮小麦及53个深色小麦材料(对照材料,包括红皮、紫皮、黑皮、蓝皮、灰皮和褐皮小麦),均由美国农业部种子资源库提供,现保存于四川农业大学小麦研究所种质资源库。502个白皮小麦材料包括主推栽培品种、地方品种、育种材料和遗传材料,来源广泛(Fehorri 25031 Kazak和Ak-Bidaj来源地资料缺失),覆盖全球六个洲(图1)。
1.2 田间种植
555个供试材料于2011年种植于四川农业大学雅安校区农场(E1),2012年种植于四川农业大学雅安校区农场(E2)、成都校区温江试验基地(E3)和都江堰校区试验基地(E4),2013年种植于四川农业大学雅安校区农场(E5)、成都校区温江试验基地(E6)和崇州现代化实验基地(E7)。双行种植,行长1.5 m,行距0.25 m,每行15株,株距0.1 m,正常田间管理。
1.3 小麦穗发芽表型鉴定
于小麦蜡熟期(穗部落黄),收取田间麦穗。每份材料随机取5~10个单株,每个单株取3~5穗,于通风、避免阳光照射环境下阴干7 d,手工脱粒并于-20 ℃条件下保存以保持其休眠特性[30]。每份材料随机抽取150粒种子,经双氧水灭菌处理后采用培养皿发芽法进行穗发芽抗性鉴定[31-32]。每份材料均检测3个重复,每个重复50粒。发芽以露白为标准,每天记录发芽数,连续记录7 d后计算发芽率(germination rate,GR)。发芽率计算公式如下:
GR=(X1+X2+X3+X4+X5+X6+X7)/50×100%
X1~X7分别表示第1天到第7天的发芽数。

图1 502个白皮小麦材料的来源分布Fig.1 Distribution of 502 white grained wheat accessions across the world
1.4 基因型鉴定
2013年,于小麦三叶期,取成都校区温江试验基地所种植小麦的幼嫩叶片,采用改良CTAB法[33]提取其基因组总DNA。用核酸蛋白测定仪(Thermo Scientific,NANODROP ONE)检测DNA纯度与浓度,并将其稀释至80 ng·μL-1,于4 ℃保存备用。
本研究搜集到已报道与小麦穗发芽抗性位点相关的SSR分子标记236对,并从GrainGenes 2.0(http://wheat.pw.usda.gov/GG2/index.shtml)查询标记位置、引物序列和PCR条件。利用30个穗发芽抗性差异较大的白皮小麦材料进行标记筛选,选择有差异的分子标记并从502个材料中选择生育期差别较小且具有代表性的303个白皮小麦材料用于后续试验。PCR反应总体积为25 μL,包括Buffer 2.5 μL、dNTPs(200 μmol·L-1) 2 μL、上下游引物(2 mmol·L-1)各0.5 μL、1.5 UTaqplus DNA polymerase 0.1 μL、50 ng·L-1基因组总DNA 1 μL、无菌水19.4 μL。所需试剂由北京天根生化公司提供。PCR反应程序为:94 ℃预变性 5 min;94 ℃变性1 min,52~60 ℃退火 1 min,72 ℃延伸1~5 min,35个循环;72 ℃延伸5~10 min。
采用8%的聚丙烯胺凝胶电泳检测PCR产物。读带方式采用多态型标记读法,同一个标记不同的等位位点,分别加不同数字以示区分,如Xgwm397有3个不同等位位点,分别标记为 Xgwm937.1 、 Xgwm937.2 、 Xgwm937.3。对每个标记所有等位位点进行读数,若某材料中无该等位位点目标片段,则将该等位位点在对应材料中记录为带型0;反之,若有该等位位点目标片段,则将该等位位点在对应材料中记录为带型1。所有原始结果拍照留档并记录于Excel表格中。
1.5 数据分析
采用Excel对穗发芽数据进行描述性统计;采用SPSS 16.0软件进行相关性[34]及方差分析;采用Structure 2.3.4软件中的Bayesian聚类法(Bayesian clustering algorithm)进行群体结构评价[35-37];采用TASSEL 3.0软件中的混合线性模型(Mixed linear model)进行关联分析[38-39],该模型将同时控制群体结构[35]和亲缘关系[40];采用Excel、R软件(R version 3.0.3)[41]及ggplot2作图。
2 结果与分析
2.1 白皮小麦穗发芽抗性鉴定结果
相关性分析结果(表1)表明,不同环境下发芽率相关系数较低,除2012年温江试验基地及2013年雅安校区农场、温江试验基地、崇州现代化实验基地的发芽率与2012年都江堰校区试验基地的发芽率显著相关(相关系数分别为0.63、0.63、0.76和0.60,P<0.01)外,其余环境之间的相关性未达到显著水平。此外,方差分析结果(表2)表明,2012年雅安校区农场的发芽率与其余6个环境下的发芽率差异均达到显著水平(P<0.01),2012年温江试验基地与2013年雅安校区农场、崇州现代化实验基地及2013年雅安校区农场与温江试验基地的发芽率差异也达到显著水平(P<0.01)。说明白皮小麦穗发芽抗性受环境影响大。
白皮小麦和深色小麦材料均在2011年雅安校区农场检测到最低的平均发芽率,分别为76.29%和50.51%,均在2013年雅安检测到最高的平均发芽率,分别为92.13%和76.90%,白皮小麦材料的总平均发芽率为85.58%,深色小麦材料的总平均发芽率为65.57%(表3)。方差分析结果表明,白皮小麦平均籽粒发芽率显著高于深色小麦(F=61.94,Fcrit=3.86,P-value=1.87E-14),说明穗发芽抗性与小麦籽粒颜色相关。
通过对502个白皮小麦材料在7个不同环境下的平均发芽率分析发现,仅1.39%的材料7个具有较强的穗发芽抗性,其平均发芽率在40%以下,74.7%的材料375个易穗发芽,其平均发芽率在80%~100%之间(图2)。其中,Osiris的平均发芽率最低(7.61%),在7个不同的环境下发芽率均低于10%,为供试白皮小麦里最强且最稳定的抗穗发芽材料,也是7个代表性抗穗发芽材料中唯一的地方品种(表4)。Vilmorin 29和Miana的平均发芽率分别为14.01%和15.82%,这两份材料在不同环境中也相对稳定,发芽率均在10%~20%之间(表4)。Kanto 107、Darwin、Magnif MG和Benefactress的平均发芽率分别为22.05%、31.72%、33.50%和36.84%,但这4个材料的穗发芽抗性易受环境影响(表4)。Kanto107在2012年温江试验基地和2013年崇州现代化基地鉴定中发芽率只有2%,但在2012年雅安校区农场鉴定结果中高于40%;Darwin在2012年雅安点鉴定中发芽率只有7.50%,但在2012年温江点鉴定中可达61.93%;Magnif MG在2013年温江点鉴定中发芽率只有11.72%,但在2013年崇州点鉴定中可达67.91%;Benefactress在2012年温江点鉴定中发芽率只有6.05%,但在2013年温江点鉴定中高达68.76%(表4)。
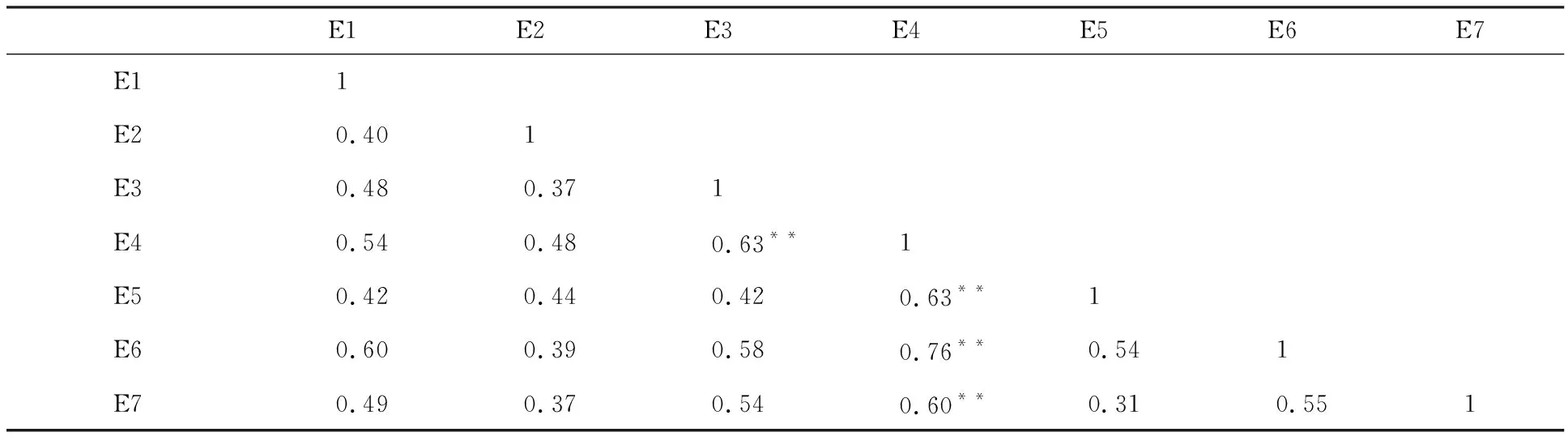
表1 白皮小麦在7个不同环境下发芽率的相关系数Table 1 Correlation coefficient of germination rates of white grained accessions in 7 environments
E1:2011年雅安;E2:2012年雅安;E3: 2012年温江;E4:2012年都江堰;E5:2013年雅安;E6:2013年温江;E7:2013年崇州;**表示在0.01水平上显著相关。下同。
E1:Ya’an in 2011; E2:Ya’an in 2012;E3: Wenjiang in 2012;E4:Dujiangyan in 2012;E5:Ya’an in 2013;E6:Wenjiang in 2013;E7:Chongzhou in 2013; ** indicated the correlation is significant at 0.01 level. The same below.

表2 白皮小麦在7个不同环境间发芽率的方差检验(F值)Table 2 Variance analysis of germination rates of white grained accessions in 7 different environments(F value)
**表示差异在0.01水平上显著。
** indicated the difference was significant at 0.01 level.

表3 白皮小麦和深色小麦在7个不同环境下穗发芽率的统计分析Table 3 Statistical analysis of germination rates of white grained and colorful grained accessions in 7 different environments %
**表示差异在0.01水平上显著。
** indicated the difference was significant at 0.01 level. WGW: White grained wheat; CGW: Colorful grained wheat.
供试材料中,除Fehorri 25031 Kazak和Ak-Bidaj来源地不确定外,其余500个均有明确的地理来源。本研究通过分析平均发芽率,以洲为单位比较不同来源地的白皮小麦穗发芽抗性差异(图3)。其中,来源于欧洲的白皮小麦平均发芽率最低,为76.51%;来源于南美洲的白皮小麦次之,平均发芽率为82.59%;来源于大洋洲的白皮小麦平均发芽率最高,为91.00%。多重比较结果表明,来自欧洲的材料平均发芽率显著低于其他5个洲,穗发芽抗性最强,其次为南美洲,来自北美洲、亚洲、非洲和大洋洲的白皮小麦穗发芽抗性差异不显著。

图2 502个白皮小麦平均发芽率的样本分布Fig.2 Sample distribution of mean germination rates of 502 white grained accessions表4 7个抗穗发芽的白皮小麦材料在不同环境下的发芽率Table 4 Germination rate of seven white grained accessions with high PHS tolerance %

-表示数据缺失。
- indicated missing values.

图注上方不同小写字母表示不同来源地小麦平均发芽率差异在0.05水平上显著。
Lowercases at the top of the figure indicated the difference is significant at 0.05 level.
图3不同来源地的白皮小麦平均发芽率比较
Fig.3Comparisonofmeangerminationratesofwhitegrainedaccessionsfromdifferentcontinents
2.2 白皮小麦遗传结构分析
首先,利用236对SSR标记对30个具有穗发芽抗性差异的(极端抗、易穗发芽)白皮小麦材料进行检测,结果筛选到71对有多态性的SSR标记。然后,利用筛选到的71对多态性SSR标记对具有代表性的303个白皮小麦材料进行基因型鉴定,共发现202个等位变异位点,其中,标记Xbarc1004、Xbarc22、Xcfd44仅鉴定到1个等位变异位点,标记Xwmc650鉴定到7个等位变异位点,其余标记鉴定到的等位变异位点介于2到5之间(表5)。
从71对SSR标记中鉴定到的202个等位变异位点,均用于303个白皮小麦群体结构分析(图4a);依据遗传结构将白皮小麦群体分为3个亚群(K=3)时,分别有40.92%、30.03%和29.04%的材料分在三个亚群中,南美洲和大洋洲材料只分为了两个亚群,非洲、亚洲、北美洲和欧洲材料均可分为三个不同的亚群;将白皮小麦群体分为4个亚群(K=4)时,有20.46%的材料组成了新的亚群,亚洲、北美洲、欧洲、南美洲和大洋洲材料均可分出新的亚群,而非洲材料仍然保持三个亚群不变;当K=5时,有8.58%的材料组成了新的亚群,非洲、北美洲和大洋洲材料保持不变,亚洲、欧洲和南美洲材料均可分出新亚群,说明群体结构变化并未稳定;当K=6时,群体结构趋近稳定,小于2%的材料组成了新的亚群,除了非洲,其他五大洲材料均可见新亚群。利用Structure Harvester(http://taylor0.biology.ucla.edu/structureHarvester/)对群体结构进行评估,结果(图4b)发现,当K=4时为最佳群体结构。由此可以看出,在不同的群体分层结构下,非洲的白皮小麦群体结构仍然单一,而来自其他地区的材料遗传结构更复杂。
2.3 抗穗发芽候选位点的关联分析
利用236 对标记检测到的202 个多样性位 点,结合穗发芽表型与不同的遗传结构,对303个白皮小麦材料进行关联分析。通过平均发芽率与各环境下发芽率分别关联到4个与15个等位变异位点(表6),其中, Xcfd44.1可在平均发芽率和两个不同的环境下关联到。在总计18个等位变异位点中,5个位点( Xbarc105.2、 Xgwm261.3、 Xgwm369.1、 Xgwm369.3和 Xgwm340.3)只能在特定的群体分层中显著关联;2个位点( Xwmc537.2和 Xwmc75.1)能在3个不同的群体分层中显著关联;其余11个位点能在4个不同的群体分层(K=3、4、5和6)同时显著关联。说明前5个位点受群体分层影响较大,而后13个位点受群体影响较小。在这13个受群体影响小的位点中,有3个位点( Xcfd102.1、 Xcfd102.2和 Xcfd102.3)只能在发芽率平均值中显著关联,有5个位点( Xwmc537.2、 Xwmc272.2、 Xbarc347.1、 Xbarc154.1和 Xbarc172.1)只能在某一个环境下显著关联,而另外5个( Xwmc24.2、 Xbarc321.2、 Xwmc75.1、 Xcfd44.1和 Xgwm397.1)则可以在两个不同的环境下显著关联,其中 Xcfd44.1还能在发芽率平均值中显著关联,这5个位点为稳定且受群体分层影响较小的位点。 Xwmc24.2可以在2011年和2012年雅安校区农场的表型鉴定中显著关联,可解释2.29%~4.02%的表型变异, Xbarc321.2可以在2013年温江校区试验基地和崇州现代化实验基地的表型鉴定中关联到,可解释6.50%~7.92%的表型变异, Xwmc75.1能在2012年雅安校区农场和温江校区试验基地的表型鉴定中关联到,可解释2.81%~4.55%的表型变异,这3个位点解释表型变异度均不足10%,为稳定的微效位点; Xcfd44.1能在2013年雅安校区农场和温江校区试验基地的表型鉴定中关联到,可解释8.55%~11.99%的表型变异, Xgwm397.1能在2013年温江校区试验基地和崇州现代化实验基地的表型鉴定中关联到,可解释7.40%~13.02%的表型变异,这2个位点最大可解释表型变异度均高于10%,且能在不同环境下关联到,为稳定的主效抗穗发芽位点;除此之外,其他关联位点可能受群体分层影响较大,或者是环境之间不能稳定重复出现,均为不稳定白皮小麦抗穗发芽位点。
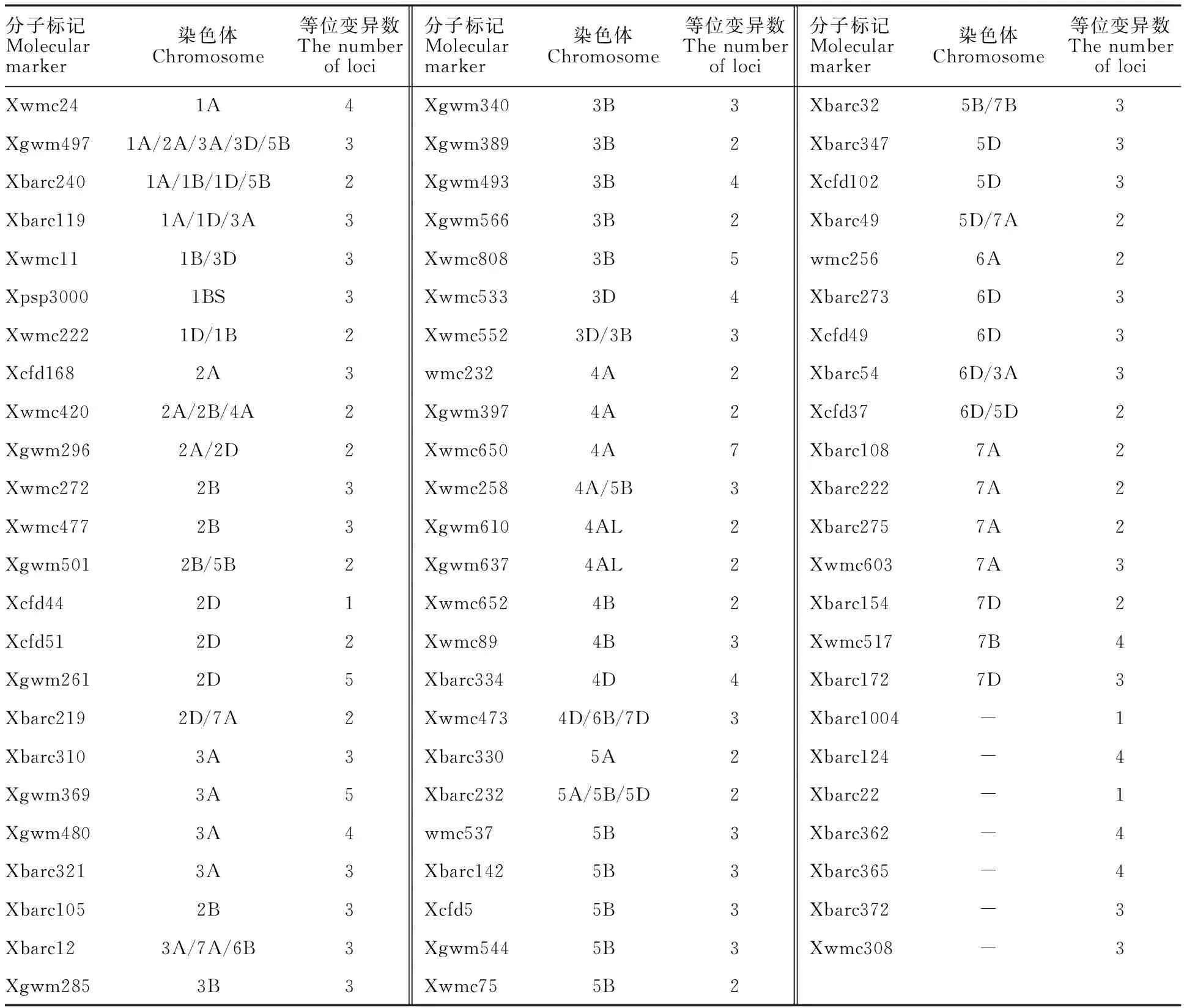
表5 71对有多态性差异的分子标记Table 5 Seventy-one polymorphism markers

AF:非洲; AS:亚洲; AU:澳洲; EU:欧洲; NA:北美洲; SA:南美洲。a:303个白皮小麦群体结构; b:不同亚群时△K的结果。
AF: Africa; AS: Asia; AU: Australia; EU: Europe; NA: North America; SA: South America.a:Structure results of the 303 white genotypes; b:△Kresults for each sub-population.

图4 白皮小麦资源群体结构分析
*表示染色体及其位置来自GrainGenes 2.0(http://wheat.pw.usda.gov/GG2/index.shtml)公共数据库;a、b、c和d分别表示群体结构为3、4、5和6。
* indicated the chromosome and detail position the locus belonged were collected from GrainGenes 2.0(http://wheat.pw.usda.gov/GG2/index.shtml);a,b,c and d indicated the number of subpopulations were 3,4,5 and 6,respectively.
3 讨 论
本研究对555个普通小麦材料进行评价,结果表明,502个白皮小麦材料在7个不同的环境下均比53个深色小麦材料的发芽率高,与前人研究结果相似。Groos等[26]认为粒色与穗发芽抗性呈正相关,颜色越深则抗性越强,红皮小麦比白皮小麦穗发芽抗性更好;巨 伟等[42]同样认为,种皮颜色与籽粒休眠性相关,影响不同品种的穗发芽抗性。20个国内白皮小麦材料,如白麦子、泰山1号、江东门和白秃头,这些材料在本研究中穗发芽抗性均较弱,与前人研究报道[43-45]相似。共筛选出7个穗发芽抗性较强的国外白皮小麦品种(Osiris、Vilmorin 29、Miana、Kanto 107、Darwin、Magnif MG和Benefactress),其中,Osiris为来自埃及的地方品种,其他6个材料均为育成品种(系)。Benefactress为来自英国的白皮小麦,家系组成为“Squarehead/Red King//Rough Chaff White”,其中,Red King为红皮小麦,Rough Chaff White和Squarehead为白皮小麦,但其穗发芽抗性水平未知,故不能从表型判断Red King的抗性来源;从关联SSR位点来看,Benefactress抗性位点由 Xwmc24.2-1A、Xbarc105.2-2B、Xwmc272.2-2B、Xgwm261.3-2D、Xgwm397.1-4A、Xwmc537.2-5B、Xcfd102.1-5D、Xbarc347.1-5D、Xbarc172.1-7D和 Xbarc154.1-7D提供,可利用这些位点对亲本进行扫描,来判断Red King的抗性来源,因此,Benefactress的抗性来源于白皮或红皮小麦亲本,还需进一步研究。Vilmorin 29由Vilmorin 23和Allies杂交选育而成,而这两个亲本籽粒均是棕黄色,推断其抗性位点与籽粒颜色相关位点无关。其他4个材料的系谱虽也清楚,但无法查询亲本信息或籽粒颜色信息,故不在此讨论。这7个白皮抗穗发芽材料,一方面可以直接或间接的作为抗穗发芽育种亲本,另一方面可以作白皮小麦抗穗发芽机理的研究对象。
关联分析是近几年发展起来的一种利用己存在的种质资源研究数量性状的新工具,可以克服传统QTL定位的不足,一方面可节约人力、物力和时间,另一方面具有更高的解析率以及更多的变异,目前己普遍应用到小麦抗穗发芽候选位点的研究之中[46-50]。结合7个不同环境下的表型数据与71对多态性分子标记的基因型鉴定,利用关联分析,在白皮小麦中关联到16对标记的18个等位变异位点与穗发芽抗性相关,包括5个稳定的等位变异位点( Xwmc24.1-1A、 Xcfd44.2-2D、 Xbarc321.2-3A、 Xgwm397.1-4A和 Xwmc75.1-5B),其中,位于染色体3A和4A上的候选基因已经成功被挖掘和克隆。Liu等[51]2008年通过构建重组自交系(RILs),在染色体3A上定位到1个主效穗发芽QTL位点 QPhs.pseru-3AS,与标记Xbarc12、Xbarc321和Xbarc369紧密连锁;Liu等[11]2010年从该重组自交系F6代中选择2个杂合株系,通过构建近等基因系,开发7个与目标性状连锁的AFLP标记,将QTL位点 QPhs.pseru-3AS进行精细定位,缩小QTL区间至1.9 cM以内;Liu等[12]2013年利用图位克隆的方法从 QPhs.pseru-3AS中克隆到一个与穗发芽相关基因,并命名为 TaPHS1。本研究关联到的Xbarc32与 QPhs.pseru-3AS紧密连锁,说明 TaPHS1也是白皮小麦抗穗发芽重要基因。与分子标记Xgwm397连锁的QTL位点phs曾在不同的研究群体中被报道过[18-23, 52-54]。Tan等[54]2006年曾利用白皮小麦AUS1408在染色体4AL上定位到标记Xgwm397与休眠性紧密连锁。Barrero等[22]2015年利用4个亲本Yitpi、Chara、Baxter和Westonia构建MAGIC群体,在3个不同的环境下,将染色体4AL定到1个QTL,与标记wsnp_Ex_c66324_64493429和CD920298连锁,位于标记Xgwm397旁边。通过在重组自交系F7代选出的5个株系构建剩余杂合群体(HIF),结合转录组测序分析数据(RNA-seq),中国春测序数据作为参考(Chromosome Survey Sequence),将目的基因定位到CSS contigs Chr4AL_7123764中,并成功克隆出ABA途径相关基因 PM19-A1和 PM19-A2。Torada等[23]2016年也在染色体上距离 PM19基因0.3 cM处发现另一个休眠相关基因MAPKinaseKinase(MKK3)。说明该QTL区间可能有2个休眠相关基因。由此可见,其他三个位点附近也应该有相应的候选基因,但至今未见基因克隆或精细定位工作。本研究中关联到的与白皮穗发芽抗性相关的SSR标记,为白皮小麦穗发芽抗性的分子标记选择辅助育种提供了理论依据。
4 结 论
通过对收集自世界主要小麦产区的502个白皮小麦资源材料在7个不同环境下进行穗发芽抗性评价,筛选出7个具有穗发芽抗性的白皮种质资源,其中,4个材料来自欧洲,另外3个材料分别来自亚洲(日本)、美洲和非洲,这些材料可用于白皮抗穗发芽机理研究和白皮小麦品种穗发芽抗性改良。此外,结合穗发芽抗性表型数据与基于穗发芽抗性相关候选位点的基因型数据,对具有代表性的303个白皮小麦进行关联分析,在平均值与各环境值下共发现18个与白皮小麦穗发芽抗性相关的等位变异位点,其中, Xwmc24.2-1A、 Xcfd44.1-2D、 Xbarc321.2-3A、 Xgwm397.1-4A和 Xwmc75.1-5B能够在不同环境中关联到,为较稳定的白皮小麦抗穗发芽位点,可以运用到白皮小麦穗发芽抗性的分子标记辅助选择育种中。
参考文献:
[1] 肖世和,闫长生,张海萍,等.小麦穗发芽研究[M].北京:中国农业科学技术出版社,2002:92-99.
XIAOS H,YAN C S,ZHANG H P,etal.Pre-Harvest Sprouting in Wheat [M].Beijing:China Agricultural Science and Technology Press,2002:92-99.
[2] 蒋国梁,肖世和.白皮小麦收获前穗发芽及品种抗性机制探讨[J].作物学报,1998,24(6):793.
JIANG G L,XIAO S H.Pre-harvest sprouting in white wheats and its resistant characteristics of cultivars [J].ActaAgronomicaSinica,1998,24(6):793.
[3] 江登阳.小麦穗发芽鉴定方法及白皮品种抗性研究[J].西藏农业科技,1991(4):36.
JIANG D Y.Evaluation methods of wheat pre-harvest sprouting and resistance progress in white wheat [J].TibetJournalofAgriculturalSciences,1991(4):36.
[4] MORRIS C F,PAULSEN G L.Review:research on pre-harvest sprouting resistance in hard red and white winter wheats at Kansas State University [R].// In: Walker-Simmons MD, Reid JJ(eds). Pre-Harvest Sprouting in Cereals. American Association of Cereal Chemists, St. Paul. MN, 1992:113-120.
[5] 张海峰,卢荣禾.小麦穗发芽抗性机理与遗传研究[J].作物学报,1993,19(6):523.
ZHANG H F,LU R H.Study on the mechanism of the resistance to preharvest sprouting and inheritance in wheat [J].ActaAgronomicaSinica,1993,19(6):523.
[6] XIAO S H,ZHANG X Y,YAN C S,etal.Germplasm improvement for preharvest sprouting resistance in Chinese white-grained wheat:an overview of the current strategy [J].Euphytica,2002,126(1):35.
[7] GRAYBOSCH R A,AMAND P,BAI G.Evaluation of genetic markers for prediction of preharvest sprouting tolerance in hard white winter wheats [J].PlantBreeding,2013,132(4):359.
[8] DERERA N F,BHATT G M,MCMASTER G J.On the problem of pre-harvest sprouting of wheat [J].Euphytica,1977,26(2):299.
[9] GORDON I L.Selection against sprouting damage in wheat.III.*Dormancy,germinative alpha-amylase,grain redness and flavanols [J].CropandPastureScience,1979,30(3):387.
[10] OSA M,KATO K,MORI M,etal.Mapping QTLs for seed dormancy and the Vp1 homologue on chromosome 3A in wheat [J].TheoreticalandAppliedGenetics,2003,106(8):1491.
[11] LIU S,BAI G.Dissection and fine mapping of a major QTL for preharvest sprouting resistance in white wheat Rio Blanco [J].TheoreticalandAppliedGenetics,2010,121(8):1395.
[12] LIU S,SEHGAL S K,LI J,etal.Cloning and characterization of a critical regulator for preharvest sprouting in wheat [J].Genetics,2013,195(1):263.
[13] NAKAMURA S,TOYAMA T.Isolation of a Vp1 homologue from wheat and analysis of its expression in embryos of dormant and non-dormant cultivars [J].JournalofExperimentalBotany,2001,52(357):875.
[14] NAKAMURA S,ABE F,KAWAHIGASHI H,etal.A wheat homolog of MOTHER OF FT AND TFL1 acts in the regulation of germination [J].ThePlantCell,2011,23(9):3215.
[15] MCCARTY D R,HATTORI T,CARSON C B,etal.The Viviparous-1 developmental gene of maize encodes a novel transcriptional activator [J].Cell,1991,66(5):895.
[16] WANG J,LIU Y,WANG Y,etal.Genetic variation of Vp1 in Sichuan wheat accessions and its association with pre-harvest sprouting response [J].Genes&Genomics,2011,33(2):139.
[17] IMTIAZ M,OGBONNAYA F C,OMAN J,etal.Characterization of QTL controlling genetic variation for pre-harvest sprouting in synthetic backcross derived wheat lines [J].Genetics,2008,178(3):1725.
[18] MARES D,MRVA K,CHEONG J,etal.A QTL located on chromosome 4A associated with dormancy in white-and red-grained wheats of diverse origin [J].TheoreticalandAppliedGenetics,2005,111(7):1357.
[19] CHEN C X,CAI S B,BAI G H.A major QTL controlling seed dormancy and pre-harvest sprouting resistance on chromosome 4A in a Chinese wheat landrace [J].MolecularBreeding,2008,21(3):351.
[20] SINGH R,MáTUS-CáDIZ M,BGA M,etal.Identification of genomic regions associated with seed dormancy in white-grained wheat [J].Euphytica,2010,174(3):391.
[21] CABRAL A L,JORDAN M C,MCCARTNEY C A,etal.Identification of candidate genes,regions and markers for pre-harvest sprouting resistance in wheat(TriticumaestivumL.) [J].BMCPlantBiology,2014,14(1):1.
[22] BARRERO J M,CAVANAGH C,VERBYLA K L,etal.Transcriptomic analysis of wheat near-isogenic lines identifies PM19-A1 and A2 as candidates for a major dormancy QTL [J].GenomeBiology,2015,16:93.
[23] TORADA A,KOIKE M,OGAWA T,etal.A causal gene for seed dormancy on wheat chromosome 4A encodes a MAP kinase kinase [J].CurrentBiology,2016,26(6):782.
[24] DONG Z D,CHEN J I E,LI T,etal.Molecular survey of Tamyb10-1 genes and their association with grain colour and germinability in Chinese wheat andAegilopstauschii[J].JournalofGenetics,2015,94(3):453.
[25] HICKEY L T,DIETERS M J,DELACY I H,etal.Screening for grain dormancy in segregating generations of dormant× non-dormant crosses in white-grained wheat(TriticumaestivumL.) [J].Euphytica,2010,172(2):183.
[26] GROOS C,GAY G,PERRETANT M R,etal.Study of relationship between pre-harvest sprouting and grain color by quantitative trait loci analysis in a white×red grain bread-wheat cross [J].TheoreticalandAppliedGenetics,2000,104:39.
[27] LIN M,CAI S,WANG S,etal.Genotyping-by-sequencing(GBS) identified SNP tightly linked to QTL for pre-harvest sprouting resistance [J].TheoreticalandAppliedGenetics,2015,128(7):1385.
[28] 张海萍,常 成,司红起,等.小麦抗穗发芽分子标记开发及育种应用[J].科技导报,2016,34(22):81.
ZHANG H P,CHANG C,SI H Q,etal.Developing of molecular marker for pre-harvest sprouting resistance and its application in wheat MAS breeding [J].Science&TechnologyReview,2016,34(22):81.
[29] XIAO S H,ZHANG H P,YOU G X,etal.Integration of marker-assisted selection for resistance to pre-harvest sprouting with selection for grain-filling rate in breeding of white-kernelled wheat for the Chinese environment [J].Euphytica,2012,188(1):85.
[30] WALKER-SIMMONS M K.Protein kinases in seeds [J].SeedScienceResearch,1998,8(2):193.
[31] 刘引祥,李冬肖,武高潮,等.一种小麦收获前穗发芽抗性鉴定的简便方法[J].中国农业科学,1993,26(1):90.
LIU Y D,LI D X,WX G C,etal.A simple method of determining the resistance to preharvest sprouting in wheat [J].ScientiaAgricultureSinica,1993,26(1):90.
[32] HUMPHREYS D G,NOLL J.Methods for characterization of preharvest sprouting resistance in a wheat breeding program [J].Euphytica,2002,126(1):61.
[33] MURRAY M G,THOMPSON W F.Rapid isolation of high molecular weight plant DNA [J].NucleicAcidsResearch,1980,8(19):4323.
[34] FIELLER E C,HARTLEY H O,PEARSON E S.Tests for rank correlation coefficients [J].Biometrika,1957,44(3/4):470.
[35] PRITCHARD J K,STEPHENS M,DONNELLY P.Inference of population structure using multilocus genotype data [J].Genetics,2000,155(2):945.
[36] FALUSH D,STEPHENS M,PRITCHARD J K.Inference of population structure using multilocus genotype data:linked loci and correlated allele frequencies [J].Genetics,2003,164(4):1576.
[37] HUBISZ M J,FALUSH D,STEPHENS M,etal.Inferring weak population structure with the assistance of sample group information [J].MolecularEcologyResources,2009,9(5):1322.
[38] WANG Y S,BARRATT B J,CLAYTON D G,etal.Genome-wide association studies:theoretical and practical concerns [J].NatureReviewsGenetics,2005,6(2):109.
[39] ZHANG Z,ERSOZ E,LAI C Q,etal.Mixed linear model approach adapted for genome-wide association studies [J].NatureGenetics,2010,42(4):355.
[40] LI M,LIU X,BRADBURY P,etal.Enrichment of statistical power for genome-wide association studies [J].BMCBiology,2014,12(1):73.
[41] R Development CORE TEAM(2011),R:A Language and Environment for Statistical Computing.Vienna,Austria :the R Foundation for Statistical Computing.ISBN:3-900051-07-0.Available online at http://www.R-project.org/.
[42] 巨 伟,郭玉平,樊 梅,等.小麦抗穗发芽研究进展[J].种子世界,2015(1):22.
JU W,GUO Y P,FAN M,etal.Progress on preharvest sprouting resistance in wheat [J].SeedWorld,2015(1):22.
[43] YANG Y,CHEN X,HE Z,etal.Distribution of Vp-1 alleles in Chinese white-grained landraces,historical and current wheat cultivars [J].CerealResearchCommunications,2009,37(2):169.
[44] BI H H,SUN Y W,XIAO Y G,etal.Characterization ofDFRallelic variations and their associations with pre-harvest sprouting resistance in a set of red-grained Chinese wheat germplasm [J].Euphytica,2014,195(2):197.
[45] CHANG C,ZHANG H P,FENG J M,etal.Identifying alleles of Viviparous-1B associated with pre-harvest sprouting in micro-core collections of Chinese wheat germplasm [J].MolecularBreeding,2010,25(3):481.
[46] JAISWAL V,MIR R R,MOHAN A,etal.Association mapping for pre-harvest sprouting tolerance in common wheat(TriticumaestivumL.) [J].Euphytica,2012,188(1):89.
[47] KULWAL P,ISHIKAWA G,BENSCHER D,etal.Association mapping for pre-harvest sprouting resistance in white winter wheat [J].TheoreticalandAppliedGenetics,2012,125(4):793.
[48] ARIF M A R,NEUMANN K,NAGEL M,etal.An association mapping analysis of dormancy and pre-harvest sprouting in wheat [J].Euphytica,2012,188(3):409.
[49] ALBRECHT T,OBERFORSTER M,KEMPF H,etal.Genome-wide association mapping of preharvest sprouting resistance in a diversity panel of European winter wheats [J].JournalofAppliedGenetics,2015,56(3):277.
[50] LIN M,ZHANG D,LIU S,etal.Genome-wide association analysis on pre-harvest sprouting resistance and grain color in US winter wheat [J].BMCGenomics,2016,17(1):794.
[51] LIU S B,CAI S B,GRAYBOSCH R,etal.Quantitative trait loci for resistance to pre-harvest sprouting in US hard white winter wheat Rio Blanco [J].TheoreticalandAppliedGenetics,2008,117:691.
[52] KATO K,NAKAMURA W,TABIKI T,etal.Detection of loci controlling seed dormancy on group 4 chromosomes of wheat and comparative mapping with rice and barley genomes [J].TheoreticalandAppliedGenetics,2001,102(6-7):980.
[53] TORADA A,IKEGUSCHI S,KOIKE M.Mapping and validation of PCR-based markers associated with a major QTL for seed dormancy in wheat [J].Euphytica,2005,143(3):251.
[54] TAN M K,SHARP P J,LU M Q,etal.Genetics of grain dormancy in a white wheat [J].CropandPastureScience,2006,57(11):1157.

