基于贪心EM的模体预测算法∗
张 斐
1 引言
当人类基因组研究进入系统测序阶段时,大量已测定的但未知功能或未经注释的DNA序列,急需可靠的自动的基因组序列注释方法和技术进行处理。
人们利用相关测序后DNA序列建立了大量转录因子(TF)数据库,为转录调控的进一步研究提供了丰富的资源,它是用计算机方法构建转录预测模型的基础。大量的数据促进了识别Motif算法和相关预测程序包的发展。目前,对识别Motif的算法研究是一个热点,它们大部分都基于一致性序列和矩阵模型。
在应用软件预测Motif时,可以运用的方法,有用不同的预测软件比较后取长补短,参考各软件的预测结果,从而提出新的预测方案,最终得到最佳的预测结果[1]。采用不同的算法来比较预测,通过预测结果对比来选择合适的算法。还可以在已有算法的基础上来进行改进,在原有序列的基础上进行比较预测,结果更具有说服力。本文就是在贪心EM算法的基础上,通过筛选优化参数修定序列的预测长度范围,从而改进算法的整体预测正确率。
2 贪心EM算法的应用基础
贪心EM算法是实现机器学习(GMM)综合学习的一种有效方法,它以递增的方式线性组合多个“弱”模型(单高斯分量),进而得到一个具有很高似然度的“强”模型(高斯混合模型),使得最后所得到的混合模型可以有效地逼近任意复杂的概率分布[2]。
2.1 混合Motif模型
首先定义一个有限字符集 Σ={α1,…,αΩ},其中 Ω= ||Σ ,对于任一序列 S=a1a2…aL( L≥1且ai=Σ),在由序列组成的字符串集Σ中,从位置1到位置L= ||S表示字符串长度。字符序列aiai+1…ai+w-1表示S中的xi开始的长度为W 的字符串。这里有n=L-W+1表示从序列S中找到的字符串的长度[3]。
序列S的概率为
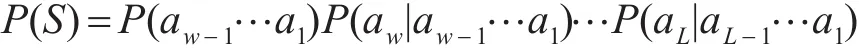
由条件概率公式,可得:

定义一个 yi=aiai+1…ai+w+2表示长度为W-1的字符串。取对数后得到似然函数L:
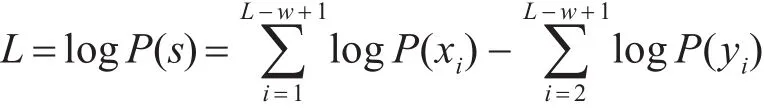
假设有N条不同的序列s={s1,…,sN},其长度分别为L1,…,LN,为了识别长度为W 的Motif,序列S中的字符串长度应为W[4]。由于每条长度为W原始序列SS有ms=Ls-W+1种的可能的组合,因而需要建立一个由n个字符串n=∑Ns=1ms组
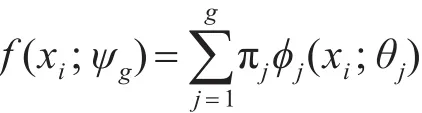
为了解决模型参数的初始化问题,出现了许多算法[5]。其中MEME就是在一次EM迭代后,用动态算法来估计基于似然函数模型的可能起始点。而贪心EM算法是在局部搜索中找到了模型的合适的参数后再进行全局搜索[5],因此本文引入了EM算法的贪心学习过程。
2.2 贪心混合学习
假定一个θg+1(xi;θg+1),将其增加到g划分的混合模型 f(xi;ψg)中。 g+1部分在Motif模型的PWMg+1中含有参数集θg+1。平衡方程如下:
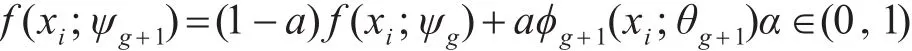
ψg+1表示一个新的参数集,它是由原来混合模型的参数集ψg、权重α和参数集θg+1组成。对ψg+1取似然得到:成的数据集X={x1,…,xn}。
对任意一个字符串xi建立一个混合Motif模型如下:
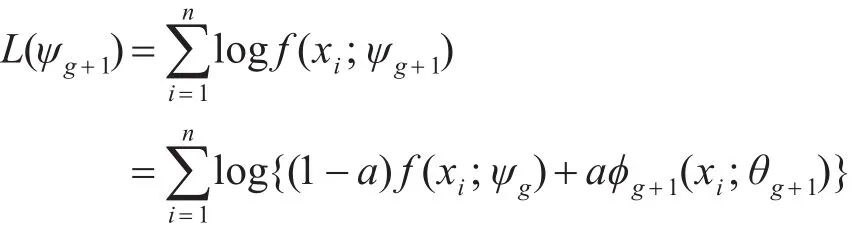
重复执行上面过程得到一个g值,最后得到接近混合分布的对数似然度,上式有两部分,第一个分量是原有混合分布 f(xi;ψg),另一个分量是φg+1(xi;θg+1)。问题就转为运用适当的方法来优化参数 a 和 θg+1,使得 ψg在最大[6]。
考虑到以上因素,本文将局部和全局搜索结合起来,再执行EM算法局部搜L(ψg+1)索来寻找a和θg+1的最大似然,这里通过学习过程可以出求混合权a和新加项分量的概率。
引入全局搜索,用泰勒(Taylor)相似a=a0来代替似然函数,我们可以通过从结果来查找最优θg+1值,在a0=0.5处展开L(ψg+1)的二阶泰勒公式,并最大化a的二次函数:
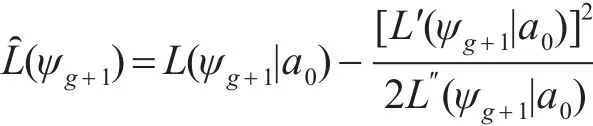
2.3 改进初始化模型参数
用搜索候选字符集的方法直接覆盖含全部字符串的数据集X=,( τ=1,…,n),这 里xτ=aτ1…aτW
[7]。这样每一个字符串 xτ就与 PWM的θτ建立了联系。
搜索候选字符串n增加了时间复杂度(o(n2)),由于每个字符串在候选参数模型中都需要计算,所以o(n2)是必须的[8]。为了减少时间复杂度,我们引入了基于Kd-树结构,通过Kd-树对EM初始化参数进行优化,从而建立了基于Kd-树的贪心EM预测算法(Production Algorithm of Kd-tree Greedy-EM,PKGE算法)。Kd-树是通过定义一个递归的二进制的K维数据集,它的根节点包括所有的数据,每一层通过检测不同的属性(关键字)值以决定选择分枝的方向,从而加快最近临近查询。选择一棵树在水平方向上一个适当的平面在垂直的方向就可以表现出主要数据的变化。从包括全部的字符串集X的根节点开始,每一步都用迭代,在每个节点上进行分隔。当计算出字符串的k位置上每一个字符的相对概率后就包含在这个节点上,在位置q上表现的最大熵值[9]:
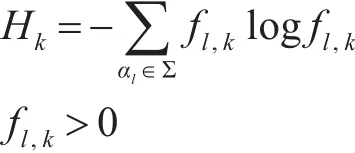
其中分隔是在搜索的字符位置q上,根据标识为“奇数”或“偶数”。生成了两个子集,在位置q上的字符串含有“偶数”和“奇数”字符,在图1中第二个字符位置出现最大熵。以上的迭代生成了含有多个节点的树,当满足其包含的字符串数量小于T时,节点就成为叶子节点[10]。树上每个节点都是一个已经初始化的字符串,每一个这样的字符串集合通过它的质心被特征化,这些叶子节点全部是由质心和相应的PWM组成。

图1 Kd-树结构
3 基于贪心EM算法的改进预测算法(PKGE算法)
设定 N条序列S={SS},长度LS= ||SS,(s=1,…,N),假设 Σ={α1,…,αΩ},长度从 Ω= ||Σ字符集中取值。算法流程如图2所示。
初始化:
1)设定长度W,在集合S中生成一个字符串X={xi}(i=1,…,n),满足n=∑Ns=1(Ls-W+1)。
2)用Kd-树来处理数据集X,通过质心C找到最终的一致性序列,利用全局搜索来定义C的相关初始参数值 θτ,τ=1,…,C[11]。
3)初始化设定 ϖτ=0,∀τ=1,…,C ,计算矩阵数量 ξτ,i。
4)初始化设定g=1,设置背景的参数等于字符的概率 αl∈ Σ ,如,,这里 fl表示字符αl在序列S中的出现频率。
参数迭代部分:
1)EM的收敛条件
2)如果 g≥2,从Motif的候选序列集合 xi(当zig>0.9)中找到与之相邻的 Ni={xj}(j=i-K,…,i+k)g≥2,并设置每个叶子节点ϖτ=1,其中τ可包含任意个xj。
3)增加 g+1部分,搜索 θτ( τ=1,…,C且ϖτ=0)并令 θˆg+1等于 θτ,用 ξτ,i来代替 φ(xi;θτ),用它的最大似然函数。再利用公式θτ=[pτl,k],中的 θˆg+1的值来计算权aˆ。
4)用 aˆ和 θˆg+1的初始值执行EM,直到执行到收敛条件 ψg+1为止[13]。
5)如果 L(ψg+1)>L(ψg),则利用 g+1划分的混合模型并重新转向1步,否则终止。直到求出Motif的最大数量,或者
算法描述了在Motif预测中的一个混合Motif模型。由于EM算法不能减小似然,所以只有当满足 L(ψg+1)>L(ψg)时才利用局部EM迭代来解决问题,从而保证了在数据集中似然函数的单调增加。算法终止的条件包含g的取值和向量集ϖ的最大值。在这里 ϖτ=1,∀τ=1,…,C[14],候选序列部分的参数空间被彻底搜索,因此其它Motif在序列大量存在的可能性是很低的。
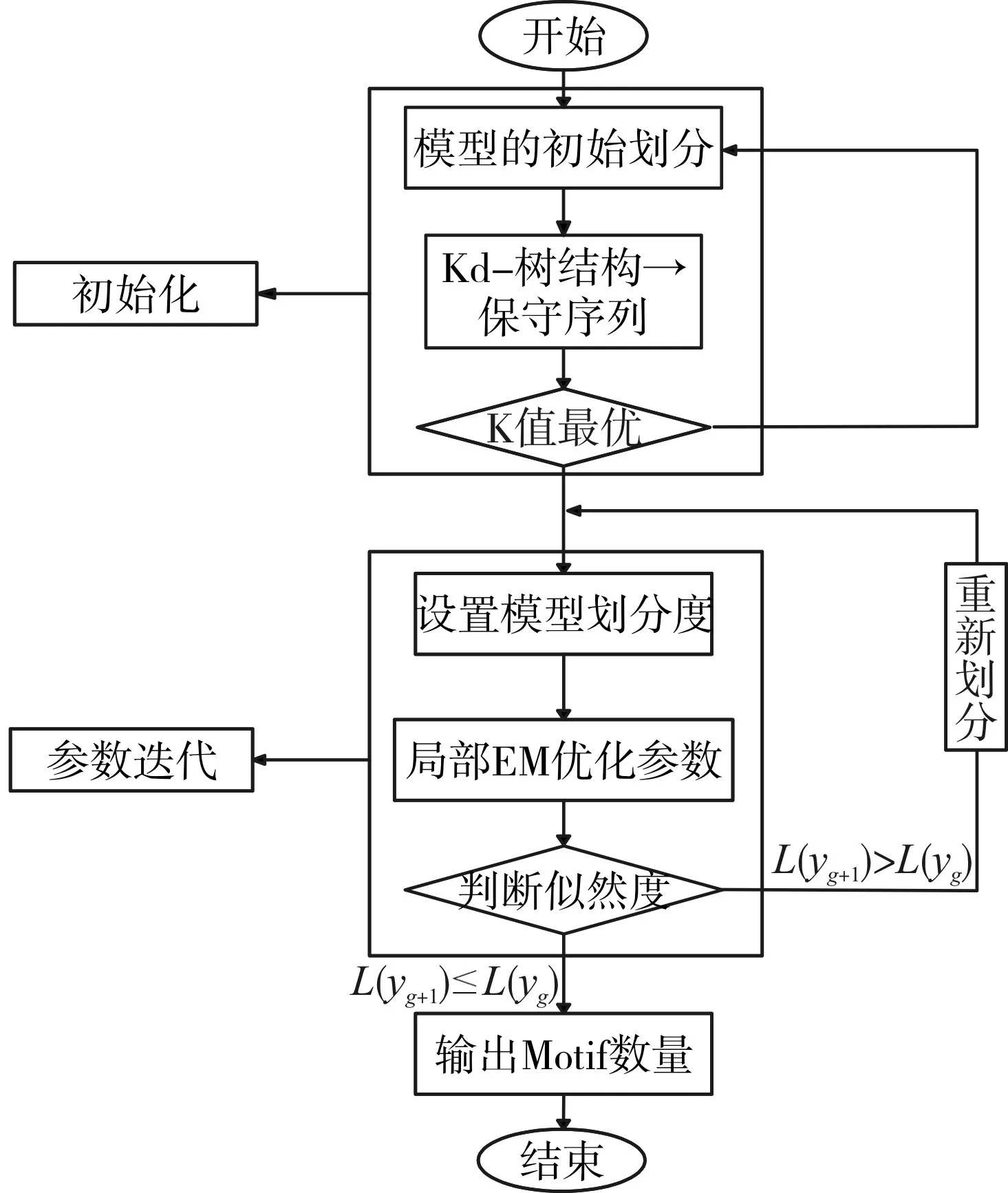
图2 PKGE算法流程图
4 算法测试结果
从PRINTS数据库下载部分蛋白质家族序列,随机产生8条长度在150bp~300bp之间[8,12]的蛋白质序列,通过多次比对设定Kd-树划分值为6,叶子节点字符串的长度设定为5,Motifs长度为10,由于选定序列源于生物数据库,虽经过人工拼接,但仍有部分片段保留,因而程序执行后仍能找到表1所示数量的Motifs。
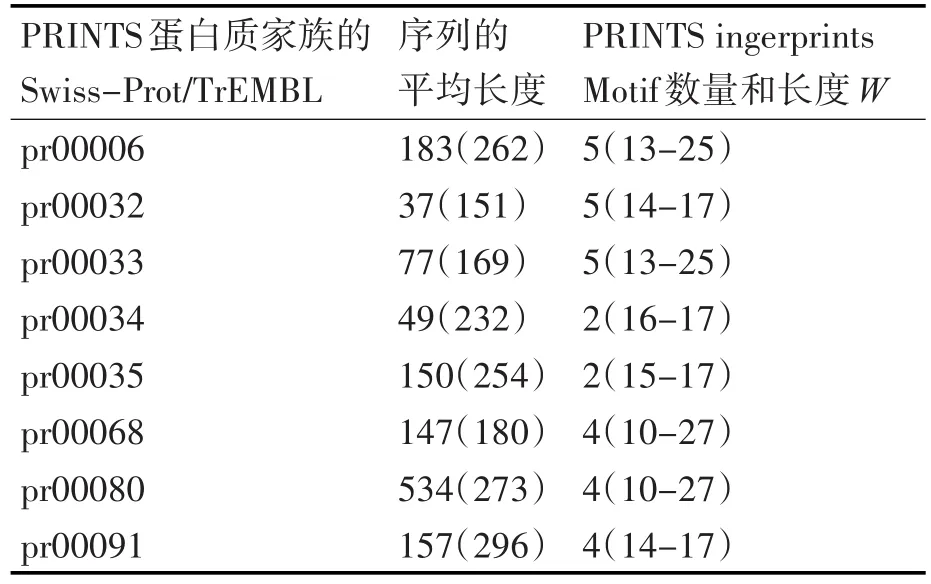
表1 预测的PROSITE蛋白质家族[15]
PKGE算法能显著降低Motif的重叠率,而且还能保证Motif候选序列参照一致的规则进行处理,此外通过迭代还减少了全局搜索的时间复杂度,与MEME算法相比,相邻的Motif字符串从数据集中剔除了,重叠的字符串被排除在候选Motif集合之外,这就相对MEME算法有了改进。PKGE算法主要是针对MEME的局限性来设计的,例如每一次输入一个新的Motif序列时需要假定这个序列是真实存在的,从而限制了MEME模型中两部分的联系,PKGE算法通过增加混合密度估计改进了这个问题。

表2 算法性能比较
5 结语
算法实现了预测性能的一定改进,但其特异性的绝对水平依然很低。对众多预测算法的评估研究表明,它们通常仅在特定的小规模数据集上表现出良好的性能,而对更为广泛和通用的数据集,算法产生了大量假阳性结果,这应该是导致此现象的直接原因,此外,限于当前的认识水平,假阳性结果中有些可能确实是尚未发现的结合位点,其功能需要后续的实验来验证。
[1]张斐,谭军,谢竞博.基于不同算法的motif预测比较分析与优化[J].计算机工程,2009,35(22):94-96.
ZHANG Fei,TAN Jun,XIE Jingbo.Comparison,Analysis and Optimization of Motif Finding Based on Different Algorithms[J].Computer Engineering,2009,35(22):94-96.
[2]王维彬,钟润添.一种基于贪心EM算法学习GMM的聚类算法[J].计算机仿真,2007,24(2):65-68.
WANG Weibin,ZHONG Runtian.A Clustering Algorithm Based on Greedy EM Algorithm Learning GMM[J].Computer Simulation,2007,24(2):65-68.
[3]J J Verbeek,N Vlassis,B Krose.Efficient greedy learning of Gaussian mixture[J].Neural Computation,2003,2(15):469-485.
[4]S K Palaniswamy,S James,H Sun,et al.AGRIS and AtRegNet.A platform to link cis-regulatory elements and transcription factors into regulatory networks[J].Plant Physiol,2006(140):818-829.
[5]MTompa,NLi,TLBailey,et al.assessing computational tools for the discovery of transcription factor binding sites[J].Nat Biotechnol,2005,23:137-144.
[6]T LBailey.Discovering Motifs in DNA and protein sequences:theappoximate common substring problem[D].San Diego:University of California,1995,12-13.
[7]T LBailey,CElkan.Unsupervised learning of multipleMotifs in biopolymers using expectation maximization[J].Machine Learning,1995,21:51-83.
[8]L BTimothy,WNadya,M Chris,et al.MEME:discovering and analyzing DNA and protein sequence Motifs[J].Nucleic Acids Research,2006,34(Web Server issue):369-373.
[9]A P Dempster,N M Laird,D B Rubin.Maximum likelihood from incomplete data via the EM algorithm[C]//J.Roy.Statist.Soc,B,1977,39:1-38.
[10]吴昕,罗静初,李伍举.基因调控元件的计算机识别和基因调控网络构建[J/OL].http://www.cbi.pku.edu.cn/chinese/documents/papers/WuX.pdf.
WU Xin,LUO Jingchu,LI Wuju.Computer recognition of regulatory elements and gene regulatory network[J/OL].http://www.cbi.pku.edu.cn/chinese/documents/papers/WuX.pdf.
[11]韩华,刘婉璐,吴翎燕.基于模体的复杂网络测度量研究[J].物理学报,2013(16):1-9.
HAN Hua,LIU Wanlu,WU Lingyan.The measurement of complex network based on motif[J].Acta Physica Sinica,2013(16):1-9.
[12]TObayashi,K Kinoshita,K Nakai,et al.ATTED-II:a database of co-expressed genes and cis elements for identifying co-regulated gene groups in Arabidopsis[J].Nucleic Acids Res,January 12,2007,35(suppl_1):863-869.
[13]覃桂敏,高琳,呼加璐.生物网络模体发现算法研究综述[J].电子学报,2009(10):2258-2265.
QIN Guimin,GAO Lin,HU Jialu.A Review on Algorithms for Network Motif Discovery in Biological Networks[J].Acta Electronica Sinica,2009(10) :2258-2265.
[14]霍红卫,郭丹丹,于强,等.(l,d)-模体识别问题的遗传优化算法[J].计算机学报,2012,35(7):1429-1439.
HUO Hongwei,GUO Dandan,YU Qiang,et al.Genetic Optimization for(l,d)-Motif Discovery[J].Chinese Journal of Computers,2012,35(7):1429-1439.
[15]宋涛.基于谱隐马尔可夫模型的蛋白质序列模体识别方法研究[D].大连:大连理工大学,2015:60-61.
SONG Tao.Research on Methods for Protein Sequence Motif Discovery Based on Profile Hidden Markov Model[D].Dalian:Dalian University of Technology,2015:60-61.

