新疆卡拉麦里山有蹄类自然保护区蒙古野驴mtDNA D-loop区的遗传多样性及系统发育研究
冯 锦 初雯雯端肖楠 胡德夫吴洪潘 葛 炎初红军*
(1.新疆大学生命科学与技术学院,乌鲁木齐,830046;2.新疆卡拉麦里山有蹄类野生动物自然保护区管理中心,乌鲁木齐,830000;3.北京林业大学自然保护区学院,北京,100083)
蒙古野驴(Equushemionus)属奇蹄目(Perissodactyla)、马科(Equidae)、马属[1],是亚洲荒漠及荒漠草原的代表性物种,属于国家I级重点保护野生动物,被列入濒危野生动植物种国际贸易公约(CITES)附录I[2]。曾广泛分布于我国甘肃、青海、新疆及内蒙古等地[3],目前分布局限在新疆卡拉麦里山有蹄类自然保护区(以下简称卡山保护区)与内蒙古乌拉特梭梭林蒙古野驴自然保护区内[2]。
国内对于蒙古野驴的研究主要集中在数量与分布[2,4-7]、行为[8-9]、食性[10-13]、生态和种群结构[14]、种群交流[15]等方面,其遗传多样性研究没有开展。
线粒体DNA(Mitochondrial DNA,mtDNA)是细胞质遗传的物质基础,与细胞核DNA相比,具有分子结构简单、无重组、进化速率快和严格遵循母系遗传等特点[16]。线粒体DNA控制区(以下简称D-loop区)又称为替代环区(displacement-loop region)或D环,是线粒体中碱基序列和长度变异最大的区域,进化速率是其他区段的5倍[17-18],具有碱基替换速率快、存在丰富的序列变异等特点,特别适合于检测种群内及种群间的遗传多样性[19-21]。
我们利用线粒体D-loop区对卡山保护区蒙古野驴的遗传多样性进行了研究,在此基础上分析了卡山保护区蒙古野驴种群濒危的可能原因和机制,从分子遗传学角度为蒙古野驴种群有效保护和遗传管理方案的制定提供了科学参考依据。
1 研究方法
1.1 研究区概况与样品采集
卡山保护区地处新疆准噶尔盆地古尔班通古特沙漠东缘,地理坐标为N44°36′~46°00′,E88°30′~90°03′,海拔高度800~1467 m,属低山荒漠、半荒漠景观。在气候上属中温带大陆性气候,其特点是冬季寒冷漫长、秋季温凉、夏季炎热短暂、春季干旱少雨。卡山保护区植被稀疏,野生动物以适应干旱和半干旱的种类占优势,大中型有蹄类动物和小型啮齿动物是卡山保护区的代表性物种,主要有蒙古野驴、野放普氏野马(Equusprzewalskii)、盘羊(Ovisammon)、鹅喉羚(Gazellasubgutturosa)、猞猁(Lynxlynx)等[22]。
2013~2015年间,对卡山保护区进行日常野外监测时发现的蒙古野驴尸体进行采样,最终所得样品共30份,其中皮张11份,肌肉组织19份。在采集过程中,对样本信息进行详细记录,采集之后立即放入-20℃冰箱储存。
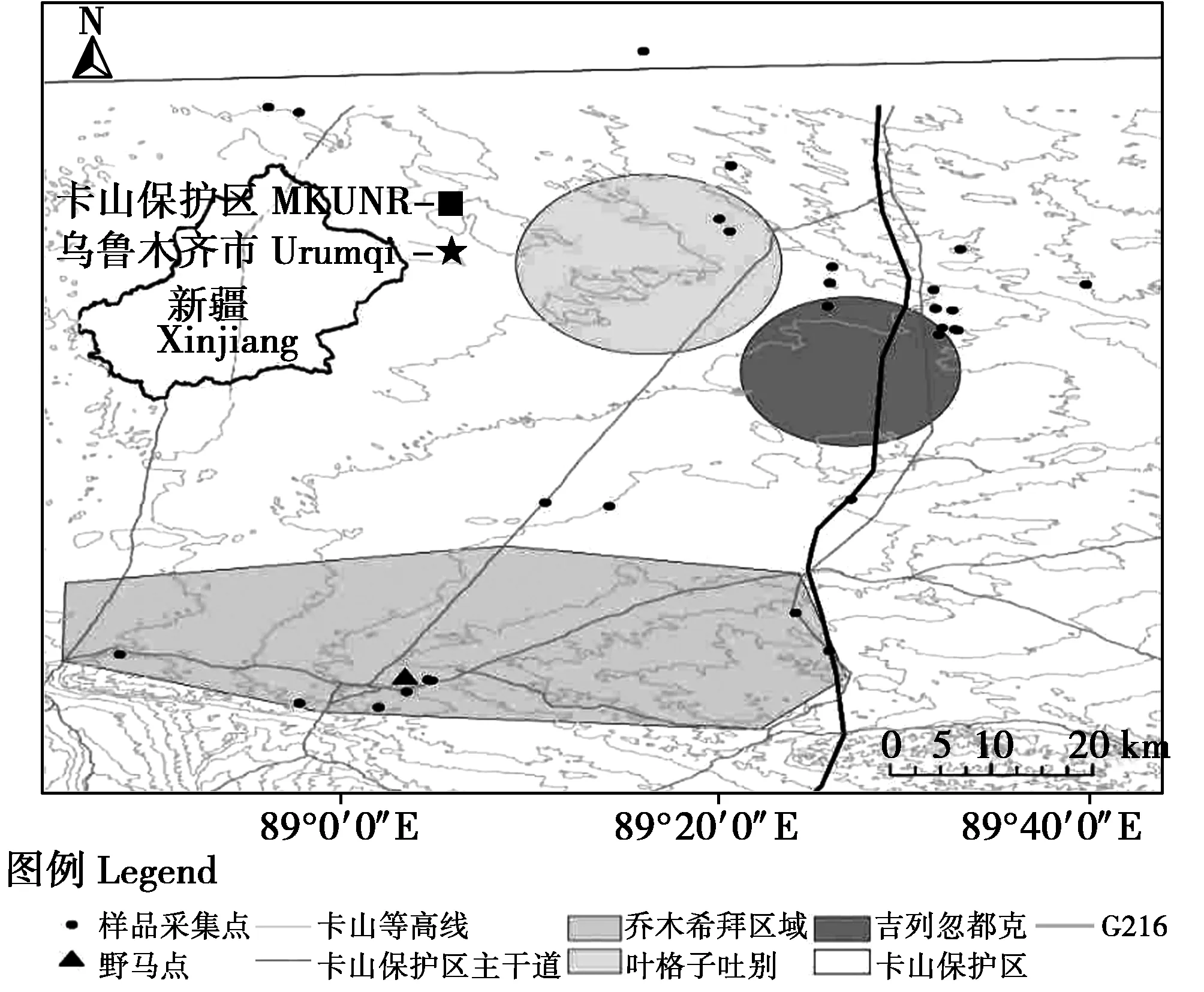
图1 卡拉麦里有蹄类自然保护区蒙古野驴采样点Fig.1 Equus hemionus sampling points in the MKUNR of Xinjiang
1.2 DNA提取、PCR扩增与测序
蒙古野驴皮张和肌肉组织mtDNA通过细胞/组织基因组DNA提取试剂盒(离心柱型)(北京百泰克生物技术有限公司)提取。根据GenBank检索的藏野驴mtDNA全序列[23],蒙古野驴mtDNA D-loop区引物通过Primer软件进行设计,由生工生物工程(上海)股份有限公司合成。
上游引物:
5′-CTTGTAAACCAGGAAAGGGGGAAAC-3′;
下游引物:
5′-ATTTAGAGGGCATTCTCACTGGGAT-3′。
PCR反应体系25 μL,其中2×TaqPCR MasterMix 12.5 μL,DNA模板1 μL,上下游引物各1 μL,ddH2O 9.5 μL。扩增过程:94℃预变性3 min,94℃变性1 min,60℃退火1 min,72℃延伸1 min,进行35个循环;最后72℃延伸10 min。PCR产物经1%琼脂糖凝胶电泳后,取扩增效果良好的样品20 μL,由北京六合华大基因科技有限公司进行双向测序。
1.3 数据分析
用DNAStar 5.0软件对所测序列进行拼接、比对并进行人工校对;运用MEGA 6.0软件的Clustal X 1.81进行多序列同源比对,统计碱基组成、转换、颠换和单倍型间Kimura参数模型的遗传距离,基于Kimura双参数距离分别用邻接法(NJ)和最大似然法(ML)等方法构建种内聚类关系;DnaSP 5.10软件进行多态位点、单倍型多样性和核苷酸多样性分析;Network 5.0软件构建单倍型网络关系图;并用 Analysis 模块进行Tajima’s D中性检验。
2 结果与分析
2.1 序列长度与碱基组成
卡山保护区采集的30个蒙古野驴样本中4个样本因保存不当或材料消耗而缺失。成功扩增并测序了26个mtDNA D-loop区848 bp的序列,T、C、A、G 等4种核苷酸的平均比例分别为25.9%(25.1%~26.3%)、30.0%(25.4%~31.0%)、27.4%(26.5%~29.2%)、16.7%(16.1%~18.6%),A+T含量53.3%,G+C含量46.7%,表明蒙古野驴mtDNA D-loop区富含碱基A+T,表现出一定的碱基偏好性。
2.2 序列的核苷酸多态性
全序列共有保守位点601个,变异位点247个,其中单一多态位点178 个,占多态位点总数的72.1 %,简约信息位点69个,占多态位点总数的 27.9 %。序列变异中碱基缺失、插入和碱基替换等均有,其中碱基替换变异类型中转换195次,颠换46次,在转换变异类型中以A/G为主,占52.2%,在颠换变异类型中以A/T为主,占60 %。蒙古野驴 mtDNA D-loop区核苷酸多样性(π)为(0.2039±0.0523)。对测定的序列进行Tajima’s D值中性检验,Tajima’ s D值为负值但不显著(D=-0.722;P>0.10),表明这些序列符合中性进化模式,蒙古野驴在整体水平上并未出现过瓶颈效应或者快速扩张等历史事件。
2.3 序列的单倍型分布
根据蒙古野驴mtDNA D-loop区序列247个变异位点,在26个样品中鉴定出23种单倍型(H1-H23),其中H3包含4条,其他22种单倍型都只包含1条序列,单倍型多样性(Hd)为(0.982±0.020),表明蒙古野驴具有丰富的单倍型类型。
2.4 种内系统发育分析
蒙古野驴23个单倍型间的 Kimura 双参数遗传距离变异范围为 0.001~1.846,平均遗传距离是0.435(表1)。基于Kimura 双参数距离采用NJ、ML法对蒙古野驴的D-loop区23个单倍型的发育树显示相同的拓扑结构(图2,图3),都显示2个主要分支。结合单倍型遗传距离可看出:A包括3种单倍型(H4,H10,H15)共3个个体,占全部单倍型的13%;分支B包括余下的20种单倍型共23个个体,占全部单倍型的87% 。
表1 蒙古野驴23个单倍型间 Kimura 双参数遗传距离

Tab.1 Equus hemionus 23 haplotype Kimura two-parameter genetic distance

图2 蒙古野驴mtDNA D-loop区23个单倍型NJ发生树Fig.2 Equus hemionus mtDNA D-loop region 23 haplotypes NJ tree
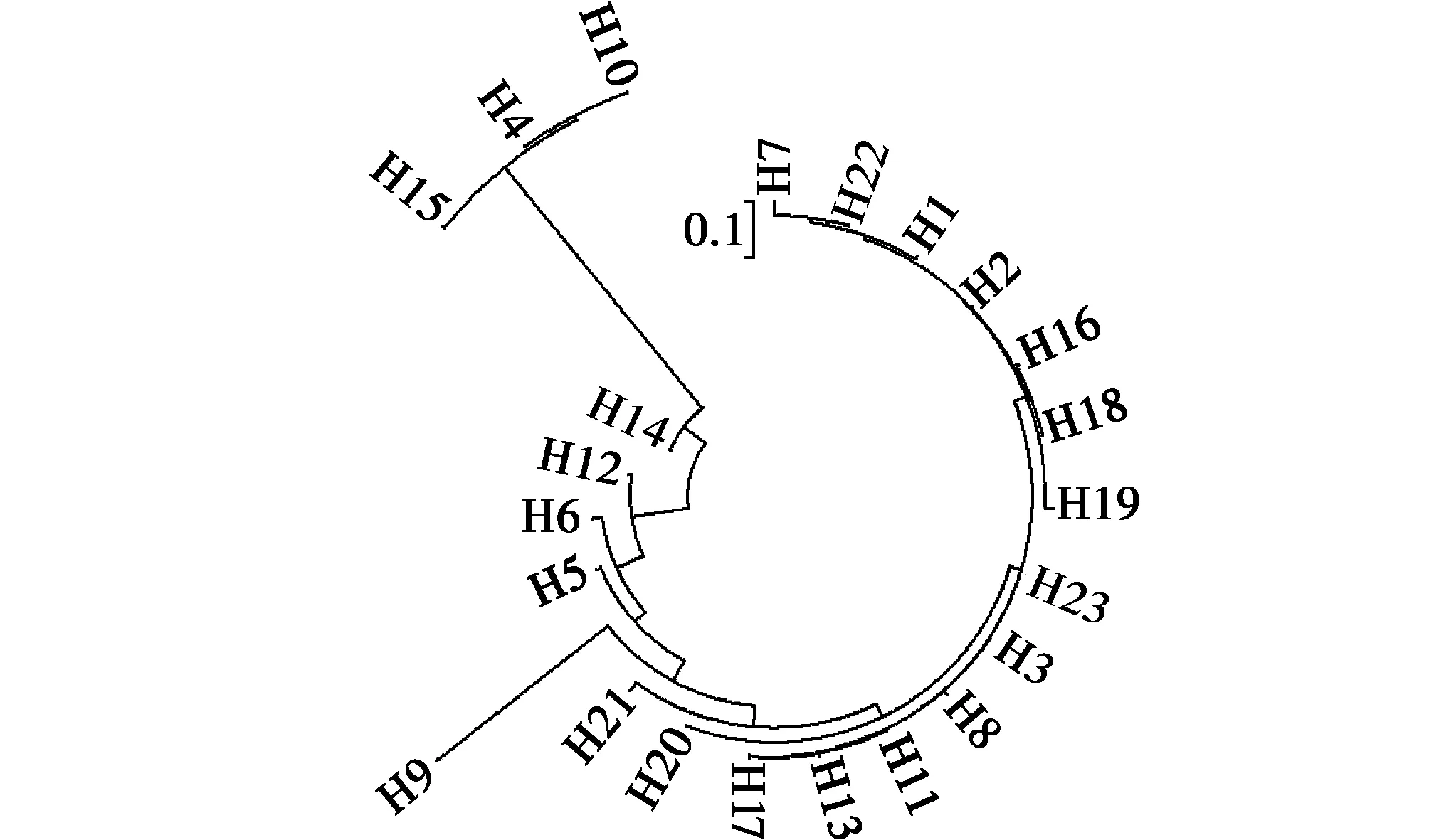
图3 蒙古野驴mtDNA D-loop区23个单倍型ML发生树Fig.3 Equus hemionus mtDNA D-loop region 23 haplotype ML tree
根据蒙古野驴 mtDNA D-loop区单倍型序列的变异位点,运用 Network 5.0 软件得出蒙古野驴单倍型网络关系图(图4)。发现23种单倍型根据演化距离满足拓扑结构分为的2个主要分支,其中单倍型H4,H10,H15相较于其他单倍型亲缘距离较远。再结合图2所示,H4、H10、H15先与H14聚在一起,然后再与H9聚在一起;而图3所示,H4、H10、H15先与H14聚在一起,然后再与H12聚在一起。从单倍型遗传距离中也可看出,分支A与分支B之间相比于同支内单倍型之间关系较远。
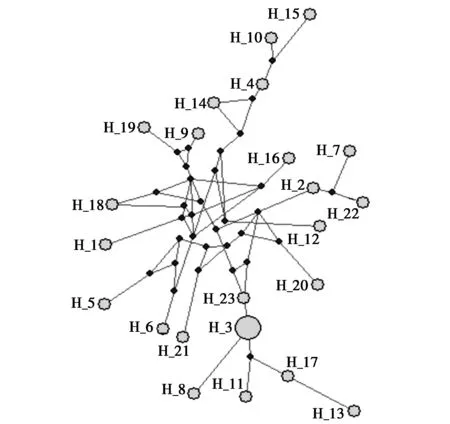
图4 蒙古野驴mtDNA D-loop区23个单倍型网络关系图Fig.4 Equus hemionus mtDNA D-loop region 23haplotypes network diagram
2.5 与其他野生马科动物的亲缘关系
将蒙古野驴序列和马科中的其他7个物种D-loop区基因序列进行比较,利用GenBank检索的其他马科野生动物的同源序列,以奇蹄目貘科(Tapiridae)的马来貘(Tapirusindicus)为外群,基于Kimura双参数距离应用最大似然法(ML)和邻接法(NJ)构建马科D-loop区基因分子系统进化树(图5,图6),进化树各分支上的数值表示1000次自举(bootstrap)检测得出对该分支的支持百分数。两种不同的分子系统进化树得到的结果相同。通过ML法得到进化树的拓扑结构中,平原斑马(Equusquagga)、山斑马(Equuszebra)和努比亚野驴(Equusasinusafricanus)形成了一个进化枝,置信度较低为32%。蒙古野驴先与藏野驴(Equuskiang)聚在一起,置信度为97%。后与细纹斑马(Equusgrevyi)三者形成一个进化枝。然后两个进化枝聚集在一起形成一支,置信度为56%。最后再与普氏野马、非洲野马(Equusferus)形成一支。马科动物内8个物种中,蒙古野驴与藏野驴的亲缘关系最近,普氏野马与非洲野马亲缘关系最近。马科动物中平原斑马、山斑马、努比亚野驴、蒙古野驴和藏野驴出现较早,其中最早出现的物种是平原斑马。
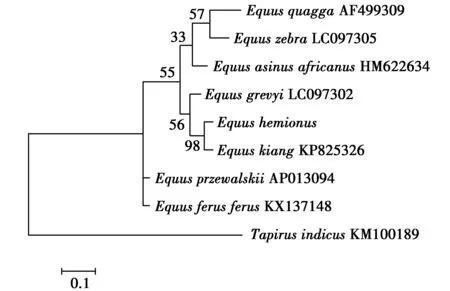
图5 基于D-loop基因序列构建的马科物种的最大似然法(ML)进化树(图中数字为自引导值)Fig.5 Maximum likelihood method(ML)evolutionary tree basedon the D-loop gene sequence constructed from equine species(The numbers in the figure are self-directed values)
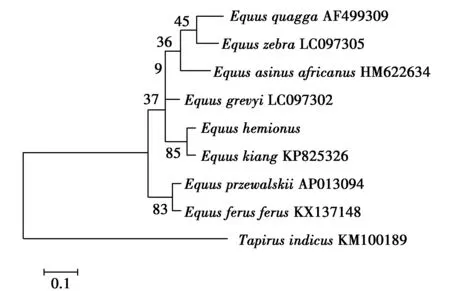
图6 基于D-loop基因序列构建的马科物种的邻接法(NJ)进化树(图中数字为自引导值)Fig.6 Adjacent(NJ)phylogenetic tree based on the D-loop genesequence (The numbers in the figure are self-directed values)
3 讨论
3.1 蒙古野驴mtDNA D-loop区的遗传多样性
本实验采用测序法,测定的26匹蒙古野驴mtDNA D-loop区848 bp的序列中,有247个变异位点,占所测核苷酸数的29.03%,序列变异中碱基缺失、插入、替换、颠换等均存在。而3个克罗地亚驴群体28匹的mtDNA D-loop区391 bp序列进行分析,共检测到36个核苷酸多态位点,约占所测核苷酸数的9.21%,其中有转换、颠换、插入位点3种类型[17]。蒙古野驴mtDNA D-loop区序列核苷酸相比克罗地亚驴突变率较高。同时我们检测的247个核苷酸多态位点中,转换195次,颠换46次,具有较强的偏向性,与mtDNA进化过程中碱基变异发生转换频率高于颠换的结果相同。
通过对卡山保护区蒙古野驴线粒体DNA D-loop区基因的测定及分析,得出核苷酸多样性(π=0.2039±0.0523)大于通过D-loop基因与Cytb基因研究[16]的蒙古野马、家马、驴核苷酸多样性(π=0.00925/0.01022),也大于王小斌等[24]研究的蒙古马核苷酸多样度(π=0.02756±0.00967)及阿地力江·卡德尔[25]研究新疆吉木萨尔繁殖中心的普氏野马核苷酸多样性(π=0.029)。其单倍型多样性(Hd=0.982±0.020),与海棠等[16]分别通过D-loop基因与Cytb基因研究的蒙古野马、家马、驴单倍型多样性(Hd=0.9356/0.400~1.000)结果大致相等,与王小斌等[24]研究蒙古马单倍型多样性(Hd=0.979±0.007)大致相等,也略小于阿地力江·卡德尔[25]研究新疆吉木萨尔繁殖中心的普氏野马单倍型多样性(Hd=1±0.00116)。说明本区域蒙古野驴比其他马科动物核苷酸多样性较高,而单倍型多样性大致相同。Khaire 等[26]利用微卫星标记研究印度野驴遗传多样性时,得出遗传多样性低的结果与我们结果不同。Nasiri-Moghadam等[27]采用微卫星标记研究波斯野驴种群遗传结构时发现人为因素是导致这种濒危亚种种群数量减少的主要原因,但对过去近百年野驴种群规模变化的评估中并未显示出突然减少,且种群遗传多样性较为稳定。我们与波斯野驴研究结果相同的是历史上蒙古野驴也曾广泛分布在蒙古高原的中西部、亚洲中部、印度次大陆和西南亚地区,即使近代文明兴起短期内导致其分布区和种群数量减少,但仍然保留了蒙古野驴丰富的遗传多样性。中性检验也证明蒙古野驴种群在过去并未发生过明显的瓶颈效应或进行快速的扩张,而是处在种群的被动平衡状态。
采用邻接法(NJ)和最大似然法(ML)构建蒙古野驴mtDNA D-loop区23种单倍型的系统发育树,拓扑结构均分为较为明显的两支。蒙古野驴单倍体间遗传距离(表1)数值差异表现23个单倍体中有3个特殊单倍体H4、H15、H10,系统发育树得到的结果一致。由于线粒体DNA具有严格的母系遗传及单倍体遗传的特性,因此相同母系个体拥有相同的单倍型[28]。由此推断,这两支蒙古野驴可能来源于具有一定遗传差异的群体,而不是少数几个关系非常近的母体。Feh等[14]利用景观生态学研究蒙古戈壁蒙古野驴生态和种群结构中提出由于戈壁公园野驴有向西或者向中国北部迁徙可能。Kaczensky等[15]利用历史记载、当前分布范围、群体遗传学及遥测数据等评估了蒙古戈壁蒙古野驴种群在自然景观、人为景观和现有保护网络通道区域的连通性,将卡山保护区与蒙古国戈壁保护区称为“跨界生态走廊”,两国边界某些围栏打开将扩大或重新连接蒙古野驴及其他哺乳动物种群之间的交流。卡山保护区位于准噶尔盆地东部,历史上记载及地理遥感信息显示在阿尔泰山以东蒙古戈壁国家公园与卡山保护区交界处并不存在较大的地理隔离,地理空间的连通性及系统进化树分析让我们猜测蒙古野驴种群有外来迁入可能。由Network中可以看到,所有单倍型起源于假想单倍型mv1,但mv1所在B支分离出去的分支中也在进化过程中出现分歧,因此符合我们对卡山保护区蒙古野驴外来迁入的猜测。
3.2 马科内系统发育地位
本研究将所得蒙古野驴D-loop区序列和利用GenBank检索的其他马科其他7个物种同源序列进行比较,发现蒙古野驴与藏野驴亲缘关系最近,而同区域生存的普氏野马与非洲野马亲缘关系最近。虽然国内尚未在分子水平上对蒙古野驴进行系统研究,以往学者[29-30]认为蒙古野驴和藏野驴只达到亚种的分化水平,现在则认为它们是亲缘关系很近的不同种[31-33]。藏野驴过去被认为是亚洲野驴的一个亚种[34],Ryder和Chemnick[35]通过比较二者的染色体和线粒体DNA证明二者并非同一物种,但证明了蒙古野驴与西藏野驴之间亲缘关系最近。Steiner等[36]和Vilstrup等[37]通过单倍型分析研究发现,藏野驴是亚洲野驴经过单倍体较大变异而形成的分支。Rosenbom等[38]在对亚洲野驴建立系统发育树发现,亚种野驴被确定为蒙古野驴和藏野驴两分支,这些分支没有进一步分化。Khaire等[39]在研究印度野驴进化过程中通过Cytb和D环构建马科动物系统发育树中,藏野驴与蒙古野驴关系最近,这与我们研究结果相同。Orlando等[40]通过非洲野驴化石与蒙古野驴和藏野驴进行形态学比较发现三者较相近,从中提取了线粒体Cytb基因和D-loop基因。在Genbank中下载马科动物Cytb基因进行比较发现非洲野驴与蒙古野驴遗传距离最小,比较D-loop基因发现非洲野驴只与蒙古野驴和藏野驴在mtDNA CR中存在共享缺失,构建系统进化树得出藏野驴与蒙古野驴关系最近的结果与我们相同。另外,系统发育树显示平原斑马最早出现,山斑马首先分裂,然后是蒙古野驴、藏野驴、努比亚野驴,然后是细纹斑马,最后是普氏野马与非洲野马。Jansen等[41]在利用马科动物mtDNA D-loop区等序列构建系统发育网络图发现马科动物分化先后顺序与我们的结果相同。Mashima[42]在对普氏野马和家马在内的4种马科动物研究中,利用mtDNA构建马科系统发生树得出普氏野马是由家马演化出来的,对于马科其他动物演化顺序与本研究大致相同。Orlando等[43]在对多种马科动物标本形态学与mtDNA基因比较后提出,将海角斑马、平原斑马、蒙古野驴及非洲野驴等归为一个物种,这在现代学分类上与我们结果不同,但在我们得出马科动物前期演化顺序基础上可将几者视为一大类。
通过分子上研究发现,开展卡山保护区蒙古野驴的遗传多样性研究尤为重要。在保护蒙古野驴多样性以及种群稳定前提下,应对卡山保护区进行合理规划,避免挤占蒙古野驴等野生动物的生存空间。另外,通过卡山保护区广大地理面积与丰富的野生动植物资源,增加保护和管理力度,保持物种繁育潜力和努力保存蒙古野驴存在最大种群数量,抵消近几十年人为因素导致蒙古野驴栖息地骤减、过度捕猎及偷猎造成数量减少的严重损失。
目前,对于卡山保护区蒙古野驴遗传多样性的研究仅限于mtDNA D-loop 区,条件具备时利用mtDNA Cytb、核DNA、Y染色体以及全基因组测序,对其进行遗传多样性、遗传分化以及发育地位等方面的研究。
致谢:本研究得到科技部科技基础性工作专项重大项目(2013FY110300),卡拉麦里山有蹄类自然保护区阿勒泰管理站中央财政森林生态效益补偿基金项目,新疆自治区科技支撑计划项目(201233131)的资助。卡山保护区阿管站蒋新春、蒙坎、李斌、白硕、艾代、蒋锋、阿尔成、叶尔江、木合买提、哈力和木合牙提等同志参加野外标本等数据收集工作;新疆大学资环学院硕士研究生王渊、刘元超、新疆大学生命学院硕士研究生杜聪聪以及新疆农业大学动科学院硕士研究生董潭成等同学参加数据收集工作;新疆卡拉麦里山有蹄类野生动物自然保护区阿勒泰管理站哈孜拜副站长、布兰科长、梁兴凯科长等同志在工作和生活上给予大力支持和帮助;新疆大学张富春教授、马纪教授、孙素荣教授和马哈木提教授等提供实验室并给予热情指导和帮助;新疆大学生命学院硕士研究生杜聪聪同学给予实验技术指导和帮助,谨致衷心感谢。

