带权疾病网络上的潜在共病关系预测
安莹 王志娜 陈先来 刘莉 李忠民 罗熹

摘 要:网络分析法将潜在的共病关系预测转化为复杂网络上的链路预测问题,而现有的基于相似性度量的链路预测方法大多仅单一地考虑某一方面的网络特征,大大影响了预测的准确性. 使用3个不同来源的真实医疗数据集分别构建了相应的带权疾病网络,并通过对不同网络结构差异性的比较,分析了现有的网络相似性度量指标的局限性. 在此基础上,提出了一种新的基于有监督分类的链路预测方法,综合多种局部和全局相似性指标作为输入特征向量,更为精确地评估节点间的相似性,从而实现潜在共病关系的有效预测. 实验结果表明,该方法能有效提高共病网络中链路预测的准确性,并且对于不同共病网络和分类算法均具有较好的稳定性和适用性.
关键词:共病;网络分析;监督学习;链路预测
中图分类号:TP399 文献标志码:A
Prediction of Latent Comorbidity Relationship in Weighted Disease Network
AN Ying1,2?,WANG Zhina1,CHEN Xianlai2,LIU Li2,LI Zhongmin2,LUO Xi3
(1. School of Information Science and Engineering,Central South University,Changsha 410083,China;
2. Institute of Information Security and Big Data,Central South University,Changsha 410083,China;
3. Department of Information Technology,Hunan Police Academy,Changsha 410138,China)
Abstract: Network analysis method transforms the prediction of potential comorbidity relationships into link prediction problems on complex network. However,most existing similarity measurement methods only consider a certain aspect of network characteristics,which greatly affects the accuracy of prediction. In this paper,three weighted disease networks are established using the real medical datasets from different sources. By comparing the structural differences of different networks,the limitations of existing network similarity indicators are analyzed. On this basis,a new link prediction method based on supervised classification is proposed,which integrates multiple local and global similarity indexes as input feature vectors in order to more accurately evaluate the similarity between nodes. Thus,the effective prediction of potential comorbidity relationships is achieved. The experimental results show that the proposed method can effectively improve the accuracy of link prediction in comorbidity network and has better stability and adaptability in different disease network and classification algorithms.
Key words: comorbidity;network analysis;supervised learning;link prediction
共病是一种或多种附加疾病与某种主要疾病在同一患者身上共同发生的现象. 由于目前针对共病的医学研究尚不完善,许多疾病之间的相互关系仍然具有很大的不确定性,这使得如何有效地分析疾病之间的关联,从而发现潜在的共病關系,预测共病的发展趋势成为当前备受关注的研究热点.
由于网络分析理论能简洁直观地展现实体对象之间的联系,已经在生物、医学、信息等领域得到了广泛应用[1-3]. 人类疾病也可以构建成一个复杂的网络系统,不同疾病之间存在着不同的共病关系. 疾病网络分析通过构建疾病网络模型,将共病关系的预测问题转化为网络上的链路预测问题,为潜在共病关系的发现以及共病发展趋势的预测提供了一种全新的研究视角. 目前,研究人员针对链路预测问题已经开展了大量的研究. 其中,最具代表性的是基于相似性的链路预测方法[4-6]. 由于这些方法中相似性的计算仅片面地考虑了网络节点或结构的部分特征,无法全面反映网络的拓扑特性,大大影响了链路预测结果的准确性. 此外,由于不同网络系统的结构具有显著的差异性,这进一步导致现有的基于相似性的链路预测方法在不同网络上的表现极不稳定,算法的泛化能力不足.
针对上述问题,本文提出一种带权疾病网络上的新型共病关系预测方法. 相比已有的研究成果,本文的主要贡献包括:
1)本文基于MIMIC-III数据集、湘雅医疗数据集、德克萨斯州健康数据集分别构建了带权共病网络,并分析和比较了3个共病网络在网络结构上的差异.
2)本文将共病关系的发现转化为共病网络上的链路预测问题. 针对现有链路预测方法考虑因素相对单一,预测准确性差,对不同网络的适应性不足等缺陷,提出了一种新的基于有监督分类的链路预测方法,综合多种局部和全局相似性指标作为输入特征向量,实现潜在共病关系的有效预测.
3)利用3个不同来源的真实医疗数据集对本文提出的方法进行验证,实验结果表明该链路预测方法有效地提高了预测的准确性,同时在不同疾病网络上均表现出了较稳定的性能.
1 相关工作
网络分析方法作为研究生物系统的有力工具,对生物信息学、医学的研究越来越重要. 近年来,研究人员提出了许多不同的生物系统网络,如蛋白质交互作用(PPI)网络、新陈代谢网络、人类疾病网络(HDN)等[7-9]. 此外,一些包含多种生物学实体的异构网络也相继出现. 例如,Sun[10]提出一种整合药物、疾病和基因的多层网络模型,旨在更直观地理解药物-疾病-基因的交互作用机制;Zhou等人[11]构建了人类疾病-症状网络,用于发现疾病症状相似性与共享基因数量间的强关联性.
链路预测(Link prediction)可用于评估已知网络中链路存在的可能性,目前已经在许多科学领域开展了大量的相关研究和应用[12-15].共病的分析研究有助于发现共存疾病间的潜在联系,实现潜在共存疾病的早期预警,对于提升患者生活质量具有重要意义. 近年来,如何通过链路预测解决疾病风险预测及潜在共病关系发现的问题成为了该研究领域关注的热点. Gül等人[16]使用疾病的共现关系构建带权网络,利用现有的链路预测方法评估疾病间的潜在关系. Kaya等人[17]使用年龄特性构建疾病进化网络,提出一种采用监督策略的链接预测方法识别疾病间的潜在联系. Shin等人[18]提出了一种基于疾病网络计算疾病共现概率的方法.
在现有链路预测方法中,研究人员大多使用基于相似度的预测算法,为节点对分配一个相似度分值来衡量节点间的相似性[19-20].基于相似性的方法大致分为基于节点相似性和基于网络结构相似性两类方法. 由于节点的相关信息获取相对困难,使得基于节点相似性的方法具有较大的局限性. 而网络的结构特征通常更易获得,因此,基于网络结构特征的链路预测方法得到了更为广泛的应用.
基于网络结构特征的链路预测方法又可被进一步划分为基于局部相似度指标和基于全局相似度指标的链路预测算法[21-22].基于网络局部结构相似度的算法具有复杂度低和性能好等优点,目前研究人员针对网络局部结构相似度的评估已经开展了大量的工作,如,Common Neighbors(CN) index[23]、Preferential Attachment index[4]、Jaccard's Coefficient[6]等. 然而,基于局部相似度指标的算法仅使用网络的局部信息,不能准确反映网络的整体结构特征,大大影响该类算法的预测准确度. 基于全局相似度的链路预测方法通过考虑网络路径信息,将分析使用的网络结构信息从较小的局部范围扩展至较大的全局范围,从而弥补上述方法的不足. 典型的基于全局相似度的链路预测算法包括Local Path index[24]、Katz index[25]等. 此外,研究人员也提出许多基于节点随机游走的算法,如Average Commute Time Index[26]、Random Walk Index Based on Cosine Similarity[27]、SimRank Index[28]等. 在上述基于网络结构的链路预测算法中,研究人员通常选取单方面的网络特征来评估节点间的相似度,并假设它们在不同的链路预测任务中都占有主导作用. 然而,大量研究表明不同网络拥有各自独特的结构特征,同一个网络的不同部分的结构特征也有所差异[29],这使得这些链路预测算法应用到不同网络上时性能差异较大,算法的适应性不佳.
本文提出一种适应不同带权疾病网络的潜在共病关系预测方法,通过综合多种网络结构特征提高相似度计算的准确性,从而实现更有效的共病关系预测,为临床辅助决策提供有力的依据.
2 带权疾病网络的构建
本文将带权疾病网络表示为:G = (V,E,W),其中,V表示网络节点的集合,E表示网络中边的集合,W为网络边上权重的集合. 在该网络中,每一个网络节点代表一种疾病,边代表两种疾病之间存在关联. 在疾病网络构建过程中,鉴于医疗数据集中患者诊断编码通常缺失率较低,因此从数据的易获取性和数据质量的角度考虑,主要利用患者就诊记录中的诊断编码,使每一种ICD编码对应疾病网络中的一个节点. 同时,根据两种疾病在同一患者的就诊记录中的共现关系来确定疾病之间的关联,疾病对的共现频次则作为其相應边上权重的衡量指标.
2.1 数据准备及网络构建
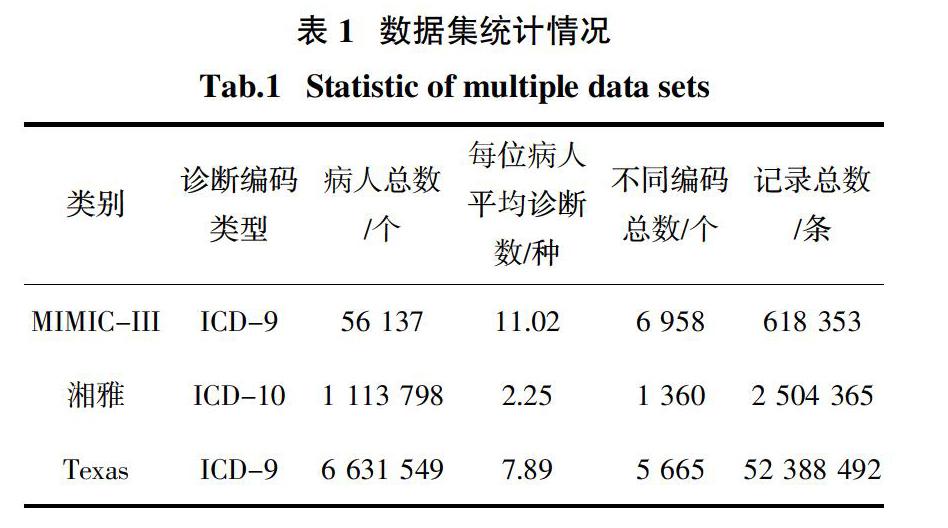
本文使用3个真实的医疗数据集,MIMIC-III(Medical Information Mart for Intensive Care III)[30]临床数据集(简称MIMIC-III)、中南大学湘雅医疗数据集[31](简称湘雅)和德克萨斯州健康数据集(Texas Health Data[32]简称Texas)来分别构建对应的疾病网络. 表1为这些数据集的统计信息.
为了确保构建的疾病网络的有效性和代表性,对原始医疗数据集进行了一定的预处理,剔除了部分无效的患者记录,具体包括:1) 清理重复的医疗记录;2)删除只包含一种疾病的患者就诊记录;3)由于疾病网络的构建工作不考虑稀有疾病,因此剔除了疾病对共现频次小于100的数据.
在完成数据清理步骤后,使用Cytoscape 3.5.0[33]来构建和可视化带权疾病网络,结果分别见图1、图2和图3. 图中节点上的标签表示节点所代表疾病的ICD编码,节点的大小与对应的患者数成正比,节点间边的宽度则与对应的权重成正比.
2.2 共病网络基础特性对比
根据构建的上述3个疾病网络,首先从网络的节点数、边数、平均度数、聚类系数等方面对各网络的基本结构特性进行了对比分析,详见表2. 由表2可知,湘雅疾病网络的节点数量和边数量最少,平均路径长度最大;MIMIC-III疾病网络的节点数量与湘雅疾病网络的相当,网络密度最大而平均路径长度最短;而德克萨斯州疾病网络,节点和边的绝对数量均远超前两者. 这说明了不同疾病网络在网络结构特征方面的明显差异.
3 疾病网络上的潜在共病关系预测
依据所构建的疾病网络,将潜在共病关系预测转化为网络上的链路预测问题. 用Eexist = {Ei}表示网络中已存在边的集合,Emiss = {Ej}表示网络中缺失边的集合,E表示网络中所有可能的边集合. 显然,它们满足下面的关系,E = Eexist∪Emiss,且Eexist∩Emiss = ?. 本文的目标是计算在给定加权疾病网络中尚未连接节点间出现新连接关系的可能性. 即,根据节点间已知的连接建立映射关系 f:{Ej}→{0,1} ,从而确定是否存在 {Ej} 中的潜在链路关系. 为此,本文在结合多种基于局部和全局网络结构特征的网络相似度指标的基础上,提出一种新的链路预测方法,实现潜在共病关系的准确预测.
3.1 相似度指标
本文从现有的基于网络拓扑结构特征的链路预测方法中选取了几种常用的相似度指标,将它们结合起来,从多个角度捕捉网络的拓扑结构特征,以提升链路预测的准确性. 为保证表述的一致性,将下文中涉及的符号统一定义如下. w(vi,vj)表示网络上节点vi和vj间连接的权重,Γ(vi)表示节点vi的邻居节点集合,a∈Γ(vi)表示a为节点vi的邻居节点.
Γ(vi)∩Γ(vj)表示节点vi与vj的共同邻居节点的集合,则z∈Γ(vi)∩Γ(vj)即为节点vi与vj的共同
邻居.
本文所选取的相似度指标主要包括:
Common Neighbors(CN)[23]. 该指标假设若两个节点共享的邻居节点越多,那么它们之间出现连接的可能性越大. 计算公式如下:
CN(vi,vj) = [][z∈Γ(vi)∩Γ(vj)]w(vi,z) + w(vj,z) (1)
Jaccard's Coefficient(JC)[6]. 该指标认为两个节点的共同邻居节点的权重之和占总邻居节点数的权
重之和比例越大,节点间越易出现新的连接. 即:
JC(vi,vj)=[][z∈Γ(vi)∩Γ(vj)]
(2)
Adamic-Adar Coefficient(AA)[5]. 該指标认为节点间的共同邻居节点越多,且这些共同邻居节点的度数较小,则出现连接的可能性越大. 即:
AA(vi,vj)=[][z∈Γ(vi)∩Γ(vj)] (3)
Preferential Attachment (PA)[4]. 该指标假设两个节点的邻居节点越多,且与邻居节点的权重之和较大,则节点间越易出现新的连接. 即:
PA(vi,vj)=[a∈Γ(vi)][]w(a,vi)× [b∈Γ(vj)][] w(b,vj) (4)
Resource Allocation Index(RA)[34]. 该指标与AA指标的区别在于对共同邻居节点与其相邻节点的权重之和的定义方式不同. 计算公式如下:
RA(vi,vj)=[][z∈Γ(vi)∩Γ(vj)] (5)
除上述几种局部相似度指标外,本文还选取了带重启动的随机游走算法(Random walk with restart algorithm,RWR)[22],来获得更大范围上的全局相似性. 其计算公式如下:
p(t+1)
= (1 - α)Sp(t)
+ αq (6)
式中:p(t)
表示第t步游走后图中的概率分布;列向量q为重启动向量,表示初始状态;S为转移概率矩阵;α为直接回到出发顶点的概率.
3.2 基于多种相似度指标的潜在共病关系预测
本文提出一种基于多种相似度指标的潜在共病关系预测方法(Latent Comorbidity Prediction based on Multiple Similarity Indexes,LCPMSI),该方法将基于不同网络结构特征的相似度指标进行整合构建特征矢量,将其输入到基于有监督学习的分类模型中,对疾病网络中尚未连边的节点对产生连接的可能性进行估计,从而实现潜在共病关系的有效预测. 在带权疾病网络中,潜在共病关系预测本质上可以看成一个二分类问题,即推断疾病节点对间是否存在
边连接. 对于给定网络G中的任意节点对(vi,vj),
E(vi,vj)表示节点对间可能的链路,l(vi,vj)是一个逻辑状态变量,用于指示网络中该节点对间是否存在连接关系. 那么,如果网络G中节点对(vi,vj)之间存在已知链路E(vi,vj),则E(vi,vj)将被标记为正样本并被置于集合Eexist中,同时l(vi,vj)将被设置为1. 否则,将E(vi,vj)设置为负样本置于集合Emiss中,并将l(vi,vj)设置为0. 然后将所有样本随机分成两部分,90%的样本作为训练集,并保证其中包含网络中90%的已存在链路,剩余的10%的样本则作为测试集.
在训练集上,任意节点对(vi,vj),根据上述6种相似度指标分别计算它们的相似度分值,并将计算得到的6种相似度分值结合起来构建相应的特征向量S(vi,vj),即:
S(vi,vj) = {S
M1(vi,vj),S
M2(vi,vj),…,S
Mn(vi,vj)}
(7)
式中:S
Mn(vi,vj)表示节点对基于指标Mn计算得到的相似度分值,Mn∈{CN,AA,JC,PA,RA,RWR}.
然后,将各节点对的特征向量输入分类器进行训练,进而实现目标节点对间链路存在性的预测.
4 实验结果及分析
4.1 实验设置及评价指标
为了验证LCPMSI算法的有效性,将其分别与采用上述6种单一相似度指标的方法以及2种简化的局部和全局相似度相结合的方法(CN+RWR和CN+PA+RWR)进行了性能比较. 实验中通过Weka软件包实现了Random Forest(RF),Random Tree(RT),Multi-Layer Perceptron(MLP),J48以及Naive Bayes 5种常见的分类器,各分类器的参数均采用Weka中的默认设置.
本文使用的评价指标包括:AUC(Area Under ROC Curve)、精准率(Precision)以及召回率(Recall). 此外,为了保证实验结果的可靠性,对训练数据进行随机打乱处理,并采用十折交叉验证对样本集进行划分. 每种预测算法均独立地执行10次,然后将其平均值作为最终的预测结果.
4.2 实验结果
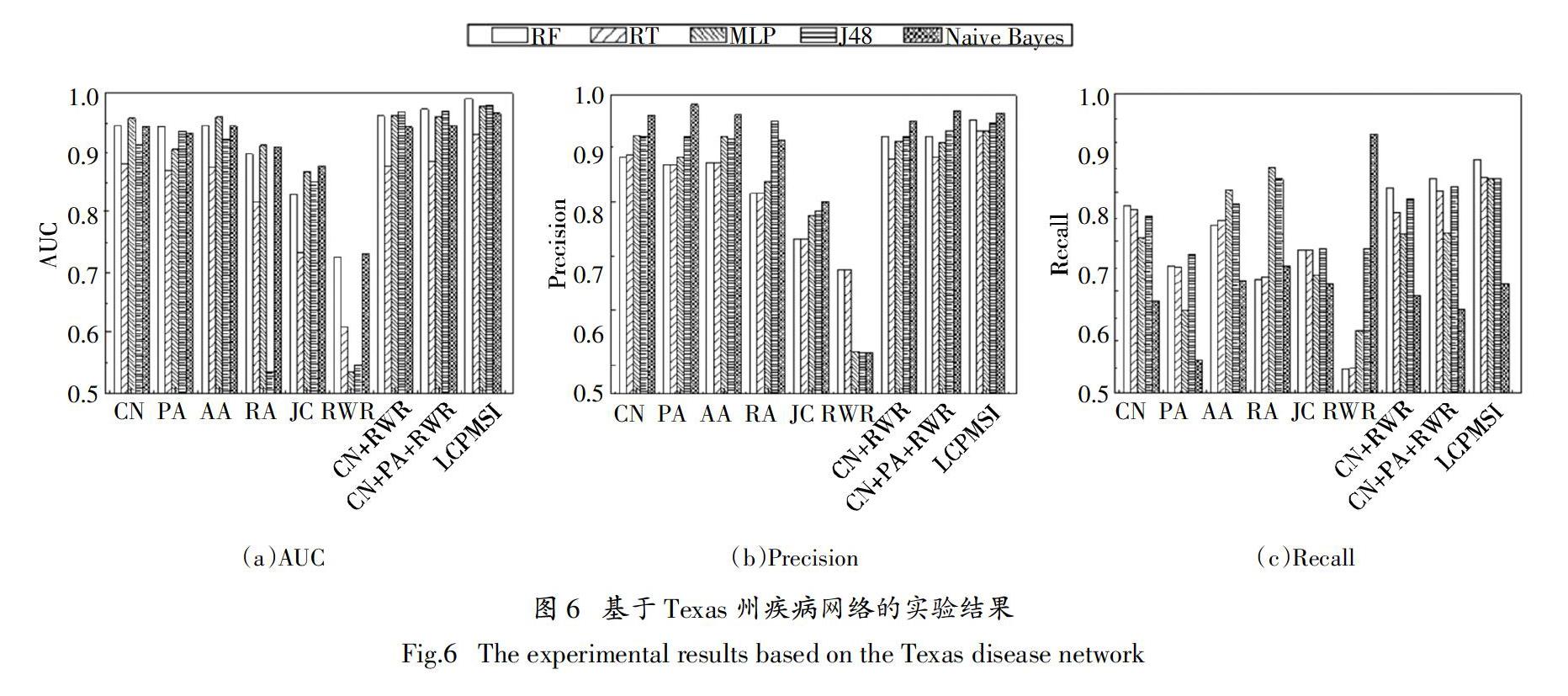
为了验证LCPMSI在共病预测中的有效性,利用已构建的MIMIC-III、湘雅和Texas疾病网络对各算法进行性能评估,结果分别如图4~图6所示.
从实验结果可看出,采用单一相似度指标的预测方法在不同疾病网络上的性能存在明显差异. 以使用RF分类器时的结果为例,按照AUC值对基于单一相似度指标的6种方法的性能进行排序,结果如下,MIMIC-III:PA>CN>AA>RA>RWR>JC;湘雅:CN>AA>JC>PA>RA>RWR;Texas:AA>CN>PA>RA>JC>RWR. 此外,当采用不同的分类器时,各相似度指标在不同疾病网络上的链路预测效果也各不相同. 以CN为例,其在MIMIC-III疾病网络中的表现最佳,AUC值达到了最高的0.963(采用Naive Bayes时),而在德州和湘雅疾病网络上CN获得的最优结果分别为0.958(采用MLP时)和0.874(采用RF时). 上述结果表明,基于单一相似度指標的链路预测方法在不同的疾病网络中的适应性较差,性能不稳定. 这正是由于上述6种单一的相似度指标仅片面地考虑了网络结构特征,缺乏对网络结构相似性的全面评估,因此在应用到不同结构特征的疾病网络上时,无法保证相似度计算的适用性,大大影响了预测算法的有效性和稳定性.
通过将基于局部和全局相似性的多个指标进行结合,预测算法的性能在不同的疾病网络上均获得了明显的提升. 如图4所示,在MIMIC-III疾病网络上,当选取CN+RWR作为特征时,AUC值达到最高值0.975(采用Naive Bayes时),比单一采用CN时的AUC提高了0.012,相较于RWR的提升幅度更是达到了0.084. 随着特征集合所包含的相似度指标的增多,预测模型在AUC值、精准率和召回率等方面的性能都逐渐有所提高. 当结合6种单一的相似度指标时,LCPMSI算法在不同的疾病网络上均获得了最佳的预测性能. 同时从不同分类器上的结果来看,LCPMSI在采用RF分类器时的性能表现最优. 如图5中湘雅疾病网络的结果所示,LCPMSI的AUC值、精准率和召回率相比CN分别相应地提高了0.072、0.105和0.131. 实验结果说明,本文提出的方法相比现有的基于单一相似度指标的链路预测算法能更好地适应不同疾病网络上的预测任务,并在预测的准确性和性能的稳定性上具有明显的优势. 这主要得益于LCPMSI对多方面网络结构特征的综合考虑,实现了对相似度的更准确把握. 同时,通过与有监督分类模型的结合,大大提高了预测算法的有效性和泛化能力.
5 结 论
共病关系预测是医学研究的热点问题. 通过构建疾病网络,可以将其转化为网络上的链路预测问题进行解决. 但现有的许多链路预测方法仅考虑了单一网络结构特征,在不同疾病网络上的性能较不稳定. 针对这一问题,本文提出了一种基于监督学习的潜在共病关系预测方法. 方法中使用基于监督学习的分类方法挖掘不同疾病网络的内部结构特征,学习每种网络结构特征的重要性,以提高算法的预测精度以及在不同疾病网络上的泛化能力. 实验结果表明,该方法有效地提高了潜在共病关系的预测性能. 同时,通过在基于不同医疗数据集构建的疾病网络上的测试结果比较,进一步证明了该方法的稳定性和适用性.
在未来的工作中,我们将进一步结合网络结构特征和语义特征来提高共病关系的预测精度. 同时,还将考虑疾病发展的时序特性,改进本文的算法以解决有向疾病网络中的链路预测问题.
参考文献
[1] ZONG N,KIM H,NGO V,et al. Deep mining heterogeneous networks of biomedical linked data to predict novel drug-target associations[J]. Bioinformatics,2017,33(15):2337—2344.
[2] CHEN H Y,LI J. A flexible and robust multi-source learning algorithm for drug repositioning[C]// Proceedings of the 8th ACM International Conference on Bioinformatics,Computational Biology,and Health Informatics. New York: ACM,2017:510—515.
[3] 侯泳旭,段磊,李岭,等. 基于疾病信息网络的表型基因搜索[J]. 软件学报,2018,29(3):721—733.
HOU Y X,DUAN L,LI L,et al. Search of gene with similar phenotype based on disease information network[J]. Journal of Software,2018,29(3):721—733.(In Chinese)
[4] BARABASI A L,BONABEAU E. Scale free networks[J]. Scientific American,2003,288(5):50—59.
[5] ADAMIC L A,ADAR E. Friends and neighbors on the web[J]. Social Networks,2003,25(3):211—230.
[6] TAN P N,STEINBACH M,KUMAR V. Introduction to data mining[M]. New Jersey: Addison-Wesley,2005:38—50.
[7] MART?N-JIM?NEZ C A,SALAZAR-BARRETO D,BARRETO G E,et al. Genome-scale reconstruction of the human astrocyte metabolic network[J]. Frontiers in Aging Neuroscience,2017,23(9):1—17.
[8] DEVREOTES P N,BHATTACHARYA S,EDWARDS M,et al. Excitable signal transduction networks in directed cell migration[J]. Annual Review of Cell and Developmental Biology,2017,33(1):103—125.
[9] JIANG Y,MA S,SHIA B C,et al. An epidemiological human disease network derived from disease co-occurrence in Taiwan[J]. Scientific Reports,2018,8(1):1—12.
[10] SUN P G. The human drug-disease-gene network[J]. Information Sciences,2015,306:70—80.
[11] ZHOU X Z,MENCHE J,BARAB?SI A L,et al. Human symptoms-disease network[J]. Nature Communications,2014,5:4212.
[12] 方陽,赵翔,谭真,等. 一种改进的基于翻译的知识图谱表示方法[J]. 计算机研究与发展,2018,55(1):139—150.
FANG Y,ZHAO X,TAN Z,et al. A revised translation-based method for knowledge graph representation[J]. Journal of Computer Research and Development,2018,55(1):139—150.(In Chinese)
[13] 陈德华,殷苏娜,乐嘉锦,等. 一种面向临床领域时序知识图谱的链接预测模型[J]. 计算机研究与发展,2017,54(12):2687—2697.
CHEN D H,YIN S N,LE J J,et al. A link prediction model for clinical temporal knowledge graph[J]. Journal of Computer Research and Development, 2017, 54(12): 2687—2697. (In Chinese)
[14] YANG J,YANG T H,WU D Z,et al. The integration of weighted human gene association networks based on link prediction[J]. BMC Systems Biology,2017,11(1):1—17.
[15] SULAIMANY S,KHANSARI M,ZARRINEH P,et al. Predicting brain network changes in Alzheimer's disease with link prediction algorithms[J]. Molecular BioSystems,2017,13(4):725—735.
[16] G?L S,KAYA M,KAYA B. Predicting links in weighted disease networks[C]//International Conference on Computer and Information Sciences (ICCOINS). Kuala Lumpur: IEEE,2016:77—81.
[17] KAYA B,POYRAZ M. Finding relations between diseases by age-series based supervised link prediction[C]//International Conference on Advances in Social Networks Analysis and Mining (ASONAM). New Jersey:IEEE,2015:1097—1103.
[18] SHIN H J. Method for providing disease co-occurrence probability from disease network[P]. US:20160350502,2016—12-01.
[19] 楊妮亚,彭涛,刘露. 基于聚类和决策树的链路预测方法[J]. 计算机研究与发展,2017,54(8):1795—1803.
YANG N Y,PENG T,LIU L. Link prediction method based on clustering and decision tree[J]. Journal of Computer Research and Development,2017,54(8):1795—1803. (In Chinese)
[20] LIBEN-NOWELL D,KLEINBERG J. The link-prediction problem for social networks[J]. Journal of the American Society for Information Science and Technology,2007,58(7):1019—1031.
[21] LEICHT E A,HOLME P,NEWMAN M E J. Vertex similarity in networks[J]. Physical Review E,2006,73(2):026120.
[22] TONG H,FALOUTSOS C,PAN J Y. Fast random walk with restart and its applications[C]//IEEE International Conference on Data Mining (ICDM). New Jersey:IEEE,2006:613—622.
[23] NEWMAN M E J. Clustering and preferential attachment in growing networks[J]. Physical Review E,2001,64(2):025102.
[24] WANG Y,ZHOU T,SHI J,et al. Empirical analysis of dependence between stations in Chinese railway network[J]. Physica A: Statistical Mechanics and Its Applications,2009,388(14):2949—2955.
[25] KATZ L. A new status index derived from sociometric analysis[J]. Psychometrika,1953,18(1):39—43.
[26] KLEIN D J,[RANDICM][′]. Resistance distance[J]. Journal of Mathematical Chemistry,1993,12(1):81—95.
[27] FOUSS F,PIROTTE A,RENDERS J M,et al. Random-walk computation of similarities between nodes of a graph with application to collaborative recommendation[J]. IEEE Transactions on Knowledge and Data Engineering,2007,19(3):355—369.
[28] JEH G,WIDOM J. Simrank: a measure of structural-context similarity[C]//The Eighth ACM SIGKDD International Conference on Knowledge Discovery and Data Mining. New York: ACM,2002:538—543.
[29] MA C,BAO Z,ZHANG H. Improving link prediction in complex networks by adaptively exploiting multiple structural features of networks[J]. Physics Letters A,2017,381(39):3369—3376.
[30] JOHNSON A E W,POLLARD T J,SHEN L,et al. MIMIC-III,a freely accessible critical care database[J]. Scientific Data,2016,3:160035.
[31] LI B,LI J B,LAN X Y,et al. Experiences of building a medical data acquisition system based on two-level modeling[J]. International Journal of Medical Informatics,2018,112:114—122.
[32] The Texas Department of State Health Services. TEXAS health and human services[EB/OL]. (2018-02-01)[2018-05-15].https://www.dshs.texas.gov/THCIC/Hospitals/Download.shtm.
[33] SHANNON P,MARKIEL A,OWEN O,et al. Cytoscape: a software environment for integrated models of biomolecular interaction networks[J]. Genome Research,2003,13:2498—2504.
[34] ADAMIC L A,ADAR E. Friends and neighbors on the web[J]. Social Networks,2003,25(3):211—230.

