N-糖酰胺酶PNGase H+ 最小糖结构作用底物的研究
胡孝春,王 婷,蔡志鹏,刘 丽,JOSEF Voglmeir
(南京农业大学食品科技学院,江苏南京 210000)
N-糖酰胺酶(Peptide-N-(N-acetyl-β-D-glucosaminyl)asparagines amidase,PNGase)是一种去糖基化酶[1]。它能催化N-糖链和天冬酰胺(Asn)残基之间β-天冬氨酰基葡糖胺键的断裂,从而将完整的N-糖链由蛋白质或多肽上释放下来[2-4]。N-糖酰胺酶作为糖组学研究的重要工具酶,在糖链的结构分析和功能研究中都发挥着重要的作用[5-7]。然而,目前常用的两种商业化N-糖酰胺酶PNGase A和PNGase F都各有不足:PNGase A可以释放所有类型的糖链,但无法作用于糖蛋白,只能作用于糖肽;而PNGase F虽然对糖蛋白和糖肽都具有活性,但无法释放植物来源的具有核心α-1,3岩藻糖修饰的N-糖链。本实验室在前期研究中,从细菌Terriglobusroseus中发现了一种新型N-糖酰胺酶PNGase H+[8]。该酶能够由原核系统重组表达,并克服了现有商业酶PNGase F和PNGase A两者的缺陷。它不仅能够作用于糖蛋白也能用于糖肽,且能够释放所有类型的N-糖链,包括具有植物特征性α-1,3岩藻糖核心结构的N-糖链,因此PNGase H+在植物来源的糖蛋白N-糖链结构分析中具有重要的应用价值[9]。
最小糖结构作用单元是N-糖酰胺酶的一个重要酶学特性,明确PNGase作用于N-糖链的具体识别位点以及催化所必须的糖链结构和糖分子数量,是深入研究N-糖酰胺酶酶催化机理的关键环节[10-12]。Altmann F发现,PNGase F能够释放的最小糖结构作用单元为与Asn相连的二乙酰壳二糖结构(N,N′-diacetylchitobiose),而PNGase A则仅需一个与Asn连接的N-乙酰葡糖胺结构,就能够识别糖链并进行水解[13]。在之前的研究中,本实验室虽然对N-糖酰胺酶PNGase H+的最适反应条件和底物特异性进行了研究,但由于缺乏合适的酶解底物,因此未对其最小糖结构作用单元进行探索。
合成具有不同糖链长度和结构的糖肽底物是研究PNGase H+最小糖结构作用单元的关键。目前常用的糖肽底物的合成方法主要有化学合成和酶法合成两种。化学合成容易破坏糖链结构,且通常伴有副产物生成[14-15],酶法具有高效性、特异的区域选择性和立体选择性的特点。Wong等[18]曾利用半乳糖基转移酶催化反应,大批量合成了半乳糖胺。因此酶法可以获得产率较高的目的产物[16-17]。本文中糖肽底物的合成拟采用糖基水解酶和糖基转移酶的方法,糖基水解酶能够特异性水解糖底物,从而获得目标产物,而糖基转移酶则能将糖基供体上的糖分子转移到糖基受体上,从而获得目标产物。
本研究拟以丹磺酰氯标记的糖肽标准品为基础,利用酶法制备不同糖单元大小的糖肽底物,并以此底物对PNGase H+的活性进行检测,得出PNGase H+的最小糖结构作用单元,从而为PNGase H+的酶催化机制研究提供依据。
1 材料与方法
1.1 材料与仪器
β-甘露糖苷酶重组质粒、E.coliBL21(DE3)感受态细胞、β-半乳糖基转移酶重组质粒、UDP-葡萄糖-4-差向异构酶重组质粒、β-N-乙酰氨基己糖苷酶、α-甘露糖苷酶 Sigma生物公司;丹磺酰氯标记的七糖单元糖肽GNGN(Dabsyl-Gly-Glu-Asn-(GlcNAc4Man3)-Arg) 由奥地利维也纳自然资源与生命科学应用大学的Thomas Dalik和Friedrich Altmann提供;异丙基硫代半乳糖苷(IPTG)、苯甲基磺酰氟、卡那霉素、Triton X-100、Ni-NTA亲和层析柱、LB液体培养基(5 g NaCl、10 g酵母提取物、10 g胰蛋白胨) 南京金丝瑞生物公司;乙腈 色谱纯,默克公司。
1416R型高速冷冻离心机 珠海黑马医学仪器有限公司;GRP-9160型恒温培养箱 上海森信实验仪器有限公司;725N型微量分光光度计One Drop OD-2000 南京五义科技有限公司;Shimadzu LC-30AD型超高效液相色谱配有SPD-20A型紫外检测器以及RF-20Axs型荧光检测器、SIL-30AC型自动进样器、CO-2000型柱温箱(恒信仪器)、LC solution工作站 岛津技迩(上海)商贸有限公司。
1.2 实验方法
1.2.1 糖肽合成相关酶的制备
1.2.1.1β-甘露糖苷酶的表达与纯化 将β-甘露糖苷酶重组质粒转化入E.coliBL21(DE3)感受态细胞[19]。挑取单菌落于5 mL含卡那霉素(50 μg/mL)的LB培养基中过夜培养,然后接种至含有400 mL LB培养基的摇瓶中扩大培养,直至菌液浓度在OD600处吸光度达到0.5左右,加入异丙基硫代半乳糖苷(IPTG)至终浓度1 mmol/L,18 ℃诱导表达22 h。4 ℃、4000 r/min离心收集菌体,将菌体沉淀重悬于10 mL裂解液(100 mmol/L NaCl,50 mmol/L Tris-HCl pH8.0,1% Triton X-100,1 mmol/L苯甲基磺酰氟)超声破碎细胞,12000 r/min、4 ℃离心20 min,收集上清,并置于80 ℃水浴锅中加热30 min,4 ℃、12000 r/min离心收集上清,并用分子截留量为30 kDa的超滤管对其进行浓缩,4 ℃保存。
1.2.1.2β-半乳糖基转移酶的表达与纯化 将β-半乳糖基转移酶重组质粒转化入E.coliBL21(DE3)感受态细胞,表达过程同1.2.1.1。使用Ni-NTA亲和层析进行纯化[20],镍柱使用5倍柱体积的平衡缓冲液(50 mmol/L Tris/HCl pH8.0,50 mmol/L NaCl)进行平衡后上样,并使用10倍柱体积的结合缓冲液(50 mmol/L Tris/HCl pH8.0,50 mmol/L NaCl,20 mmol/L咪唑)冲洗去除非特异结合的蛋白,再用洗脱缓冲液(50 mmol/L Tris/HCl pH8.0,50 mmol/L NaCl,500 mmol/L咪唑)洗脱重组蛋白,280 nm波长检测柱下液,收集蛋白峰即纯化后的重组酶,4 ℃保存[21]。
1.2.1.3 UDP-葡萄糖-4-差向异构酶的表达与纯化 将UDP-葡萄糖-4-差向异构酶重组质粒转化入E.coliBL21(DE3)感受态细胞,表达和纯化过程同1.2.1.2,4 ℃保存。
1.2.2 丹磺酰氯标记糖肽底物的酶法合成
1.2.2.1 五糖单元糖肽的制备 取GNGN(10 pmol/μL)1 mL作为初始底物,加入1 μLβ-N-乙酰氨基己糖苷酶和250 μL pH为7.0的Tris/HCl缓冲溶液,振荡混匀,置于37 ℃培养箱中反应16 h后,金属浴中95 ℃灭活5 min结束反应[22],得五糖单元糖肽。
1.2.2.2 三糖单元糖肽的制备 以1.2.2.1中反应获得的产物作为底物,取1 mL该底物置于EP管中,加入1 μLα-甘露糖苷酶和250 μL pH为5.5的柠檬酸缓冲溶液,振荡混匀,置于37 ℃培养箱中反应16 h后,金属浴中95 ℃灭活5 min结束反应,得三糖单元糖肽。
1.2.2.3 二糖单元糖肽的制备 以1.2.2.2中反应获得的产物作为底物,取1 mL该底物置于EP管中,加入10 μLβ-甘露糖苷酶和200 μL 400 mmol/L pH为7.4的2-氨基-2-甲基-1,3-丙二醇(AMPD)缓冲溶液,振荡混匀,于75 ℃金属浴中反应10 min后,95 ℃灭活5 min结束反应,得二糖单元糖肽。
1.2.2.4 单糖单元糖肽的制备 以1.2.2.3中反应获得的产物作为底物,取400 μL该底物置于EP管中,加入1 μLβ-N-乙酰氨基己糖苷酶和100 μL pH为7.0的Tris/HCl缓冲溶液,振荡混匀,置于37 ℃培养箱中反应16 h后,金属浴中95 ℃灭活5 min结束反应,得单糖单元糖肽。
1.2.2.5 新三糖单元糖肽的制备 以1.2.2.3中反应获得的产物作为底物,取5 μL该底物置于EP管中,加入5 μL MgCl2(10 mmol/L)、1 μL MnCl2(100 mmol/L)、8 μLβ-半乳糖苷转移酶、8 μL UDP-葡萄糖-4-差向异构酶及5 μL pH为7.4的AMPD(400 mmol/L)缓冲溶液,振荡混匀,置于37 ℃培养箱中反应16 h后,金属浴中95 ℃灭活5 min结束反应,得糖结构不同的新三糖单元糖肽。
1.2.3 丹磺酰氯标记糖肽底物的检测 利用超高液相色谱对1.2.2所制备糖肽进行检测。色谱柱:Phenomenex Kinetex® 1.7 μm C18 100Å 150×2.10 mm;流动相A:超纯水(含0.1%甲酸(v/v)),流动相B:乙腈(含0.1%甲酸(v/v));流速:0.25 mL/min;检测波长:UV/Vis 436 nm;进样量:30 μL;柱温常温。洗脱程序:0~1 min B 5%,1~30 min B 5%~50%,30~31 min B 50%~95%,31~32 min B 95%,32~33 min B 95%~5%,33~40 min B 5%,50 min结束程序。
糖肽底物定量分析:利用Labsolutions 软件进行分析,以糖肽GNGN(10 pmol/μL)为外标,对制备底物的峰面积积分,计算底物浓度[13]。
1.2.4 N-糖酰胺酶PNGase H+对不同糖肽底物酶解活性的测定
1.2.4.1 N-糖酰胺酶PNGase H+的表达纯化 N-糖酰胺酶PNGase H+的表达及纯化步骤表达和纯化过程同1.2.1.2,4 ℃保存。
1.2.4.2 PNGase H+对不同糖肽底物酶解活性的检测 分别以五糖单元、三糖单元、二糖单元、单糖单元、三糖新单元糖肽作为反应底物,检测PNGase H+的酶解活性,反应体系:2 μL糖肽底物、2 μL PNGase H+、2 μL pH为2.6的柠檬酸缓冲溶液和4 μL超纯水;不加酶底物对照组:2 μL糖肽底物、2 μL pH为2.6的柠檬酸缓冲溶液和6 μL超纯水;产物对照组:2 μL Dabsyl-Gly-Glu-Asn-Arg、2 μL pH2.6的柠檬酸缓冲溶液和6 μL超纯水,振荡混匀,分别置于37 ℃恒温培养箱中反应16 h后,于金属浴中95 ℃灭活5 min结束反应,12000 r/min常温离心机中离心1 min,利用超高液相色谱进行检测,色谱条件、试验步骤同1.2.3。
1.3 数据处理
采用Microsoft Excel Office 2010数据整理,绘图软件采用Adobe illustrator进行色谱图绘制。
2 结果与分析
2.1 丹磺酰氯标记的糖肽底物的合成
核心五糖结构(由2个N-乙酰葡糖胺及3个甘露糖构成)是所有种类N-糖链的共有结构,因此合成具有该糖单元结构以及在此基础上产生的递减糖单元结构的特殊糖肽类底物,是N-糖酰胺酶识别糖单元结构研究的基础。本实验以丹磺酰氯标记的七糖单元糖肽Dabsyl-Gly-Glu-Asn-(GlcNAc4Man3)-Arg(GNGN)为起始底物,经β-N-乙酰氨基己糖苷酶、α-甘露糖苷酶、β-甘露糖苷酶依序处理生成相应的五糖、三糖和二糖单元糖肽,再进一步使用β-N-乙酰氨基己糖苷酶处理产生单糖单元糖肽,所得产物分别由高效液相色谱检测,结果如图1所示。
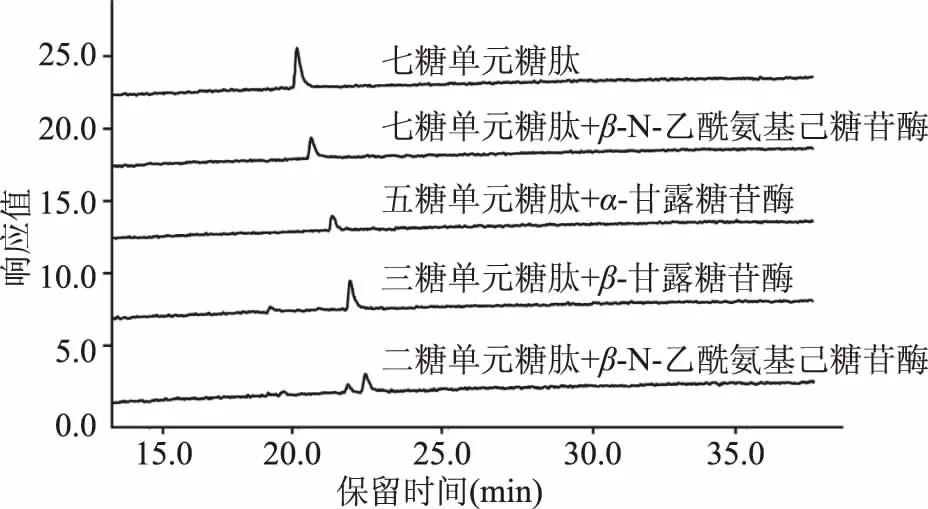
图1 五糖、三糖、二糖和单糖单元丹磺酰氯标记糖肽底物液相色谱检测图
七糖单元糖肽底物液相保留时间为21 min,经β-N-乙酰氨基己糖苷酶作用后,核心甘露糖结构上的末端β-N-乙酰氨基葡萄糖被完全水解,生成五糖单元糖肽Dabsyl-Gly-Glu-Asn-(GlcNAc2Man3)-Arg,其色谱峰出峰位置发生后移(保留时间21.5 min),所得产物纯度为100%,浓度为8.3 pmol/μL。该五糖单元糖肽在α-甘露糖苷酶的作用下,末端的α-1,3和α-1,6连接甘露糖被完全解离,生成三糖单元糖肽Dabsyl-Gly-Glu-Asn-(GlcNAc2Man)-Arg,其色谱峰位置后移至22 min,所得产物纯度为100%,浓度为7.0 pmol/μL。随后在β-甘露糖苷酶的作用下,与二乙酰壳二糖相连的β-甘露糖被解离,生成二糖单元糖肽Dabsyl-Gly-Glu-Asn-(GlcNAc2)-Arg,保留时间为22.5 min,所得产物纯度为100%,浓度为6.5 pmol/μL。最后再次经β-N-乙酰氨基己糖苷酶的作用,生成单糖单元糖肽Dabsyl-Gly-Glu-Asn-(GlcNAc)-Arg,部分色谱峰后移至23 min,所得产物纯度约为70%,浓度为4.5 pmol/μL。
此外,为了保证最小糖单元结构研究的准确性,本实验还以二糖单元糖肽Dabsyl-Gly-Glu-Asn-(GlcNAc2)-Arg为底物,通过β-半乳糖基转移酶的作用,生成了一种非自然存在的三糖单元糖肽Dabsyl-Gly-Glu-Asn-(GlcNAc2Gal)-Arg。根据液相色谱图2所示,二糖单元糖肽Dabsyl-Gly-Glu-Asn-(GlcNAc2)-Arg的色谱峰液相保留时间为22.5 min,经β-半乳糖基转移酶反应,生成新三糖单元糖肽Dabsyl-Gly-Glu-Asn-(GlcNAc2Gal)-Arg(保留时间22 min),该三糖单元糖肽的纯度为100%,浓度为6.5 pmol/μL。
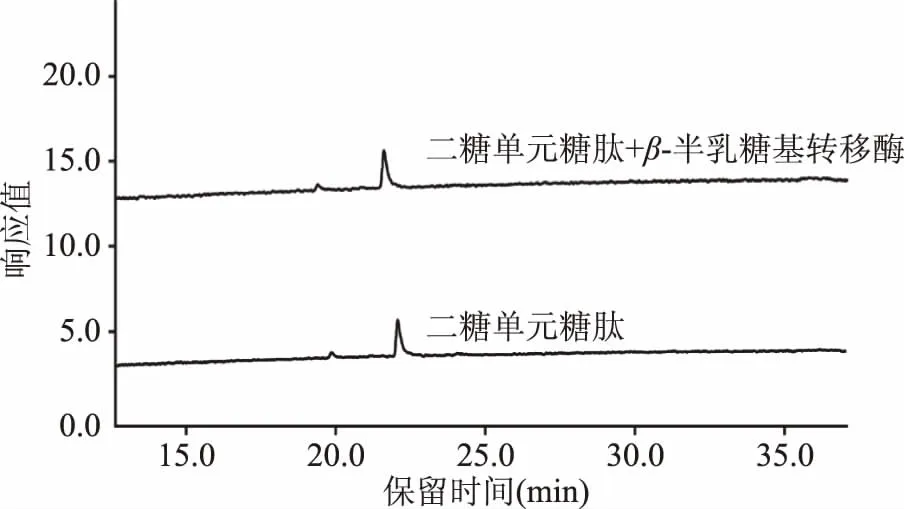
图2 新三糖单元丹磺酰氯标记糖肽底物色谱检测图
通过上述反应,制备了五种不同的糖单元糖肽,其名称、结构和浓度如表1所示。这五种糖单元糖肽都是以核心五糖结构为基础,在特异性糖苷酶的作用下,糖单元的数量或结构发生改变,从而合成了目标糖肽底物,为后续测定PNGase H+最小糖结构作用底物的探究提供了基础。

表1 丹磺酰氯标记糖肽底物结构及浓度
2.2 N-糖酰胺酶PNGase H+对不同丹磺酰氯标记糖单元糖肽底物的酶解作用
2.2.1 PNGase H+对五糖单元糖肽的酶解作用 以五糖单元糖肽Dabsyl-Gly-Glu-Asn-(GlcNAc2Man3)-Arg为底物,检测 PNGase H+对该底物的酶解效果。反应16 h后,产物通过液相色谱进行检测,并与丹磺酰氯标记的Dabsyl-Gly-Glu-Asp-Arg的液相图谱进行比较,确定PNGase H+对五糖单元糖肽的酶解效果,检测结果如图3所示。底物五糖单元糖肽的色谱峰(保留时间21.5 min)发生完全偏移,生成的产物色谱峰(保留时间24.5 min)与丹磺酰氯肽段Dabsyl-Gly-Glu-Asp-Arg完全一致,从而判定PNGase H+能够将五糖单元从糖肽上完全解离下来。已往对PNGase H+的底物特异性研究显示,PNGase H+能够作用于高甘露糖型、复杂型和杂合型所有种类的N-糖链[23],而核心五糖作为这些N-糖链的共有结构,PNGase H+也应对其具有酶解效果,本试验结果进一步验证了该结论。
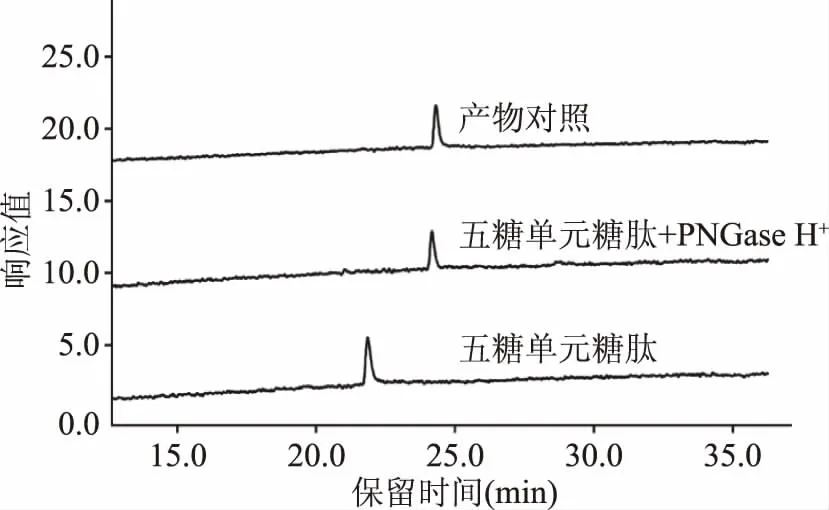
图3 PNGase H+对五糖单元糖肽酶解作用液相色谱图
2.2.2 PNGase H+对三糖单元糖肽的酶解作用 以三糖单元糖肽Dabsyl-Gly-Glu-Asn-(GlcNAc2Man)-Arg为底物对PNGase H+的酶解作用进行测定,结果如图4所示。反应16 h后,底物三糖单元糖肽的色谱峰(保留时间22 min)发生完全偏移,生成的产物色谱峰(保留时间24.5 min)与丹磺酰氯肽段Dabsyl-Gly-Glu-Asp-Arg完全一致,从而判定PNGase H+能够将三糖单元糖肽上的N-糖链完全解离。在反应底物的制备过程中,通过α-甘露糖苷酶的作用,N-糖链结构五糖核心末端两个α-连接的甘露糖被解离,只留下与核心N-乙酰壳二糖相连的β-甘露糖。这种三糖单元糖肽虽然不具备完整的五糖核心,但也是自然界存在的一种N-糖链结构,如曾有相关报道在大豆糖蛋白的N-糖链分析中鉴定出该结构[24]。试验结果表明,即使消去了五糖核心的末端两个α-连接的甘露糖,PNGase H+依旧能够很好地识别,并释放该糖链结构。
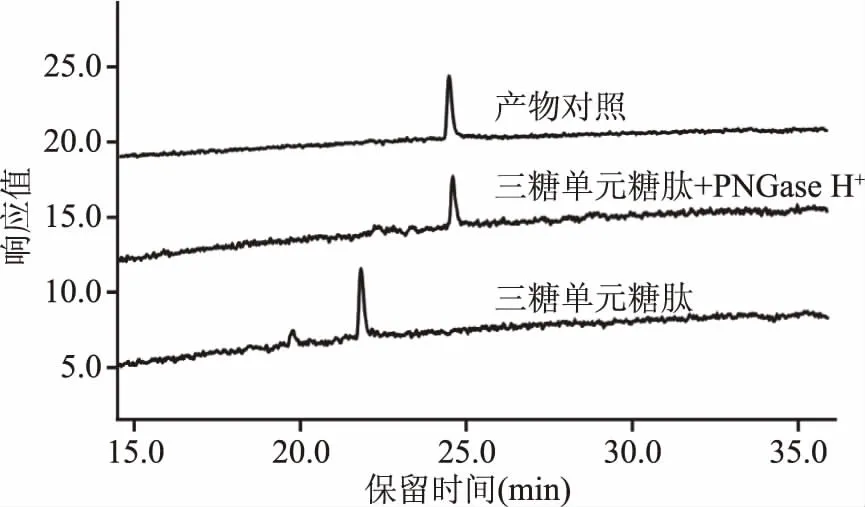
图4 PNGase H+对三糖单元糖肽酶解作用液相色谱图
2.2.3 PNGase H+对二糖单元糖肽的酶解作用 以二糖单元糖肽Dabsyl-Gly-Glu-Asn-(GlcNAc2)-Arg为底物对PNGase H+对酶解作用进行了测定,结果如图5所示。反应16 h后,底物二糖单元糖肽的色谱峰(保留时间22.5 min)发生完全偏移,生成的产物色谱峰(保留时间24.5 min)与丹磺酰氯肽段Dabsyl-Gly-Glu-Asp-Arg完全一致,从而判定PNGase H+能够将二糖单元糖肽上的N-糖链完全解离。该二糖单元底物在五糖核心结构基础上,通过α-甘露糖苷酶和β-甘露糖苷酶的共同作用,所有甘露糖基均被解离,只留下核心N-乙酰壳二糖。试验结果表明,即使只剩核心N-乙酰壳二糖结构,PNGase H+依旧能够很好地识别并释放该糖链结构,该结果与已往的报道一致。如Altman等发现,PNGase F和PNGase A均能水解连接有N-乙酰壳二糖的六肽底物[13]。晶体结构研究也证明,N-乙酰壳二糖能够结合在PNGase F的活性中心[25]。此外Fan等发现,PNGase F对含有苯丙氨酸(Phe)氨基酸残基的N-乙酰壳二糖糖肽活性极低[26]。这说明除了糖链结构与糖单元数量,肽段的氨基酸序列对于N-糖酰胺酶的酶解作用也有一定影响,而本实验使用的糖肽底物中不含有苯丙氨酸,不会对PNGase H+的活性检测造成影响。
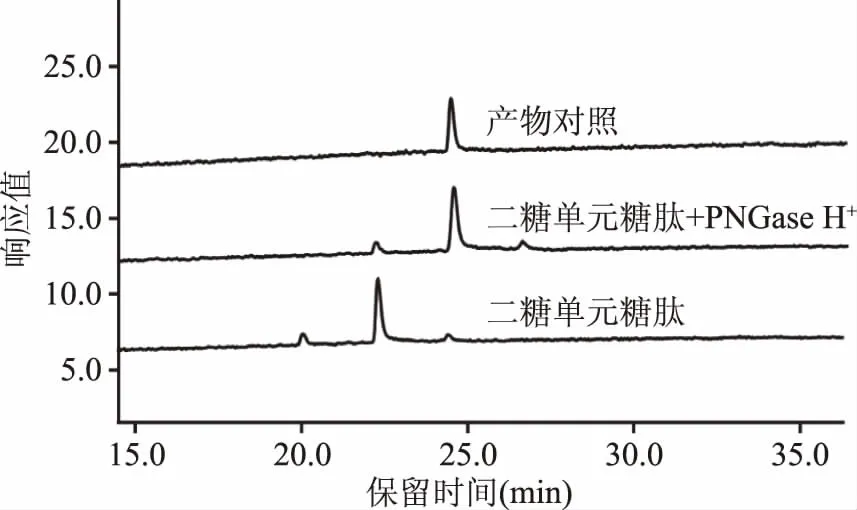
图5 PNGase H+对二糖单元糖肽酶解作用液相色谱图
2.2.4 PNGase H+对单糖单元糖肽的酶解作用 以单糖单元糖肽Dabsyl-Gly-Glu-Asn-(GlcNAc)-Arg为底物对PNGase H+的酶解作用进行了测定,结果如图6所示。前期制备的单糖单元糖肽纯度仅为70%,其中还含有30%的二糖单元糖肽,加入PNGase H+反应16 h后,可见保留时间为22.5 min的二糖单元糖肽的色谱峰完全消失,同时保留时间24.5 min检测到丹磺酰氯肽段Dabsyl-Gly-Glu-Asp-Arg,而单糖分子糖肽的底物色谱峰(保留时间为23 min)峰高和峰面积均无明显变化,表明PNGase H+对单糖分子糖肽活性极弱,无法将N-乙酰葡糖胺从单糖分子糖肽上解离下来。PNGase F的晶体结构研究中显示,酶活性中心谷氨酸残基118位(Glu118)能够与第二位N-乙酰葡萄糖胺6′位的氧形成氢键,从而提高对糖链的结合能力,由此可见第二位的N-乙酰葡萄糖胺对PNGase F的活性具有重要影响[25]。而本试验结果表明,PNGase H+与PNGase F相似,能够作用的最小糖链结构为N-乙酰壳二糖,而其酶活性中心是否存在氨基酸残基能够与第二位N-乙酰葡萄糖胺形成氢键,还需进一步的晶体结构研究验证。
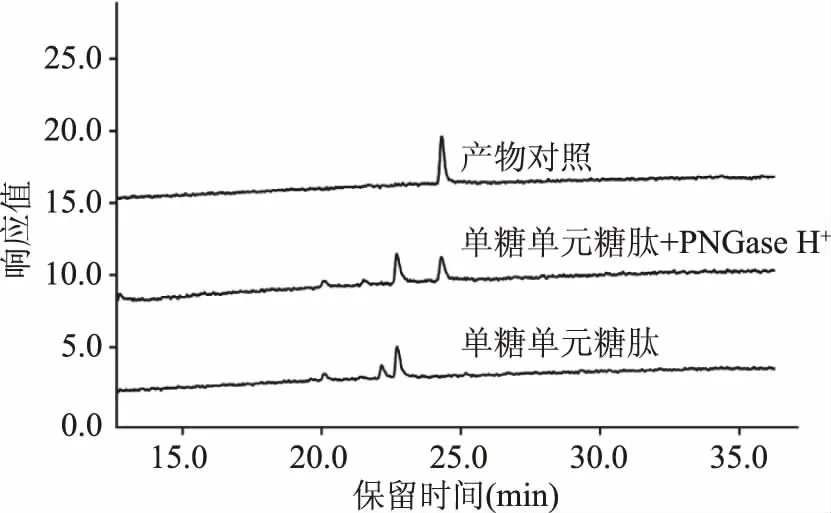
图6 PNGase H+对单糖单元糖肽酶解作用液相色谱图
2.2.5 PNGase H+对三糖新单元糖肽的酶解作用 为了进一步验证N-乙酰壳二糖为PNGase H+的最小糖结构作用单元,以人工合成的非天然三糖单元糖肽Dabsyl-Gly-Glu-Asn-(GlcNAc2Gal)-Arg为底物对PNGase H+的酶解活性进行了测定,结果如图7所示。反应16 h后,底物三糖单元糖肽的色谱峰(保留时间22 min)发生完全偏移,生成的产物色谱峰(保留时间24.5 min)与丹磺酰氯肽段Dabsyl-Gly-Glu-Asp-Arg完全一致,从而判定PNGase H+能够将该三糖单元糖肽上的N-糖链完全解离。自然存在的N-糖链构象中,与核心N-乙酰壳二糖相连的糖基一般是甘露糖而非半乳糖[27]。然而PNGase H+一样能够高效的水解释放该种糖链,说明第三位的半乳糖基并未影响糖链与PNGase H+活性中心的结合,进一步验证了PNGase H+的最小糖结构作用单元为N-乙酰壳二糖的推断。
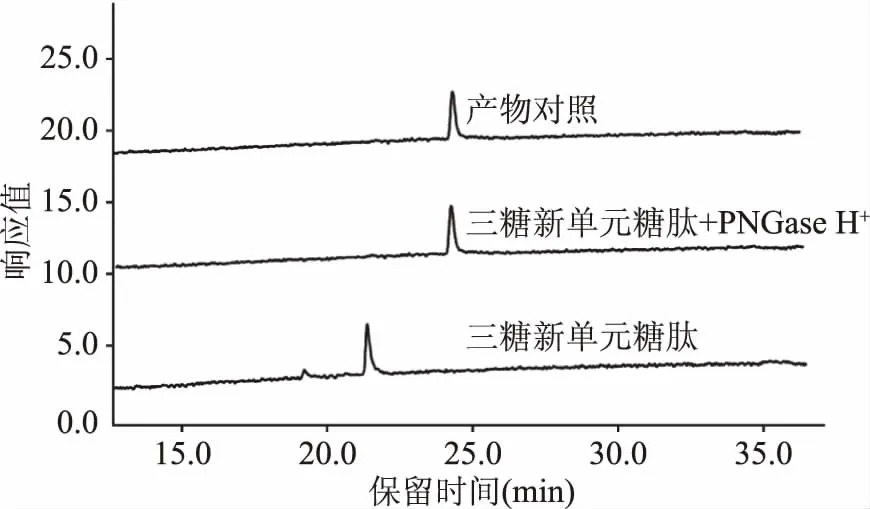
图7 PNGase H+对三糖新单元糖肽酶解作用液相色谱图
3 结论
本研究首先利用生物学的方法成功表达纯化出了β-半乳糖苷酶和β-半乳糖基转移酶,然后以磺酰氯标记的复杂型七糖单元糖肽标准品为初始底物,利用酶法合成获得了5种不同结构的糖肽底物,以合成的底物为基础,通过高效液相色谱来检测PNGase H+对底物的酶解效果,从而得出N-糖酰胺酶PNGase H+的最小糖结构作用底物,实验表明,利用酶法成功制备出了5种不同结构的糖肽底物Dabsyl-Gly-Glu-Asn-(GlcNAc2Man3)-Arg、Dabsyl-Gly-Glu-Asn-(GlcNAc2 Man)-Arg、Dabsyl-Gly-Glu-Asn-(GlcNAc2)-Arg、Dabsyl-Gly-Glu-Asn-(GlcNAc)-Arg和Dabsyl-Gly-Glu-Asn-(GlcNAc2Gal)-Arg,浓度分别为8.3、7.0、6.5、4.5、6.5 pmol/μL。PNGase H+不仅能够作用于糖单元数为二、三和五的三种不同的天然构象糖肽,且能够作用于人工合成的非天然构象的三糖单元糖肽Dabsyl-Gly-Glu-Asn-(GlcNAc2Gal)-Arg,但不能酶解只具有单个N-乙酰葡糖胺的糖肽Dabsyl-Gly-Glu-Asn-(GlcNAc)-Arg,由此推断PNGase H+的最小糖结构作用单底物为带有N-乙酰壳二糖的糖肽。

