基于LIDC/IDRI影像数据库的专家诊断信息解读软件的开发
王婧璇,林 岚*,赵思远,吴水才
(1.北京工业大学生命科学与生物工程学院,北京 100124;2.北京工业大学软件工程学院,北京 100124)
0 引言
肺癌是发病率和死亡率增长最快、对人群健康和生命威胁最大的恶性肿瘤之一[1]。2018年2月,国家癌症中心发布了最新一期的全国癌症统计数据:我国恶性肿瘤发病率和死亡率居首位的均是肺癌,按发病例数排位,每年发病约78.1万例;按死亡例数顺位,每年死亡病例约62.6万[2]。而肺癌早期发现是可治愈的,“早期发现、早期诊断、早期治疗”是降低肺癌死亡率的重要措施。因此,肺癌的早期诊断越来越受到广大群众及医务人员的重视。欧美发达国家已经开展了对肺癌高危人群的早期筛查[3]。目前,肺癌早期排查最有效的方法之一是进行CT检查[4]。早期肺癌往往表现为肺部恶性结节,只要对肺部恶性结节做到及时发现并进行鉴别诊断,即可有效筛选出早期肺癌。因此,基于CT影像的计算机辅助恶性结节的自动检测是极为必要的[5-6]。随着计算机辅助诊断(computer aided diagnosis,CAD)技术的发展,越来越多的医生利用CAD系统对肺部CT图像进行诊断,这不仅可以减轻医生的工作负担,还可以提高诊断的速度和准确率。
肺结节的分割与检测离不开参考标准数据集的支持,良好的标准数据集可推动肺部疾病研究中算法的发展。LIDC/IDRI(Lung Image Database Consortium/Image Database Resource Initiative)是世界上最大的公开肺结节影像数据库,为该领域的研究者提供了一个权威的参考平台[7]。LIDC/IDRI影像数据库包含来源于1 010个患者的1 018套全肺CT扫描图像(扫描层厚0.6~5 mm,512×512像素)和对结节的详细诊断描述等信息[8-9]。其中CT影像被存储于DICOM文件中,而与结节相关的专家诊断信息则采用数据描述和交换的通用格式——XML进行数据传输[10-11]。但XML是一种重量级数据格式,数据编码复杂,直接可读性差,对其进行解析需要耗费研究者大量的精力,同时还存在数据查询检索困难,且没有简易的方法建立结节标注信息与CT影像间的联系等问题。
针对LIDC/IDRI影像数据库使用中存在的一些局限,研究者们在这方面做了大量的有益探索。顾晓晖等[12]通过对LIDC/IDRI影像数据库中诊断信息的提取,构建了一个Access数据库,方便用户实现结节信息的查询。王伟胜等[13]使用.NET FrameWork3.5中的关键类技术实现对XML的信息查询。林红利等[14]基于SQL Server数据库,采用Visual C++编程语言开发了一套基于LIDC数据库进行数据检索的肺癌图像数据系统。Wang等[15]建立了统一的数据系统,将不同格式的源数据文件中包含的信息提取并集成到一个新的、统一的数据模型中,并在该数据模型的基础上,总结了结节的几何特征和诊断特征的统计分布。Lin等[16]为了提高DICOM数据模型的可读性、改善其结构、促进DICOM文件中数字成像和通信的共享,提出了DICOM数据模型的XML模式表示方法。这些探索性研究在一定程度上推动了LIDC/IDRI影像数据库的使用,但LIDC/IDRI影像数据库构建的目的是为肺结节的检测与诊断提供一个算法优化和比较的基础平台,而上述研究更多集中在数据库功能上,主要实现数据的检索,并不能方便地导入其他开发平台进行进一步的智能算法验证与开发,且其一般情况下仅能单机使用,开放性和共享性差。
本研究针对LIDC/IDRI影像数据库数据表示中存在的不足,剔除其中包含的一些冗余信息,优化数据表示结构;并对关键诊断信息进行解析,转换为JSON和MAT格式,方便机器学习开发平台对数据的直接读取;同时还设计了一个可视化界面,实现对结节标注的显示。
1 软件开发
1.1 软件架构
基于LIDC/IDRI影像数据库的专家诊断信息解读软件采用自底向上的开发方法,由子类继承父类属性,使类与类之间存在关联,其软件架构如图1所示。软件接受异构类型的输入数据即文本数据和图像数据,其分别用于底层架构中的诊断信息解析模块和诊断信息展示模块,为实现信息统计、专家标注查询和信息分析功能打下基础。
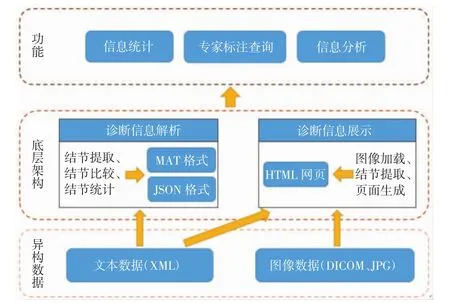
图1 基于LIDC/IDRI影像数据库的专家诊断信息解读软件架构
1.2 软件运行及开发环境
基于LIDC/IDRI影像数据库的专家诊断信息解读软件的开发和调试硬件配置:联想ThinkPad Intel(R)Core(TM)i3-2328M,CPU2.2GHz×4 核,4GBRAM,硬盘500 GB。采用JetBrains公司的PyCharm 2017为软件开发平台,并调用MATLAB接口进行MAT解析格式开发,基于Python编程语言、JavaScript网页编程语言、HTML和CSS完成各功能模块。软件运行环境为64位操作系统,可在Windows 7、Windows 8、Windows 10操作系统,Mac OS系统及Linux系统中运行。
2 软件整体设计
2.1 优化诊断数据结构
XML是一种用于标记电子文件使其具有结构性的标记语言。放射专家把LIDC/IDRI影像数据库中结节的详细情况标注并存储在XML文件中,其中包含专家阅片区域、结节的病理学特征、结节诊断结果等信息[7]。在XML首个标签中包含了序列实例号和检查实例号2个比较重要的信息,其他信息如服务日期、服务时间和版本号等则可以忽略。在专家阅片区域,可以查询到服务放射专家编号、结节信息和非结节的点坐标信息。其中,最受关注的是结节标签中包含标识结节的唯一编号、大结节病理学特征和结节感兴趣区域。而大结节的感兴趣区域由结节像素的点坐标一一描述,即用点表示轮廓,但该方法无法将大结节的形状直观地展示出来,且一个点坐标需占用XML文件的一行,繁多的点坐标使得XML文件长度达到了上万行。这种冗余的信息和复杂的数据存储结构会给相关研究带来一定的困难。
XML是包括多层嵌套的数据结构,其与树模型下节点间的结构关系直接对应,因此本研究中将XML数据转化成一个元组树的数据结构。将XML的每个标记表示成一个Python元组数据类型并进行封装,然后再以“对象名·属性名”面向对象的方式来访问各类诊断信息。基于此,本研究中采用面向对象的方法来完成对诊断信息结构的优化,采用模块化的方法来完成软件开发。
2.2 诊断信息解析
目前,机器学习技术被大量用于肺癌检测与诊断中,但适于机器学习平台调用的肺癌数据比较少,对于转换后的LIDC/IDRI影像数据库中的专家诊断信息都存储在大型数据库中,其不能承载大数据,且开放性和共享性较差,因此需要开发出方便导入机器学习平台的数据。为了解决上述问题,本研究中将解析过程分为结节(nodule)、公用工具(utilities)和信息统计(statistic_case)三大类,其结构设计如图2所示。
首先对结节进行分解,将一些功能独立、内聚强、相互之间耦合较松散的模块抽象出来,得到一些通用的属性和操作并加以封装,进而建立结节基类,并用于定义结节特征与信息处理的主要函数接口,其包含的多个派生类数据结构随结节种类的不同而稍有差别。但是这些派生类的数据结构及其函数、方法和过程的定义存在很大相似性,因此,可以以构件重用的形式有针对性地对诊断信息进行结构化的表达和组织,最大限度地进行代码和数据结构的重用和共享。结节基类中各派生类的详细介绍见表1,它们共同用以完成各类结节的特征提取与信息处理。

表1 结节基类中的各派生类
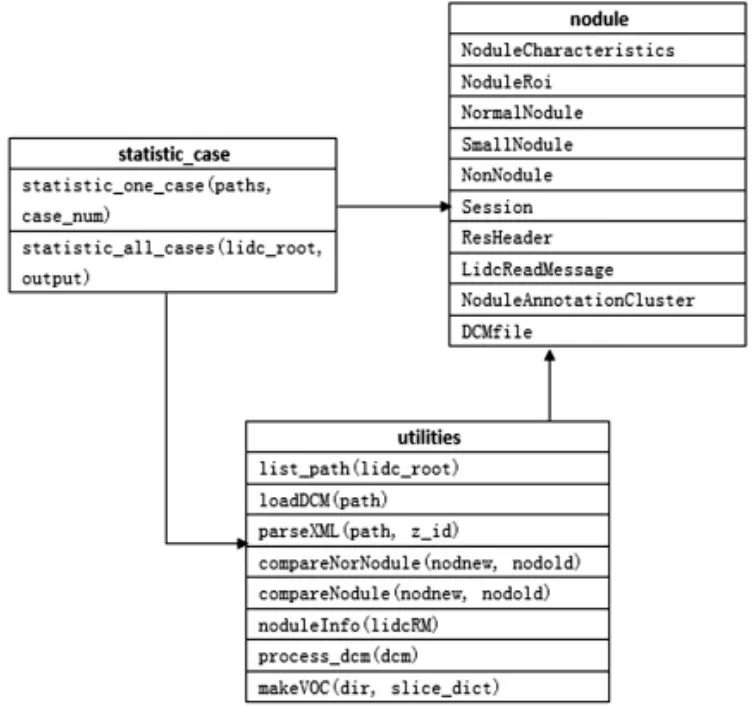
图2 XML解析类之间的关系
公用工具类中包含文件路径处理函数list_path、DICOM图像导入函数loadDCM、XML文件解析函数parseXML、结节比较函数compareNorNodule、非结节比较函数compareNodule、结节信息函数noduleInfo、图像处理函数process_dcm等。
信息统计类中主要包含统计各医生标注的结节信息和全部对象的结节信息2个函数,每个函数均是一个功能完备的子模块。
2.3 可视化界面设计
通过阅读和分析文本类解析信息并不直观,为了使研究者更直接地学习到专家诊断信息,本文结合DICOM和XML设计并实现了诊断信息可视化界面。可视化展示是一种更为直观的显示方式,在对XML和DCIOM图像分析的基础上,结合HTML、CSS和JavaScript等前端开发技术来实现专家标注结节的展示模块。该模块主要通过对生成界面类(createhtml)、结节类(nodule)和页面模板类(html)函数进行调用,从而实现结节可视化的功能,结节可视化程序类之间的关系如图3所示。首先,肺CT图像中的信息全部以DICOM格式进行存储,且每张图像包含103个字段,可视化程序中将调用其中的几个关键字段。其次,需要调用保存在XML格式中的专家标注结果。最后,实现DICOM和XML间对应关系的匹配。具体来说,在DICOM和XML中均用StudyInstanceUID字段表示检查实例号,用SeriesInstanceUID字段表示一次检查的序列号;而切片在z方向的相对位置在DICOM中用SliceLocation字段表示,在XML中用imageZposition字段表示,但其实际含义是一致的。根据以上3个关键字段可以实现诊断信息与医学图像间的关联,从而实现诊断信息的可视化展示。
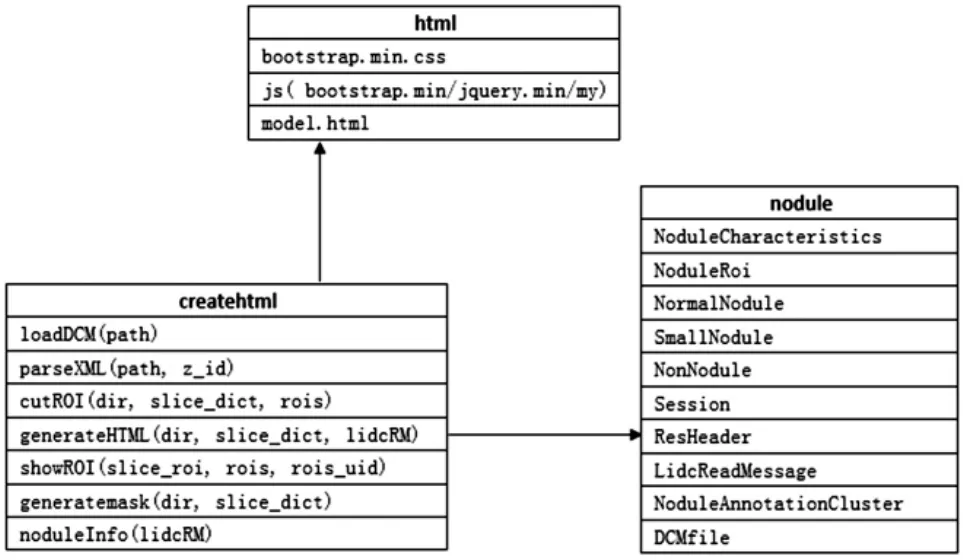
图3 结节可视化程序类之间的关系
3 软件功能实现
3.1 诊断信息解析模块
诊断信息解析模块剔除了原有XML文件中的冗余内容,使优化后的诊断数据结构更加清晰,数据间的表示关系更加规范,提高了查询速度。该模块实现了诊断信息的解析,并生成了适用于机器学习平台的数据:将XML数据格式以“对象名·属性名”形式转换成MAT和JSON格式(部分结果如图4所示)。这不仅便于机器学习平台对数据进行直接读取、分析、集成与使用,还可以选择性地展开所需信息,关闭无用信息。

图4 诊断信息的格式转换
3.2 诊断信息展示模块
诊断信息展示模块即可视化界面模块,实现了诊断信息的展示。图5为专家诊断信息的可视化界面,可以直观地将结节信息反映在DICOM图像中。图中显示了4名专家的诊断结果,每位专家对于结节3种情况(3 mm以上大结节、3 mm以下小结节和非结节)的勾画可以通过红、绿和蓝3种颜色框清晰地展现。如果需要按照专家或者结节类别进行查看,则可以勾选对应的选择框。

图5 结节可视化展示界面
4 结语
本研究在开发与设计中充分利用了模块化、构件重用和面向对象的思想,实现了结节基类和派生类的设计,并将其应用于结节信息提取和标注可视化的显示功能,软件整体的可重用性、可维护性及可读性好,结构清晰。同时,为了与机器学习平台更好地集成,本软件还将数据转换为常用机器学习平台可以直接使用的格式,便于与智能化分析系统的集成。整个软件基于Python编程语言开发,拥有强大的标准库和第三方库,从而具有跨平台的可移植性、开放性和共享性。当然,本文构建的原型软件还不完善,仍有很多地方需要改进,如解析和显示还是2个单独的模块,并没有集成为一个完整的系统。同时,尽管诊断数据被转换为常用机器学习平台可直接读取的格式,但还没有实现其自动化诊断分析的集成工作,后期将会对上述问题进行进一步的研究与完善。

