两株灰黄霉素产生菌的18S rDNA和ITS序列测定与分析
陆娇娇 畅瑛 李波 施碧红



摘 要:灰黄霉素产生菌原始菌株4541和突变株D-756在形态与生理生化上具有很大差异。为了解两菌株的遗传背景差异,明确其系统发生关系,该研究分别克隆并测定了突变株D-756与出发菌株4541的ITS和18S rDNA基因序列,进行同源性分析并分别构建系统发育树。由ITS序列构建的进化树显示,突变株D-756及野生株4541与Genebank中的另外3株灰黄青霉、1株荨麻青霉菌株聚为一簇;由18S rDNA序列构建的进化树中突变株D-756、出发菌株4541与Genebank中的3株灰黄青霉菌株也聚为一支。研究结果表明,在分子水平上出发菌株4541及其突变株D-756属于灰黄青霉种。
关键词:灰黄青霉;ITS序列;18S rDNA;系统发育树
中图分类号:Q754 文献标志码:A 文章编号:0253-2301(2019)10-002
DOI: 10.13651/j.cnki.fjnykj.2019.10.002
Abstract: There were significant differences in morphology, physiology and biochemistry between the original strain 4541 and the mutant strain D-756 of griseofulvin producing strains. In order to understand the genetic background differences between the two strains and make clear their phylogenetic relationships, the ITS and 18S rDNA gene sequences of the mutant strain D-756 and the original strain 4541 were cloned and determined in this study. Then, the homology analysis was conducted and the phylogenetic trees were constructed respectively. The evolutionary tree constructed from the ITS sequence showed that the mutant strain D-756 and the wild strain 4541 were clustered together with the other 3 strains of Penicillium griseofulvum and 1 strain of Penicillium urticae in Genebank. The mutant strain D-756 and the original strain 4541 in the evolutionary tree constructed by 18S rDNA sequence were also clustered with the other 3 strains of Penicillium griseofulvum in Genbank into one branch. The results showed that the original strain 4541 and the mutant strain D-756 belonged to penicillium griseofulvum at the molecular level.
Key words: Penicillium griseofulvum; ITS sequence; 18S rDNA; Phylogenetic tree
灰黃霉素是一种有效的聚酮类抗真菌抗生素,可用于治疗皮肤癣菌引起的浅层真菌感染,在农业上也用于防治多种植物病原菌,灰黄霉素还能在体外抑制肝炎病毒的复制[1-2]。近年来发现灰黄霉素可抑制癌细胞中心体聚集,从而形成多级纺锤体,导致癌细胞分裂抑制而死亡[3]。鉴于灰黄霉素的医用和农用价值,人们对灰黄霉素高产菌株的选育工作一直持续进行。本研究采用的D-756是吴松刚等[1]用野生菌展青霉Penicillium patulum 4541经13轮复合诱变获得的灰黄霉素高产突变株。突变株D-756与出发野生菌株4541相比,孢子形态由野生菌的绿色变为白色,灰黄霉素的合成能力也大幅提高[4-5]。但目前就两者的遗传背景差异知之甚少,明确诱变系谱中菌株的系统发生关系是进行后续遗传研究的基础。
传统的真菌分类主要以形态学观察结合生理生化特征分析为主,但真菌的种类繁多复杂,种属间的形态学差异不明显,生理生化性状也会发生交叉重叠,且容易因温度、湿度、土壤等生活环境的改变而变得不稳定,因此传统的方法进行种属间鉴定时有出现同名异物或异物同名的情况[6]。随着真菌分类学的发展,一些影响较大或在早期的文献中普遍的种名,因种的概念的变化,现已成为其他种的异名,如展青霉Penicillium patulum、荨麻青霉Penicillium urticae现均是灰黄青霉Penicillium griseofulvum Dierckx的异名[6]。在关于灰黄霉素早期的研究报道中,有不少来自展青霉或荨麻青霉的报道,包括本研究的菌株D-756和4541也存有这种情况[1,4-5]。现代分子鉴定技术为传统真菌分类方法提供了有力补充。ITS和18S r DNA分子鉴定具有快速、准确、微量、方便的优点,能够从分子水平为真菌的分类、鉴定提供重要依据和丰富的遗传信息,已广泛用于真菌属种间的系统学研究[7]。本研究利用2对真菌通用引物ITS-1、ITS-4和S1、S2分别扩增了突变株D-756及其野生出发菌株4541的ITS和18S rDNA序列,并进行测序分析,测得的序列提交GenBank,并分别基于ITS和18S rDNA序列进行GenBank数据库同源性检索比对和系统发育树的构建,以了解两株灰黄霉素产生菌的亲缘关系,为进一步明确其归属、确认这两株菌的分类地位以及为后续的遗传研究提供信息数据。
1 材料与方法
1.1 菌株来源
灰黄霉素产生菌原始菌株4541和突变株D-756由福建师范大学吴松刚教授提供;大肠杆菌JM109为本实验室保存。
1.2 主要试剂药品
LA Taq DNA聚合酶、Ex Taq DNA 聚合酶、SolutionI连接酶、pMD18-T Vector购于TaKaRa公司;Ezup柱式真菌基因组DNA抽提试剂盒、SanPrep柱式DNA胶回收试剂盒、SanPrep柱式质粒DNA小量抽提试剂盒、氨苄青霉素购于生工生物工程技术服务(上海)有限公司;真菌ITS基因通用引物ITS1和ITS4及18S rDNA通用引物由生工生物工程技术服务(上海)有限公司合成。
1.3 主要培养基
察氏培养基:蔗糖30 g·L-1、氯化钾0.5 g·L-1、硝酸钠3 g·L-1、磷酸二氢钾1 g·L-1、硫酸亚铁0.01 g·L-1、硫酸镁0.5 g·L-1、琼脂20 g·L-1。LB液体培养基(LB固体培养基加入1.5%琼脂):胰蛋白胨 10 g·L-1、酵母粉5 g·L-1、NaCl 10 g·L-1,pH 7.2。种子培养基、发酵培养基参照文献[4]。
1.4 引物设计
1.4.1 真菌ITS通用引物 真菌ITS通用引物设计如下:
ITS-1:5′-TCCGTAGGTGAACCTGCGG-3′;
ITS-4:5′-TCCTCCGCTTATTGATATGC-3′。
1.4.2 18S rDNA通用引物 18S rDNA通用引物设计如下:
S1:5′-CCAACCTGGTTGATCCTGCCAGTA-3′;
S2:5′-CCTTGTTACGACTTCACCTTCCTCT-3′。
1.5 试验方法
1.5.1 两菌株灰黄霉素合成能力的比较 突变株D-756和原始出发株4541经过14 d摇瓶发酵后,采用比色法分别测定两株菌发酵液的灰黄霉素效价[8]。
1.5.2 基因组DNA提取 分别收集突变株D-756和出发菌株4541的菌体,用Ezup柱式真菌基因组DNA抽提试剂盒提取总DNA。
1.5.3 ITS和18S rDNA片段的PCR扩增 分别以提取的菌株D-756和4541的基因组DNA为模板,采用真菌ITS的通用引物ITS1和ITS4进行PCR扩增。PCR扩增体系(50 μL)为:10×Ex PCR buffer 5 μL、dNTP Mix (2.5 mmol·L-1) 4 μL、引物ITS1 2 μL、引物ITS4 2 μL、模板DNA 1 μL、Ex Taq 酶0.5 μL、ddH2O 35.5 μL。PCR条件:95℃预变性5 min,94℃变性40 s,55℃退火35 s,72℃延伸45 s,进行30次循环后,72℃延伸10 min,4℃保存。
以基因组DNA为模板,采用真菌18S rDNA的通用引物S1和S2进行PCR扩增。PCR扩增体系(50 μL)为:10×LA PCR buffer 5 μL、dNTP Mix (2.5 mmol·L-1) 4 μL、S1上游引物2 μL、S2下游引物2 μL、模板DNA 1 μL、LA Taq 酶0.5 μL,ddH2O 35.5 μL。PCR条件:95℃预变性5 min,94℃变性40 s,55℃退火35 s,72℃延伸2 min,进行30次循环后, 72℃延伸10 min,4℃保存。
用1%琼脂糖凝胶电泳检测扩增产物,用DNA胶回收试剂盒回收目的产物。
1.5.4 ITS、18S rDNA序列的测定 将上述胶回收纯化的PCR产物与载体pMD 18-T连接,并转化大肠杆菌JM109,菌落PCR鉴定阳性克隆。PCR体系(20 μL):10×LA PCR buffer 2 μL、dNTP Mix (2.5 mmol·L-1) 1.6 μL、上游引物0.8 μL、下游引物0.8 μL、LA Taq 酶0.2 μL、ddH2O 14.6 μL。挑取阳性克隆过夜培养,培养液用质粒DNA小量抽提试剂盒提取质粒DNA送至生工生物工程技术服务(上海)有限公司测序。
1.5.5 系统发育树构建 测序获得的结果提交Genbank获取登录号,并在NCBI中进行blast检索同源序列,选取相似度高的序列,用Clustal X(1.83)软件进行多序列比对后,采用MEGA 6.0软件以邻近相接法(Neighbor-joining,NJ)构建系统进化树,自展数为1000。
2 结果与分析
2.1 D-756和4541的灰黄霉素效价
突变株D-756是由野生出发菌株4541经13轮诱变育种筛选出的灰黄霉素高产突变株,两者除菌落形态上由野生型的灰绿色变成突变型的白色外[1],其灰黄霉素的合成能力也有了大幅度的提高。分别将菌株
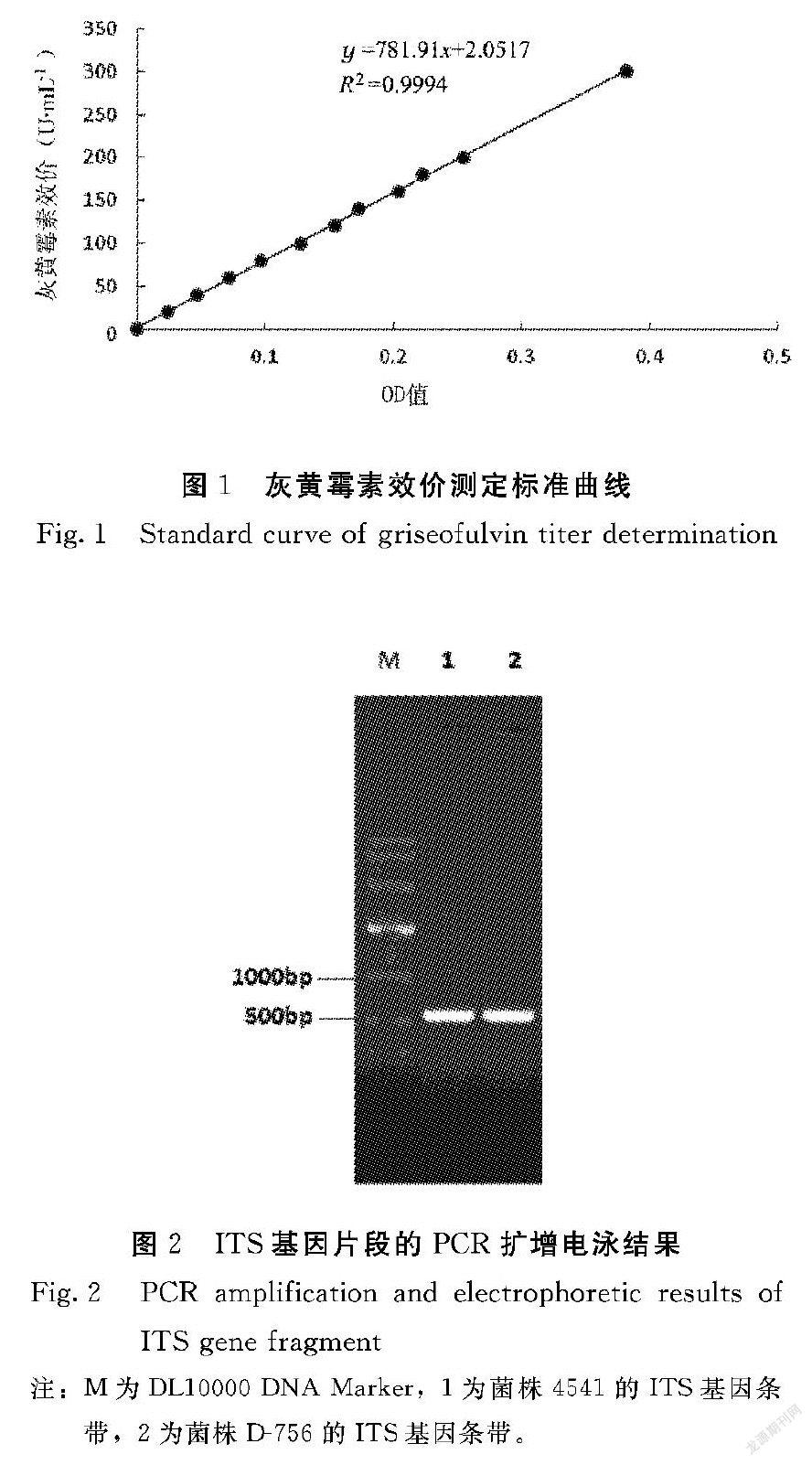
D-756和4541进行摇瓶发酵14 d,用比色法测定灰黄霉素效价,参照标准曲线(图1),获得D-756和4541的灰黄霉素平均效价分别为13788 U·mL-1和180 U·mL-1,突变株D-756的灰黄霉素效价是出发菌株的76倍。
2.2 ITS基因序列片段的PCR扩增
不同真菌的ITS长度不一样,一般在500~800 bp[9]。以提取的基因组DNA为模板PCR扩增的ITS片段如图2所示,目的条带的位置大約在550 bp处,与预期相符且条带明亮清晰无杂带。
2.3 18S rDNA基因序列片段的PCR扩增
真菌的18S rDNA基因大小约为1.8 kb[10],由PCR扩增的18S rDNA片段如图3所示,目的条带的位置约在1710 bp处,大小符合预期,且条带明亮清晰,可用来构建重组质粒。
2.4 ITS及18S rDNA序列的测定
将上述ITS及18S rDNA的PCR产物纯化后,克隆到pMD18-T载体后测序。出发菌4541和突变株D-756的ITS序列均为545 bp(登录号:MK843252、MN046363)。经Bioedit软件序列比对,发现两者的ITS序列完全相同,这说明突变株D-756虽然是4541经13轮复合诱变而来,但其ITS区域高度保守;菌株4541和D-756的18S rDNA序列长度分别为1711 bp(登录号:MN044711)和1710 bp(登录号:MK841479),序列比对结果表明,两菌株的18S rDNA序列9个碱基位点差异,说明4541经过13轮诱变处理,其18S rDNA序列有少数位点发生突变,但两者的序列相似性仍大于99%。
2.5 ITS序列和18S rDNA序列构建系统发育树
基于ITS序列的检索分析,选取同源性较高的包括青霉属、曲霉属、新萨托菌属等属的20条序列(相似性在90.51%~100.00%),并以1株Monascus eremophilus为外类群,由MEGA 6.0构建ITS系统发育树(图4),图4结果显示菌株4541和D-756与Genbank中的另外3株灰黄青霉菌株P.griseofulvum(AF034452.1、NR103692.1、GU566212.1)及1株荨麻青霉P.urticae(AF514301)聚为一簇,并且与这些序列的相似性均≥99%。Landeweert[11]等提出ITS鉴定原则,供试菌株的ITS序列与GenBank数据库中的序列检索比对,序列相似性≥99%,可以鉴定为相同种。
基于18S rDNA序列检索分析,选取了同源性较高的19条序列(相似性在96.49%~99.76%)(包括青霉属、曲霉属、副球孢子菌属等),以1株Paracoccidioides brasiliensis为外类群,构建18S rDNA进化树(图5),结果表明菌株4541和D-756与Genbank中的另外3株灰黄青霉菌株(JN886804.1、EF607282.1、JN886803.1)聚为一簇,其序列间相似性均≥99%。由图4~5可以看出,出发菌株4541和突变菌株D-756与Genebank中的灰黄青霉其他菌株均聚为一簇,说明他们属于灰黄青霉种。
3 结论与讨论
微生物分类方法有很多种,依据的分类标准也有所不同,真菌的分类主要依靠营养体以及子实体的形态特征来判断,但是这两个特征很容易受环境的影响发生改变[12]。并且,随着越来越多的真菌种类被人们发现,传统的形态学分类方法已经不能精确地进行种属分类鉴定。如今,分子生物学的飞速发展,可以在分子水平上确定物种的分类和进化关系,是对傳统微生物分类方法的有力补充。ITS区域是真菌非编码区,包括ITS1和ITS2两部分,承受的选择压力较小,具有较丰富的位点变异信息。18S rDNA是真核生物的核糖体小亚基RNA的编码DNA,在进化上十分保守。18S rDNA和ITS都可以作为真菌种属鉴定的分子标记,目前在国内外应用广泛。
灰黄青霉P.griseofulvum 4541(早期文献为展青霉P.patulum 4541)及其诱变获得的高产突变株D-756在形态上有显著差异,出发菌株4541的孢子呈灰绿色,突变株D-756的孢子为白色[1,5]。此外突变菌株D-756较出发菌株4541而言,灰黄霉素的产量大幅提升(提高76倍)。为探究两菌株的分子水平差异,本研究分别克隆并测定了菌株4541和D-756的ITS及18S rDNA序列,结果显示两者的ITS序列完全相同,其18S rDNA序列相似性亦大于99%。
将菌株4541和D-756的ITS及18S rDNA序列分别与GenBank数据库中的同源性较高的相应序列构建系统进化树,发现菌株4541和D-756均与GenBank中其他灰黄青霉P.griseofulvum及荨麻青霉P.urticae聚为一簇,且它们之间的序列相似性大于99%,说明菌株4541和D-756都属于灰黄青霉种,在分子水平上进一步确定了原始菌株4541及其突变株D-756的分类地位以及它们之间进化关系。该结果也论证了国内外将早期报道的展青霉P.patulum、荨麻青霉P.uritcae归入灰黄青霉Penicillium griseofulvum异名的事实[7,2,13]。同时结果也表明,突变株D-756与原始菌株4541表型差异较大,可能是由于菌株在多代的诱变过程中为了适应生存,相关基因发生了改变,但确实是由原始菌株突变而来,不存在菌种污染,为后续全面的分子遗传研究储备信息数据。
在青霉属真菌的开发应用过程中,由于历史的条件限制,常把青霉与非青霉弄混淆,加之不同的研究者常赋予同一个菌不同的名字,青霉属真菌的命名有些混乱[6]。灰黄霉素原始产生菌株4541及其突变株
D-756在早期文献为展青霉Penicillium patulum[1,4-5],在《中国真菌志》中已将荨麻青霉Penicillium urticae、展青霉Penicillium patulum归为灰黄青霉Penicillium griseofulvum的异名[6]。本研究结果与《中国真菌志》中的有关灰黄青霉分类阐述一致。研究结果也表明了基于ITS和18S rDNA序列的分子鉴定可以快速有效地进行种属鉴定,是传统真菌分类方法的有效补充。
参考文献:
[1]吴松刚,郝家骐,戚晓玉,等.Penicillum patulum 4541耐前体突变株的选育[J].遗传,1980,2(4):16-18.
[2]CHOOI Y H ,CACHO R,TANG Y.Identification of the Viridicatumtoxin and Griseofulvin Gene Clusters from Penicillium aethiopicum[J].Chemistry & Biology,2010,17(5):483-494.
[3]REBACZ B,LARSEN T O,CLAUSEN M H,et al.Identification of Griseofulvin as an Inhibitor of Centrosomal Clustering in a Phenotype-Based Screen[J].Cancer Research,2007,67(13):6342-6350.
[4]吴松刚,鲍运森.灰黄霉素高产菌株特性的研究[J].微生物学通报,1983,10(5):204-205.
[5]吴松刚,许艳萍,吴芷萍,等.灰黄霉素产生菌(Penicillium patulum 4541)的选育[J].医药工业,1979(2):14-20.
[6]孔华忠.中国真菌志第三十五卷.[M].北京:科学出版社,2007.
[7] IWEN P C,HINRICHS S H,RUPP M E.Utilization of the internal transcribed spacer regions as molecular targets to detect and identify human fungal pathogens[J].Medical Mycology,2002,40(1):87-109.
[8]陳钧鸿,徐玲娣.抗生素工业分析[M ].北京:中国医药科学技术出版社,1991:326-329.
[9]李玉龙,董婧,王彬,等.基于18S和ITS-5.8S rDNA基因序列的白色霞水母(Cyanea nozakii)的分子鉴定与检测[J].海洋与湖沼,2016,47(1):158-165.
[10]林子杰.三种绿藻的ITS和18SrDNA序列及系统发育分析[D].苏州:苏州大学, 2011.
[11]LANDEWEERT R,LEEFLANG P,KUYPER T W,et al.Molecular identification of ectomycorrhizal mycelium in soil horizons[J].Applied and Environmental Microbiology,2003,69(1):327-333.
[12]陈凤毛.真菌ITS区序列结构及其应用[J].林业科技开发,2007,21(2):5-7.
[13]BANANI H, MARCET-HOUBEN M,BALLESTER AR,et al.Genome sequencing and secondary metabolism of the postharvest pathogen Penicillium griseofulvum[J].BMC Genomics,2016,17:19-33.
(责任编辑:林玲娜)

