肝细胞肝癌转录组学的初步结果分析
姚佳

【摘 要】目的:通过对肝细胞肝癌的转录组学研究,从而对肝细胞肝癌的基因组的异质性和生物学机制有一个更加全面的理解。方法:对10例肝细胞肝癌患者,对癌及癌旁组织进行转录组测序。结果:每个样本大量的表达差异的长链非编码RNA(范围为54到69)和差异表达的基因(范围为2971到6910)。共2873个具有明显表达差异的基因,其中上调1666个,下调1207个(差异基因标准:FDR<0.001, Foldchang≥2, 存在于5对样本以上)。对差异基因的功能分析结果显示:差异基因主要集中在细胞周期、细胞粘附、脂代谢和炎症等功能类和PPRA、P450的代谢、P53和DNA的复制等信号通路。此外,每个样本发现了大量的肿瘤特异性突变,其中22.2%位于基因的编码区,大部分(42.6%)为T:A >C:G这一类型的突变。结论:对差异基因的分析显示主要集中于细胞周期、PPRA和P53等信号通路,表明这些信号通路在肝癌的发生发展中起到关键作用。对于肿瘤特异性突变的分析,高频的T:A >C:G这一突变类型的发生,我们推测基因突变主要发生在RNA编辑这一过程中,这表明RNA编辑对肝癌的发生发展有相当大的影响。大量的新的基因事件的发生,说明肝癌的发生发展机制比以往的理解更为复杂。
【关键词】肝癌;转录组测序;RNA编辑
【中图分类号】R158 【文献标识码】A 【文章编号】1004-7484(2020)09-0288-02
肝细胞肝癌占了原发性肝脏肿瘤的80—90%,居全世界肿瘤发病率的第五位,肿瘤相关性死亡的第三位[1]。肝细胞肝癌是一种具有高度侵袭性的肿瘤,尽管采用了以手术切除为主的一系列综合治疗手段,但是肝癌患者的长期生存率仍然较低[2-3]。肝细胞肝癌是一种异质性疾病,由于基因、表观遗传和转录组水平的改变,使得肝细胞肝癌的形态学和临床表现多样性[4] 这些遗传多样性严重影响了肝癌的诊断、治疗及预后。
对于肝细胞肝癌分子水平的转录组研究成为了近年研究的热点,并且对于进一步筛选肝癌诊断及复发转移相关的分子、揭示肿瘤的发生发展机制和寻找新的靶向治疗位点显示出了不可估量的潜力。本研究围运用二代测序技术,对肝细胞肝癌进行深入测序,以寻找新的肝癌诊断及复发转移的相关分子和通路,对肝细胞肝癌的分子生物学机制有一个更加全面和深入的理解为目标。
1方法及材料:
1.1 收集本院 2015 年 1 月至 2018 年 12月接受肝癌肝切手术的男性病人,均经病理组织学证实为原发性HCC,且均为HBV相关性肝癌,无合并HCV等其他肝炎病毒感染。临床病理学资料完整,包括年龄、性别、术前AFP水平、有无血管侵犯及远处转移、病理分化分级、肿瘤大小、数目等。术前均未行抗肿瘤治疗,手术切除的组织立即冷冻于-80 ℃以用于提取RNA。
1.2 主要试剂:美国Invitrogen 公司Trizol总RNA提取试剂;上海生工生物工程有限公司 DEPC、琼脂糖;美国Illumina公司DNase I,oligo(dT),fragmentation buffer,random hexamers,缓冲液、dNTPs、RNase H和DNA polymerase I,polyA, QiaQuick PCR试剂盒;其他化学试剂:氯仿、异丙醇、无水乙醇、甲醇、等均为国产分析试剂。
1.3实验方法:
1.3.1转录组测序:RNA-Seq 由深圳华大基因公司所做,简要步骤如下: 分别提取样本的总RNA,使用 DNase 消化 DNA 后富集mRNA,用打断试剂将mRNA打断成短片段并以此为模板合成 cDNA, 使用试剂盒纯化双链 cDNA 再进行末端修复、加 A 尾, 连接接头后选择合适的片段大小进行PCR扩增,构建好的文库用 Agilent 2100 Bioanalyzer 质检合格后,使用 Illumina HiSeqTM 2500 测序仪进行测序,上样量为 50 ng。
1.3.2测序数据质量评估及处理
测序得到的原始数据为raw data,接下来数据处理的步骤:(1)去除含adaptor的reads;(2)去除N的比例大于5%的reads;(3)去除低质量reads(质量值Q≤15的碱基数占整个read的50%以上)。得到Clean data后,我们使用MAQ 软件将clean reads 与参考序列进行比对,每个read的碱基错配不能大于5个。
1.3.3差异表达基因(DEGS)的筛选
对基因进行表达分析,基因表达量使用 FPKM( Fragments Per kb per Million reads) 方法計算,以校正的 P 值( false discovery rate, FDR) ≤0. 05、差
异倍数的对数绝对值( ︱log2FoldChange︱,︱FC︱) ≥2为阈值,筛选癌和癌旁的DEGS。1.3.4 差异表达基因的 GO 分析和 KEGG 富集筛选出 DEGs 后,我们对 DEGs 进行 GO 和 KEGG 富集分析。
2结果:
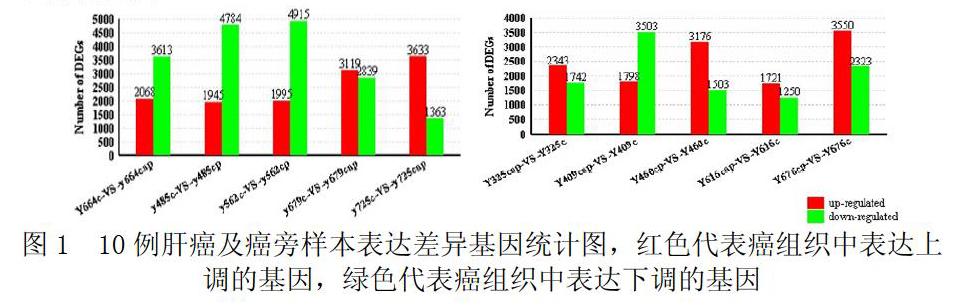
2.1 表达差异:(图1)
我们选择10例肝癌样本及相应的癌旁组织进行转录组测序(RNA-seq),10对样本平均生成26 355 902 (range: 25 580 346-27 540 350) reads和2.37(range: 2.30-2.48)Gb的测序数据。采用MAQ软件,平均92.76% (range: 89.37-97.02%)的reads能够比对到人类基因组(UCSC version hg19)。通过比对肿瘤及其配对癌旁组织的转录本,我们筛选到每对肝癌样本中具有表达差异的基因(2971 to 6910)。
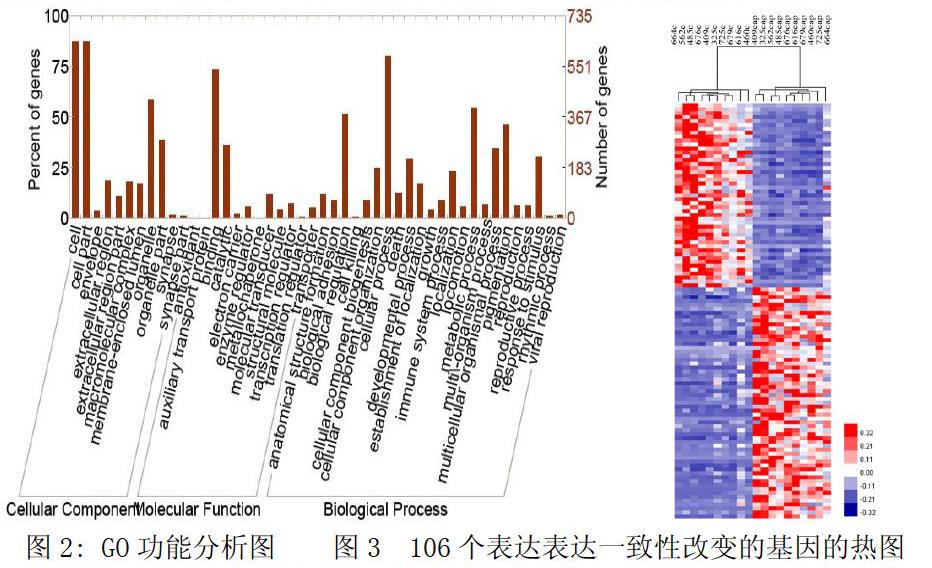
2.2差异基因及GO和pathway分析
通过将10对样本的read比对到基因组后,计算他们的表达量,用RPKM来衡量他们的表达量。通过将癌和癌旁组织的RPKM值进行比较,计算他们的表达差异。将10对样本的差异基因进一步按以下条件筛选:FDR ≤ 0.001,foldchang≥ 2,上调或者下调在5对样本以上。共获得2873个基因,随后进行GO和pathway的分析,结果表明,差异基因主要集中于细胞周期的调控、细胞粘附、类固醇及脂类代谢、依赖于DNA的修复及炎症免疫反应等功能类和P53、细胞周期,PPRA,DNA修复等信号通路,具体结果见图2及表1。随后对于10对样本中一致上调或下调的106个基因进行聚类,见图3
2.3 SNP及肿瘤特异性突变:
每个肝癌样本平均找到12909(范围为6877-27970)个肿瘤特异性点突变,这些突变位点22.2%位于基因编码区域(CDS区),其中946个为非同义突变。其中42.6%为T:A >C:G(图4)。
讨论:
从分子生物学的角度讲,肝癌和其他肿瘤一样,是一种复杂的疾病[4-5]。过去的几十年的研究已经证明,基因的改变在肝癌的发生发展因此起着重要的作用[6-8]。对于肝癌的基因学特征的更加详细的了解,能够促进肝癌在诊断,治疗及预后等方面发展。本研究中,我们对10例肝癌及其癌旁组织进行了转录组测序,通过这些数据及联合分析,我们首先分析了肿瘤及癌旁组织基因的表达差异,其中有很多基因已经被证实与肝癌的发生有关,例如血管内皮生长因子(VEGF),磷脂酰肌醇蛋白聚糖3(GPC3)[9-11]。同样,也发现了很多新的未被注释的基因被找到,这说明基于转录组测序来寻找表达差异的基因是一种更加高效的研究方式。在这些信号通路中,大部分已经被报道证实与肝癌的发展具有密切的关系,例如细胞周期的信号通路、P53、PPAR等[12]。同时,差异基因在一些基本的代谢途径中被富集,例如,类固醇的代谢、脂肪酸的代谢。特别有趣的是,细胞色素P450这一信号通路,许多该家族的基因在肝癌组织中明显的下调,这与先前关于肝癌组织中细胞色素P450分子的活性降低及低表达相一致[13]。总而言之,转录组测序揭示肝癌的发生涉及到多条关键的细胞通路,包括细胞生长相关的通路,代谢相关的过程及免疫相关的调剂通路。
人类的基因是由多个外显子中穿插着内含子来构成。初级转录本RNA通过RNA的剪切的过程,内含子被去除,成为一个成熟的mRNA.通过选择性剪切的机制,一个初级转录本能够生成多个mRNA[14]。RNA的选择性剪切起着重要的生物功能,例如膜结合蛋白IgM和分泌蛋白IgD 是由同一个初级转录本经过选择性剪接所产生[15]。已有许多基于基因组的基础上,对于RNA选择性剪切的研究,这些研究表明人类35-39%的基因至少有一个选择性剪切异构体[16-19]。一些研究显示,RNA选择性剪切频繁的发生于肿瘤相关的基因,例如EGFR,CD44,NER[20-22]。本研究基于基因组学的基础上,寻找与肝癌相关的选择性剪切事件,作为肝癌诊断的一个潜在的肿瘤标志物。突变位点22.2%位于基因编码区域(CDS区)其中42.6%为T:A >C:G,对于这一现象,合理的解释为这种类型的点突变发生在RNA编辑过程中。RNA编辑过程中将腺嘌呤(A)改变为次黄嘌呤核苷酸(I),而后次黄嘌呤核苷酸被翻译为鸟嘌呤核苷酸(G)[23]。
针对转录组数据的分析,发现了大量的新的基因事件的发生,包括选择性剪接、肿瘤特异性突变等,说明肝癌的发生发展機制比以往的理解更为复杂。这一较为全面的肝癌转录组水平的研究,为我们进一步研究肝癌发病的分子机制提供了重要的线索和参考价值。
参考文献
[1]Lau WY, Lai EC. Hepatocellular carcinoma: current management and recent advances. Hepatobiliary Pancreat Dis Int 2008; 7: 237-257.
[2]Zheng SS, Xu X, Wu J et al. Liver transplantation for hepatocellular carcinoma: Hangzhou experiences. Transplantation 2008; 85: 1726-1732.
[3]Lo CM, Fan ST. Liver transplantation for hepatocellular carcinoma. Br J Surg 2004; 91: 131-133.
[4]Feo F, Frau M, Tomasi ML et al. Genetic and epigenetic control of molecular alterations in hepatocellular carcinoma. Exp Biol Med (Maywood) 2009; 234: 726-736.
[5]Zucman-Rossi J, Laurent-Puig P. Genetic diversity of hepatocellular carcinomas and its potential impact on targeted therapies. Pharmacogenomics 2007; 8: 997-1003.
[6]Acevedo LG, Bieda M, Green R, Farnham PJ. Analysis of the mechanisms mediating tumor-specific changes in gene expression in human liver tumors. Cancer Res 2008; 68: 2641-2651.
[7]Woo HG, Park ES, Lee JS et al. Identification of potential driver genes in human liver carcinoma by genomewide screening. Cancer Res 2009; 69: 4059-4066.
[8]Hoshida Y, Nijman SM, Kobayashi M et al. Integrative transcriptome analysis reveals common molecular subclasses of human hepatocellular carcinoma. Cancer Res 2009; 69: 7385-7392.
[9]Chiang DY, Villanueva A, Hoshida Y et al. Focal gains of VEGFA and molecular classification of hepatocellular carcinoma. Cancer Res 2008; 68: 6779-6788.
[10]Midorikawa Y, Ishikawa S, Iwanari H et al. Glypican-3, overexpressed in hepatocellular carcinoma, modulates FGF2 and BMP-7 signaling. Int J Cancer 2003; 103: 455-465.
[11]Nakatsura T, Yoshitake Y, Senju S et al. Glypican-3, overexpressed specifically in human hepatocellular carcinoma, is a novel tumor marker. Biochem Biophys Res Commun 2003; 306: 16-25.
[12]Iakova P, Timchenko L, Timchenko NA. Intracellular signaling and hepatocellular carcinoma. Semin Cancer Biol 2011; 21: 28-34.
[13]Morgan ET. Down-regulation of multiple cytochrome P450 gene products by inflammatory mediators in vivo. Independence from the hypothalamo-pituitary axis. Biochem Pharmacol 1993; 45: 415-419.
[14]Hastings ML, Krainer AR. Pre-mRNA splicing in the new millennium. Curr Opin Cell Biol 2001; 13: 302-309.
[15]Early P, Rogers J, Davis M et al. Two mRNAs can be produced from a single immunoglobulin mu gene by alternative RNA processing pathways. Cell 1980; 20: 313-319.
[16]Huang YH, Chen YT, Lai JJ et al. PALS db: Putative Alternative Splicing database. Nucleic Acids Res 2002; 30: 186-190.
[17]Modrek B, Resch A, Grasso C, Lee C. Genome-wide detection of alternative splicing in expressed sequences of human genes. Nucleic Acids Res 2001; 29: 2850-2859.
[18]Brett D, Hanke J, Lehmann G et al. EST comparison indicates 38% of human mRNAs contain possible alternative splice forms. FEBS Lett 2000; 474: 83-86.
[19]Mironov AA, Fickett JW, Gelfand MS. Frequent alternative splicing of human genes. Genome Res 1999; 9: 1288-1293.
[20]Wikstrand CJ, Hale LP, Batra SK et al. Monoclonal antibodies against EGFRvIII are tumor specific and react with breast and lung carcinomas and malignant gliomas. Cancer Res 1995; 55: 3140-3148.
[21]Naor D, Nedvetzki S, Golan I et al. CD44 in cancer. Crit Rev Clin Lab Sci 2002; 39: 527-579.
[22]Saito H, Nakatsuru S, Inazawa J et al. Frequent association of alternative splicing of NER, a nuclear hormone receptor gene in cancer tissues. Oncogene 1997; 14: 617-621.
[23]Bass BL. RNA editing by adenosine deaminases that act on RNA. Annu Rev Biochem 2002; 71: 817-846.
基金資助:
2017浙江省教育厅课题(Y201738263)

