比较基因组学分析鉴定影响藏山羊耳朵大小的遗传位点
钟 杰,李 利,宋天增,张红平,郭家中
(1.四川农业大学 动物科技学院,成都 611130;2.西藏自治区农牧科学院 畜牧兽医研究所,拉萨 850009)
作为山羊的一个品种标准性状,耳型主要包括耳朵(耳廓)大小、形状和直立性。根据耳型,现有山羊品种可简单地划分为直立耳和长垂耳两类;其中,大多数山羊品种的耳型为直立耳,例如,萨能山羊、吐根堡山羊、济宁青山羊等[1]。而著名的长垂耳类型山羊品种则包括波尔山羊和努比亚山羊等[2]。另外,原产于西班牙,现主要饲养于美国的拉曼查山羊,其耳朵极小;但该品种其他性状正常,尤其是奶用性能突出[3]。总的来说,哺乳动物的耳朵发育是一个受到多基因协同调控的复杂过程。已有研究表明,成纤维细胞生长因子基因家族(Fibroblast growth factor,FGF)的一些成员是调控哺乳动物耳朵发育的关键信号[4-6]。例如,FGF10通过调控Wnt、BMP信号通路,参与到小鼠内耳的半规管、前庭和耳蜗发育[7]。另外,同源框基因家族(Homeobox,HOX)的一些基因也在哺乳动物耳朵发育的过程中发挥重要作用。Kaur等[8]利用转基因小鼠模型发现,利用肌动蛋白的启动子特异性干扰HOX-2.2基因表达,会导致小鼠耳朵发育为畸形的小耳。在畜禽上,Qiao等[9]在二花脸和沙子岭猪的杂交后代中,发现HOXA1基因的突变(c.451delinsT>C)与猪的外耳畸形显著地关联。最近,Wei等[10]通过全基因组分析揭示出MSRB3基因与绵羊耳朵大小相关;Kumar等[11]利用SNP芯片在7个巴基斯坦山羊品种中,也发现MSRB3基因与山羊耳朵大小相关联。但总体而言,目前对山羊耳型变异的遗传机制研究仍较少。
藏山羊养殖是藏区畜牧业的重要组成部分;依据生态类型,藏山羊可分为高原型与山谷型[12]。传统上,由于地理和交通原因,不同地区藏山羊群体间基因交流并不频繁。Li等[13]利用26个微卫星标记对12个藏山羊群体进行遗传研究,发现来自不同生态区的群体间存在明显的遗传分化。近年来,在中国山羊遗传改良工作中,通过引入其他地区的优良品种与地方品种杂交,取得了较好的效果[14-15]。在藏山羊遗传改良工作中,其他山羊品种被引入藏区[16-17],导致一些地区的藏山羊群体遗传结构进一步发生变化。上述研究表明,青藏高原地区的藏山羊群体结构值得深入研究。
1 材料与方法
1.1 试验材料
在西藏自治区仲巴县帕江乡某羊场分别收集来自同一群体的正常耳型(n=30)和小耳型(n=30)藏山羊的耳长、耳宽等表型数据。用φ=75%的酒精对羊耳进行消毒处理后,使用耳号钳采集耳组织样品,用于基因组DNA的提取与分析。
1.2 DNA提取与高通量测序
分别从两种耳型(正常耳组和小耳组)藏山羊中,各选择20个个体的耳朵样本,利用DNA提取试剂盒(TIANamp Genomic DNA Kit)提取山羊基因组DNA;质量检测合格后,分别将正常耳和小耳组不同个体的DNA样品等量混合,形成2个DNA池。将混合后的DNA样品送至北京诺禾致源科技股份有限公司;构建高通量测序文库,并采用Illumina HiSeq X10测序仪进行双末端 (2×150)测序。
1.3 原始读段质量控制与比对
首先,过滤掉原始数据中包含接头的读段,然后使用Trimmomatic[18](v0.36)过滤低质量读段(参数:LEADING:20,TRAILING:20,SLIDINGWINDOW:4:20,MINLEN:50)。使用BWA[19](v0.7.12)将高质量读段比对到山羊参考基因组[20](assembly ARS1,https://asia.ensembl.org/index.htm)。使用Picard(v2.10.6,http://broadinstitute.github.io/picard/)去除重复读段,然后使用GATK[21](v3.8-0)进行局部重比对和碱基质量值校正。
1.4 SNPs的检测与质量控制
为了更准确地分析藏山羊群体特征,本研究还使用前期获得的5个山羊群体的测序数据[22]。应用Varscan[23](v2.4.3)检测波尔山羊(BE)、美姑山羊(MG)、金堂山羊(JT)、南江黄羊(NJ)、藏山羊(措勤,ZZ)以及正常耳组的藏山羊(NE)、小耳组藏山羊(SE)共7个混池样品的SNPs(参数:mpileup2snp-min-coverage 15-min-reads2 2-min-avg-qual 30-min-var-freq 0.01);然后对原始SNPs进行过滤,只保留最小等位基因频率(MAF)> 0.05和缺失基因型个体数量≤ 1的SNPs。另外,从7个群体的SNPs数据集里提取并合并正常耳和小耳组的SNPs数据,重新计算等位基因频率,过滤掉MAF<0.05的位点,得到仲巴县藏山羊群体的SNPs,最后使用SnpEff[24]对SNPs进行注释。
1.5 全基因组选择信号的检测与基因富集分析
参考已有研究[22],采用滑动窗口的方法(窗口长度为100 kb,步长为25 kb),在全基因组范围内,计算正常耳组和小耳组藏山羊之间的群体分化指数(FST)以及期望杂合度比值(HPratio)。将分布在前5%窗口的FST值和HP比值对应窗口的交集区域定义为受选择区域。根据注释结果,与受选择区域重合的基因被确定为受选择 基因。
利用Enrichr[25]对筛选出的受选择基因进行MGI哺乳动物表型富集分析,筛选与哺乳动物耳朵发育相关的条目(P<0.05)。最后,计算相关选择区域内SNPs位点的等位基因频率差异(△AF=|AFSE-AFNE|,AFSE和AFNE分别是小耳组和正常耳组的突变型等位基因频率);参考已有研究[26],选择△AF>0.8的SNPs位点进一步鉴定影响藏山羊耳型的遗传变异。
1.6 主成分分析
使用GCTA[27](v1.26.0)进行主成分分析,探讨本研究中7个山羊品种的遗传组成特点。
2 结果与分析
2.1 耳长和耳宽表型值特征
由图1-B所示,正常耳组藏山羊的耳朵长度、宽度均值分别为12.3 cm和6.4 cm;小耳组藏山羊耳朵长度、宽度均值分别为5.1 cm和4.0 cm。t检验结果表明,正常耳组的耳长、耳宽均极显著高于小耳组(P<0.01)。
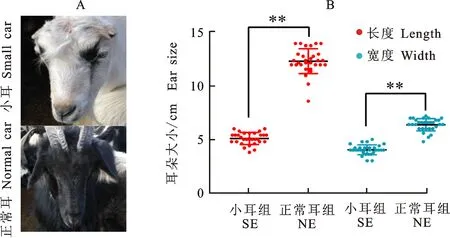
A. 藏山羊小耳与正常耳 Tibetan goats with normal and small ear size;B. 两种耳型的耳长与耳宽的比较 Comparison of the ear length and width between the two types of ear;红点和蓝点分别代表耳长和耳宽 Red and blue points represent the ear length and width,respectively;SE和NE分别为小耳组和正常耳组 SE and NE represent the small ear group and normal ear group,respectively.“**”表示t检验差异极显著(P<0.01) The “**” indicate significant difference underttest(P<0.01)
图1 两种耳型藏山羊耳长与耳宽的比较
Fig.1 Comparison of ear length and width between Tibetan goats andother two different ear types
2.2 读段比对和SNPs检测
基于正常耳组藏山羊构建的测序文库共包含421 120 994条高质量读段,测序深度约为22.1×;基于小耳组藏山羊构建的测序文库共包含345 842 498条高质量读段,测序深度约为21.2×。序列比对结果显示,正常耳组和小耳组分别有414 280 705和340 355 762条高质量读段比对到山羊参考基因组,比对率分别为98.38%和 98.41%。
但是不同于祖母所代表的墨西哥传统女性,赛利亚意识到依靠男性并不能真正使自己摆脱困境,并鼓励女性实现自身价值,这是她不同于墨西哥传统女性的女性觉醒。
基于混池测序数据,首先在7个山羊群体中共检测到6 959 360个高质量SNPs。进一步在仲巴藏山羊群体中检测到3 662 933个SNPs(表1),SNPs的平均密度为2.82个/kb。

表1 仲巴县藏山羊29条常染色体的SNPs数量分布及密度Table 1 Distribution and density of SNPs across 29 autosomes in Tibetan goats from Zhongbacounty
2.3 选择信号的鉴定与受选择基因的富集分析
如图 2 所示,依据5%的阈值,共有4 899 个窗口的FST值大于0.12,其中2号染色体的 56.475~56.575 Mb区域在两种耳型藏山羊群体间分化程度最高(FST=0.36)。基因注释结果显示,与该窗口重合的基因包括STAT1和GLS。HP比值的计算结果显示,共有4 918 个窗口的HP比值大于1.21(前5%的阈值),最大值为 5.26。结合FST与HP比值,最终共有894 个窗口被确定为受选择区域。根据基因注释结果,在受选择区域内总共鉴定到 372 个受选择基因;其中,25% 的基因(93 个)位于7号染色体,并且在这93 个基因中,56%的基因(52 个)位于45.05~59.76 Mb区域内。

图2 两种耳型藏山羊全基因组的选择信号分布Fig.2 Genome-wide distributions of selection signals betweenTibetan goats andother two different ear types
如图3所示,通过对 372 个受选择基因进行富集分析,共鉴定到10 个可能与哺乳动物耳朵发育相关的表型条目(P<0.05),具体包括:破骨细胞数量减少(MC4R、SPARC、CAMK4、RGS10、CCR2);普通髓系祖细胞形态异常 (TSTA3、KMT2A、STAT1、TNFRSF9、TYK2);背根神经节形态异常(UCHL1、KMT2A、TCOF1、NDN、GDAP1);皮肤易脱落(ERRFI1、SPINK5、CYP7A1);小型骨形态异常(FAM210A、ENAM、SLC4A4、PPARD);中线腭架融合(TCOF1、PDGFC、GLI2);骨强度降低(FAM210A、BBX、PPARD);成骨细胞分化受损(SATB2、PPARD);颞骨形态异常(TCOF1、SATB2);额骨形态异常(TCOF1、SATB2、GLI2)。

图3 与哺乳动物耳朵发育相关的表型条目Fig.3 MGI-MP terms related to ear development in mammals
2.4 选择信号区域内SNPs位点的等位基因频率差异
如图4所示,在全基因组FST值最大的滑动窗口(chr2:56.475~56.575 Mb)内共有77个SNPs位点,其中30 个的等位基因频率在两种耳型藏山羊之间差异较大(△AF>0.5),其中1个位于GLS基因内含子的SNP位点(chr2:56559507,C>T)的△AF高达0.87。
对于7 号染色体,共有1 884 个SNPs 位点的△AF>0.5,10 个SNPs位点的△AF>0.8,其中7 个均在45.05~59.76 Mb区域内;该区域内△AF的峰值位于SPINK5基因上游约5 kb处,对应SNPs位点(c.-4283A>G、c.-4295G>A)的△AF分别为0.9和0.83(图5)。

图4 2号染色体56.475~56.575 Mb区域内SNPs位点的等位基因频率差异Fig.4 Allele frequency differences of SNPs in the region of 56.475-56.575 Mb on chromosome 2

图5 7号染色体SNPs位点的等位基因频率差异Fig.5 Allele frequency differences of SNPs onchromosome 7
2.5 主成分分析
主成分分析的结果显示(图6),第一和第二特征向量分别解释25.95%和19.04%的遗传变异,7个山羊群体聚集成4个簇。其中,来自仲巴县的正常耳(NE)组和小耳(SE)组藏山羊聚在一起;来源于四川盆地的金堂黑山羊(JT)、南江黄羊(NJ)、美姑山羊(MG)聚类为一簇;而波尔山羊(BE)和来自西藏措勤县的藏山羊(ZZ)均未与其他群体聚类到一起。

图6 7个山羊群体的主成分分析Fig.6 Principal component analysis of seven goat populations
3 讨 论
耳型是现代家畜品种的一个重要表型性状;家畜耳型的特征主要表现在耳朵大小、形状和直立性三方面。尽管有关山羊耳型变异的研究已有报道[3,11];但总体来说,人们对山羊耳型变异的分子机制仍知之甚少。本研究基于全基因组SNPs信息,利用比较基因组学的分析方法,首先发现,2号染色体的56.475~56.575 Mb基因组区域在两种耳型藏山羊之间遗传分化程度最高,并且多个位点表现出较高的等位基因频率差异。虽然GLS基因与该区域重合部分最多,但目前未见GLS基因与耳朵发育有直接或间接关联的报道。
值得注意的是,在染色体水平上,本研究鉴定到的受选择基因在7号染色体上分布最多,且主要集中在45.05~59.76 Mb区域。而Brito等[3]利用努比亚山羊、波尔山羊、萨能山羊等长耳、中等耳长的山羊品种作为对照,鉴定到7 号染色体的48.5~57.3 Mb区域是影响拉曼查山羊小耳的一个基因组区域,这与本研究结果较一致。已有研究表明,该区域内存在多个与哺乳动物耳朵发育相关的基因;例如,SPINK5基因通过调控皮肤表皮蛋白分解速度参与皮肤形成过程[28]。而ANXA6基因通过调控ERK-MAPK信号通路的活性来调节终末软骨细胞分化[29]。Xiang等[30]利用小鼠模型发现,敲除POU4F3基因将导致内耳毛细胞的退化。已有研究表明,FGF10基因调控小鼠内耳发育[7],而NDST1基因在FGF信号转导过程中发挥作用[31]。因此,推测NDST1基因间接参与小鼠内耳的形成。简言之,基于拉曼查山羊与本研究中小耳型藏山羊相似的小耳表型,该基因组区域可以被认为是影响山羊耳型变异的遗传位点,但是具体的因果突变仍需进一步研究。另外,在猪和绵羊上报道的与耳型变异相关的基因HOXA1[9]、MSRB3[10]在本研究中并未被鉴定到,表明在哺乳动物中存在多种耳型变异机制。
虽然生活在青藏高原及其毗邻地区的固有山羊品种均被称为藏山羊[32],但青藏高原幅员辽阔,不同地区的藏山羊群体在体型、毛色、角型等性状上表现出显著的区别。基于微卫星标记的研究表明,藏山羊具有丰富的遗传多样性,但来自不同生态区的藏山羊群体间存在明显的遗传分化[13,33]。利用外显子组测序技术,Song等[34]比较了2个西藏绒山羊品种和7个低海拔绒山羊品种的群体分化指数,结果表明,西藏绒山羊与低海拔绒山羊之间的遗传分化程度更高。最近,Deng等[35]通过对线粒体D-loop区的分析,发现拉萨、曲水、萨嘎等10个地区的藏山羊群体间遗传分化明显。在本研究中,仲巴藏山羊与金堂黑山羊、南江黄羊等低海拔品种的遗传距离更近,与措勤地区的藏山羊较远,表明上述两个地区藏山羊群体的遗传组成差异较大。

