阿拉伯岩芥AP2基因家族的生物信息学分析
付春 刘晓伟 王玲

摘要 AP2基因作为一种调控植物花发育的功能基因,参与花分生特性建立。利用生物信息学方法对阿拉伯岩芥AP2蛋白的理化性质、高级结构和系统进化关系等进行了详细的预测和分析。结果表明,除了成员AA40G00375、AA54G00162为稳定蛋白,其余AP2蛋白均为不稳定蛋白,且大多数蛋白定位在细胞核。只有成员AA8G00350属于分泌蛋白,其余均为非分泌蛋白,且均不属于跨膜蛋白。磷酸化位点分析表明AP2蛋白的生物功能主要是通过在丝氨酸(Ser)位点进行磷酸化来实现的。AP2蛋白的二级结构以无规则卷曲为主,各个成员的二级结构元件组成含量大小排列顺序为无规则卷曲>α-螺旋>延伸链>β-转角。其中14个AP2蛋白成员有2个保守结构域,其余均有1个保守结构域。系统进化树表明阿拉伯岩芥AP2基因家族与拟南芥、小麦有较近的亲缘关系。这些结果可为进一步研究其生物学功能提供一定的参考依据。
关键词 阿拉伯岩芥;AP2基因;生物信息学
Abstract AP2 gene,as a functional gene regulating plant flower development,is involved in the establishment of floral meristems.The physicochemical properties,advanced structure and phylogenetic relationship of AP2 proteins from Aethionema arabicum were predicted and analyzed in detail by bioinformatics.The study shows that apart from the stable proteins AA40G00375 and AA54G00162,the other AP2 proteins are unstable,and most of them were located in the nuclear.Only member AA8G00350 belongs to secretory proteins,the rest are nonsecretory proteins,and they are not transmembrane proteins.Analysis of phosphorylation sites shows that the biological function of AP2 protein is mainly achieved by phosphorylation of serine (Ser) site.The secondary structure of AP2 protein is dominated by random coil,and the content of secondary structural elements of each member is presented in the order of random coil > alpha helix > extended strand > beta turn.Fourteen members of AP2 protein have two conserved domains,and the rest have one conserved domain.Phylogenetic tree shows that AP2 gene family of A.arabicum is closely related to that of Arabidopsis thaliana,Triticum urartu.These results provides some references for further study of its biological function.
Key words Aethionema arabicum;AP2 gene;Bioinformatics
阿拉伯岩芥(Aethionema arabicum)是十字花科巖芥菜属半灌木植物,短周期,快速开花的一年生植物,较适合生长在砂质土壤中,喜热植物[1-2]。阿拉伯岩芥是在4—6月完成其生命周期,在陡峭的上坡上缺少植被覆盖,阿拉伯岩芥可以承受强紫外线的照射,在炎热的夏季,寒冷的冬季它都可以比其他植物表现更好,具有耐旱和耐寒的特点[3-4]。果实异形性被定义为同一花序上产生的果实和种子的大小、颜色、形状、休眠与萌发都不同[5]。短周期和异态表型可能是其适应不可测的本地环境的增长趋势[5]。GLS是十字花科植物中防御病原体和食草动物的一套新的代谢产物,除了它们的防御功能,GLS也可以作为引诱剂,通常用于芥末味和抗癌基因活性研究,对于增加十字花科植物的多样性有一定的价值[6]。
在植物的体内存在大量的转录因子[7],转录因子(transcription factor)指能够与基因启动子区的顺式作用元件(Cis-acting element)相互作用,进而激活或抑制目标基因转录的一类蛋白,也被称为反式作用因子[8]。转录因子不仅在植物的生长发育、形态建成等方面发挥着重要作用,而且在植物抗逆性及次生代谢等方面也起着重要的调控作用。转录因子根据其DNA结构域的不同,被划分为bZIP、MYB、AP2/EREBP、WRKY和NAC等几个大家族[9-10]。作为植物最大转录因子家族之一,AP2/EREBP转录因子广泛存在于植物中。AP2/EREBP家族又被分为5个亚族:AP2、ERF、RAV、CBF/DREB和Soloist [11-12]。AP2转录因子家族成员至少含有一段60个左右氨基酸构成的AP2保守结构域[13-14],除了在拟南芥、水稻、玉米和番茄等植物中,还在蓝藻、线虫和病毒中也发现了具有AP2结构域和位点特异核酸内切酶的蛋白。所以有人认为当今植物中的AP2基因起源于细菌或者病毒基因的横向转移,AP2结构域可能来自后来进化为叶绿体的原始蓝细菌的内共生[15]。AP2转录因子可通过响应乙烯、赤霉素、脱落酸、细胞分裂素和生长素的调节[16-24],直接或间接参与种子发育过程、花和果实等器官的形态建成等植物发育的多个进程[25-26];除了初生代谢,AP2转录因子还在植物次生代谢尤其是在调控药用植物主要药用活性成分(如青蒿素、紫杉醇和木质素)合成方面效果显著[27]。同时,有报道称拟南芥AP2基因具有正向调节抗灰霉病的功能,AP2基因在植物应对高盐、干旱、缺氧、低温等非生物胁迫方面具重要功能,为植物的耐受性研究提供新契机[28-37]。
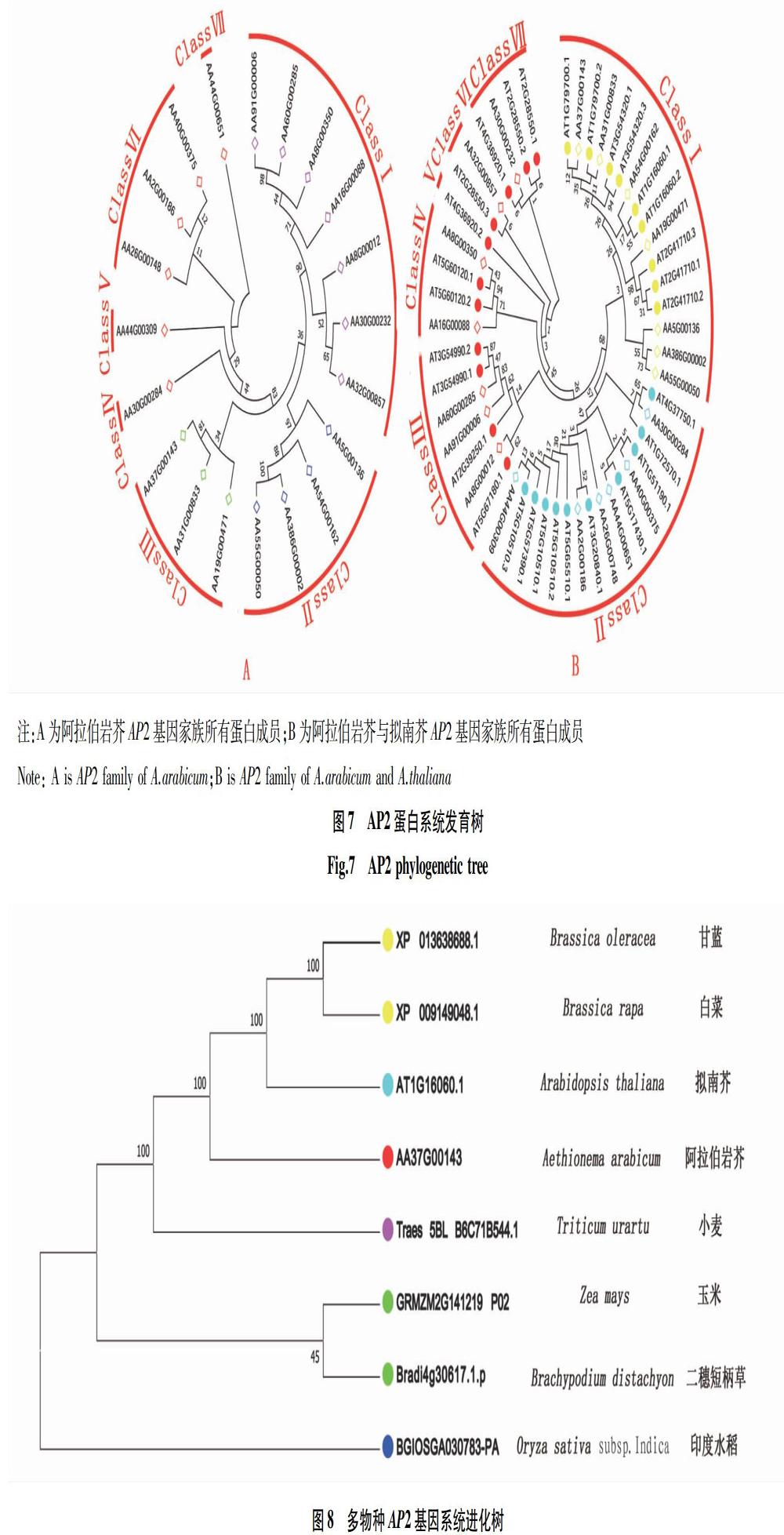
2.8 阿拉伯岩芥AP2基因系统进化树构建
对阿拉伯芥AP2基因家族的所有蛋白成员系统发育进化关系分析表明,阿拉伯岩芥AP2蛋白家族分为7组,组 Ⅰ 有7个成员,成员数最多,组 Ⅱ 有4个成员,组Ⅲ和组Ⅵ有3个成员,其余组都只有1个成员。第Ⅶ组成员AA44G00651是最原始的一支,第 Ⅰ 组成员AA91G00006和AA60G00285是距离原始亲缘最远的一支,其进化速度最快(图7A)。
阿拉伯岩芥与拟南芥AP2蛋白的系统进化分析表明,阿拉伯岩芥与拟南芥亲缘关系很近。整体共分为7个组,组 Ⅰ 共16个基因成员,阿拉伯岩芥成员有7个,拟南芥成员有9个,其中拟南芥成员AT1G79700.1是进化速度最快的1个;阿拉伯岩芥成员AA37G00143与其处在同一地位,亲缘关系最近。组 Ⅱ 共有16个成员,阿拉伯岩芥成员有6个,拟南芥成员有10个,其中成员AT4G37750.1与成员AA30G00284、成员AA40G00375与成员AT1G51190.1、成员AA44G00651和AT5G17430.1、成员AA2G00186和AT3G20840.1、成员AA44G00309和AT5G10510.3有较近的亲缘关系,同为姊妹关系。组Ⅲ共有7个成员,阿拉伯岩芥成员有3个,拟南芥成员有4个,其中只有成员AA8G00012和AT5G67180.1的亲缘关系最近,可能为亲缘关系较近的一类蛋白。组Ⅳ共有4个成员,阿拉伯岩芥成员数量和拟南芥成员数量相等,都是2个;组Ⅴ只有1个拟南芥成员;组Ⅵ共有2个成员,阿拉伯岩芥和拟南芥成员各1个;组Ⅶ共有4个成员,阿拉伯岩芥成员只有1個,其余均为拟南芥成员,其中拟南芥成员AT2G28550.1是最原始的一支(图7B)。
阿拉伯岩芥AP2基因家族成员AA37G00143与拟南芥等多个物种AP2成员分子进化关系分析表明,阿拉伯岩芥与其他成员都有较高的亲缘关系。其中阿拉伯岩芥成员AA37G00143与拟南芥成员AT1G16060.1和小麦成员Traes-5BL-B6C71B544.1的亲缘关系最近,与印度水稻的亲缘关系最远。从十字花科分类的角度分析,阿拉伯岩芥与拟南芥亲缘关系最近,可能与其同属于十字花科有关,但是与甘蓝的亲缘关系最远;从双子叶植物与单子叶植物分类分析,阿拉伯岩芥亦是与拟南芥和小麦亲缘关系最近,然而与印度水稻的亲缘关系最远,可能与它们分别属于单双子叶植物有关(图8)。
3 讨论与结论
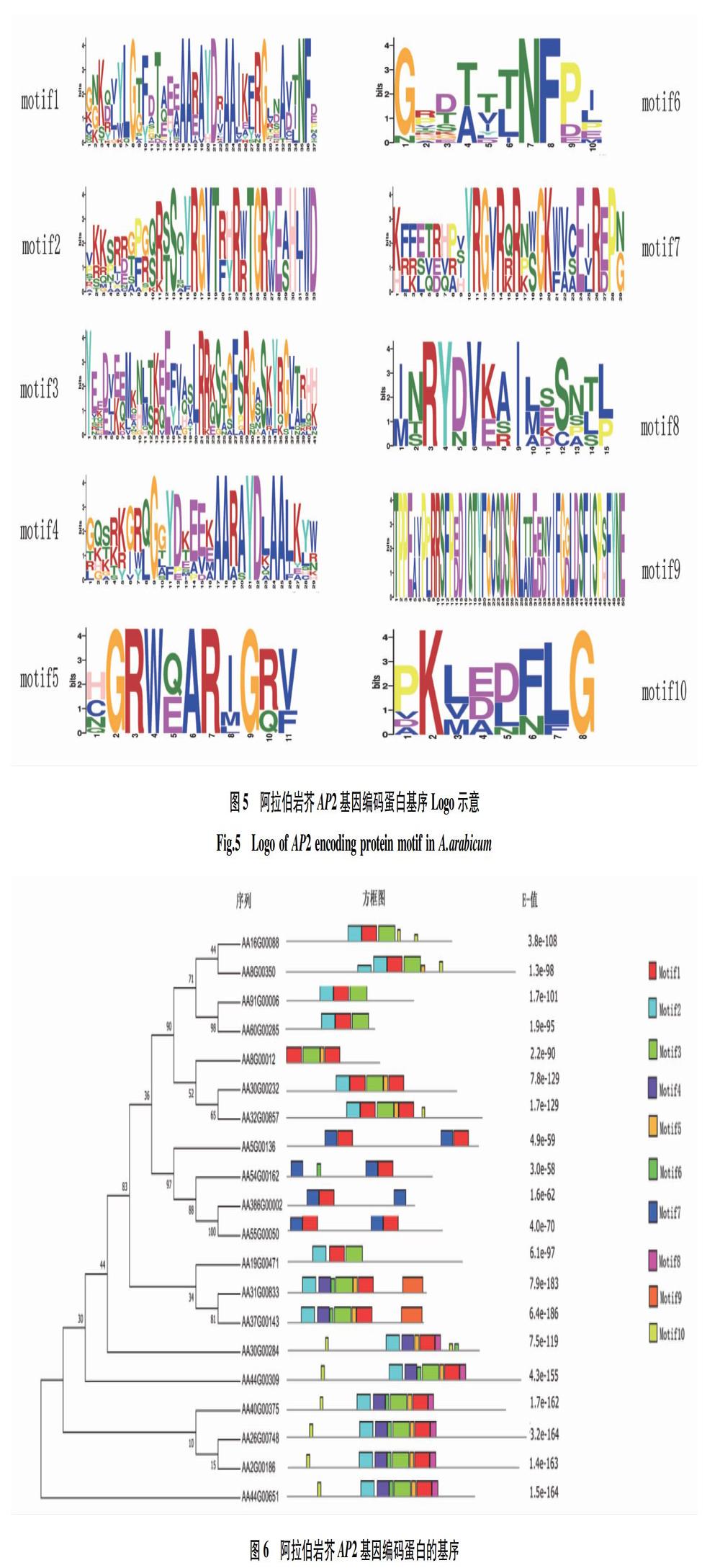
郭慧等[8]对甘蓝AP2/ERF转录因子的研究分析表明甘蓝AP2蛋白质结构为不稳定性蛋白。该研究中阿拉伯岩芥AP2基因编码蛋白之间存在差异性,且为不稳定蛋白,这与郭慧等[8]对甘蓝AP2蛋白结构研究结果相同。谢藤等[72]对新疆紫草AP2/ERF转录因子进行生物信息学分析表明其亚细胞定位存在于细胞核,不存在跨膜域,不属于分泌蛋白。该研究表明阿拉伯岩芥AP2蛋白亚细胞主要存在于细胞核,跨膜域分析显示所有编码蛋白都不属于跨膜蛋白,成员AA8G00350属于分泌蛋白,跨膜域分析与谢藤等[72]的研究结果相同,但亚细胞定位分析和信号肽分析结果与其不同,不同的原因可能与不同物种蛋白的结构组成存在差异性有关。赵金玲等[73]对杨树AP2/ERF基因家族性质的研究表明杨树AP2/ERF家族蛋白二级结构都以随机卷曲为主,其二级结构特点与阿拉伯岩芥AP2基因编码蛋白的二级结构特点相似,都以无规则卷曲结构为主,且阿拉伯岩芥AP2蛋白各个成员的二级结构元件组成含量大小排列顺序都表现为无规则卷曲>α-螺旋>延伸链>β-转角,模拟生成三级结构的图像与二级结构预测的结果相一致。不同物种之间其AP2家族成员数量各有不同,例如拟南芥为30个,玉米为54个,甘蓝为43个。但是不同物种之间的AP2基因具有一定的保守性,都含有特定的AP2结构域[30,71]。该研究显示阿拉伯岩芥AP2基因编码蛋白20个成员都含有AP2保守结构域,其中14个成员有2个保守结构域,其余均只有1个保守结构域。植物为了克服生长发育中不利影响,自身会在体内形成各种应答机制,有些蛋白会在非生物胁迫下作为保护因子调控植物细胞的稳定,进而提升其抗逆性[71,74]。该研究中阿拉伯岩芥AP2蛋白整体成员表现为亲水性蛋白,这可能是利于阿拉伯岩芥增强抗逆性的因素。YRG和RAYD元件都已被证实参与了DNA或其他转录因子的结合。研究证实,AP2中的一个转录因子WXP1与苜蓿和拟南芥的抗旱性都有关[71,75-77]。基因序列对比表明阿拉伯岩芥AP2基因序列含有YRG元件和RAYD元件,推测阿拉伯岩芥抗干旱功能也与其有关。庄静[78]对白菜与拟南芥进行研究分析建构进化树表明,白菜与拟南芥有较近的亲缘关系。该研究中阿拉伯岩芥与多个物种建构系统进化树表明阿拉伯岩芥AP2基因家族与拟南芥、小麦等都有较近的亲缘关系,完善了庄静[78]的系统进化分析结果。
该研究结果表明,阿拉伯岩芥AP2基因家族编码蛋白不同成员之间存在差异,其中只有成员AA40G00375、AA54G00162为稳定蛋白,其余均为不稳定蛋白,其大多数蛋白的亚细胞定位以细胞核为主,只有成员AA8G00350属于分泌蛋白,其余为非分泌蛋白,且所有蛋白为亲水性蛋白,不属于跨膜蛋白,都含有至少1个AP2结构域,通过丝氨酸位点处进行磷酸化来实现功能。其二级结构较整齐,都以无规则卷曲为主,各个成员的二级结构元件组成含量大小排列顺序都表现为无规则卷曲>α-螺旋>延伸链>β-转角,三级结构的图像与二级结构预测的结果相一致。阿拉伯岩芥AP2基因家族与拟南芥、白菜、玉米、小麦等都有较近的亲缘关系,其中阿拉伯岩芥成员AA37G00143与拟南芥成员AT1G16060.1和小麦成员Traes_5BL_B6C71B544.1的亲缘关系最近,与印度水稻的亲缘关系最远。阿拉伯岩芥与拟南芥的亲缘关系最近,可能与其同属于十字花科有关,而与印度水稻的亲缘关系最远,可能与它们分别属于单双子叶植物有关。该研究结果为进一步研究阿拉伯岩芥AP2基因家族在其生长发育过程中的生物学功能提供了一定的参考依据。
[21] KONG X P,TIAN H Y,YU Q Q,et al.PHB3 maintains root stem cell niche identity through ROSresponsive AP2/ERF transcription factors in Arabidopsis[J].Cell reports,2018,22(5):1350-1363.
[22] GU C,GUO Z H,HAO P P,et al.Multiple regulatory roles of AP2/ERF transcription factor in angiosperm[J].Botanical studies,2017,58(1):1-8.
[23] JISHA V,DAMPANABOINA L,VADASSERY J,et al.Overexpression of an AP2/ERF type transcription factor OsEREBP1 confers biotic and abiotic stress tolerance in rice[J].PLoS One,2015,10(6):1-24.
[24] LIU Z N,KONG L J,ZHANG M,et al.Genome-wide identification,phylogeny,evolution and expression patterns of AP2/ERF genes and cytokinin response factors in Brassica rapa ssp.pekinensis[J].PLoS One,2013,8:1-15.
[25] 徐倩,殷學仁,陈昆松.基于乙烯受体下游转录因子的果实品质调控机制研究进展[J].园艺学报,2014,41(9):1913-1923.
[26] YU Y,DUAN X B,DING X D,et al.A novel AP2/ERF family transcription factor from Glycine soja,GsERF71,is a DNA binding protein that positively regulates alkaline stress tolerance in Arabidopsis[J].Plant molecular biology,2017,94(1):509-530.
[27] WUDDINEH W A,MAZAREI M,TURNER G B,et al.Identification and molecular characterization of the switchgrass AP2/ERF transcription factor superfamily,and overexpression of PvERF001 for improvement of biomass characteristics for biofuel[J].Frontiers in bioengineering and biotechnology,2015,3:1-21.
[28] KITOMI Y,ITO H,HOBO T,et al.The auxin responsive AP2/ERF transcription factor CROWN ROOTLESS5 is involved in crown root initiation in rice through the induction of OsRR1, a typeA response regulator of cytokinin signaling[J].Plant journal,2011,67(3):472-484.
[29] 闻可心,刘雪梅.AP2功能基因在植物花发育中的重要作用[J].生物技术通报,2010(2):1-7.
[30] 张计育,王庆菊,郭忠仁.植物AP2/ERF类转录因子研究进展[J].遗传,2012,34(7):835-847.
[31] 季爱加,罗红梅,徐志超,等.药用植物转录因子AP2/ERF研究与展望[J].科学通报,2015,60(14):1272-1284.
[32] DOSSA K,WEI X,LI D H,et al.Insight into the AP2/ERF transcription factor superfamily in sesame and expression profiling of DREB subfamily under drought stress[J].BMC Plant Biology,2016,16(1):1-16.
[33] LIU Q,KASUGA M,SAKUMA Y,et al.Two transcription factors,DREB1 and DREB2,with an EREBP/AP2 DNA binding domain separate two cellular signal transduction pathways in droughtand lowemperatureresponsive gene expression,respectively,in Arabidopsis[J].The plant cell,1998,10(8):1391-1406.
[34] XU Z S,ChEN M,LI L C,et al.Functions and application of the AP2/ERF transcription factor family in crop improvement[J].Journal of integrative plant biology,2011,53(7):570-585.
[35] SAKUMA Y,LIU Q,DUBOUZET J G,et al.DNAbinding specificity of the ERF/AP2 domain of Arabidopsis DREBs,transcription factors involved in dehydrationand coldinducible gene expression[J].Biochemical and biophysical research communications,2002,290(3):998-1009.
[36] LI H,WANG Y,WU M,et al.Genomewide identification of AP2/ERF transcription factors in cauliflower and expression profiling of the ERF family under salt and drought stresses[J].Frontiers in plant science,2017,8:946.
[37] MAWLONG I,ALI K,SRINIVASAN R,et al.Functional validation of a droughtresponsive AP2/ERF family transcription factorencoding gene from rice in Arabidopsis[J].Molecular breeding,2015,35(8):1-14.
[38] PARK C S,GO Y S,SUH M C.Cuticular wax biosynthesis is positively regulated by WRINKLED4,an AP2/ERF-type transcription factor,in Arabidopsis stems[J].The plant journal,2016,88(2):257-270.
[39] THAMILARASAN S K,PARK J I,JUNG H J,et al.Genomewide analysis of the distribution of AP2/ERF transcription factors reveals duplication and CBFs genes elucidate their potential function in Brassica oleracea[J].BMC Genomics,2014,15(1):1-13.
[40] WANG X H,HAN H Y,YAN J,et al.A new AP2/ERF transcription factor from the oil plant Jatropha curcas confers salt and drought tolerance to transgenic tobacco[J].Applied biochemistry and biotechnology,2015,176(2):582-597.
[41] YANG Y Q,DONG C,LI X,et al.A novel AP2/ERF transcription factor from Stipa purpurea leads to enhanced drought tolerance in Arabidopsis thaliana[J].Plant cell reports,2016,35(11):2227-2239.
[42] 周媛媛,董曉静,吕娅,等.拟南芥分子生物学研究进展[J].中国农学通报,2018,34(30):56-62.
[43] 綦洋,王柬钧,桑园园,等.大白菜YUCCA基因家族的鉴定与生物信息学分析[J].江苏农业科学,2019,47(3):49-54.
[44] 袁明月,楼轶,张竞,等.禾谷镰刀菌AP蛋白家族的生物信息学分析[J/OL].基因组学与应用生物学,2019-03-01[2019-08-16].http:kns.cnki.net/kcms/detail/45.1369.Q.20190301.0942.002.html.
[45] 张书芹,乐愉,武斐.陆地棉GRF基因家族的鉴定和生物信息学分析[J].分子植物育种,2019,17(12):3817-3824.
[46] 白羽聪,李翔宇,程占超,等.毛竹PIN基因家族的鉴定与生物信息学分析[J].分子植物育种,2019,17(16):5238-5247.
[47] 逄洪波,潘英杰,李嘉琦,等.向日葵Hsp70基因家族成员的全基因组鉴定与生物信息学分析[J].分子植物育种,2019,17(18):5918-5929.
[48] 郭栋,杜媚,周宝元,等.玉米CCT基因家族的鉴定与生物信息学分析[J].植物遗传资源学报,2019,20(4):1001-1010.
[49] 狄建军,孙佳欣,杨智慧,等.蓖麻PLA2α基因启动子的克隆及生物信息学分析[J].分子植物育种,2019,17(15):4961-4966.
[50] 何焱,李忠玥,李卿,等.藏药翼首草角鲨烯合成酶的生物信息学分析[J/OL].基因组学与应用生物学,2019-01-14[2019-08-16].http:∥kns.cnki.net/kcms/detail/45.1369.q.20190111.1501.002.html.
[51] 赵思华,尹晗,吕昊哲,等.大豆GmTAA1-1基因的生物信息及时空表达分析[J].分子植物育种,2019,17(21):6946-6952.
[52] 吴桂梅,赵小峰,汪瑶,等.大鼠单核细胞趋化蛋白-1基因的生物信息学分析[J].安徽农业科学,2019,47(1):93-95.
[53] 张西燕,付玉荣,伊正君.结核分枝杆菌PE_PGRS47蛋白的生物信息学分析[J].中国病原生物学杂志,2019,14(1):44-49.
[54] 徐小钦,杜雪,李国勤,等.白羽王鸽卵清蛋白关联蛋白Y基因(OVALY)克隆与生物信息学分析[J].浙江农业学报,2019,31(5):704-708.
[55] 冯立娟,尹燕雷,杨雪梅,等石榴查尔酮合成酶蛋白的生物信息学分析[J].山东农业科学,2019,51(2):7-12.
[56] 宋雪,秦彩艳,霍金龙,等.版纳微型猪近交系CPN10克隆、亚细胞定位、组织表达和生物信息学分析[J].云南农业大学学报,2019,34(2):255-262.
[57] 姜铮,王芳,何湘,等.蛋白质磷酸化修饰的研究进展[J].生物技术通讯,2009,20(2):233-237.
[58] 李杰,杭柏林,宁春妹,等.牛源cathelicidin类抗菌肽的生物信息学分析[J].河南科技学院学报(自然科学版),2019,47(1):40-45.
[59] 龙芳,李绍鹏,李茂富.7种植物ALAD基因的生物信息学分析[J].基因组学与应用生物学,2013,32(6):802-814.
[60] 韩长志.禾谷炭疽菌RGS蛋白生物信息学分析[J].微生物学通报,2014,41(8):1582-1594.
[61] 宋志忠,杨顺瑛,金曼,等.杨树基因组AMT转运蛋白的生物信息学特性(英文)[J].基因组学与应用生物学,2011,30(6):641-648.
[62] 郭溆,罗红梅,陈士林.灵芝GLUGT1基因的全长cDNA克隆和生物信息学分析[C]∥中药与天然药高峰论坛暨全国中药和天然药物学术研讨会论文集.北京:中国药学会,2012.
[63] 刘金鸽.擬南芥AtDPBF4基因克隆及生物信息学分析与表达研究[D].兰州:兰州理工大学,2013.
[64] 张加强,刘慧春,周江华,等.植物赤霉素氧化酶GA20ox基因的生物信息学分析[J].分子植物育种,2019,17(15):4986-5002.
[65] 皮博艺,阮颖,黄勇.植物串联CCCH 锌指蛋白RR-TZF家族研究进展及生物信息学分析[J].分子植物育种,2019,17(7):2171-2177.
[66] 唐跃辉,包欣欣,王健,等.小桐子Dof基因家族生物信息学与表达分析[J].江苏农业学报,2019,35(1):15-25.
[67] 王雁,王小贝,陈海江,等.桃GLV多肽受体PpRGI基因家族的生物信息学及表达分析[J].园艺学报,2019,46(5):855-863.
[68] 袁梦如,杨杨,张磊,等.沙柳SpsTAC2基因克隆、生物信息学及组织特异表达分析[J].分子植物育种,2019,17(7):2144-2151
[69] 王丽珊.拟南芥和水稻Cel基因家族的生物信息学分析[J].闽西职业技术学院学报,2019,21(1):101-106.
[70] 郭娟,彭秋菊,佘明明,等.辣椒生育酚环化酶(TC)基因克隆和生物信息学分析[J].分子植物育种,2019,17(10):3171-3179.
[71] 阮先乐,张杰,张福丽,等.甘薯AP2基因家族的生物信息学分析[J].分子植物育种,2017,15(6):2066-2072.
[72] 谢腾,王升,周良云,等.新疆紫草AP2/ERF转录因子的电子克隆和生物信息学分析[J].中国中药杂志,2014,39(12):2251-2257.
[73] 赵金玲,姚文静,王升级,等.杨树AP2/ERF转录因子家族生物信息学分析[J].东北林业大学学报,2015,43(10):21-29.
[74] 张丽丽,李博,谭燕华,等.玉米转录因子基因ZmASR家族的生物信息学分析[J].分子植物育种,2016,14(4):803-811.
[75] OKAMURO J K,CASTER B,VILLARROEL R,et al.The AP2 domain of APETALA2 defines a large new family of DNA binding proteins in Arabidopsis[J].Proceedings of the national academy of sciences,1997,94(13):7076-7081.
[76] ZHANG J Y,BROECKLING C D,BLANCAFLOR E B,et al.Overexpression of WXP1,a putative Medicago truncatula AP2 domaincontaining transcription factor gene,increases cuticular wax accumulation and enhances drought tolerance in transgenic alfalfa (Medicago sativa)[J].Plant journal,2005,42(5):689-707.
[77] LIN R C,PARK H J,WANG H Y.Role of Arabidopsis RAP2.4 in regulating lightand ethylenemediated developmental processes and drought stress tolerance[J].Molecular plant,2008,1(1):42-57.
[78] 庄静.大白菜和甘蓝型油菜AP2/ERF家族转录因子的克隆与分析[D].南京:南京农业大学,2009.

