基于转录组数据的青蛤微卫星标记开发与验证
方 军,沈彦鹏,张雷雷,李腾腾,邵艳卿*
(1.浙江省海洋水产养殖研究所、浙江省近岸水域生物资源开发与保护重点实验室、 中国水产科学研究院海洋贝类工程技术研究中心,浙江 温州 325005; 2.温州医科大学,浙江 温州 325016)
青蛤(Cyclinasinensis) 俗称环文蛤,隶属双壳纲、帘蛤目、帘蛤科,是我国一种重要养殖经济贝类。随着青蛤养殖方式多样化、养殖技术不断成熟,越来越多育种群体及家系被培育出来,为了避免由于常年人工养殖导致遗传结构单一,同时也为保护青蛤自然种群不被人工多代繁育的养殖群体所污染,需要通过有效的分子手段评价种质资源和遗传多样性。然而有关青蛤微卫星标记十分有限,仅见董志国等(2011)通过磁珠富集法从基因组中开发SSR标记的报道[1]。目前迫切需要开发具有共显性遗传、多态性丰富、易于规模化检测、检测成本低等优势的分子标记,以开展青蛤种群遗传多样性分析、遗传育种等研究。
在众多水生生物分子标记中,微卫星标记又称简单序列重复(Simple Sequence Repeat, SSR),由1~6个核苷酸串联重复片段构成,与其他分子标记相比,具有共显性遗传、高度多态杂合且稳定性好、易操作、检测成本低等优点,目前已广泛应用于系谱分析、遗传图谱构建、遗传多样性研究等多项领域。李炼星(2017)通过对选育的缢蛏(Sinonovaculaconstricta)后代进行微卫星分析[2],结果表明连续6代在亲本间有近亲关系或者亲本数量有限情况下,人工选育缢蛏苗种遗传多样性显示明显降低。连续累代群体选育工作使得缢蛏选育系的遗传特性纯化,可为后续选育工作中通过不同种群、家系间杂交或者增加亲本数量来提高遗传多样性和生长性状、耐病性、适应环境变化等目标性状;韩学凯等(2015)利用509个微卫星标记构建了三角帆蚌(Hyriopsiscumingii)覆盖19个连锁群[3],1 940.3 cM(cM为厘摩尔根)的遗传连锁图谱,为三角帆蚌分子标记辅助育种打下基础。可见微卫星标记在海洋贝类遗传育种方面应用之广泛。
因此,本研究在通过转录组测序获得生物学数据信息基础上,拼接组装获得大量含SSR的EST(Expressed Sequence Tag)序列,并研究这些EST-SSR在青蛤EST中的分布特征,设计并合成部分引物,对浙江灵昆青蛤群体进行遗传结构分析,以期为青蛤种质资源保护、遗传多样性评价等提供有力工具。
1 材料与方法
1.1 材料
实验用青蛤60只,于2016年5月采自浙江省温州灵昆,活体运回实验室,解剖剪取闭壳肌及斧足,保存于-80℃冰箱中备用。
1.2 方法
1.2.1 SSR序列的获得及引物设计 根据青蛤cDNA文库的IlluminaHiseq测序分析工作,利用MISA (Microsatellite Identification Tool)对基因转录本进行SSR检测,找到SSR标记后,使用Primer3软件完成SSR引物设计,参数均设为默认。随机选取81条序列的引物送上海生工公司进行合成。
1.2.2 DNA制备、PCR反应及产物检测 基因组DNA的提取参照海洋生物组织基因组DNA提取试剂盒(北京艾德莱生物科技有限公司)的方法提取。
PCR反应体系为20μL∶1 μL模板DNA (30 ng/μL),0.1 μL Taq酶(5 U/μL),2 μL 10×PCR buffer (Mg2+plus),1.6 μL dNTP Mixture (2.5 mmol/μL ),0.4 μL正反引物对(10 μmol/L),14.5 μL超纯水。PCR反应程序为:94 ℃预变性5 min后,进入30个PCR循环:94 ℃变性45 s,复性45 s,72 ℃延伸45 s;最后72 ℃下延伸7 min,并将反应产物于4 ℃下保存。用1.5%琼脂糖凝胶检测扩增产物,鉴定有扩增产物后再用8%非变性聚丙烯酰胺胶电泳检测,EB银染、Bio-Rad Universal Hood II拍照。
1.2.3 多态性检测及数据分析 根据电泳图谱,参照引物设计时预期片段的大小统计条带,采用POPGENE32软件计算青蛤微卫星引物的期望杂合度(Expected Heterozygosity,He)、观察杂合度(Observed Heterozygosity,Ho)、有效等位基因数(Effective Number of Allele,Ne)和哈代-温伯格平衡值(Hardy-Weinberg Equilibrium,WE)。应用PIC-ALC软件计算多态信息含量(Polymorphic Information Content,PIC)。
2 结果与讨论
2.1 青蛤SSR分布特点
利用MISA将去冗余后得到115 441条Unigene序列进行检测得到SSR位点12 418个,主要为单核苷酸重复、三核苷酸重复和双核苷酸重复。如图1所示,单核苷酸重复数量最多,共有8 422个,占所有微卫星位点的67.82%,在单核苷酸重复的微卫星引物中,以A/T重复的引物最多,共有4 106个,占所有单核苷酸重复的48.75%,其次是三核苷酸重复,共有1 501个,占所有微卫星位点的12.09%。而其中重复单元为(TTG/AAC)n的位点数量最多,共有171个,占三核苷酸重复的微卫星总数的11.39%。双核苷酸重复共有1 419个,占所有微卫星位点的11.43%,其中AT/TA重复的位点最多,共有721个,占双核苷酸重复微卫星的50.81%。另外还有部分四核苷酸、五核苷酸、六核苷酸重复以及混合型重复共1 076个,占所有SSR位点的8.66%。
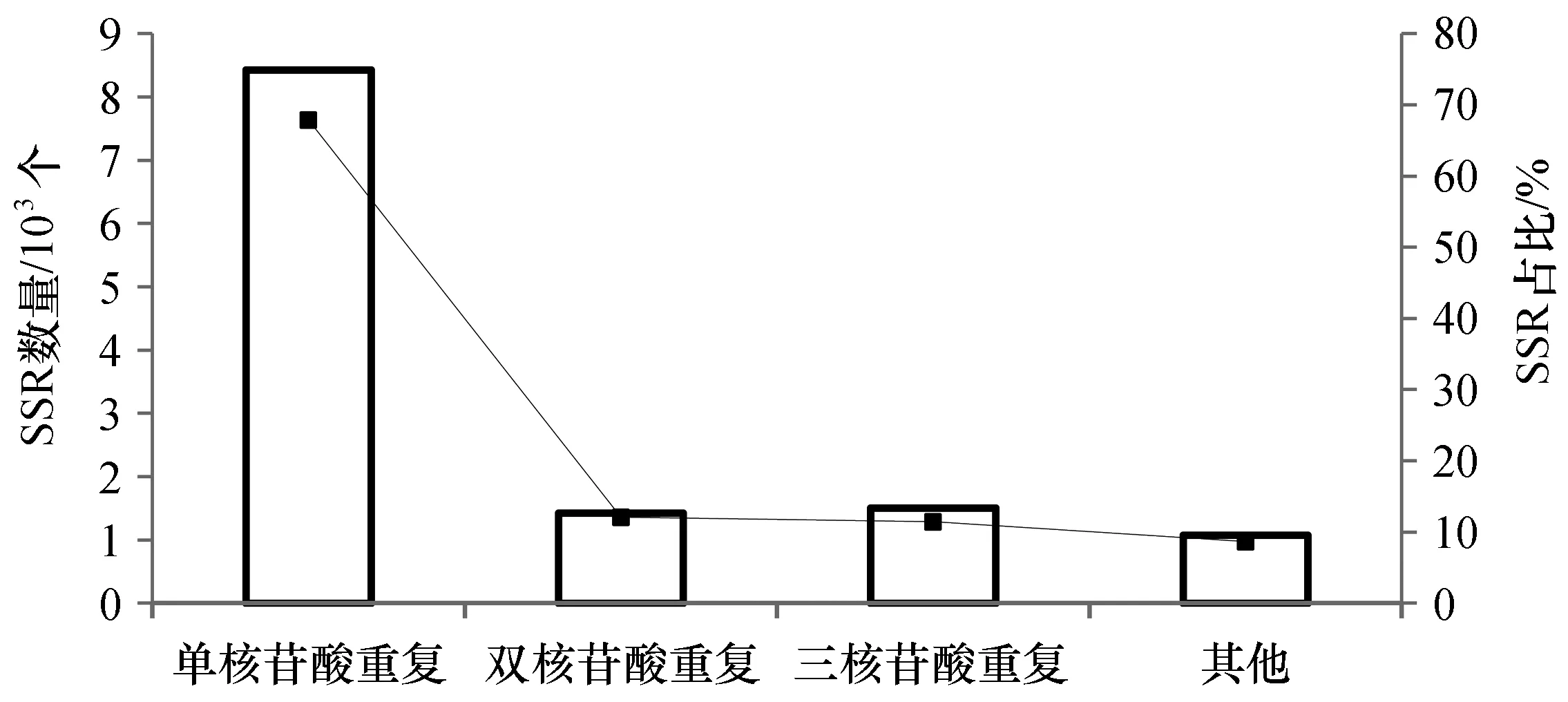
图1 青蛤 EST-SSR重复类型及所占比例Fig.1 Distribution of microsatellite sequences in the tested clam
2.2 青蛤SSR多态性引物筛选
利用Primer3软件,设计了81对SSR引物,以8个灵昆青蛤的基因组DNA为模板,进行PCR扩增,退火温度上下波动5 ℃左右为梯度,对81对EST-SSR引物进行了初步筛选。研究发现扩增出稳定的可重复条带的引物为48对,占引物总数的59.26%;通过非变性聚丙烯酰胺凝胶电泳结合EB染色检测,81对SSR引物中有27对表现出个体多态性,占引物总数的33.33%。多态性引物占有效引物的比例为56.25%。27对多态性引物中,11个双核苷酸重复,4个三核苷酸重复,3个四核苷酸重复,2个五核苷酸重复,1个六核苷酸重复,5个混合型重复。27对引物序列等详细信息如表1所示。

表1 青蛤27对微卫星引物序列及其参数Tab.1 Primer sequences and parameters of the 27 SSRs for C.sinensis
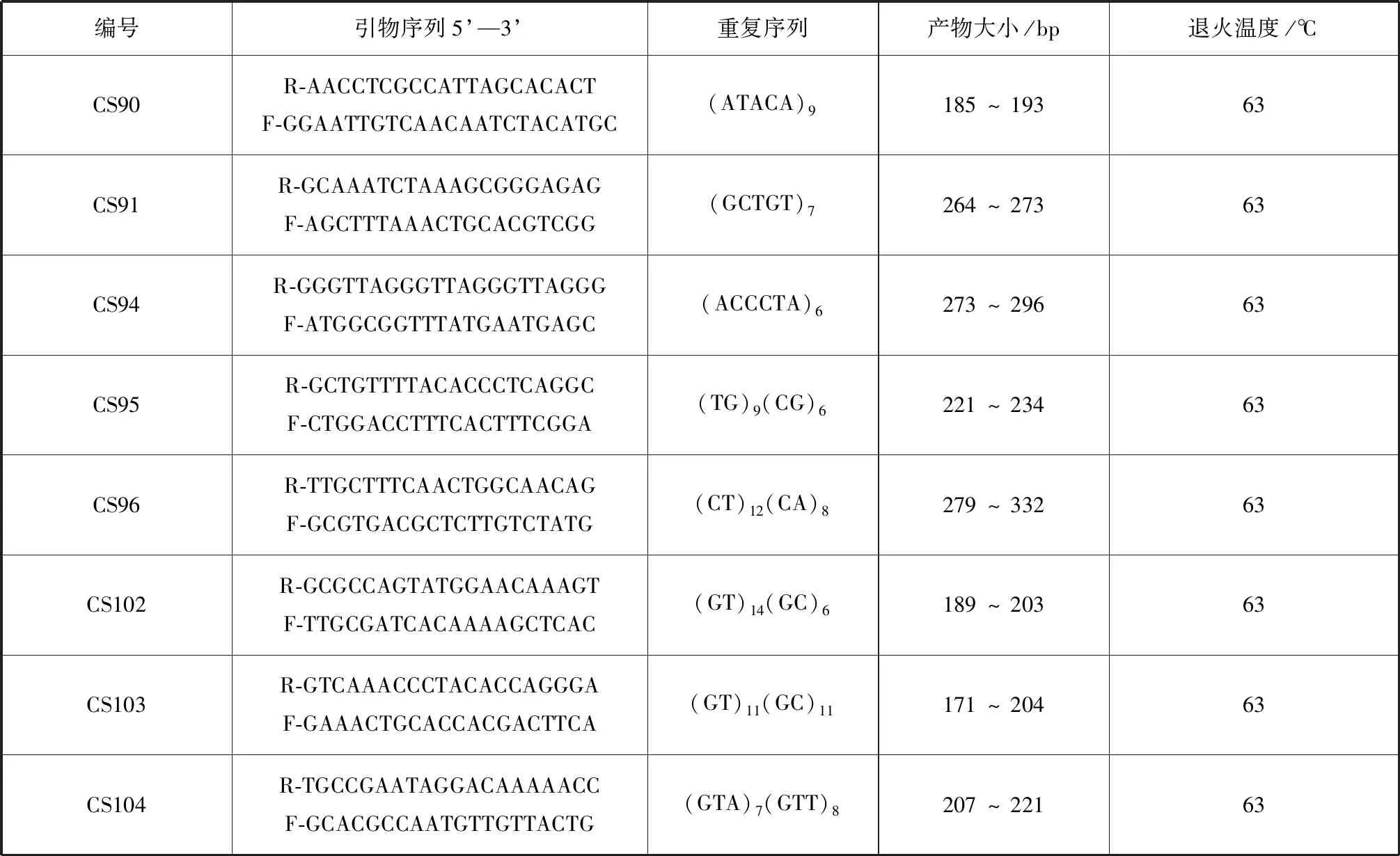
续表1
2.3 灵昆青蛤群体遗传变异分析
用筛选出的27对微卫星引物对30只灵昆青蛤群体进行多态性检测,图2为引物CS83在青蛤中的扩增检测结果。
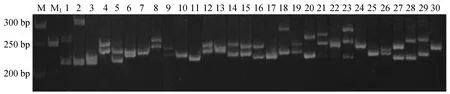
图2 引物CS83在青蛤30个个体中的扩增Fig.2 PCR results of EST-SSR primer CS83 amplified in 30 labelled clams 从左到右依次是M:50 bp DNA ladder marker,M1:DNA marker DL2000,1~30号个体样品。
30个灵昆青蛤群体中,用27对引物扩增共获得99个等位基因,每个位点产生等位基因数Na为2~6个,平均每个位点产生3.67个,其中CS78位点的等位基因数量最多(6个);有效等位基因数Ne为1.03~4.89,所有位点大小在116~330 bp之间。青蛤灵昆群体的各遗传参数见表2,观测杂合度Ho为0.000 0~0.678 6,其中Ho ̄值最高的是引物CS80,最低的是引物CS96。期望杂合度He为0.033 0~0.808 5,其中He值最高的是引物CS61,最低的是引物CS71。经过软件分析,各位点遗传多态信息含量PIC在0.032 3~ 0.764 5之间,最高的是引物CS83,最低的是引物CS71。除了CS71、CS78、CS80、CS85、CS90、CS91、CS94、CS104这8对引物之外其余均显著偏离Hardy-Weinberg平衡(p<0.01)。
2.4 讨论
目前,微卫星标记开发主要基于基因组文库和转录组文库,转录组微卫星标记EST-SSR和基因组来源的微卫星标记相比,具有种间通用性好,基因功能相关,开发成本低且周期短等优点[4]。在双壳纲贝类中,已成功开发利用的微卫星标记有李云峰等(2010)通过对虾夷扇贝(Patinopectenyessoensis)外套膜和肾脏组织构建cDNA文库进行的多态性EST-SSR筛选[5];李红蕾等(2003)使用RepeatMasker软件在栉孔扇贝(Chlamysfarreri)的6 935条ESTs中发现了42条微卫星序列,并在设计的7对引物中发现有6对能够产生扩增产物[6];史松富等(2013)利用3 000条泥蚶(Tegillarcagranosa)单拷贝序列设计50条引物,得到24个多态性SSR位点等[7]。

表2 青蛤群体中各微卫星位点分析统计结果Tab.2 Statistical results of microsatellite loci in the tested clams
2.4.1 青蛤微卫星分布 本研究通过对浙江灵昆青蛤进行转录组测序,发现在青蛤转录组序列中单核苷酸重复的微卫星数量远高于其他类型,其中A重复与T重复总量占所有微卫星数量的65.19%,这与王忠良等(2015)使用MISA软件对马氏珠母贝(Pinctadamartensi)转录组测序数据分析得到的EST-SSR主要特征相吻合[8]。在多核苷酸重复微卫星中,三核苷酸重复(12.09%)略高于双核苷酸重复(11.43%);而四核苷酸(2.29%)、五核苷酸(0.16%)、六核苷酸重复(0.03%)三者都较双核苷酸重复要少;此外还观测到部分混合型重复微卫星(6.18%)。这种三核苷酸重复出现频率较高的分布特征与泥蚶[9]以及三角帆蚌[10]类似。另外,在所有类型的微卫星中,A/T出现的频率均高于C/G,这种分布特征同样出现于红鳍东方鲀(Takifugurubripes)[11],三疣梭子蟹(Portunustrituberculatus)[12]等海洋动物的报道中,倪守胜等(2018)认为微卫星A/T含量高的原因可能是微卫星序列A/T含量高,则退火温度Tm值降低,DNA链容易解开,通过DNA重组机制和复制滑动机制,产生富含AT重复类型的机率更高[13]。也有研究表明海洋生物在甲基转移酶的催化下DNA中的GC两个核苷酸胞嘧啶C被选择性添加甲基,甲基化的胞嘧啶C很容易经过脱氨基作用突变成胸腺嘧啶T,在DNA复制及转录过程中使得G/C突变A/T,而少量的GC又是维持DNA热力学稳定性所必需,因此微卫星序列中G/C含量远远不如A/T[14]。
2.4.2 微卫星位点多态性 经过PCR扩增以及聚丙烯酰胺凝胶电泳,自81对引物中筛选出27对多态性引物,多态性引物占33.33%。观测杂合度Ho为0.000 0~0.678 6,期望杂合度He为0.033 0~0.808 5,平均观测杂合度为0.292 9;平均期望杂合度为0.585 0。其中大部分位点的观测杂合度低于期望杂合度,CS71观测杂合度等于期望杂合度,只有CS80观测杂合度高于期望杂合度。产生这个结果的可能原因之一是养殖群体经过多代人工繁育和近亲繁殖使得杂合度显著低于野生种群,这种情况普遍出现在养殖贝类群体中,如皱纹盘鲍(Haliotisdiscus)[15]、厚壳贻贝(Mytiluscoruscus)[16]等。另一个可能导致观测杂合度低于期望杂合度的原因是未扩增的PCR无效等位基因的存在,因为未扩增的杂合子被作为纯合子计算,导致该位点观测到的杂合度低于实际情况[17],这同时也可能是本实验中多数位点显著偏离Hardy-Weinberg平衡(p<0.01)的原因。另外,牛东红等(2010)认为微卫星位点多态性越高,等位基因数越多,则所需的样本量也越大,当样本量>30时,杂合度趋于稳定[18];而鲁翠云等(2008)通过对镜鲤(Cyprinuscarpio)养殖群体样本量和微卫星标记多态性水平之间的相关性分析认为当样本量>40时,杂合度变动范围变小,并出现平台现象,得到当使用微卫星进行群体遗传评估时,最适样本量为40~50个的结论[19]。本实验中所有的微卫星位点选取的样本数均为30,为牛东红等人认为的最小样本量,小于鲁翠云等人认为的最适样本量,故而多数位点显著偏离Hardy-Weinberg平衡(p<0.01)也可能是由于样本量不足所导致。
遗传多态信息含量PIC指的是一个亲本为杂合子,另一亲本为不同基因型的概率,是一个多态性遗传标记可提供信息量的度量。在本实验中,遗传多态信息含量PIC为0.032 3~ 0.764 5之间,平均遗传多态信息含量为0.515 9。Bostein等(1980)提出,当PIC>0.50时,该位点属于高度多态位点;当0.25 本实验利用青蛤的转录组数据,识别出大量微卫星位点,并成功筛选出27个青蛤多态性EST-SSR标记,其中19个为高度多态性位点。这些微卫星多态性位点的获得,为进一步开展青蛤种质资源评估、群体遗传多样性分析、遗传图谱构建等提供了新的有效分子标记。3 结论

