阿拉尔垦区棉花根系组织及根际土壤细菌多样性分析
阿依妮萨·阿卜力海提 代先兴 李猛 王 阿卜杜萨拉木·如则麦麦提 郝海婷


摘要:以阿拉尔垦区棉花为试材,采用高通量测序方法研究棉花根系组织和根际土壤的细菌群落组成及其生态功能。结果显示,棉花根际土壤细菌丰富度指数(Chao1)和多样性指数(Shannon)显著大于根系组织内生细菌丰富度指数(Chao1)和多样性指数(Shannon)。棉花根际土壤中细菌的各分类水平(门、纲、目、科、属)数量大于棉花根系组织中的数量。物种注释结果表明,根系组织和根际土壤两个微环境优势菌群明显不同。棉花根系组织和根际土壤优势菌群不一样,对生物防治菌种筛选有一定的指导作用。
关键词:阿拉尔垦区;棉花;根系组织;根际土壤;细菌多样性;高通量测序方法
中图分类号:S562. 文献标识码:A 文章编号:2095-3143(2020)03-0020-07
DOI:10.3969/j.issn.2095-3143.2020.03.003
Bacterial Diversity of Cotton Root Tissue and Rhizosphere Soil
in Alar Reclamation Area
Ayinisa·Abolihaiti, Dai Xianxing, Li Meng, Wang Zhe, Abodusalamu·Ruzemaimaiti, Hao Haiting
(College of Plant Science, Tarim University., Alar,Xinjiang 843300, China)
Abstract: The composition and ecological function of bacterial community in root tissue and rhizosphere soil were studied by high-throughput sequencing, the cotton in Araer Reclamation area was took as experimental material. The results showed that the bacterial richness index (Chao1) and diversity index (Shannon) of cotton rhizosphere soil were significantly higher than those of endophytic bacterial richness index (Chao1) and diversity index (Shannon). the number of bacteria classification levels (doors, classes, orders, families, genera) were larger than that in the root tissue of cotton. The results of species annotation showed that the dominant bacterial population of root tissue and rhizosphere soil were significantly different. The dominant bacterial population in cotton root tissue was different from rhizosphere soil, which could guide the selection of biological control bacteria.
Key words: Alar reclamation area; Cotton; Root tissue; Rhizosphere soil; Bacterial diversity; High throughput sequencing method
0 引言
新疆作为我国最大的棉花生产基地,在棉花种植面积、总产、单产、商品调拨量连续25年位居全国第一[1]。自2014年棉花目标价格改革试点工作实施以来,各棉花生产主体在品质意识方面有所增强。一些部分团场、当地轧花厂严格质量标准,在收购价格上建立优质优价的导向[2]。然而,随着近5年新疆气候的改变,尤其是降水增多和寒流提前,棉花“三病”(立枯病、枯萎病和黄萎病)[3-4]也随之加重,但还没有引起足够的重视。农药品种落后,防治被动是严重影响棉花产量和品质的主要问题[5]。
目前,利用传统的分离技术只能代表一部分可培养微生物,无法分离到棉花中所有微生物。近年来,Illumina 高通量测序技术因通量高、检测全面、准确等优点能够更加真实地揭示微生物种群结构,为获取更多种类的微生物资源提供了依据。但目前国内有关棉花根系内生菌与根际土壤微生物的菌群特征却鲜有报道,因此,作者采用Illumina Mi Seq高通量测序技术,对棉花根系组织以及根际土壤两个微环境细菌的16S r RNA基因序列进行分析,从而更好地揭示棉花根系组织以及根际土壤细菌的种类组成及菌群多样性的分布,为进一步开发植物促进生长和病害防治的生防菌资源提供依据。
1 材料和方法
1.1 采样地概括
研究地點位于新疆阿拉尔市农业科学研究所棉田,该田土质条件好,灌排通畅。该地区属于温带大陆性气候,日照充足,气候干旱少雨,昼夜温差大,阿拉尔地区年平均气温为10.5℃,年平均最高气温为18℃;年平均最低气温为3℃。
1.2 材料与取样方法
棉花品种采用新陆中37号,采样时间为7月,棉花正处于花蕾期,选取棉田健康植株,采集棉花根系及根际粘上的土壤。迅速将土样保存于带有冰袋的保温箱中,带回实验室,冻存于装有10 kg干冰的泡沫盒中,封口,邮寄给北京诺禾致源生物信息科技有限公司。
1.3 实验流程
DNA的提取、试验上机流程、原始测序数据分析等与郝海婷,等[10]人的方法相同,但稍作了修改,由北京诺禾致源生物信息科技有限公司负责。具体步骤如下。
1.3.1 DNA的提取
采用CTAB或SDS方法对样本的基因组DNA 进行提取,之后利用琼脂糖凝胶电泳检测DNA的纯度和浓度,取适量的样本DNA于离心管中,使用无菌水稀释样本至1 ng/μl。以稀释后的基因组DNA为模板,根据测序区域的选择,使用带Barcode的特异引物,即“New England Biolabs”公司的 Phusion? High-Fidelity PCR Master Mix with GC Buffer,和高效高保真酶进行PCR,确保扩增效率和准确性。
引物对应区域为16 S V4区引物(515F和806R),鉴定细菌多样性为PCR产物的混样和纯化,PCR产物使用2%浓度的琼脂糖凝胶进行电泳检测;根据PCR产物浓度进行等量混样,充分混匀后使用2%的琼脂糖凝胶电泳检测PCR产物,对目的条带使用qiagen公司提供的胶回收试剂盒回收产物。
1.3.2 文库构建和上机测序
使用TruSeq? DNA PCR-Free Sample Preparation Kit建库试剂盒进行文库构建,构建好的文库经过Qubit和Q-PCR定量,文库合格后,使用NovaSeq6000 进行上机测序。
1.3.3 原始测序数据分析
根据Barcode序列和PCR扩增引物序列从下机数据中拆分出各样本数据,截去Barcode和引物序列后使用FLASH(V1.2.7,http://ccb.jhu.edu/software/FLASH/)[11]对每个样本的reads进行拼接,得到的拼接序列为原始Tags数据(Raw Tags);拼接得到的Raw Tags,需要经过严格的过滤处理[12]得到高质量的Tags数据(Clean Tags)。
1.3.4 OTU聚类和物种注释
利用Uparse软件(Uparse v7.0.1001,http://www.drive5.com/uparse/)[13]对所有样本的全部Effective Tags进行聚类,默认以97%的一致性(Identity)将序列聚类成为OTUs(Operational Taxonomic Units),同时会选取OTUs的代表性序列,依据其算法原则,筛选的是OTUs中出现频数最高的序列作为OTUs的代表序列。 对OTUs序列进行物种注释,用Mothur方法与SILVA132(http://www.arb-silva.de/)[14]的SSUrRNA数据库[15]进行物种注释分析(设定阈值为0.8~1.0),获得分类学信息并分别在kingdom(界)、phylum(门)、class(纲)、order(目)、family(科)、genus(属)、species(种)水平上统计各样本的群落组成。
1.3.5 样本复杂度分析(Alpha Diversity)
使用Qiime软件(Version 1.9.1)计算Chao1,Shannon指数,使用R软件(Version 2.15.3)绘制稀释曲线。
Chao - the Chao1 estimator (http://scikit-bio.org/docs/latest/generated/skbio.diversity.alpha.chao1.html#skbio.diversity. alpha.chao1);
Shannon - the Shannon index (http://scikit-bio.org/docs/latest/generated/skbio.diversity.alpha.shannon.html#skbio.diversity.alpha.shannon)。
2 结果与分析
2.1 测序数据统计及 Alpha 多样性统计
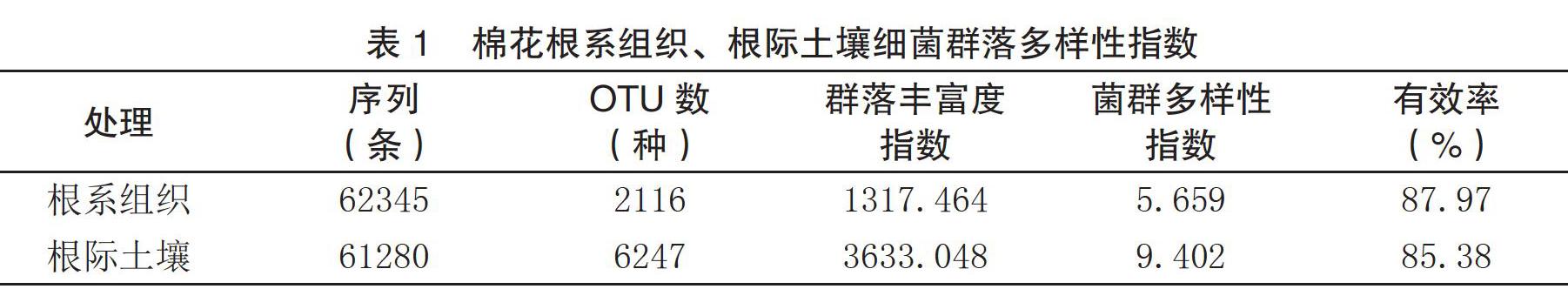
通过对棉花根系和根际土壤不同取样点进行微生物多样性测序分析,结果显示,根系组织(a1)和根际土壤(a2)可用于后续分析的有效序列分别为62345和61280條。利用QIIME软件对相似度大于97%进行分类单元(OTU)聚类,a1和a2两个样本得到OTUs分别为2116和6247种,测序覆盖率大于85%。
对OTU进一步分析可知(表1),棉花根际土壤和根系Chao1指数分别为3633和1317,相差2.7倍;棉花根际土壤Shannon指数为9.4,而棉花根系Shannon指数为5.7,相差1.6倍。相对棉花根系,棉花根际土壤细菌群落具有较高的丰富度(Chao1)和多样性(Shannon),说明两种样本的菌群多样性差异显著。
2.2 棉花根系和根际土壤微生物群落组成
应用RDP classifier软件分析棉花根际土壤和根系微生物群落组成显示:根际土壤中各分类水平数量分别为:门37种,纲45种,目93种,科170种,属289种;根系组织中各分类水平数量分别为:门20种,纲26种,目65种,科110种,属175种。从数量上来看,根际土壤样本各分类水平均大于根系组织各分类水平。
在门水平上,由图1可以看出,根际土壤中变形菌门、芽单胞菌门是棉花根际土壤主要的优势菌群,所占比例分别为49.7%、11.5%。放线菌门、酸杆菌门、拟杆菌门、厚壁菌门、硝化螺旋菌门、蓝细菌门、绿弯菌门在棉花根际土壤中所占比例均大于1%。
棉花根系组织中放线菌门、变形菌门是主要的优势菌群,所占比例分别为50.9%、43.7%。拟杆菌门所占比例大于1%,蓝细菌门的比例接近1%。
在属水平上,由图2可知,棉花根系和根际土壤样本中有144种共有属。根际土壤中有145种特有属,根系中有31种特有属,特有属种类相差4.6倍。
分析144种共有属表明,棉花根际土壤中假单胞菌属、沙雷氏菌属,未鉴定的酸杆菌(unidentified_Acidobacteria)、鞘氨醇单胞菌属、芽孢杆菌属、未鉴定的蓝藻细菌(unidentified_Cyanobacteria)、葡萄球菌属、溶杆菌属是主要优势菌,所占比例均大于1%。棉花根系组织中肠杆菌属所占比例大于10%,链霉菌属、假黄单胞菌属、未鉴定的根瘤菌(unidentified_Rhizobiales)、鞘脂菌屬、未鉴定的根瘤菌(unidentified_Rhizobiaceae)、鞘脂单胞菌属、类固醇杆菌属、黄杆菌属、沃斯菌属、假氨基杆菌属为主要菌群,所占比例均大于1%。
在种水平上(图3),根际土壤中的优势种是粘质沙雷菌、佛雷德里克斯堡假单胞菌,所占比例均大于1%。根系组织的优势种为Rhizobiales bacterium_GB3、Rhizobium helanshanense和解聚乙二醇鞘氨醇盒菌,所占比例均大于1%。
2.3 棉花根系组织和根际土壤的细菌功能预测分析
根系组织内的细菌在功能方面主要涉及到化学异养(chemoheterotrophy)和需氧化学异养(aerobic chemoheterotrophy)。根据结果预测,根际土壤细菌涉及的功能种类显著多于根系。主要包括反硝化(denitrification)、亚硝酸铵化(nitrite ammonification)、硝酸铵化(nitrate ammonification)、亚硝酸盐呼吸(nitrite respiration)、硝酸盐呼吸(nitrate respiration)、硝酸盐还原(nitrate reduction)、硝化作用(nitrification)。从功能预测结果也反映了根系组织内生菌群落和棉花根际土壤环境中微生物群所扮演的角色不同。
3 讨论
高通量测序技术区别于传统的分离培养方法,能高效、系统地了解微生物的全貌。本研究采用高通量测序技术对棉花根系组织和根际土壤微生物进行测序和分类,发现根际土壤微生物群落指数Chao1和Shannon均高于根系组织内生物群落指数Chao1和Shannon,分别约高2.7倍和1.6倍,说明棉花根系组织微环境和接触的根际土壤微环境存在很大差异。
对各个分类水平分析发现,棉花根际土壤微生物多样性在门、纲、目、科和属水平上都大于棉花根系组织内生菌的种类。且棉花根系组织和根际土壤两个不同的微环境,优势菌群显著不同。在门水平上,棉花根际土壤中优势菌群主要是变形菌门,而棉花根系组织的优势菌群主要是变形菌门和放线菌门。棉花根际土壤中大于1%的菌群有7个,分别为放线菌门、酸杆菌门、拟杆菌门、厚壁菌门、硝化螺旋菌门、蓝细菌门和绿弯菌门;而根系组织中只有拟杆菌门,可见棉花根系组织和根际土壤两个微环境下微生物群落显著不同,说明棉花根系组织和根际土壤两个微环境菌群所行使的功能不同。
对种水平的进一步分析表明,棉花根际土壤中优势种是粘质沙雷氏菌、佛雷德里克斯堡假单胞菌。有研究表明,粘质沙雷氏菌具有促进植物生长作用[16-17];佛雷德里克斯堡假单胞菌具有将土壤中的不溶性磷源溶解成为可溶性、并能促进植物生长等作用[18-19]。这说明这两种菌对棉花的生长起到了积极地作用。根系组织的优势种为R.bacterium_GB3、R.helanshanense和解聚乙二醇鞘氨醇盒菌。R.bacterium_GB3和R.helanshanense都属于根瘤菌,具有固氮的作用。解聚乙二醇鞘氨醇盒菌为可产铁载体,可以向寄主植物提供可利用的铁元素,也可与环境中病原菌竞争可利用的铁而抵抗病原菌,从而达到促进植物的生长的作用[20]。由此可见,这3种内生菌主要在棉花固氮、提供微量元素、抵抗病原菌方面起到一定的作用。综合以上结果认为,尽管棉花根系组织和根际土壤中微生物群落显著不同,但是对于棉花的健康生长都起到很积极的作用,也为进一步挖掘和开发新的植物生长促进剂和病害防治资源提供依据。
4 致谢
感谢新疆生产建设兵团第一师农业科学研究所武刚同志对本研究的帮助!
参考文献
[1] 汪烨. 我国棉花种植继续向新疆集中[J]. 农经,2020(03):53-55.
[2] 梁亚军,李雪源,郑巨云,等. 新疆2019年棉花产业情况概述及存在问题与策略[J]. 棉花科学,2020,42(1):14-20.
[3] 徐娅,谢成建,杨星勇. 棉花黄萎病生物防治研究进展[J]. 安徽农业科学,2019,47(02):18-22.
[4] 乔艳艳,杨兆光,杨磊,等. 棉花黄萎病菌的拮抗生物菌研究浅述[J]. 棉花科学,2016,38(6):19-23.
[5] 龚平. 新疆棉花生产中存在的问题及改进措施[J]. 中国棉花,2018,45(04):37-38.
[6] 李秀英,赵秉强,李絮花,等. 不同施肥制度对土壤微生物的影响及其与土壤肥力的关系[J]. 中国农业科学,2005,38(8):1591-1599.
[7] 戈子龙,张泽金,周爱梅,等. 基于高通量测序与培养方法分析新鲜佛手与老香黄中的细菌多样性[J]. 食品与发酵工业,2020,46(03):250-256.
[8] Degnan P H , Ochman H . Illumina-based analysis of microbial community diversity[J]. The ISME Journal,2012,6(1):183-194.
[9] Caporaso J G , Lauber C L , Walters W A , et al. Ultra-high-throughput microbial community analysis on the Illumina HiSeq and MiSeq platforms[J]. The ISME Journal,2012,6(8):1621-1624.
[10] 郝海婷,王若愚,赵霞,等. 基于高通量测序技术的堆肥对兰州百合根际微生物多样性的影响[J].西北农业学报,2017,26(03):437-447.
[11] Mago? Tanja, Salzberg S L.FLASH: fast length adjustment of short reads to improve genome assemblies[J]. Bioinformatics,2011,27(21):2957-2963.
[12] Bokulich, Nicholas A, Subramanian,et al. Quality-filtering vastly improves diversity estimates from Illumina amplicon sequencing[J]. Nature Methods,2013,10(1):57-59.
[13] Haas B J, Gevers D, Earl A M,et al. Chimeric 16S rRNA sequence formation and detection in Sanger and 454-pyrosequenced PCR amplicons[J]. Genome Research,21(3):494-504.
[14] Edgar R C. UPARSE: highly accurate OTU sequences from microbial amplicon reads. Nature methods,2013,10(10):996-998.
[15] Wang Q , Garrity G M , Tiedje J M , et al. Na?ve Bayesian Classifier for Rapid Assignment of rRNA Sequences into the New Bacterial Taxonomy[J]. Applied and Environmental Microbiology,2007,73(16):5264-5267.
[16] 陽洁,秦莹溪,王晓甜,等. 广西药用野生稻内生细菌多样性及促生作用[J]. 生态学杂志,2015,34(11):3094-3100.
[17] Selvakumar G,Mohan M,Kundu S,et al.Cold tolerance and plant growth promotion potential of Serratia marcescens strain SRM (MTCC 8708 ) isolated from flowers of summer squash (Cucurbita pepo) [J]. Letters in applied microbiology,2008,46(2):171-175.
[18]曾庆伟,温心怡,吴小芹. 1株Pseudomonas frederiksbergensis JW-SD2的解磷特性及解磷条件研究[J]. 微生物学杂志,2016,36(01):11-16.
[19] 林英,司春灿,韩文华,等. 解磷微生物研究进展[J]. 江西农业学报,2017,29(02):99-103.
[20] 孙磊,邵红,刘琳,等. 可产生铁载体的春兰根内生细菌多样性[J]. 微生物学报,2011,51(02):189-195.
收稿日期:2020-5-13
基金项目:大学生创新训练项目(2019016);塔里木大学校长基金博士项目(TDZKJC201801)。
通信作者:郝海婷,m18919046163@163.com。

