DNA基因序列快速比对算法
史传杰

摘 要:随着人类基因组计划(HGP)的实施,产生了大量的有关生物体核酸、蛋白质等数据,如何处理这些呈指数增长的数据,成为一个难题。其中,基因序列比对是生物信息学中最基本的研究方法之一,科学家通过序列比对,来研究DNA序列的功能、结构以及物种进化信息。本文中提介绍了几种基因比对算法,并比较了几种算法各自的优点和缺点。
关键词:DNA序列对比,算法;
Abstract: with the implementation of human genome project (HGP), a large number of biological nucleic acid and protein data have been produced. Among them, gene sequence alignment is one of the most basic research methods in bioinformatics. Scientists use sequence alignment to study the function, structure and evolution information of DNA sequence. In this paper, three gene comparison algorithms are introduced, and their advantages and disadvantages are compared.
Key words: DNA sequence comparison, algorithm;
1 引言
人類基因组计划(HGP)是美国在1990年提出实施的,自那以后,科学家已经获取了大量的蛋白质、基因序列的数据。而随着基因序列数据的激增,使用高效率的算法找出序列之间的相似性和同源性已是迫在眉睫的问题。
序列对比指的是运用某种数学算法或模型,将两个或多个序列排列在一起进行比对,比较两条或多条序列碱基,标明其相似之处。DNA序列对比指的是比较两条或多条DNA序列,展示其相似性和同源性。
目前,国内外DNA双序列对比的算法主要有三种:动态规划算法、点阵法和词或k串法[1]。用比较两条序列之间的相似性的算法有Smith-Waterman算法;用于多条序列之间的比对的算法有HMM算法;用于搜索拥有相似性或者同源性的算法有Blast算法、Fasta算法等。以下,我将介绍几种算法:动态规划算法、点阵法、基于隐马尔可夫模型算法、智能计算等。
2 DNA双序列比对算法
2.1打分矩阵
在建立的打分矩阵中,替换矩阵和空位罚分对矩阵得到的结果有直接影响。基因序列经过碱基对或碱基片段的插入、替换或删除等,会产生一系列的变化,因此必须对每个碱基对的插入、替换或删除进行运算预置得分值,而替换矩阵[2]则由这些预置的得分值构成。其中,最基本的打分矩阵是等价矩阵。
例如,如果两个碱基匹配,得分为 1,否则,得分为 0[3]。如下图:
连续空位罚分的公式为:W + S x (K-1)。其中,K为连续出现的空位个数,W为起始罚分,S为延伸罚分。
2.1.1 动态规划算法:
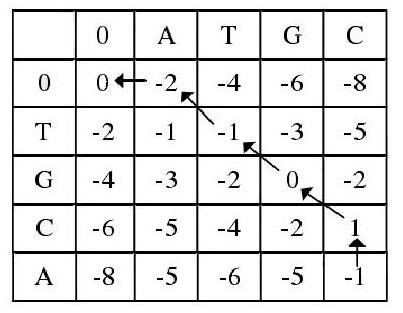
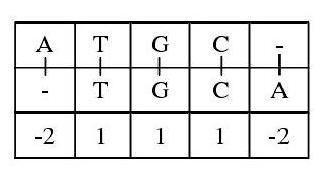
动态规划算法[4]通常被用于双序列的比对,假设存在两条序列为ATCG和TGCA,则以两条序列为横纵坐标,组成一个矩阵。矩阵的求解包括一个或多个大小为(m+1)×(n+1)的表格。 表中的单元格[i,j]与单元格[i-1,j-1]和[i-1,j],[i,j-1]有关。其中,m、n表示两条序列的长度,i表示行,j表示列。如下图所示:
可以得到最优序列:
动态规划算法的空间复杂度为O(m + n),时间复杂度为O(mn)[5]。动态规划算法是一种最优算法。但与此同时,动态规划算法的不足之处也十分明显:时间复杂度、空间复杂度高。
2.1.2 点阵法:
在使用点阵法时,需要先建立里一个矩阵。以两条序列M和序列N分别为矩阵的横轴和纵轴。点阵法是从横轴序列M的第一个字符开始,沿着矩阵的第一行向第二个字符移动,若纵轴序列N为相同字符,则用阴影标记出来。当第一行比较完成后,到第二行比较,以此类推,直至标记完成整个矩阵。矩阵中的阴影部分表示了两条序列所有可能的匹配。在图中,斜对角线方向的一连串的阴影部分表示为相似区域,而对角线以外一些孤立的阴影部分表示随机匹配的序列,没有任何生物学意义。
假设序列M=CCTGAATCGA,序列N=CCTGAAGCAC
如下图:
优点:点阵法可以直观的表示出两个序列之间所有可能的匹配。
缺点:点阵法得到的比对结果不够精确,并且点阵法只适用于较短的两个序列,而面对如今数据量庞大的生物序列数据明显存在着缺陷[6]。
3 DNA多序列比对算法
3.1渐进比对
渐进比对算法[7]是将多序列比对转化为双序列比对,之后将双序列比对的结果进行整理,进而得到最终的结果。
目前大多数多序列比对算法都是采用了渐进比对算法。渐进比对算可以简述为有n个序列需要比对。先比较最近的两条序列,然后将两条序列比对的结果和接下来的一条进行比对。
例如:第一步:将x序列和x+1序列条先行进行比对,得到结果y序列;
第二步:将y序列和x+2序列进行比对,得到y1序列;
重复上述第二步过程,直到比较完所有需要比对的序列。
如果序列条数n相对较小,则初始比对中产生的错误越小。反之,序列条数n越大,则在初始比对中产生的错误多,甚至序列无法继续比对。
3.2迭代比对、启发式对比
迭代比對算法是基于局部最优思想的比对算法。迭代比对算法是用动态规划的方法来改正渐进算法中发生的偏差。以此来得到较高的得分。相对于迭代对比算法,启发式算法同样使用动态规划算法,但启发式算法得出的结果是最优的多序列比对结果。
4基于隐马尔可夫模型的算法
隐马尔可夫模型可以被用于多序列基因的测定。隐马尔可夫模型可以用于模拟生物DNA基因序列的插入、缺失、突变以及匹配的过程。生物的DNA序列可以看成有一个原始的DNA序列,经过若干基因序列或片段的插入、突变、删除而得到。因此隐马尔可夫模型在生物DNA基因序列比对中得到了广泛的应用。
在DNA的多重序列对比中,可以将隐马尔可夫模型看做在DNA序列上前进,从某个状态开始进入插入或删除或匹配。如果插入或匹配,则产生一个新的碱基序列;如果删除,则返回到前一个碱基。当这个状态结束后,则进行到下一个状态,在下一个状态中重复如上操作。
当马尔可夫链从开始状态到结束状态后,我们便可以得到一条由A、G、C、T四个字母组成的基因序列和一条看不见的状态序列。
优点:模型中的每一个位置都考虑了所有的残基的分布,能够有效地修复DNA序列演变中的残基的缺失。
缺点:隐马尔可夫模型需要大量的相似的DNA序列进行比对,需要占有大量的资源。
5智能计算
在DNA基因对比的智能计算方面,常见的算法有粒子群算法(PSO),遗传算法(GA),模拟退火算法(SA)。[8]
5.1粒子群算法
粒子群算法在设计上比较简单,计算量也相对较小,收敛速度较快。相对于其他算法,对适应函数没有求导的要求,因此计算更加简单效率。但是这种算法由于搜索的随机性不够,容易陷入局部最优。
5.2遗传算法
遗传算法初始值范围较广,避免了局部最优的问题,并且减少了计算次数,从而降低了运算难度。但此算法容易由于过早的收敛性而得到一个局部最优。此外,初始值的范围设定也决定了计算的难度。
5.3模拟退火算法
模拟退火算法是围绕着“产生新的序列→计算当前的序列与新的序列之差→判断是否接受新的序列→接受或舍弃新的序列”这个过程迭代的。这种算法存在的问题是计算的效率低,速度慢。
3 结论
本文主要介绍了几种用于DNA基因序列比对的算法。随着人类基因组计划(HGP)的进行与实施,由此而来的大量DNA序列等遗传物质等待着科学家们通过序列比对,来研究DNA序列的功能、蛋白质的结构以及物种进化得信息。本文总结并希望为研究人员提供合适的比较分析工具提供参考。
参考文献:
[1]曹雪卉.DNA词序列比对及应用[D]. 哈尔滨工业大学,硕士学位论文 2013:1-2
[2]Paten B, Earl D, Nguyen N, et al. Cactus: Algorithms for genome multiple sequence alignment[J]. Genome research, 2011, 21(9): 1512-1528.
[3]Aniba M R, Poch O, Marchler-Bauer A, et al. AlexSys: a knowledge-based expert system for multiple sequence alignment construction and analysis[J]. Nucleic acids research, 2010, 38(19): 6338-6349.
[4]Sarkar S, Kulkarni G R, Pande P P, et al. Network-on-Chip hardware accelerators for biological sequence alignment[J]. Computers, IEEE Trans actions on, 2010, 59(1): 29-41.
[5]唐玉荣,汪懋华.基于动态规划的快速序列比对算法[R].生物数学学报2005.8:207-212
[6]Sery O. Enhanced Property Specification and Verification in BLAST[J]. Fundamental Approaches to Software Engineering, Proceedings,2009(5503):456-469

