盖子环替换及定点突变提高菜豆环氧化物水解酶对邻甲基苯基缩水甘油醚的催化性能
章晨,朱秀秀,李闯,邬敏辰
(1 江南大学药学院,江苏无锡214122;2 江南大学生物工程学院,江苏无锡214122;3 江南大学无锡医学院,江苏无锡214122)
手性环氧化物是一类重要的药物砌块,广泛运用于医药、农药等领域[1]。如(S)-环氧苯乙烷是合成抗HIV 病毒药、杀虫剂等的重要中间体[2];(R)-oMGE 是合成β1-肾上腺素受体拮抗剂的重要原材料[3]。目前主要是通过化学合成以及生物酶催化拆分外消旋(racemic,rac-)环氧化物来获取单构型的环氧化物。化学合成中会使用大量的重金属,不仅在生产中毒害大,也会对环境造成极大的破坏。相比之下,生物酶法由于其低毒性和经济性,被广泛接受[4]。如环氧化物水解酶(epoxide hydrolases,EH,EC 3.3.2.-),能够催化外消旋环氧化物对映或归一性水解为相应的邻二醇,且广泛存在于植物、动物以及微生物体内[5]。
近年来,环氧化物水解酶的挖掘及改造已成为研究热点。如许建和团队[6-7]从绿豆中扩增出2条编码EH 的基因,并导入大肠杆菌(Escherchia coli,E.coli),成功地实现了植物来源环氧化物的异源表达,所得的重组酶VrEH1和VrEH2能对映归一性水解对硝基环氧苯乙烷,生成的(R)-对硝基苯乙二醇的对映体过量值(enantiomeric excess,ee)分别为70.0%和84.8%。又如朱勍团队[8]从苹果轮纹病菌中挖掘出一种EH,能够动力学拆分苯基缩水甘油醚类环氧化物,其中,对rac-oMGE对映体比率(enantiomeric ratio,E)达到48.6。但目前发现的大多数野生EH因为E、ee或活性低等原因,无法满足工业生产的需求。随着蛋白质分子改造技术的日益成熟,改造EH 从而改善其性质的研究也屡见报道。如Reetz等[9]通过定点饱和突变技术将一种黑曲霉EH 对rac-oMGE的E值由4.6提高到160。
实验室前期从菜豆中挖掘出两种菜豆环氧化物水解酶PvEH1和PvEH2,均能对映选择性水解racoMGE[9]。现拟通过分子改造提高PvEH2 的催化性能,使其水解rac-oMGE 时既能保留单构型环氧化物,又能生成较纯的单构型邻二醇。先同源模拟PvEH1 和PvEH2 的三维结构,然后替换PvEH2 分子结构中的盖子环区域,再利用分子对接技术选取距离底物8Å(1Å=0.1nm)以内的非保守氨基酸,将其全部突变为丙氨酸,得到了一个最优突变子Pv2Pv1K176A,并采用分子对接模拟分析其区域选择性提高的原因,最后用E. coli/pv2pv1K176A催化高浓度rac-oMGE。
1 实验材料与方法
1.1 菌株与质粒
E. coli BL(DE3)和E. coli JM109,保存于本实验室;克隆质粒pUCm-T,上海Sangon公司;表达质粒pCold Ⅱ,美国Novagen公司。
1.2 试剂与仪器
1L LB 培养基:胰蛋白胨10g、酵母提取物5g和NaCl 10g(固体培养基另加20g 琼脂粉),pH 调至7.0。质粒提取试剂盒(无锡康为世纪公司)。rac-oMGE(上海TCI 公司)。全温摇瓶柜HYG-A(江苏太仓实验设备厂);高速冷冻离心机5804R(德国Eppendorf 公司);实验所用酶均购于上海Sangon生物技术有限公司。液相色谱OD-H手性柱(250mm×4.6mm×5μm)购自大赛璐药物手性技术(上海)有限公司。
1.3 PvEH2的结构分析
在Swiss-Prot Protein 数据库(http://www.ebi.ac.uk/swissprot/)中搜索与PvEH2 同源性最高且结构和功能已知的蛋白分子,以此蛋白三维结构为模板在SWISS-MODEL 网 站(https://swissmodel.expasy.org/)中模拟出PvEH1和PvEH2的三维结构。利用PyMOL软件(https://pymol.org/2/)对它们的三维结构进行可视化分析。
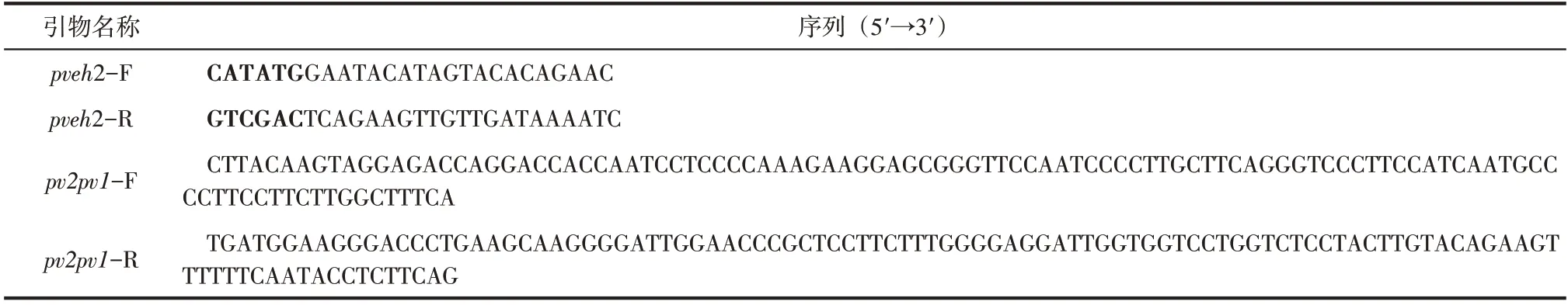
表1 FPCR所用引物
1.4 PvEH2的盖子环替换
本节所用引物如表1所示,由无锡亦欣生物公司合成。提取含有PvEH2 编码基因的大肠杆菌转化子质粒(pCold Ⅱ-pveh2),并作为模板,通过三轮融合聚合酶链式反应(FPCR)获得目的基因pv2pv1。第一轮以pveh2-F 和pv1pv2-R 作为引物,在DNA聚合酶PrimerSTAR HS DNA polymerase作用下进行PCR:98℃变性3min,30 个循环(98℃,30s;50℃,30s和72℃,40s),72℃充分延伸10min,对PCR产物进行琼脂糖凝胶电泳检测并割胶回收。再以回收产物为模板,pveh2-R 和pv2pv1-F 为第二轮引物,其他条件不变进行扩增、检测和割胶。最后以第一轮和第二轮产物作为第三轮PCR 的上下模板,pveh2-F和pveh2-R为引物,其他条件不变,获得盖子环被替换的EH 编码基因pv2pv1。连接pUCm-T 载体,转化E. coli JM109 并用氨苄西林(Amp)筛选阳性克隆子送至上海Sangon 生物技术有限公司进行DNA 测序验证,测序成功的转化子命名为pUCm-T-pv2pv1,用Nde I 和Sal I 双酶切,最终连接至pCold Ⅱ,转入E. coli BL21(DE3),经Amp 抗性筛选、验证并且测序正确的重组菌株命名为E.coli/pv2pv1。
1.5 全细胞酶的诱导及催化特性分析
将获得的菌接种(接种量为1%)于2mL含0.1μg/L Amp 的LB 培养基中,于37℃、220r/min 条件下培养过夜;取2mL 培养液转接于100mL 含0.1μg/L Amp 的LB 培养基中,培养至OD600(在600nm 波长处的吸光值)为0.6~0.8时,加入0.1mL 500mmol/L的异丙基硫代半乳糖苷(IPTG) 至终浓度为0.5mmol/L,14℃诱导16h后离心收集湿菌体。
称取0.1g 湿菌体悬浮于1mL 磷酸钠缓冲液(100mmol/L,pH=7.0)中,取0.3mL 100g/L菌悬液加到含0.65mL磷酸钠缓冲液的2mL 环氧树脂(EP)管中,20℃孵育5min 后加入50μL rac-oMGE(200 mmol/L,溶剂为甲醇)至终浓度为10mmol/L,20℃恒温震荡反应10min,取150μL 样品用1mL 乙酸乙酯萃取,有机相经无水硫酸镁干燥。样品采用高效液相色谱仪(Waterse2695)、手性液相色谱柱(OD-H)和紫外检测器(Waters2489)进行分析。高效液相色谱条件为:流动相为正己烷∶异丙醇=80∶20(体积比),柱温30℃,流速为0.8mL/min,检测波长为220nm。(R)-oMGE、(S)-oMGE、(R)-oTPD和(S)-oTPD 的保留时间分别为6.69min、8.03min、8.83min和9.82min。
在上述测定条件下,酶活单位(U)定义为每分钟消耗1μmol 的rac-oMGE 所需的酶量。全细胞比活力(U/g)计算公式如活性=C0×v×c/(t×m)所示。其中c为全细胞催化rac-oMGE的转化率,%;C0为底物初始浓度,mmol/L;t 为反应时间,min;v 为反应体积,mL;m为湿细胞质量,g。
底物的ees=(RS-SS)/(RS+SS)×100% 和产物eep=(SP-RP)/(SP+RP)×100%。其中RS和SS分别代表(R)-oMGE和(S)-oMGE的峰面积;RP和SP表示(R)-oTPD和(S)-oTPD 的峰面积。酶的对映选择性用E 来评价,E=ln[(1-c)(1-ees)]/ln[(1-c)(1+ees)],E值越高,则对映选择性越高[10]。
区域选择性系数αS和βR(图1)可由线性回归方程eep=(βS-αR)×ees×(1-c)/c+αR+βS-1进行计算[11]。eep和ees×(1-c)/c 定义为因变量和自变量。通过测定酶催化rac-oMGE 水解过程中不同时间点的c、ees和eep,对上述线性方程进行拟合,从而算得αR,βS。
1.6 Pv2Pv1的丙氨酸突变
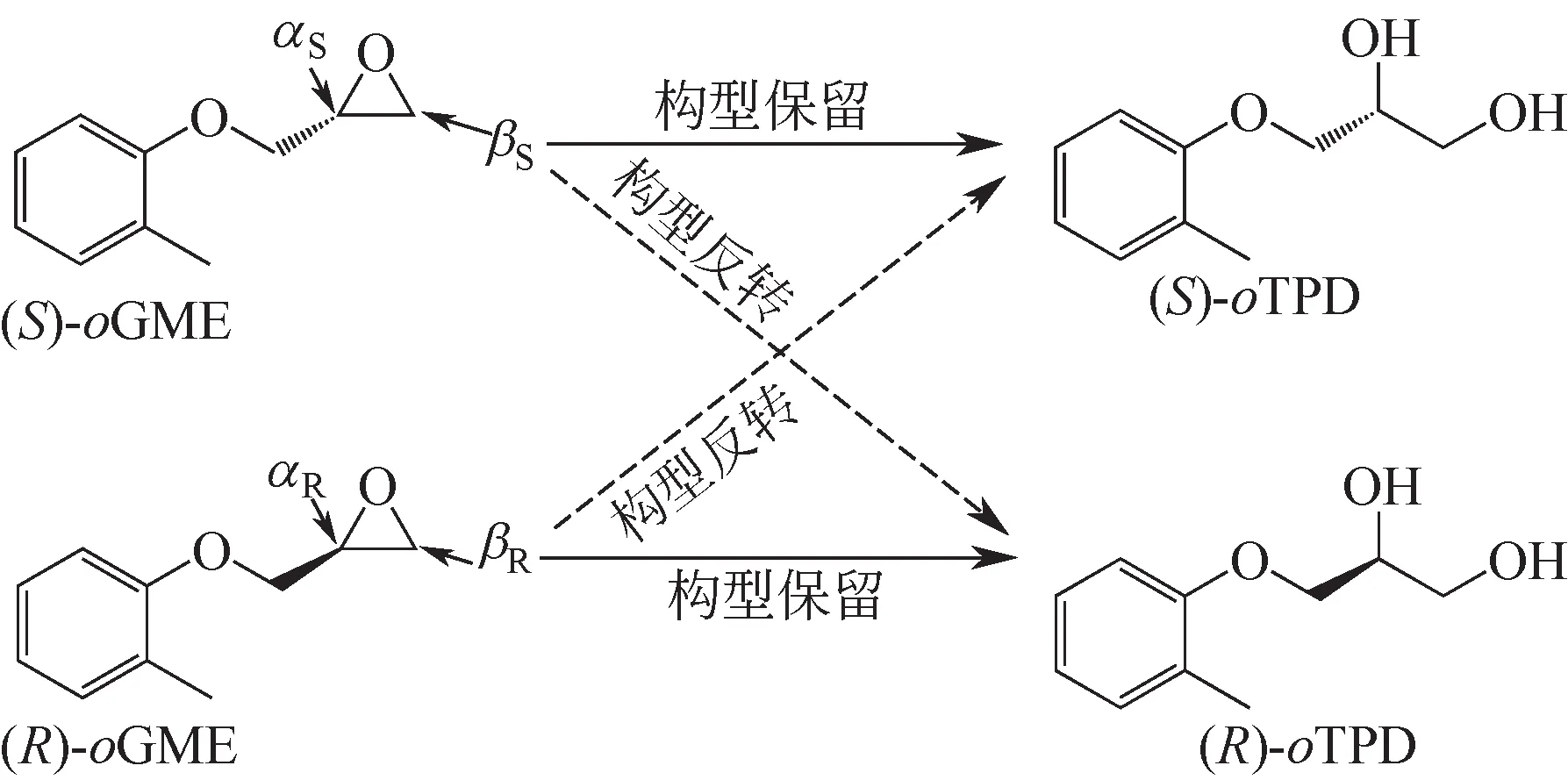
图1 区域选择性的示意图
将Pv2Pv1 和另外4 种植物来源的EH 的一级结构进行同源序列比对,找出非保守氨基酸(同源性≤60%)。根据1.3节方法对Pv2Pv1同源建模,再通过AutoDock4.2 软 件(http://autodock.scripps.edu)进行分子对接,用PyMOL 找出距离(R)-oMGE 8Å以内的非保守氨基酸,并通过两轮全质粒PCR 将这些氨基酸突变为丙氨酸(A)。根据相关文献设计突变引物[10]。一轮PCR所用的上游引物为设计的突变引物,下游引物为通用引物pCold Ⅱ-R,预变性95℃,4min;变性98℃,10s;退火55℃,5s;延伸72℃,1min;从变性到延伸循环30 次,最后充分延伸,72℃,10min。二轮PCR 以一轮PCR 产物为引物,全质粒为模板,除延伸时间变为5min,其他条件同第一轮。产物经限制性内切酶DpnI 消化后导入E. coli BL21(DE3),培养12h 后对转化子进行菌液PCR验证,并对阳性转化子进行测序,将测序结果正确的转化子命名为E.coli/pv2pv1突变位点。
1.7 分子对接分析
用ChemDraw Ultra 12.0 软 件 (http://www.cambridgesoft.com/)构建(R)-oMGE的三维结构。参照1.3 节方法模拟Pv2Pv1 和Pv2Pv1K76A的三维结构模 型。采 用AutoDock 4.2 将PvEH2、Pv2Pv1 和Pv2Pv1K76A分别与(R)-oMGE 进行分子对接,通过Gromacs(http://www.gromacs.org/)进行分子动力学模拟,将生成的结合能最低的构象作为最佳构象,用PyMOL分析所得构象。
1.8 E.coli/pv2pv1K76A水解高浓度rac-oMGE
为了确定E.coli/pv2pv1K76A水解动力学拆分racoMGE的最大浓度,在10mL EP管中,用200mg/mL的E.coli/pv2pv1K76A催化不同浓度(80~180mmol/L)的rac-oMGE,反应体系2mL,25℃反应2~8h。测定ees、eep、YP和YS。在确定最大反应浓度后,放大反应体系至10mL,在25℃条件下用200mg/mL的E.coli/pv2pv1K76A水解150mmol/L 的rac-oMGE,定时进行取样监测,参照方法1.5用HPLC测定残余的(R)-oMGE、(S)-oMGE 及生成的(R)-oTPD、(S)-oTPD 的浓度,并计算ees、eep、YP和YS,绘制反应进程曲线,同时计算时空产率[space-time yield, STY,表示在一定的反应条件下,单位体积单位时间获得目的产物总量,g/(L·h)[11]]。
2 实验结果与讨论
2.1 PvEH2和PvEH1的三维结构分析
实验室前期挖掘了两种菜豆环氧化物水解酶,PvEH1 和PvEH2,它们均能对映选择性水解racoMGE,优先水解S构型,保留R构型。当S构型的底物水解完全时(ees>99%),所生成的(S)-oTPD的eep仅有8.3%和53.6%,其中全细胞酶E. coli/pveh1 对rac-oMGE 的活性为157.2U/g,远远高于E.coli/pveh2的4.1U/g[12-13]。
以PvEH2一级结构为模板,在SWISS-MODEL网站找到一个同源性最高(80.06%)且功能已知的绿豆环氧化物水解酶(VrEH1,PDB∶5XMD.2),并以它为模板模拟PvEH2、PvEH1的三维结构。结果显示,PvEH2 和PvEH1 均属于α/β 水解酶家族,催化三联体为Asp-His-Asp 和两个Tyr 质子供体分别位于中心结构域和盖子结构域(图2)。其中盖子环是盖子结构域中的一段可变环,在不同的EH中,它的结构差异很大,因而对EH的催化性质影响很大[14]。PvEH2和PvEH1的盖子环在结构上有很大的差异,如图2(b)。基于全细胞酶E.coli/pveh1的高酶活,期望通过盖子环替换改善E.coli/pveh2 的酶活及其他催化性质。

图2 PvEH1和PvEH2的三维模拟结构比对

表2 E.coli/pveh2及/pveh2cl1的相关催化性质
2.2 Pv2Pv1的催化性质分析
按1.4 节方法获得了重组菌E. coli/pv2pv1,诱导表达收菌后,按1.5 节方法对其相关催化性质进行分析,其活性几乎没有发生变化,E值由23.0下降到15.1,当底物的ees>99%时,其产物(S)-oTPD的eep由E. coli/pveh2 的58.3%提高到75.5%,通过对区域选择性系数的分析发现,出现该现象的原因是αR由3.8%提升至42.4%。保留的(R)-oMGE 的产率(Ys) 有所降低,但是产物(S)-oTPD 的产率(Yp)由42.9%提高至58.4%(表2)。表明盖子环替换后的Pv2Pv1,有一定的能力使(R)-oMGE 翻转生成(S)-oTPD。
2.3 丙氨酸突变位点的选择
运 用AutoDock 4.2 对Pv2Pv1 和(R)-oMGE 进 行分子对接,其结果如图3(a)所示。距离底物8Å 以内的氨基酸残基共计有32个[图3(c)中“*”表示]。与其他4 种植物来源的EH 序列比对,除去包括催化三联体和两个质子供体在内的17个保守位点和4个相对保守位点,一共有11 个非保守氨基酸位点:
I105、W129、P137、K175、L176A、N179、L187、P203、I238、S266 和M273[图3(b)]。据报道,突变EH中的非保守氨基酸,能很好地改善其催化性质,如Li等[14]通过突变PvEH3中的非保守氨基酸,将产物对氯苯基乙二醇的eep由85.1%提高至93.2%,Kong等[15]通过在BmEH的催化活性中心附近进行丙氨酸突变,大大地提高了其对α-萘基缩水甘油醚的活性和对映选择性。因此,本研究选择将以上11个非保守氨基酸突变为丙氨酸。
2.4 丙氨酸突变子的催化特性分析
按1.6节方法获得11个丙氨酸突变子,用全细胞催化反应,按1.5 节方法测定各突变子对racoMGE 的催化特性。如表3 所示,11 个突变子中,E.coli/pv2pv1K176A的催化性能综合表现最为优越,与E.coli/pv2pv1 相比,催化性能得到明显提升,活性由4.2U/g提高至8.7U/g,提高了2.1倍。当S构型的底物刚好反应完全时,R 构型的邻二醇的eep由75.5% 提高至80.3%,产率也由58.4% 提高至61.9%。经测定,区域选择性系数βS=4.6%,αR=52.2%,αR相对于Pv2Pv1提高了9.8%。

表3 丙氨酸突变子对rac-oMGE的催化特性分析
2.5 分子对接分析区域选择性提高原理
为了从分子水平了解Pv2Pv1及Pv2Pv1K176A区域选择性系数αR提高的原理,运用AutoDock 4.2 将PvEH2、Pv2Pv1、Pv2Pv1K176A与(R)-oMGE 进 行 对接,对接后的局部放大图如图4。根据Reetz等[16-17]关于EH催化机理的研究:位于活性中心的天冬氨酸的O原子通过攻击环氧环上的C原子,从而使环氧环断开,其中,天冬氨酸中的氧到环氧环中Cα原子的空间距离dα是一个关键参数,dα越短,Cα越容易受到O 原子的攻击,其αR值越大。PvEH2 的dα=4.5Å,经过盖子环的替换后,dα变为3.1Å,在上述的实验检测中,其对应的αR值由3.8%提高到42.4%,丙氨酸突变后dα进一步缩短为2.9Å,αR进一步变为52.2%。对接分析结果和实验测量结果相符。
2.6 E.coli/pv2pv1K176A水解高浓度rac-oMGE
为确定E.coli/pv2pv1K176A能水解rac-oMGE 的最大浓度,使用200mg/mL菌悬液分别拆分80mmol/L、100mmol/L、120mmol/L、150mmol/L 和180mmol/L的rac-oMGE。当E. coli/pv2pv1K176A水解150mmol/L的rac-oMGE时,5.0h水解完全,得到(R)-oMGE和(S)-oTPD 的产率YS和YP分别为32.5%和59.1%,相应对应体纯度ees>99%,eep=80.9%。由于EH 存在底物或产物抑制的现象[4],当rac-oMGE 的浓度达到180mmol/L 时,即使反应时间延长至8.0h,ees仍然仅为70.3%。因此,确定E. coli/pv2pv1K176A水解rac-oMGE的最大底物浓度为150mmol/L。

图3 Pv2Pv1与4种植物EH一级结构的多序列比对

图4 PvEH2、Pv2Pv1和Pv2Pv1K176AL与(R)-oMGE的分子对接模拟(1Å=0.1nm)
用200mg/mL E. coli/pv2pv1K176A在10mL 体系中水解150mmol/L rac-oMGE。其水解反应进程图5所示。在反应5.0h 后,(S)-oMGE 完全水解完(ees>99%),得到(R)-oMGE的产率为32.5%,高于AnEH的29%,但低于TlEH 的40%,得到(R)-oMGE 的产率为32.7%,高于AnEH的29%,但低于TlEH的40%,STY为1.6g/(L·h),分别是AnEH和TlEH的6.7和2.4倍。(S)-oTPD 的产率为60.1%,eep为80.4%,均高于TlEH,STY为3.3g/(L·h),是TlEH的3.5倍[18-19]。

图5 200mg/mL E.coli/pv2pv1K176A水解150mmol/L rac-oMGE的进程图
3 结论
(1)本研究通过FPCR,构建了一种盖子环替换的杂合酶Pv2Pv1,通过分析其催化性质,Pv2Pv1在水解rac-oMGE时,当S构型的底物水解完全时,得到的产物(S)-oTPD的产率和eep都有明显升高。
(2)获得11 个丙氨酸突变子,其中,E. coli/pv2pv1K176的酶活,(S)-oTPD的产率,eep都有进一步的提升。通过分子对接分析发现相对于PvEH2 和Pv2Pv1,Pv2Pv1K176有着最短的dα,从而解释了其区域选择性系数αR提高的原因。
(3)用E.coli/pv2pv1K176水解高浓度rac-oMGE,发现其具有应用于工业化生产的潜能,同时该酶也存在一定的缺陷,如酶活和产物(S)-oTPD 的eep还有进一步的提升空间,今后可以通过定向进化的手段对其催化性能进行进一步的优化和改造。

