栽培大豆和野生大豆线粒体基因组密码子使用偏性的比较分析
唐向民 杨守臻 陈怀珠 孙祖东 赖振光 曾维英 韦清源

摘 要:為分析栽培大豆和野生大豆线粒体基因组的密码子使用特征差异,该文以其线粒体基因组编码序列为研究对象,比较其密码子偏性形成的影响因素和演化过程。结果表明:(1)栽培大豆和野生大豆线粒体基因组编码区的GC含量分别为44.56%和44.58%,说明栽培大豆和野生大豆线粒体编码基因均富含 A/T 碱基。(2)栽培大豆和野生大豆线粒体基因组密码子第1位、第2位GC含量平均值与第3位GC含量的相关性均呈极显著水平,说明突变在其密码子偏性形成中的作用不可忽略;PR2-plot 分析显示,在同义密码子第3位碱基的使用频率上,嘌呤低于嘧啶; Nc-plot分析中Nc比值位于-0.1~0.2区间的基因数占总基因数的95%以上;突变和选择等多重因素共同作用影响了大豆线粒体基因组编码序列密码子使用偏性的形成。(3)有20、21个密码子分别被确定为栽培大豆和野生大豆线粒体基因组编码序列的最优密码子,其中除丝氨酸TCC密码子外均以A或T结尾。综上结果认为,栽培大豆线粒体密码子偏性的形成受选择的影响要高于野生大豆,这可能是栽培大豆由野生大豆经长期人工栽培驯化的结果。
关键词:栽培大豆,野生大豆,线粒体基因组,密码子使用偏性,最优密码子
中图分类号:S565.1
文献标识码:A
文章编号:1000-3142(2020)07-0926-09
Abstract:The characteristic of codon usage in mitogenome of wild and cultivated soybean was studied and compared to explore the main factors affecting codon usage bias and codon evolution. The results were as follows:(1) The GC content in the coding region of mitochondrial genome of cultivated soybean and wild soybean was 44.56% and 44.58%,respectively,which indicated that the coding sequences of soybean mitogenome was rich in A/T base. (2) PR2-plot analysis showed that the third base of codon usage was biased,where pyrimidine was used more frequently than purine; Nc-plot analysis revealed that the number of genes with Nc ratio in the range of -0.1-0.2 accounts for more than 95% of the total number of genes. The above-mentioned results showed that the multiple factors such as mutation and selection affected the formation of codon usage bias in soybean mitogenome. GC1 and GC2 value was significantly correlated with GC3,suggesting that codon bias in the mitogenome of cultivated and wild soybeans was mainly affected by mutation. (3) In addition,20 and 21 codons were identified to be the optimal codons for the mitogenome of cultivated and wild soybeans,with most of the preferred synonymous codons ending with A or T base. The formation of mitochondrial codon bias of cultivated soybean was more affected by selection than that of wild soybean,which may be the result of long-term artificial cultivation and domestication of cultivated soybean.
Key words:Glycine max,Glycine soja,mitogenome,codon usage bias,optimal codon
同义密码子使用偏好性是指编码序列中同义密码子被生物体偏爱使用的不同频率(Qiu et al.,2011;Hanson et al.,2018)。由于生物界通用密码子的简并性,每个氨基酸至少对应1种密码子,最多有6种对应的密码子。同义的三联体密码子通常不是被随机使用的,这种偏好性确保了最优密码子可以与数量最多tRNA基因的反密码子配对(Sun et al.,2009)。同时避免了氨基酸的错掺,减少了翻译加工差错(Wei et al.,2014)。研究同义密码子使用的偏好性以及导致其形成的因素,可以帮助更好地了解生物基因组的特征、分子进化以及生态适应性等。之前关于陆地植物全基因组和叶绿体基因组密码子偏性的相关研究较多(Mazumdar et al.,2017),但植物线粒体基因组的密码子使用模式及其相关作用力尚未得到很好的研究。
作为一种细胞核外的遗传系统,线粒体基因组具有重排进化快、叶绿体DNA插入等结构特点。目前,对线粒体基因组的研究主要体现在对基因的结构和功能、基因表达的时空调控、核质互作、分子进化规律、物种的起源进化以及线粒体的起源等生物学领域的研究(夏玉玲等,2008;Kong et al.,2013)。 mtDNA已被广泛应用于物种鉴定、种内母系演化、种间系统发育、种群系统进化及遗传多样性分析等方面的研究(Galtier et al.,2009; Wei et al.,2014; 农全东等,2019; 李平等,2019)。高等植物的线粒体基因组具有RNA编辑和密码子偏好性等特殊的表达方式(李玉秋等,2011)。了解植物线粒体基因组密码子的使用偏好,可以更好地了解其线粒体基因组进化过程。
大豆是豆科大豆属一年生草本植物。一年生野生大豆(Glycine soja)为栽培大豆(G. max)的祖先种,是开展栽培大豆遗传育种工作的宝贵种质。栽培大豆是从野生大豆经过人工改良驯化和长期定向选择逐渐积累有益农艺性状演化而成的。虽然栽培大豆和野生大豆的线粒体全基因组已完成测序(Chang et al.,2013;Sajjad et al.,2018),但关于两者的mtDNA编码基因的密码子使用模式比较研究尚未见报道。比较研究野生大豆和栽培大豆mtDNA编码基因的密码子偏好性,能够更好地揭示两个同属物种的线粒体基因表达系统差异和进化差异。本研究拟以大豆属的野生大豆和栽培大豆线粒体基因组为研究对象,对其密码子使用特征进行系统分析,揭示影响其密码子偏性形成的主要因素,并确定最优密码子。通过比较两个同属物种对线粒体编码序列密码子使用的差异,以期为大豆在人工驯化进程中线粒体表达系统遗传变异的深入研究奠定理论基础。
1 材料与方法
1.1 序列材料
在NCBI数据库检索一年生野生大豆和栽培大豆的线粒体基因组序列,其GenBank登录号分别为NC_039768.1和NC_020455.1。分别下载2个物种的线粒体基因组所有编码序列,剔除重复的编码序列以及小于300 bp的编码序列,并经开放阅读框研判,符合条件的余下编码序列用于本研究的偏性分析。
1.2 方法
1.2.1中性绘图分析 分别统计野生大豆和栽培大豆线粒体基因组各CDS在密码子第1位、第2位和3位的GC含量,以GC3(第3位GC含量)为横坐标,以GC2(第2位GC含量)和GC1(第1位GC含量)的平均值为纵坐标(计为GC12)绘制二维散点图,以分析三联体密码子三个位置碱基组成的相关性(Sueoka,1988)。若GC12与GC3的相关性不显著,则表明密码子第1位、第2位与第3位碱基使用存在差异,选择压力对密码子偏性影响较大;若GC12与GC3显著相关,表明密码子第1位、第2位与第3位碱基使用无差异,密码子偏性的形成受突变的影响大。
1.2.2 相对同义密码子使用度分析 应用CodonW软件计算获得各编码基因的有效密码子数(Nc值)、密码子偏爱指数(codon bias index,CBI)、最優密码子使用频率(frequency of optimal codons,Fop)。利用CAIcal在线服务器对大豆线粒体基因组编码序列的相对同义密码子使用度(relative synonymous codon usage,RSCU)进行分析(Puigbo et al.,2008)。若RSCU=1,表明该密码子的使用无偏好性;若RSCU>1,表明该密码子的使用频率大于同义密码子使用的平均频率;若RSCU<1,则表明低于平均频率。
1.2.3 Nc-plot绘图分析 以GC3s为横坐标,Nc值为纵坐标,作散点图,以探讨碱基组成对密码子偏好性的影响;以仅由碱基组成决定密码子偏好性时的理论值作标准曲线,标准曲线反映了在突变压力下的Nc和GC3s的函数关系(Wright et al.,1990)。理论Nc值计算公式为Nc=2+GC3s+29/[GC3s 2+(1-GC3s)2]。
1.2.4 奇偶偏好分析 PR2(parity rule 2,PR2)分析,为避免由密码子第3位碱基的A/T 或T/A 和G/C 或C/G的突变不均衡,仅选择4种密码子编码的氨基酸,即丙氨酸、亮氨酸、脯氨酸、丝氨酸、苏氨酸、缬氨酸、精氨酸和甘氨酸,进行密码子第3位上4种碱基组成的分析。以每个基因A3/(A3+T3)值作纵坐标,G3/(G3+C3)值作横坐标,以平面散点图展示各基因碱基组成。中心点代表C=G且A=T,由中心点向坐标点发出的矢量则表示偏倚程度和方向(Sueoka,1999)。
1.2.5 最优密码子分析 以CDS的Nc值为偏性标准,将大豆线粒体基因组编码基因中Nc值居于最低和最高两极的10%基因分别构成高、低表达组,统计两组的RSCU值。当两组间ΔRSCU(高表达组RSCU值-低表达组RSCU值)大于0.08密码子定义为高表达密码子。将整体RSCU >1的密码子确定为高频率密码子。同时满足上述两种条件的密码子定义为最优密码子(Wang et al.,2018)。
2 结果与分析
2.1 密码子使用特征
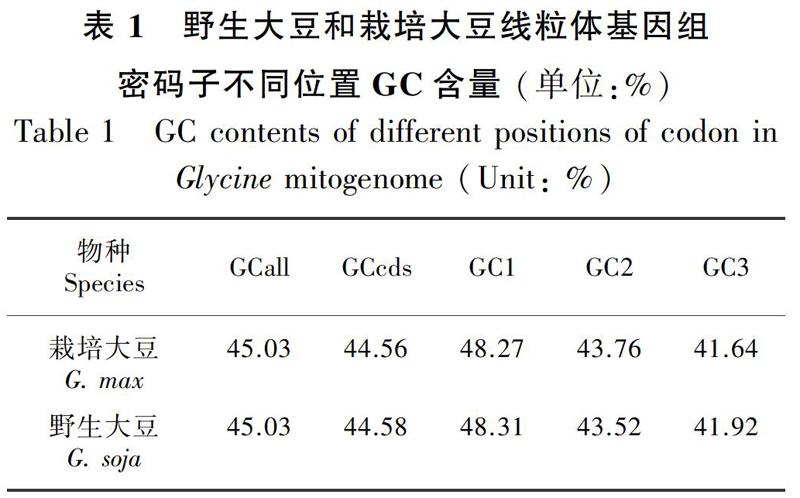
栽培大豆和野生大豆在线粒体基因组编码区的碱基组成上基本一致,其总体GC含量分别为44.56%和44.58%。它们的整个线粒体基因组GC含量均为45.03%。栽培大豆和野生大豆的线粒体编码基因在密码子三个不同位置的碱基GC含量也有所差异,密码子第1位碱基的 GC 含量最高,分别为 48.27%、48.31%,第2位分别为 43.76%、43.52%,第3位分别为 41.64%、41.92%,呈现出GC1>GC2> GC3的趋势(表1)。这表明两个物种线粒体编码基因富含 A、T 碱基。
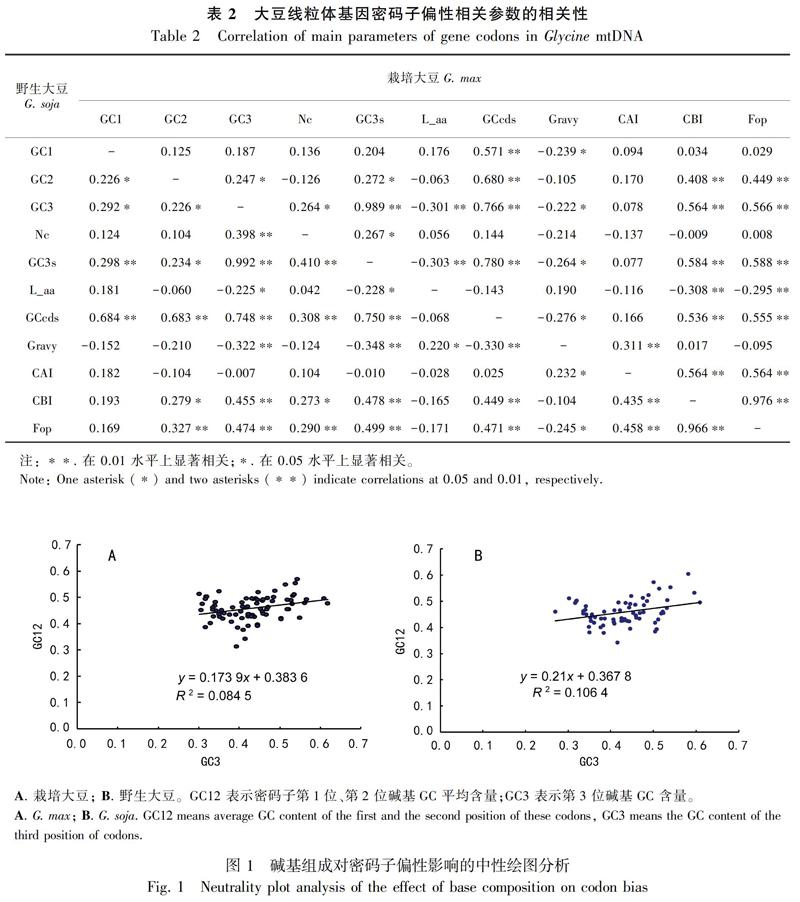
栽培大豆和野生大豆的Nc与GC3分别呈显著相关和极显著相关,与GC1和GC2的相关性均未达显著水平,说明其线粒体编码基因密码子第3位上的碱基组成对密码子偏性影响较大。线粒体基因组编码序列的GC1、GC2和GC3,在野生大豆中呈两两显著相关,表明其密码子第1位、第2位和第3位的碱基组成相似;而在栽培大豆中GC1与GC2及GC3的相关性均未达显著水平,仅GC2和GC3显著相关,表明其密码子第1位与第2位、第3位的碱基组成不同,第2位和第3位的碱基组成相似。在野生大豆中,Nc还与线粒体基因组编码基因的GC含量(GCcds)及最优密码子使用频率(Fop)呈极显著相关,与CBI呈显著相关;而在栽培大豆中,Nc与GCcds及Fop均不显著(表2)。这表明两种大豆属植物线粒体基因组密码子偏性的影响因素有所差异。
2.2 中性繪图分析
栽培大豆和野生大豆线粒体基因组各基因的中性绘图分析(图1)结果显示,各基因坐标点未沿对角线趋势分布。栽培大豆线粒体CDS的GC12分布在0.312 5~0.568 0,GC3分布在0.301 6~0.619 0;一年生野生大豆线粒体CDS的GC12含量分布在0.342 3~0.604 5,GC3分布在0.270 0~0.609 5。两个物种GC12和GC3的相关系数分别为0.326和0.329,双侧检验表明其相关性达极显著水平;回归曲线斜率分别为0.1739和0.21。这说明密码子第1位、第2位与第3位碱基的组成和变异模式相似,突变对基因密码子偏好性的形成有重要影响,其他因素(例如自然选择)可能对密码子偏好性形成的影响较小。栽培大豆线粒体密码子偏性的形成受选择的影响高于野生大豆,这可能是大豆长期人工栽培驯化的结果。
2.3 Nc与GC3s 的关联分析
以GC3s为横坐标,各基因Nc值为纵坐标绘制Nc-plot曲线,以进一步判断突变或选择等因素对大豆线粒体基因组编码序列密码子偏好性形成的影响。根据(Nc预期-Nc实际)/Nc预期的公式计算Nc比值(表3)。栽培大豆和野生大豆Nc比值分布在-0.1~0.1 区间的线粒体基因占70%以上,这些基因对应的散点集中分布在标准曲线附近,其Nc实际值和预期值差异较小,表明这些线粒体基因的密码子偏好性的形成主要受突变因素的影响;其余基因散点偏离于标准曲线下方(图2),Nc实际值和预期值之间相差较大,表明这些基因的密码子偏好形成更多地受选择的影响。
2.4 PR2-plot分析
采用PR2-plot方法分析栽培大豆和野生大豆线粒体基因组CDS的四种同义密码子第3位碱基组成(图3)。图3结果表明,散点并不是均匀地分布于PR2平面图中的四个区域内,50%以上散点位于平面图的左下区域,表明在同义密码子第3位碱基的使用频率上,A低于T,G低于C。若密码子使用偏好性仅由突变影响,则同义密码子第3位4种碱基的使用频率理论值相等,即A=T,C=G。A 和 T 以及 G 和 C 的使用频率不均衡性,表明两种大豆线粒体基因组密码子的使用偏好性由突变和选择等因素共同影响。
2.5 最优密码子分析
对栽培大豆和一年生野生大豆线粒体基因组各编码序列的Nc值进行排序,分别从两极各选出8 个基因构建高、低表达组。按组计算各基因同义密码子的RSCU值,并根据对高表达密码子和高频率密码子的定义,最终确定了栽培大豆和野生大豆TTT、TTA、CTT、ATT、GTA、TCC、CCT、ACT、ACA、GCT、CAA、AAT、AAA、GAT、GAA、CGT、AGA、GGT18个共有的最优密码子。此外,确定了CAT和CGA为栽培大豆的最优密码子;GTT、TGT和GGA为野生大豆的最优密码子。以上最优密码子中以A结尾有9个,以T结尾的有13个,以C结尾的有1个,没有以G结尾的最优密码子。暗示了大豆线粒体基因组偏好于使用A或T结尾的密码子(表4)。
3 讨论与结论
根据内共生理论,线粒体由ɑ-蛋白质细菌祖先进化(Roger et al.,2017)。作为半自主型的细胞器,线粒体是细胞进行有氧呼吸的主要场所,其有自身的遗传表达系统,但大多数定位于线粒体的功能蛋白(>95%)是由核基因编码的(Giegé et al.,2005)。由于其含有相对独立的细胞质遗传信息,故线粒体基因组亦在揭示物种间亲缘关系和物种进化等方面具有重要价值。通过比较核基因和线粒体基因在密码子使用模式上的差异,可以预测真核生物核糖体在细胞内的定位。此外,研究线粒体密码子的使用特征可以更好地了解线粒体基因组的进化过程及主要影响因素。
同义密码子的使用偏好性是物种长期演化的结果,形成这种偏好性的主要动力是自然选择和突变(Duret,2002)。突变和选择等多重因素共同影响了大豆线粒体基因密码子使用偏好性的形成。比较栽培大豆和野生大豆的ENC-plot、中性绘图和PR2-plot等分析,结果暗示了它们具有相似的密码子使用模式,这是因为两个物种在进化和亲缘关系上较近(Grantham et al.,1980)。但两个物种在密码子使用模式上也存在一定的差异。按同一标准判断它们有相同的最优密码子,也有各自不同的最优密码子。
本研究确认了栽培大豆的20个和野生大豆的21个最优密码子。其中18个最优密码子为两个物种共有,且多以A或T碱基结尾。栽培大豆和野生大豆的线粒体编码基因在密码子第3位的GC含量分别为41.64%、 41.92%。这与许多高等植物的叶绿体基因组第3位密码子偏好使用A或T碱基相似(Zhou et al.,2008;尚明照等,2011;傅建敏等,2017;原曉龙等,2019)。而与线粒体基因不同,大豆的核基因最优密码子被确定为UCC和GCC(张乐等,2011),暗示了大豆的核基因和线粒体基因在密码子使用偏性上的差异。按照同样的最优密码子计算方法,经比较分析,普通野生稻和栽培大豆共有的线粒体基因组最优密码子为15个,普通野生稻和野生大豆共有的线粒体基因组最优密码子为16个(金刚等,2019)。推测这并非偶然,而是植物线粒体基因组表达系统在进化中保守性的体现。尚需对其他高等植物的线粒体密码子进行归纳研究后得出结论。而非最佳密码子可能更多地与其二级结构相关(Chaney et al.,2017)。按照现代的植物细胞质雄性不育(cytoplasmic male sterility,CMS)发生理论,细胞质雄性不育的发生与线粒体基因组的重组变异有关(Tang et al.,2017)。普通野生稻线粒体基因组中细胞质雄性不育相关基因CW-orf307与其线粒体基因组的密码子偏好性有所不同,TCT、CCT、ACA、TTT和CAT等在线粒体基因组中被认定的最优密码子在CW-orf307基因中的使用频率并不高,暗示了CW-orf307独特的起源(金刚等,2019)。但野生大豆中的相关情况尚需对其线粒体基因组中的细胞质雄性不育基因进行鉴定并分析后才能确定。本研究通过分析比较野生大豆和栽培大豆线粒体基因组编码基因的密码子使用特征,将有助于进一步加深对大豆属植物的线粒体基因表达系统进化的理解。
参考文献:
CHANEY JL,STEELE A,CARMICHAEL R,et al.,2017. Widespread position-specific conservation of synonymous rare codons within coding sequences[J]. PLoS Comput Biol,13(5):e1005531.
CHANEY JL,CLARK PL,2015. Roles for synonymous codon usage in protein biogenesis[J]. Ann Rev Biophys,44(1):143-166.
CHANG S,WANG Y,LU J,et al.,2013. The mitochondrial genome of soybean reveals complex genome structures and gene evolution at intercellular and phylogenetic levels[J]. PLoS ONE,8(2):e56502.
FU JM,SUO YJ,LIU HM,et al.,2017. Analysis on codon usage in the chloroplast protein-coding genes of Diospyros spp.[J]. Nonwood For Res,35(2):38-44.[傅建敏,索玉静,刘慧敏,等,2017. 柿属植物叶绿体蛋白质编码基因密码子用法[J]. 经济林研究,35(2):38-44.]
GALTIER N,NABHOLZ B,GLEMIN S,et al.,2009. Mitochondrial DNA as a marker of molecular diversity:A reappraisal[J]. Mol Ecol,18(22):4541-4550.
GIEGé P,SWEETLOVE LJ,COGNAT V,et al.,2005. Coordination of nuclear and mitochondrial genome expression during mitochondrial biogenesis in Arabidopsis[J]. Plant Cell,17(5):1497-1512.
PUIGBO P,BRAVO IG,GARCIA-VALLVE S,2008. CAIcal:a combined set of tools to assess codon usage adaptation[J]. Biol Direct,3:38.
QIU S,ZENG K,SLOTTE T,et al.,2011. Reduced efficacy of natural selection on codon usage bias in selfing Arabidopsis and Capsella species[J]. Genome Biol Evol,3:868-880.
ROGER AJ,MUOZ-GóMEZ SA,KAMIKAWA R,2017. The origin and diversification of mitochondria[J]. Curr Biol,27(21): R1177-R1192.
SAJJAD A,LATIF KA,AHMED AH,et al.,2018. The first complete mitochondrial genome of wild soybean (Glycine soja)[J]. Mitochondrial DNA Part B,3(2):527-528.
SUEOKA N,1999. Translation-coupled violation of Parity Rule 2 in human genes is not the cause of heterogeneity of the DNA G+C content of third codon position[J]. Gene(Amsterdam),238(1):1-58.
SUEOKA N,1988. Directional mutation pressure and neutral molecular evolution[J]. Proc Natl Acad Sci USA,85(8):2653-2657.
SUN Z,WAN D,MURPHY RW,et al.,2009. Comparison of base composition and codon usage in insect mitochondrial genomes[J]. Genes Genom,31(1):65-71.
TANG H,XIE Y,LIU YG,et al.,2017. Advances in understanding the molecular mechanisms of cytoplasmic male sterility and restoration in rice[J]. Plant Reprod,30(4):179-184.
WANG B,LIU J,JIN L,et al.,2010. Complex mutation and weak selection together determined the codon usage bias in bryophyte mitochondrial genomes[J]. J Integr Plant Biol,52(12):1100-1108.
WANG L,XING H,YUAN Y,et al.,2018. Genome-wide analysis of codon usage bias in four sequenced cotton species[J]. PLoS ONE,13(3):e0194372.
WEI L,HE J,JIA X,et al.,2014. Analysis of codon usage bias of mitochondrial genome in Bombyx moriand its relation to evolution[J]. BMC Evol Biol,14(1):262.
WEI SJ,LI Q,VAN ACHTERBERG K,et al.,2014. Two mitochondrial genomes from the families Bethylidae and Mutillidae:Independent rearrangement of protein-coding genes and higher-level phylogeny of the Hymenoptera[J]. Mol Phylogenet Evol,77:1-10.
WRIGHT F,1990. The ‘effective number of codons used in a gene[J]. Gene,87(1):23-29.
XIA YL,2003. The study on mitochondrial genome of B. mandarina in China[D]. Chongqing:Southwest Agircultural University: 2-10.[夏玉玲,2003. 中国野桑蚕线粒体基因组研究[D]. 重庆:西南农业大学:2-10.]
YUAN XL,LI YQ,ZHANG JF,et al.,2019. Analysis of codon usage bias in the chloroplast genome of Dalbergia odorigera[J]. Guihaia:1-9[2019-11-20]. https://kns.cnki.net/kcms/detail/45.1134.Q.20190918.1651.1008.html.[原晓龙,李云琴,张劲峰,等,2019. 降香黄檀叶绿体基因組密码子偏好性分析[J]. 广西植物:1-9[2019-11-20]. https://kns.cnki.net/kcms/detail/45.1134.Q.20190918.1651.1008.html.]
ZHANG L,JIN LG,LUO L,et al.,2011. Analysis of nuclear gene codon bias on soybean genome and transcriptome[J]. Acta Agr Sin,37(6):965-974.[张乐,金龙国,罗玲,等,2011. 大豆基因组和转录组的核基因密码子使用偏好性分析[J]. 作物学报,37(6):965-974.]
ZHOU M,LI X,2009. Analysis of synonymous codon usage patterns in different plant mitochondrial genomes[J]. Mol Biol Rep,36(8):2039-2046.
ZHOU M,LONG W,LI X,2008. Patterns of synonymous codon usage bias in chloroplast genomes of seed plants[J]. Fores Stud China,10(4):235-242.
(责任编辑 何永艳)

