基于转录组序列的羊肚菌EST-SSR标记开发与遗传多样性分析
马杰 屈雯 陈春艳 王磊 马维 刘针杉 马俊 杨珊 丁丽 高强 孙勃


摘要:利用羊肚菌(Morchella spp.)轉录组数据开发EST-SSR(表达序列标签-简单重复序列)标记,并对33份羊肚菌材料进行遗传多样性分析。结果表明,转录组测序共获得73 781条Unigene序列,其中25 461条Unigene序列中含有41 814个简单重复序列(Simple sequence repeats, SSR)位点,SSR位点发生频率为34.51%,平均分布距离为2.51 kb。优势重复序列类型为单核苷酸,占总SSR位点数量的51.39%,其次为三核苷酸和二核苷酸,分别占总SSR位点数量的28.04%和13.35%。A/T、AG/TC、AGC/TCG分别是单核苷酸、二核苷酸、三核苷酸的优势重复基元。以33份羊肚菌和1份酵母菌为材料,从61对SSR引物中筛选出15对多态性引物,经扩增得到130条多态性条带,多态性比例为100%。遗传多样性分析结果显示,平均每个位点的等位基因数(Na)为4.866 7个,平均有效等位基因数(Ne)为2.124 2个,平均多样性指数(I)为0.958 3,平均观察杂合度(Ho)为0.191 1,平均期望杂合度(He)为0.495 1,平均Neis基因多样性指数为0.483 5,表明筛选出的15对SSR引物的扩增产物具有较好的遗传多样性。通过非加权组平均法(Unweighted pair-group method with arithmetic means, UPGMA)聚类分析,可将33份羊肚菌材料分为4类。试验结果可为羊肚菌的种质资源鉴定、品种鉴别和分子标记辅助育种等研究提供依据。
关键词:羊肚菌;分子标记;EST-SSR;转录组;遗传多样性
中图分类号:S646.701文献标识码:A文章编号:1000-4440(2020)05-1282-09
Abstract:Expressed sequence tag-simple sequence repeat (EST-SSR) markers were developed using transcriptome data of Morchella spp., and the genetic diversities of 33 varieties of Morchella spp. were analyzed. The results showed that a total of 73 781 Unigene sequences were obtained using transcriptome sequencing. There were 41 814 simple sequence repeats (SSR) loci in 25 461 Unigene sequences, the occurrence frequency of SSR locus was 34.51%, the average distribution distance was 2.51 kb. Single nucleotide was the advantage repetitive sequence type, which accounted for 51.39% of the total SSR loci, followed by tri-nucleotides and di-nucleotides, which accounted for 28.04% and 13.35%, respectively. A/T, AG/TC, AGC/TCG were the predominant repetitive motifs in single nucleotides, di-nucleotides, tri-nucleotides, respectively. 15 pairs of polymorphic primers were selected from 61 pairs of SSR primers, using 33 varieties of Morchella spp. and one variety of yeast as material. 130 polymorphic bands were obtained through amplification, with a polymorphic rate of 100%. Results of genetic diversity analysis showed that the average number of alleles (Na) was 4.866 7, the average number of effective alleles (Ne) was 2.124 2, the average diversity index (I) was 0.958 3, the average observed heterozygosity (Ho) was 0.191 1, the average expected heterozygosity (He) was 0.495 1, and the average Neis gene diversity index was 0.483 5, indicating that the amplified products of the selected 15 pairs of SSR primers had a good genetic diversity. 33 varieties of Morchella spp. could be divided into four groups by unweighted pair-group method with arithmetic means (UPGMA) cluster analysis. The results can provide basis for germplasm resources identification, variety identification and molecular marker assisted breeding of Morchella spp.
Key words:Morchella spp.;molecular marker;expressed sequence tag-simple sequence repeats (EST-SSR);transcriptome;genetic diversity
羊肚菌(Morchella spp.)属于子囊菌门(Ascomycata),盘菌纲(Pezizomycetes),盘菌目(Pezizales),羊肚菌科(Morchellaceae),羊肚菌属(Morchella),由于其菌盖表面凹凸不平,形如羊肚,故名羊肚菌[1]。羊肚菌肉质脆嫩、风味独特、味道鲜美、营养丰富[2],在全球范围内主要分布在法国、德国、美国、印度、中国等地,在中国主要分布在中西部地区,如贵州、云南、四川等地。
简单重复序列(Simple sequence repeat, SSR),又称微卫星DNA(Microsatellite DNA),是一类由16个碱基组成的基序串联重复而成的DNA序列[3]。SSR因具有共显性、数量丰富、多态性高、重复性好、易检测、操作简单等优点[4]而被广泛应用于植物遗传多样性分析[5]、遗传图谱构建[6]及亲缘关系研究[7]等领域。根据SSR的来源不同,可以将其分为基因组SSR和EST(Expressed sequence tag,EST,表达序列标签)-SSR两大类型,与基因组SSR标记相比,由于EST-SSR是基于EST序列开发的,来源于表达的基因组区域,可以直接反映相关基因的多样性,因而在不同物种之间具有良好的通用性[8-10]。此外,由于转录组序列多集中在功能基因上,因此基于转录组数据开发的SSR标记有利于后期与重要农艺性状进行关联分析,从而为分子标记辅助育种节约时间和成本。
目前,EST-SSR在蔬菜[7]、果树[11]、花卉[12]等许多植物中都有应用,但尚未见将其应用于羊肚菌的相关报道。本研究利用转录组数据开发出羊肚菌EST-SSR标记,并对其多样性进行分析,旨在筛选适合羊肚菌SSR标记分析的核心引物,为羊肚菌种质资源遗传多样性评价和亲缘关系分析提供引物。
1材料与方法
1.1试验材料
本试验采集了33份羊肚菌材料和1份酵母菌材料作为提取DNA模板的原材料,材料的相关信息见表1。
1.2试验方法
1.2.1转录组测序采集不同品种羊肚菌的各组织,进行等量混合后送至北京诺禾致源公司进行转录组测序。
1.2.2基因组DNA的提取采集各个材料样品,用液氮冷冻后研磨,采用改良的十六烷基三甲基溴化铵(CTAB)法提取样品的基因组DNA[13],并于-20 ℃储存备用,用1.0%琼脂糖凝胶电泳检测DNA的质量。
1.2.3引物设计与合成采用MISA软件(http://pgrc.ipk-gatersleben.de/misa/misa.html)对羊肚菌转录组的Unigene基因进行搜索。分别设定单核苷酸、二核苷酸、三核苷酸、四核苷酸、五核苷酸、六核苷酸的最少重复搜索次数为10次、6次、5次、5次、5次、5次。用Primer 3进行引物设计,引物序列长度为18~27 bp,预计扩增产物长度为100~300 bp,G+C含量为40%~60%,退火温度为54~61 ℃。随机挑选出设计的61对引物送至生工生物工程(上海)股份有限责任公司进行合成。
1.2.4PCR擴增及检测20 μl PCR反应体系如下:1 μl DNA模板,10 μl 2×EasyTaq PCR SuperMix for PAGE,各1 μl上游、下游引物,用去离子水补足体积至20 μl。PCR反应扩增条件如下:95 ℃预变性 2 min;98 ℃变性10 s,49~54 ℃退火30 s,72 ℃延伸15 s,32个循环;72 ℃延伸10 min。初筛用1.5%琼脂糖凝胶电泳进行检测,复筛用8%非变性聚丙烯酰胺凝胶电泳进行检测。
1.2.5数据统计采用人工读带的方法,将聚丙烯酰胺凝胶电泳图上可重复的清晰条带的记为“1”,在同一位置条带较弱或无条带的记为“0”,由此建立原始数据矩阵。用POPGENE 1.31和PIC-CALC软件进行SSR位点的遗传多样性分析,用NTSYS-PC 1.0软件进行供试材料的聚类分析。
2结果与分析
2.1羊肚菌转录组中SSR位点的分布及特点
通过对羊肚菌的73 781条Unigene序列(总长度约为104 910 kb)进行搜索发现,在25 461条Unigene序列中含有41 814个SSR位点,其中7 306条Unigene含有2个或2个以上SSR位点。整体上看,SSR位点的发生频率为34.51%,即平均每2.51 kb出现1个SSR。单核苷酸重复是主要类型,占总SSR位点数的51.39%;其次是二核苷酸重复、三核苷酸重复,分别占总SSR位点数的13.35%、28.04%;四核苷酸重复、五核苷酸重复和六核苷酸重复类型数量较少,分别占总SSR位点数的4.56%、1.10%和1.56%。在所有SSR位点中,重复10次的SSR位点数最多,共有6 658个,占总数的15.92%;重复5次的SSR位点数次之,共有6 396个,占总数的15.30%;重复6次的SSR位点共有5 426个,占总数的12.98%(表2)。
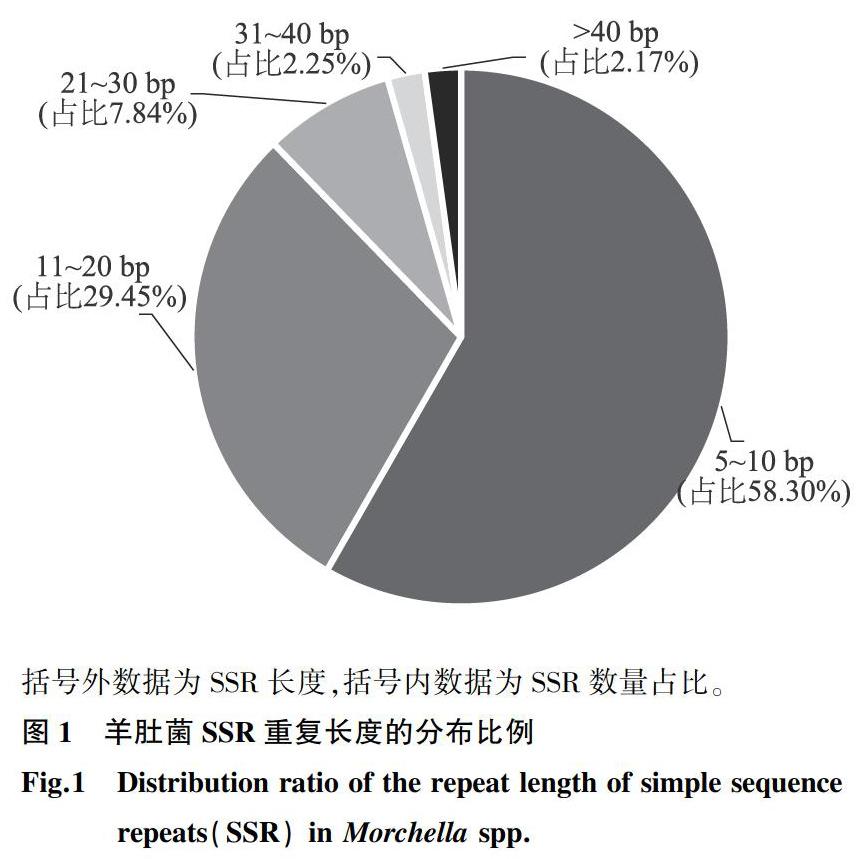
羊肚菌的平均SSR位点长度为18.63 bp,其中六核苷酸重复序列的平均长度最长,达到38.94 bp。羊肚菌的SSR长度范围为5~114 bp,其中长度为5~10 bp的重复序列数最多,共有24 377个,占总SSR位点数的58.30%;长度为11~20 bp的重复序列数次之,共有12 313个,占总SSR位点数的29.45%;长度大于40 bp的重复序列数最少,仅占总SSR位点数的2.17%(图1)。
在本试验中,利用15对SSR引物对来自7个省份的33份羊肚菌材料和1份酵母菌材料进行PCR扩增,结果表明,扩增条带的多态性比例为100.0%,高于黑木耳(95.5%)[24]、野生香菇(95.9%)[25]、真姬菇(81.2%)[26]。15对SSR引物扩增产物的遗传多样性分析结果表明,这些羊肚菌材料具有丰富的遗传多样性。徐锐[25]利用34对高多态性引物对94个香菇菌株进行遗传多样性分析,结果表明,I、Ho、PIC的平均值分别是0.89、0.24、0.40。张跃新等[24]研究发现,黑木耳SSR标记的平均PIC、I分别为0.216、0.455,这些结果都略低于本试验结果,表明本试验开发的羊肚菌的SSR多态性较好。一些真菌的遗传多样性高于本试验结果,如灰树花的平均PIC为0.52[15],金针菇的平均PIC为0.801 9[21],这些现象可能与物种的差异有关。本研究通过对33份羊肚菌材料进行聚类分析,结果共分为4大类,来自贵州毕节的11个材料在这4类中均有分布,说明这些来自贵州毕节的羊肚菌材料受所在地区羊肚菌基因渗透的影响较小。而来自云南昆明、贵州贵阳和四川成都的大部分材料都各自聚类在同一小组,说明这些材料在区域内没有发生明显的遗传分化,由此表明聚类分析结果与地理位置有一定关系。
4结论
本研究基于转录组数据成功开发了羊肚菌SSR位点,共筛选获得15对核心引物,并将其成功应用于33个羊肚菌的遗传多样性和聚类分析中。研究结果丰富了羊肚菌EST-SSR标记引物库,为今后羊肚菌的种质资源鉴定、物种鉴别、分子标记辅助育种、亲缘关系分析等研究提供了引物支持和实践参考。
參考文献:
[1]谢占玲,谢占青. 羊肚菌研究综述[J].青海大学学报(自然科学版), 2007,25(2):36-40.
[2]倪淑君,张海峰. 我国羊肚菌的产业发展[J].北方园艺, 2019(2): 165-167.
[3]杨梦婷,黄洲,干建平,等. SSR分子标记的研究进展[J].杭州师范大学学报(自然科学版), 2019, 18(4): 429-436.
[4]GARCIA-LOR A, CURK F, SNOUSSI-TRIFA H, et al. A nuclear phylogenetic analysis: SNPs, indels and SSRs deliver new insights into the relationships in the ‘true citrus fruit trees group (Citrinae, Rutaceae) and the origin of cultivated species[J].Annals of Botany,2013,111(1): 1-19.
[5]苏一钧,王娇,霍恺森,等.甘薯引进种SSR遗传多样性分析[J]. 江苏农业学报, 2018, 34(5):984-997.
[6]BHATTACHARJEE M, PRAKASH S H, ROY S, et al. SSR-based DNA fingerprinting of 18 elite Indian varieties of sesame (Sesamum indicum L.)[J]. Nucleus, 2020, 63(1): 67-73.
[7]林珲,薛珠政,裘波音,等. SSR标记在茄子杂交种纯度检验中的应用[J/OL].分子植物育种,2020. http://kns.cnki.net/kcms/detail/46.1068.S.20200310.1700.006.html.
[8]王玲玲,陈东亮,黄丛林,等. SSR分子标记技术在植物研究中的应用[J].安徽农业科学,2017,45(36):123-126,130.
[9]POWELL W, MACHRAY G C, PROVAN J. Polymorphism revealed by simple sequence repeats[J].Trends in Plant Science,1996,1(7):215-222.
[10]SCOTT KD, EGGLER P, SEATON G, et al. Analysis of SSRs derived from grape ESTs[J]. Theoretical and Applied Genetics, 2000,100(5): 723-726.
[11]文舜,陈秀萍,郑少泉.龙眼EST-SSR标记开发及无患子科5个属种质遗传多样性分析[J].园艺学报,2019, 46(7): 1359-1372.
[12]贺 丹,吴芳芳,张佼蕊,等.牡丹转录组SSR信息分析及其分子标记开发[J].江苏农业学报,2019,35(6):1428-1433.
[13]孙立夫,张艳华,裴克全. 一种高效提取真菌总DNA的方法[J]. 菌物学报, 2009, 28(2): 299-302.
[14]李明卫. 大型真菌黑松露EST-SSR信息分析[D]. 临安: 浙江农林大学, 2012.
[15]杨杨. 灰树花EST-SSR引物开发及遗传多样性研究[D]. 长春: 吉林农业大学, 2016.
[16]李强,陈诚,熊川,等.杏鲍菇转录组数据SSR位点的生物信息学分析[J]. 应用与环境生物学报, 2017, 23(3): 454-458.
[17]孟清,谢占玲,戴大日,等.青海小海绵羊肚菌M12-10转录组SSR信息分析及其分子标记开发[J].青海大学学报, 2019, 37(6): 1-10.
[18]刘伟,蔡英丽,何培新. 粗柄羊肚菌转录组的SSR分布和序列特征分析[J].轻工学报, 2017, 32(2): 33-39.
[19]鲁铁. 蒙古白丽蘑的遗传多样性及其保育学研究[D]. 长春: 吉林农业大学, 2018.
[20]万志兵,王小龙,管宏伟,等. 杨树锈菌表达序列微卫星分析及EST-SSR标记开发[J].东北林业大学学报, 2012, 40(6): 76-80.
[21]金群力,沈颖越,蔡为明,等. 金针菇基因组SSR位点分析及标记开发[J].食用菌学报,2016,23(2): 12-19.
[22]王莹,陈明杰,汪虹,等. 美味牛肝菌全基因组SSR位点的分布规律研究[J].菌物学报, 2015,34(2): 204-214.
[23]徐凯,张劲松,唐传红,等. 灵芝基因组SSR位点分布与分析[J].上海农业学报, 2014, 30(5): 32-37.
[24]张跃新,胡伟,郝艺铭,等. 北方地区黑木耳部分主栽品种与野生菌株遗传多样性SSR分析[J].中国食用菌, 2019, 38(5): 44-48.
[25]徐锐. 野生香菇数量性状与SSR分子标记的关联分析[D].武汉: 华中农业大学, 2013.
[26]董岩,陈辉,赵明文,等. 真姬菇栽培菌株的ITS和SSR分析[J].上海农业学报, 2009, 25(3): 59-64.
(责任编辑:徐艳)

