蜂窝状数字PCR 微阵列荧光图像的信息提取
李树力,李金泽,郭 振,3,朱文艳,3,周连群,3*,张芷齐,3*
(1. 上海大学 通信与信息工程学院,上海200444;2. 中国科学院苏州生物医学工程技术研究所 中国科学院生物医学检验技术重点实验室,江苏 苏州215163;3. 中国科学技术大学,安徽 合肥230026)
1 引 言
数字聚合酶链式反应(digital Polymerase Chain Reaction,dPCR)技术在循环肿瘤DNA 检测、稀有突变检测和拷贝数变异分析等核酸定量分析中有着广泛的应用[1-2]。1999 年,Kinzler 和Vogelstein 等 提 出 了 数 字PCR 的 概 念[3]。 数 字PCR 实验包含3 个环节,即样本的分散、PCR 扩增、荧光信号的采集与数据分析。 在数据分析时,利用泊松分布可以精确计算得到原始样本的模板拷贝数[4-5]。影响最终结果的关键在于对荧光信号的采集与处理,即读取各个反应单元的荧光信号并判断其阴性或阳性。
随着光刻与MEMS 技术的发展[6],芯片式数字PCR 得以广泛应用,解决了微升级样品到纳升级反应单元的精准化分问题[7]。 芯片式数字PCR 相比于液滴式数字PCR,图像信息获取更方便。数字PCR 芯片图像的特征是由大量的形状类似的样点组成的阵列,它们之间的排列一般都是规则的,处理这种微阵列图像一般有三个步骤:样点定位、样点分割和信号强度提取[8-10]。其中,样点定位是微阵列图像分析中最基本、最重要的步骤,准确的样点定位大大提高了后续分割与量化步骤的效率。Saeid 等[11]提出一种局部阈值网格化的方法。Nguyen 等[12]提出一种基于小波检测、主动轮廓分割的方法。李铁军等[13]利用一种基于四阶矩阵的对比度增强的方法,提高样点定位的准确性。Belean 等[14]提出一个基于偏微分方程的微阵列网格对齐的方法。牟颖等[15]使用最大类间方差法来提取信息。这些方法均基于投影法,容易实现,加以改进也可以达到不错的定位结果,但是它们只适用于矩形排列方式的微阵列芯片图像。而近些年,出现了一些类似蜂窝状的六边形排布方式的数字PCR 芯片[16]。与矩形网格相比,蜂窝状网格在芯片上的利用率更高,在相同的面积里,蜂窝状的排布方式可以设计更多的样点位。对于这种非正交排列的阵列图像,投影法无法分隔所有的样点。Genpix[17]和ScanAlyze[18]等软件包使用固定模板的方法,预先设置了几种不同排布方式的模板,分析数据前,让用户手动选择,然后将模板对准嵌套在采集到的芯片图像上,利用设定好的坐标定位每个样点的位置。这种方法的局限性在于:图片处理中需要人工参与,无法处理和预设模板排列方式不同的芯片。 Giannakeas 等[19]提出了一个生长同心六边形算法,利用Voronoi 图[20]对样点定位。这种方法可以处理蜂窝状排布方式的芯片图像,但是由于是逐点定位,计算量过于庞大,不适用于处理数据量大的数字PCR 芯片图像[21-22]。由相机拍摄采集到的芯片图像除了存在光照分布不均匀的问题,其样点的信号强度一般也非常低,这导致图像整体灰度值集中在一个比较低的范围,信号与背景的对比度低。如果不进行特定的运算处理,直接采用全局固定阈值分割的方法,无法准确地分割出每个有效样点。有一些方法对图像对比度进行了改进[23],其中最简单和有用的是自适应直方图均衡化方法[24]。然而,这些方法都试图将原始图像分割成小块并调节增强程度,必然涉及到参数设置和阈值搜索,信号和亮度会被破坏。
针对目前任意排布方式的数字PCR 芯片,本文提出基于形态学的三通道蜂窝状荧光图像样点寻址算法及数字PCR 图像信息提取方法。该方法包含三通道图像配准、样点的寻址定位(Automatic Hexagon Microarray Addressing Algorithm,AHMAA)、样点分割、信息提取以及对样点的筛选,能够快速、准确地获取芯片多个通道的信息。
2 实验材料
实验采用Applied Biosystems™QuantStudio ™3D的数字PCR 芯片[25]。该芯片是一种基于硅基基片的表面疏水孔壁亲水的毛细管微阵列芯片,使用硅基底板制造并在其上蚀刻有约20 000 个大小均一的微米级反应孔。芯片上的微孔以蜂窝状网格的方式排列,微孔直径为60 μm,孔间隔15 μm,结构如图1 所示。使用数字PCR 检测系统在明场下拍摄芯片整体图像,芯片尺寸为10 mm×10 mm,其中局部放大部分是使用扫描电镜拍摄的芯片图像,可以清晰地观察到六边形结构。
PCR 实验使用到的试剂主要有质粒DNA(生工生物工程股份有限公司),TaqManTMLiquid Biopsy dPCR Assay(Thermofisher),Quant-Studio ™3D Digital PCR Master Mix(Thermofisher)等。
芯片图像由本实验室自主研制的数字PCR检测系统采集。本系统主要基于CMOS 成像原理,有多个荧光通道,可同时采集多个通道的荧光信号。本文使用的数据是5 块不同的PCR 芯片 ,分 别 在FAM ,VIC ,ROX(校 正)3 个 通 道采集图片,每张图片的规格为8 位2 048×2 448 pixel。

图1 数字PCR 芯片结构Fig. 1 Structure of digital PCR chip
3 图像处理
为了快速准确地处理三通道蜂窝状堆叠的微孔阵列芯片图像,本文提出基于形态学的三通道蜂窝状荧光图像样点寻址算法及数字PCR 图像信息提取方法。图2 展示了本文的整个数字PCR 芯片图像处理流程。
该方法主要分为四个步骤,第一步:三通道的图像配准,针对不同通道的图像进行配准融合,使样点排布整齐;第二步:样点寻址(AHMAA),通过增强图像的对比度,选取有效样点区域,基于改进伽马算法去除光照分布不均效应,基于形态学算法对紧密排列样点进行识别,对微阵列芯片图像进行单元定位;第三步:样点分割,以样点坐标为中心,选择符合样点结构形状的区域作为信息提取的区域;第四步:样点的信息提取,提取每个样点的生物分子荧光信息。
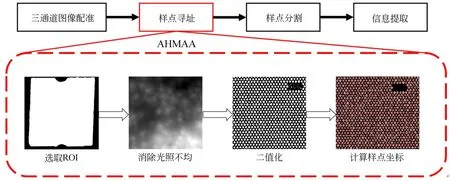
图2 数字PCR 微阵列荧光图像信息提取流程Fig. 2 Flow chart for information extraction of digital PCR microarray fluorescence image
3.1 三通道图像配准
实验室研发的数字PCR 图像采集系统可以同时采集芯片在3 个不同波段的荧光图像,为了同时获取多个通道的图片信息,本文将不同通道的图片进行融合处理。由于图像采集系统中存在光学误差,同一个样点在不同通道里的位置并不是完全重合的,有几个像素的偏差,进而导致后续获取样点数据的不准确。为了解决这一问题,本文采用了基于互信息的多模态图像配准方法,将不同通道的样点对齐。互信息量度是用于测量两个变量之间的相关性的信息理论技术。算法使用来自两个图像的像素采样的联合概率分布来测量一组像素的值映射到另一图像中的相似值的确定性,该信息是图像相似程度的定量度量。高互信息意味着两个分布之间的不确定性(熵)大大降低,表明图像更好地被配准[26]。
3.2 样点寻址
图像配准完成后,得到一张包含多个通道信息的融合图片,由于多个通道的图像已经配准,所以对其中一个通道的图像做寻址,得到样点坐标可以直接用于其他通道的图像。为了准确地定位样点,针对芯片图像存在的问题,本文对图像进行了以下几步处理:
(1)提高图像整体对比度,选择ROI;
(2)去除光照不均影响;
(3)增强样点孔内外的对比度、二值化;
(4)计算统计样点的位置信息。
图像采集系统采集到的芯片图像,除了芯片部分,还包含了芯片载具的部分,需要将芯片以外的部分都去掉。本文在提取边缘之前,先对图像直方图进行均值化处理,增强芯片区域与背景之间的对比度;然后,模糊处理,去除芯片区域内细小边缘的干扰;最后边缘提取,得到准确的外边界。
针对上一步提取的芯片主体区域,本文使用基于形态学的方法,去除芯片内部的连通区域;采用多次提取边缘并减去的方法,将整个芯片主体区域的边缘部分去掉。最终得到只含有有效样点的区域,作为下一步处理的感兴趣区域(Region of Interest,ROI)。
在采集芯片图像的过程中,由于受到激发光源大小、镜头视野等外部因素的影响,芯片上的光照不均匀,主要表现为芯片中间区域的光照强度比芯片边缘区域的光照强度更高[27]。这会导致样点的分割结果不理想,阴阳性样点的分类结果不准确等问题。
本文使用改进的基于二维γ 函数的光照校正算法来校正芯片的光照分布。该算法的主要思想是提取图片的光照分量信息,通过校正使得光照过亮的区域整体亮度降低,光照过暗的地方亮度得到提高,提升整块芯片的光照亮度均匀性。为了防止直接在RGB 3 个通道做校正导致图像色彩失真,选择在HSV 色彩空间进行处理[26],其中色调(H)、饱和度(S)、亮度(V)是相互独立的。
算法主要步骤如下:
(1)输入芯片图像IRGB,将图像从RGB 空间转化到HSV 空间IHSV,单独对亮度(V)处理;
(2)多尺度高斯卷积,赋予不同尺度不同的权值,得到芯片图像的光照分量(gaus);
(3)根据二维γ函数公式校正,得到校正后的亮度(V ′);
(4)将校正后的亮度(V ′)与原图的色调(H)、饱和度(S)重新合成,得到新的图像I ′HSV;
(5)将图像从HSV 空间转化到RGB 空间,得到I ′RGB。
二维γ函数公式为:

为了增强样点与周围环境,即芯片上孔内外的对比度,本文提出一种基于顶帽变换与底帽变换增强对比度的算法。顶帽变换与底帽变换都是基于数学形态学的图像处理方法[28]。
灰度图像f的顶帽变换定义为f减去其开运算结果,即:

在设置合适尺寸的构造元素后,可以得到比较理想的孔内与孔外的图像信息。在得到顶帽与底帽变换之后的图像后,将二者做差,可以大幅增强样点孔内与孔外的对比度。这种方法可以保证只有样点被增强,而不受噪声的干扰。
对上一步结果进行二值化处理,去掉面积过小与过大的连通域,得到一幅包含所有有效样点的二值化点阵。计算统计所有连通域的中心点的坐标,将它作为样点的位置信息,至此样点的寻址定位步骤完成。
3.3 样点分割
理论上,微孔阵列芯片的每个样点孔都有固定的空间结构和形状,而使用AHMAA 算法得到的定位结果是每个样点的中心点。当我们得到了每个样点的中心坐标,则以定位点坐标为中心,划取一个样点大小的正六边形区域作为信息采集区域。这种直接分割的方法比先网格化定位,再对所有网格小碎片里的样点分割的方法更快速、准确。
3.4 信息提取
计算每个六边形区域内所有像素点的均值,作为样点的荧光强度值。统计得到所有样点的荧光信号值后,本文采用基于K 均值的分类方法对数据分类,得到芯片的阴阳性点数目信息。
4 结果与讨论
图3 是通过AHMAA 算法最终定位的结果。图中视野内所有样点无论是阳性还是阴性,都被成功地识别并准确地分割出来。

图3 样点定位结果Fig. 3 Result of spot addressing
图4 展示了荧光通道1 与荧光通道2(FAMvs. VIC)的二维聚类散点图。根据计算得出的阈值将图像分为4 个象限,并将样点分为4 类,分别代表FAM 阳性与VIC 阴性、FAM 阳性与VIC阳 性 、FAM 阴 性 与VIC 阴 性 、FAM 阴 性 与VIC阳性。
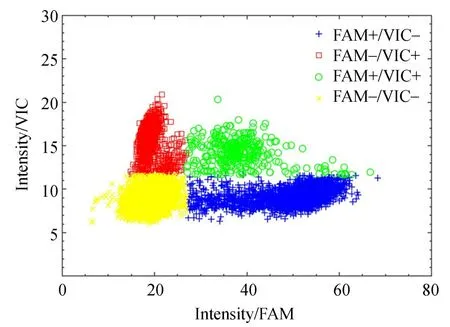
图4 二维聚类散点Fig. 4 Two-dimensional clustering scatter plots
4.1 三通道图像配准
由于没有一个准确描述两幅图像对齐性的量,因此很难对配准结果进行定量分析。通常通过可视化的结果来定性地判断配准效果。图5 显示了三通道融合图像配准前后的效果(彩图见期刊电子版)。在配准前,由红色和绿色代表的两张不同通道的图片有明显的错位现象,导致同一个样点在不同通道图片上的坐标位置不同;配准后,绿色的圆圈与红色的圆重合了。
4.2 对比度增强的作用
为了提取芯片的主体区域,需要准确地识别芯片的边缘范围,直接使用传统的边缘提取方法,会受到大量的样点边缘信息干扰。所以,需要先提高图像的整体对比度。
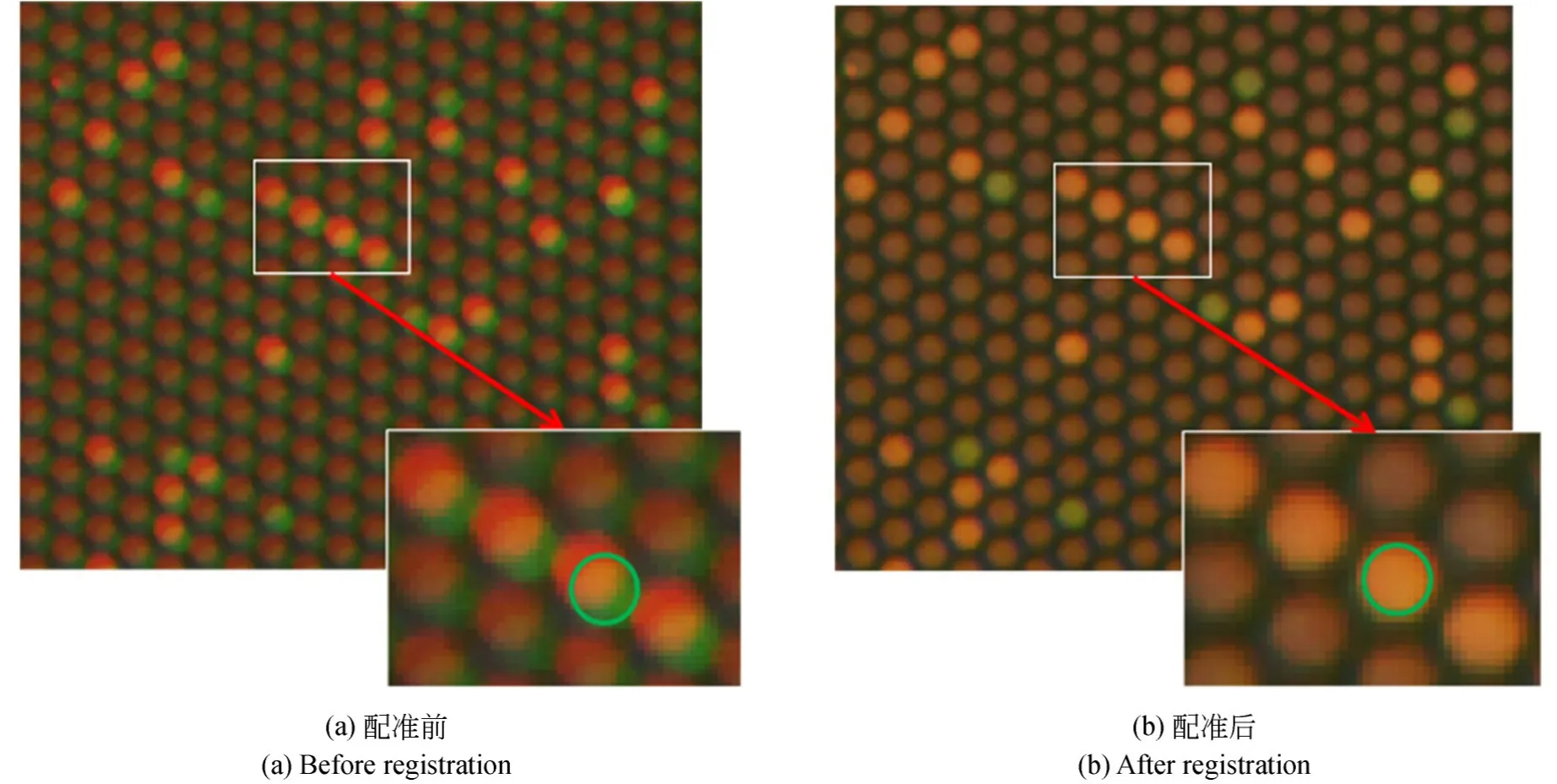
图5 三通道芯片的图像配准Fig. 5 Image registration of multi-channel chip
图6 展示了整体对比度增强前后,图片的灰度直方图分布。原图的灰度值集中在0~100,图片整体亮度非常暗,而且几乎分辨不出芯片区域的边界;处理后图片的灰度值均匀地分布在0~255,边界非常明显,为后续ROI 的提取打下了很好的基础。
4.3 光照不均匀对结果的影响
在实际实验中,经常遇到一些光照不均匀的图片,如果不加以处理,则会对后续样点的识别和定位有很大影响;除此之外,光照不均也会对获取到的样点信号值有影响,导致亮度高的区域信号值偏高,影响阴阳性分类结果。
图7 显示了去除光照不均影响前后的差别,图7(a)是待处理的图片,图7(b)是没有经过处理的图像分割结果,可以从放大的局部细节7(e),7(f)中看到,在原图中过暗或过亮的区域,都无法很好地分割和识别样点。图7(c)是图7(a)的光照分量图,在做了去除光照不均的处理后,分割结果如图7(d)所示,可以观察到局部细节图7(g)和图7(h),分割结果得到了很好的改善。
4.4 去除无效样点的必要性

图6 灰度直方图Fig. 6 Gray histograms

图7 去除光照不均影响前后图像的分割效果Fig. 7 Segmentation results before and after removing effects of uneven illumination
实验中,芯片上经常会出现如图8 中的连通区块。这导致大片的样点呈阳性表征,这些点就是典型的伪阳性点,会对后续生物学统计结果产生巨大的误差,所以在芯片图像处理的过程中,需要去除掉这一部分的点。使用本文提出的算法可以有效地去除这些点。图8 中显示的是2 组图片,其中,(a),(c)是采集到的原图,(b),(d)是最终选择的点的区域,可以清楚地看到,箭头标记的问题区域被识别并去除了。
4.5 定位时间及精度

图8 去除无效的样点Fig. 8 Removal of invalid spots
为了验证算法的定位效率,本文在一块完整的数字PCR 芯片上随机截取4 组局部图片,每组图片分别有100 张,4 组图片分别含有约10 个,100 个,1 000 个,10 000 个样点。对每张图片,分别用Voronoi 定位方法与本文提出的AHMAA定位算法进行计算,得出每张图片运算的时间,并对每组数据计算其均值与标准差,画出图9 的误差条形图。当计算约有10 个样点的图片时,Voronoi 耗 时 约0. 297 s,AHMAA 耗 时 约0. 022 s;当计算约有100 个样点的图片时,Voronoi 耗时约0. 968 s,AHMAA 耗时约0. 028 s;当计算约有1 000 个样点的图片时,Voronoi 耗时约17. 099 s,AHM AA 耗 时 约0. 100 s;当 计 算 约 有10 000 个样点的图片时,Voronoi 耗时约1 230. 163 s,AHMAA 耗时约0. 649 s。

图10 样点间距的一致性Fig. 10 Consistency of distance between spots

图9 样点定位耗时对比Fig. 9 Comparison of spots location time
为了探究算法定位的准确性,本文计算了样点之间的距离。 理论上,对于精加工的微孔芯片,每个相邻孔之间的距离都是固定的,而实际上,通过光学系统拍摄得到图像上样点的间距也是不会有太大误差的。所以,如果通过本文的算法计算得出的样点位置坐标足够精确,由此计算得出的样点之间的距离应该也是一个相对固定的值。
如图10 所示,在芯片中间区域随机选取了1 000 片方形碎片,每块碎片都包含约20 个样点,使用AHMAA 算法取得每个样点的位置信息,计算得出这20 个点与点之间的欧式距离。在这样的小碎片中,点与点之间大约有24 种不同的距离。其中,相邻点之间的距离最短,对角线两端两个点之间的距离最长。本文计算统计了所有的距离,然后将这1 000 块碎片的相同距离的数据放在一起,计算每种距离的CV 值。最后得出结果,CV 值最大的是相邻点之间的距离,为2. 69%,间隔一个点之间的距离CV 值为1. 43%,随着距离的增大,CV 值逐渐减小。 图10(b)是截取的前10 组距离的CV 值。 为了验证最终采集数据的准确性,本文对5 张数字PCR芯片样品,分别使用标准仪器系统软件和本实验室自行研制的图像采集系统及AHMAA 算法处理,得到有效填孔数与拷贝数,结果如表1~表2 所 示 。 这5 张 芯 片 中 ,编 号 为C0HGWS、C0HS51 的只含有FAM 染料,编号为C0HY06的只含有VIC 染料,编号为C0HUN8、C0I6MQ的是同时含有FAM 与VIC 染料的芯片。 与标准仪器的结果相比,通过AHMAA 算法处理得出的有效填孔数目结果的误差率在0. 35%~3. 03%,平均相差 1. 208%,准确率达到98. 792%;计算得到的拷贝数与之相比,误差率在0. 57%~8. 31%,平均误差率为3. 8%,准确率达到96. 2%。

表2 拷贝数Tab. 2 Number of copies

表1 有效填孔数Tab. 1 Effective number of holes to be filled
5 结 论
为了解决蜂窝状排列方式的数字PCR 芯片图像对比度低、光照不均匀、样点定位困难等问题,本文提出了基于形态学的图像处理方法。该方法对一组包含约20 000 个样点的数字PCR 芯片的三通道图像,平均运算时间约为20 s,定位最小样点间距离误差CV 值小于2. 69%。对5 组不同拷贝数的数字PCR 芯片,应用AHMAA 算法处理统计得出的有效样点数目结果与标准仪器[1]相比,误差率在0. 35%~3. 03% 之间,平均相差1. 208%;计算得到的拷贝数相比,误差率在0. 57%~8. 31%,平均误差率为3. 8%。 实验结果表明,本文方法在样点寻址的时候,使用了AHMAA 算法,可以准确定位蜂窝状排布方式的样点,而其他方法使用基于投影法的算法,只能对矩形排布方式的样点进行寻址定位。与Voronoi 定位方法相比,处理相同数量样点的图片时,时间相差3 个数量级。 在计算的准确度方面,与使用了预设模板的标准仪器相比,样点识别数精度达到98. 79%,拷贝数计算准确度达到96. 2%。该方法实现了对蜂窝状排布方式图像的快速准确处理,能在尽可能短的时间内为用户提供准确的样点荧光强度数据,为后续拷贝数计算等生物统计学研究提供了精确的原始数据。

