高粱钾离子通道Shaker蛋白家族的鉴定及生物信息学分析
欧阳浩 蒋君梅 杜巧丽 方远鹏 李向阳 谢鑫


摘 要:Shaker蛋白家族是植物体摄入钾元素的重要通道,其对植物环境耐受性发挥着重要的作用。本研究在高粱全基因组中鉴定了Shaker蛋白家族成员,并对其进行生物信息学分析。结果表明,高粱中共鉴定到9个Shaker家族成员,共可分为5个亚族,且处于同一个亚族的基因结构以及蛋白质结构相似;亚细胞定位显示,这些成员均定位于细胞质膜上;共线性分析结果表明,高粱Shaker蛋白家族在禾本科的进化历程中相对保守;组织特异性及模拟干旱表达结果显示,Shaker成员具有明显的组织特异性与干旱应答差异。本研究有望为进一步研究高粱Shaker蛋白家族的生物学功能提供理论基础。
关键词:高粱;Shaker家族;生物信息学分析
中图分类号:S514 文献标识码:A
基金项目:国家自然科学基金(31801691,32060614);贵州省科技计划(黔科合支撑[2019]2408);贵州省高层次留学人才创新创业择优资助项目([2018]02);大学生“SRT计划”项目(贵大SRT字[2019]329)
文章编号:1008-0457(2021)05-0001-09 国际DOI编码:10.15958/j.cnki.sdnyswxb.2021.05.001
Identification and Bioinformatics Analysis of Shaker Protein Family of Potassium Channels in Sorghum
OUYANG Hao1,JIANG Junmei1,2,DU Qiaoli1,FANG Yuanpeng1,LI Xiangyang2,XIE Xin1*
(1.Key Laboratory of Agricultural Microbiology,College of Agriculture,Guizhou University,Guiyang,Guizhou 550025,China;2.Key Laboratory of Green Pesticide and Agricultural Bioengineering,Ministry of Education,Guizhou University,Guiyang,Guizhou 550025,China)
Abstract:Shaker protein family is an important channel of potassium uptake for plant,which plays an important role in plant environmental tolerance.Here,members of the Shaker protein family were identified in the whole genome of sorghum and thenanalyzed by bioinformatics.The results showed that a total of 9 Shaker members were identified in sorghum,which could be divided into 5 subfamilies.Gene structure and protein structure of members of the same subfamily were similar.The results of subcellular localization showed that all these members were located on the cytoplasmic membrane.Based on the results of collinear analysis,the Shaker protein family of sorghum was relatively conservative in the evolution of Gramineae.According to the results of tissue specificity and simulated drought expression showed that Shaker members had significant differences in tissue specificity and drought response.In words,this study is expected to provide a theoretical basis for further study of the biological function of sorghum Shaker protein family.
Keywords:sorghum;Shaker family;bioinformatics analysis
鉀作为植物细胞内主要的无机成分之一,占植株总干重的10%,在植物多种生命活动中具有重要意义[1-4]。现有研究表明,钾离子可以提高植物对干旱、盐碱等非生物胁迫的耐受性,同时也可以抵抗真菌等生物胁迫[5]。由于正常土壤中钾离子的含量远低于植物细胞质中含量[6],植物的生长需要根系从土壤溶液中吸收大量的钾离子,并分布到整个植物中,以维持各个器官的正常运转,植物钾离子通道Shaker家族在这个过程中起到了至关重要的作用。
Shaker蛋白家族是通道蛋白中功能保守的一种,该蛋白家族广泛存在于各种植物中[7-8]。拟南芥(Arabidopsis thaliana)中的Shaker家族由9个成员组成[9-10],根据其功能特点可划分为5个亚组:内向整流通道I(Inward Rectifying Channel I):AKT1、AKT5、SPIK;内向整流通道II(Inward Rectifying Channel II):KAT1、KAT2;弱内向整流通道(Weakly Inward Rectifying Channel):AKT2;参与向内整流电导形成的调节亚基(Regulatory Subunit Involved in Inwardly Rectifying Conductance Formation):KC1(KAT3);以及向外整流通道(Outward Rectifying Channel):SKOR、GORK[11-13]。Shaker家族成员具有4个特殊的蛋白结构域,分别是植物特异性的跨膜钾离子通道结构域:Ion_trans_2、存在于在离子通道与cNMP依赖性激酶中的结构域:cNMP、含有多个重复短序列的结构域:ANK以及富含疏水性和酸性残基的钾离子通道的二聚化结构域:KHA[12]。正是因为这四种结构域的相互协作,Shaker蛋白可以通道形成异源四聚体结构[13],可以使植物调节不同细胞中的钾离子转运活性[8]。在拟南芥中,9个钾离子通道中有7个成员具有内向整流特性,而外向整流通道仅有两个成员,分别为:中柱外向整流型通道(SKOR)和保卫细胞外向整流型钾离子通道(GORK)[14]。同时,Shaker蛋白家族成员具有明显的组织特异性;例如,拟南芥AtAKT1、AtKC1主要在植物根中表达[14-15],而AtSPIK则主要在萌发的花粉管中表达,AtSKOR主要在拟南芥的根茎组织中表达,AtGORK主要在保卫细胞中表达[16]。
高粱(Sorghum bicolor (L.) Moench)具有喜温、抗旱、耐贫瘠等优良特点。在食用、药用、酿酒以及饲料制作等方面有着重要意义,但其严重环境胁迫仍影响其生产[17-18]。高粱作为重要的经济作物及C4植物,对其开展Shaker通道的系统分析,对高粱及其他C4植物抗逆研究具有指导价值。然而,目前Shaker通道的系统分析局限于拟南芥、水稻(Oryza sativa)、紫花苜蓿(Medicago truncatula)等C3植物中,但包括高粱、小米在内的C4植物中Shaker通道的系统分析尚且缺乏。本研究通过生物信息学的方法对高粱Shaker家族进行了全基因组鉴定,染色体定位分析,基因结构分析,系统进化发育分析,为进一步探究高粱钾离子通道Shaker蛋白家族提供基础。
1 材料与方法
1.1 高粱Shaker蛋白家族成员的鉴定
拟南芥Shaker蛋白家族的序列来源于拟南芥数据库(https://www.arabidopsis.org/index.jsp)。利用生物信息分析软件TBtools 1.0的Simple HMM Search功能[19]。根据已报道的拟南芥Shaker家族成员作为蛋白源序列建立的比對序列徽标(PF00520、PF07885、PF00027、PF11834)检索EnsemblPlants数据库,最终得到符合搜索条件的Shaker成员。然后利用在线网站SMART和NCBI-CD检测所获取序列的保守蛋白质结构域,进一步筛选,最终获得高粱Shaker蛋白家族全部成员。
1.2 高粱Shaker蛋白家族成员的理化性质及亚细胞定位预测
用ExPASy-ProtParam Tool预测SbShakes的基本理化性质;根据Softberry(http://www.csbio.sjtu.edu.cn/bioinf/Cell-PLoc-2/)在线网站进行SbShakers蛋白的亚细胞定位预测。
1.3 高粱Shaker蛋白家族系统进化树构建以及亚族分组确定
利用CLUSTALW将拟南芥、二穗短柄草(Brachypodium distachyon)、水稻(Oryza sativa)、马铃薯(Solanum tuberosum)、玉米(Zea mays)以及高粱的Shaker蛋白序列进行多序列比对分析。并通过邻接法(Neighbour-Joining,NJ)在MEGA 7.0中构建系统进化树,自展值(Bootstrap)设定为1000[20]。并使用在线网站Evolview对进化树进行美化。
二穗短柄草、水稻、马铃薯、玉米的Shaker基因家族的序列来源于EnsemblPlants(https://plants.ensembl.org/)植物数据库。
1.4 高粱Shaker蛋白家族成员保守基序及基因结构分析
利用MEME在线网站(https://meme-suite.org/)预测分析SbShakers蛋白保守功能基序,再结合TBtools对高粱Shaker家族成员进行基因结构分析。
1.5 高粱Shaker蛋白家族成员染色体定位分析
利用生物信息分析软件TBtools 1.0的Show Genes on Chromosome功能,对已经鉴定的高粱中的SbShakers进行染色体定位分析。
1.6 高粱Shaker蛋白家族成员共线性分析
利用TBtools 1.0软件MCScanX功能对高粱Shaker成员进行成员进化的共线性分析,研究不同物种间Shaker家族成员的同源性。
1.7 高粱Shaker蛋白家族的组织及胁迫表达分析
通过在线网站高粱功能组学数据库(http://structuralbiology.cau.edu.cn/sorghum/index.html)收集SbShakers成员的组织特异性以及环境胁迫处理表达数据,建立高粱Shaker蛋白家族表达热图进行分析。
2 结果与分析
2.1 高粱Shaker蛋白家族鉴定结果分析
运用生物信息学方法对蛋白结构域筛选验证后,从高粱全基因组中鉴定到9个Shaker家族成员,根据位于染色体位置顺序依次命名为:SbShaker1~SbShaker9(表1)。
高粱Shaker家族的氨基酸序列长度介于601 aa(SbShaker4)与919 aa(SbShaker1)之间;分子量介于65.80 kDa(SbShaker4)与100.65 kDa(SbShaker1)之间;等电点介于5.95(SbShaker9)和8.63(SbShaker3)之间;等电点在7左右,说明该家族可能是一类中性蛋白;在对Shaker家族进行其亚细胞位置预测后发现,该家族成员全部位于细胞质膜上,这与Shaker家族通道蛋白的功能特点相符(表1)。应用SMART 和NCBI-CD中分析结构域,发现4个保守蛋白结构域,它们分别为Ion_trans_2结构域、cNMP结构域、ANK结构域和KHA结构域。其中Shaker3、Shaker4、Shaker5不存在ANK结构域,这一结果与拟南芥Shaker家族成员中的AKT1、KAT3、KAT1相同。说明ANK结构域在物种间遗传中并不是非常保守,同时也说明了高粱与拟南芥Shaker蛋白家族结构的相似性。
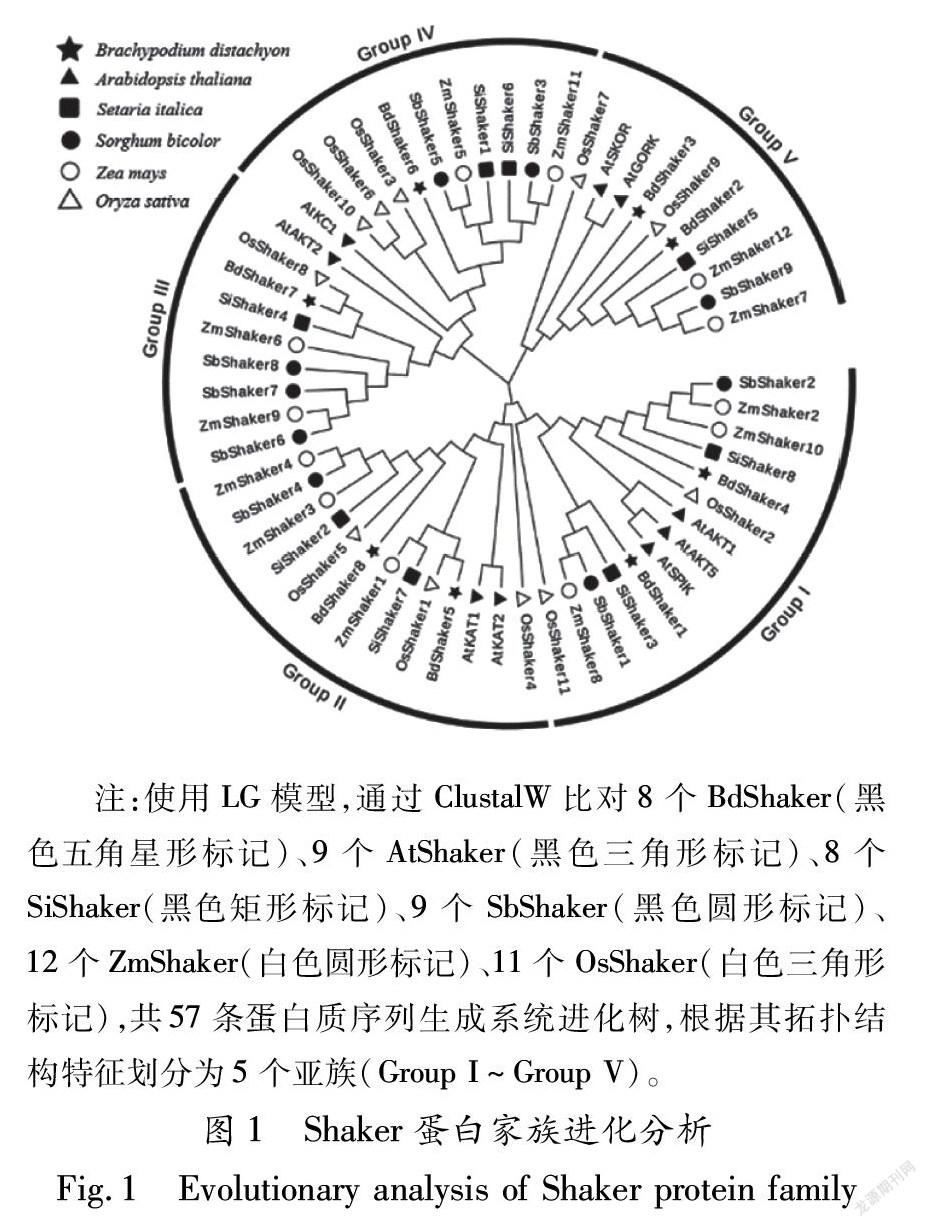
2.2 高粱Shaker蛋白家族系统进化分析
为了进一步验证高粱Shaker蛋白家族系统进化情况,构建高粱与拟南芥、二穗短柄草、水稻、马铃薯、玉米Shaker蛋白家族系统进化树(图1)。其
中I亚族成员为:Sbshaker1、Sbshaker2,II亚族成员为:Sbshaker5,III亚族成员为:Shaker6、Shaker7、Shaker8,IV亚族成员为:Shaker3、Shaker4;Ⅴ亚族成员为:Shaker9。与拟南芥不同的是,在拟南芥Shaker蛋白家族中Ⅴ亚族成员有两个,分别为SKOR和GORK。这两个基因在拟南芥中作为向外整流通道而存在。因此可以推测出,高粱钾离子通道中的外整流通道与拟南芥进行了两个不同的进化历程。除此之外,在I、II、IV亚族的成员个数也与拟南芥中亚族成员不一致。由此可见,高粱中Shaker通道的亚族成员在禾本科植物的不断进化中,可能发生了不保守遗传。
2.3 高粱Shaker蛋白家族保守基序及基因结构分析
对高粱Shaker蛋白家族进行了蛋白保守结构基序分析,共预测到18个保守基序(Motif),同时对这些基序进行注释,结果显示Motif 1、Motif 3对应Ion_trans_2结构域、Motif 2、Motif 5对应cNMP结构域,Motif 4、Motif 10对应ANK结构域,Motif 6、Motif 9对应KHA结构域;此外在I亚族中Shaker2的cNMP结构域中多了Motif 8;III亚族中每一个成员的cNMP结构域都多了Motif 15;IV亚族成员都不存在ANK结构域;II亚族成员中Ion_trans_2结构域中多了Motif 14;Ⅴ亚族中的KHA结构域缺少Motif 6。分析结果表明Shaker家族成员的结构域并不是完全保守,高粱Shaker蛋白家族可能在物种进化中发生某些功能的缺失和增加(图2)。
对高粱Shaker蛋白家族结构分析可知,9个成员中SbShaker4不含有非蛋白编码区,全为蛋白编码区段,剩余成员既含有编码区段,又含非编码区段。其中,SbShaker2、SbShaker5、SbShaker9均含有11个编码区;SbShaker6、SbShaker7含有6个编码区;SbShaker1含有9个编码区;SbShaker3含有7個编码区;SbShaker8含有8个编码区;与系统发育分析结果一致的是,聚集在同一亚族中的SbShakers成员,具有相同或相似外显子/内含子结构和基因长度(图2)。这也表明了SbShakers基因结构具有保守性。其次,Shaker家族基因结构中其内含子数目为0~10个,说明即使是相同亚类家族成员之间,其外显子和内含子的数量也存在较大的差异(图2)。
2.4 高粱Shaker蛋白家族染色体定位分析
通过生物信息学分析软件TBtools对9个高粱Shakers家族基因进行染色体定位分析。结果显示:9个基因主要分布在2、3、4、9和10号染色体上;Shaker1分布在2号染色体上端,Shaker2、Shaker3处于3号染色体下端,Shaker4位于4号染色体上端,Shaker5位于4号染色体下端,Shaker10处于10号染色体上端。其中III亚族成员(Shaker6、Shaker7、Shaker8)全部位于9号染色体下端。而其他亚族成员不均等分布在其他染色体上。可预测III亚族的成员(SbShaker6、SbShaker7、SbShaker8)履行着相近或相同的生物学功能(图3)。
2.5 高粱Shaker家族成员共线性分析
为深入研究不同物种间Shaker蛋白家族成员的同源性,利用模式植物:水稻,禾本科植物:二穗短柄草、高粱、玉米、谷子,绘制了5种Shaker基因家族共线图(图4)。共发现43对共线性基因。其中,每一个植物与另一物种的Shaker成员都出现了一个基因与多个基因或者多个基因与单个基因呈共线关系。可得出,在这些物种的进化过程中发生了多个基因拷贝,其次通过对各个物种间的共线性传递的数量以及每个物种之间Shaker成员的数量可以分析出,高粱Shaker蛋白家族在禾本科的进化历程中相对保守。
2.6 高粱Shaker蛋白家族的组织及胁迫表达分析
对高粱Shaker蛋白家族9个成员在胚芽(Embryos)、花分生组织(Floral meristem)、花(Flowers)、营养分生组织(Vegetative meristem)、根(Root)、叶(Shoot)6个部位进行组织表达分析(图5)。结果显示,高粱在不同组织部位中的表达量差异显著。I亚族成员:SbShaker1、SbShaker2表达差异较大,SbShaker1几乎在上述部位中不表达,SbShaker2却在除了花器官中的组织中都有表达,特别是在花分生组织表达量最高;II亚族成员:SbShaker4在芽、胚芽、花分生组织中少量表达;III亚族成员:SbShaker6、SbShaker7、SbShaker8在上述组织中表达差异较大。其中,SbShaker6在上述组织中均表达,而且在营养分生组织、芽、花中的表达量最高;SbShaker7在花中少量表达;SbShaker8在六个组织部位均不表达。IV亚族成员SbShaker3、SbShaker5在各组织中均表达,由此得出该亚族成员主要在成熟器官中表达。其中V亚族成员SbShaker9在根部、花中的表达量最高,这与拟南芥、水稻中报道的类似。
此外,对SbShakers进行激素脱落酸(Abscisic acid,ABA)、聚乙二醇模拟干旱(Polyethylene Glycol,PEG)分析(图6)。结果显示,I亚族成员: SbShaker1、SbShaker2在高粱的根与叶中表达量有所不同。SbShaker1在脱落酸、聚乙二醇处理下基因表达量变化极不显著,SbShaker2在脱落酸、聚乙二醇处理,在根中表达量下调,而在叶中表达出现少量上调趋势;II亚族成员:SbShaker5在高粱根中的表达,基本不受脱落酸与聚乙二醇的影响,但在叶中SbShaker5表达量却显著提高;III亚族成员:SbShaker7、SbShaker8在脱落酸与聚乙二醇处理,表达不显著,但SbShaker6在脱落酸处理,其基因表达量显著下调。IV亚族成员SbShaker3、SbShaker4在脱落酸与聚乙二醇处理表达也不明显。V亚族成员SbShaker9的表达量则与环境中脱落酸与聚乙二醇呈正相关,且通过外源施加脱落酸还会导致SbShaker9的基因表达量出现明显的上升的趋势。
3 结论与讨论
钾是一种重要的矿质元素,在植物体内的各项生命活动中都发挥重要作用,如膨胀度调节、细胞伸长、气孔和叶片运动以及酶活性调节方面[1-3,21]。其中Shaker蛋白家族是一类维持植物体内钾离子稳态的重要通道蛋白,该家族在植物体进行环境抗逆方面发挥着极其重要的作用[22-24]。本研究对高粱Shaker蛋白家族进行全基因组鉴定,并从进化、基因表达等方面进行系统分析。在高粱中共鉴定到9个成员,并按基因及蛋白结构特征,分为5个亚家族(亚族I~亚族V)。前人的研究结果显示,拟南芥和马铃薯(Solanum tuberosum)共有9个Shaker蛋白;水稻和甘薯(Ipomoea batatas)有11个;梨(Pyrus × bretschneideri)有8个,说明高粱的Shaker家族成员在物种进化过程中相对保守[25-28]。但与拟南芥、水稻的成员分布不同的是:高粱中鉴定到Shaker通道的III亚族中有3个(Shaker6、Shaker7、Shaker8),而在拟南芥中仅有1个(AtAKT2);在V亚族中拟南芥有2个成员(AtGORK、AtSKOR),而高粱中仅有SbShaker9,该结果与豆科模式植物苜蓿(Medicago truncatula)类似[29-30],说明不同的物种为了适应不同环境,对Shaker通道进行了基因拷贝或系统性缺失。
Shaker蛋白通道广泛存于在植物的各个组织细胞中,具组织表达特异性[9-11]。AKT1是从吸收钾离子缺陷酵母突变体功能互补中所克隆出的钾离子通道基因,主要在拟南芥根中表达,它能响应外界钾离子浓度的变化来调节植物根中生长所必需的钾元素量,进而影响生长素在根中的重新分配[31-32]。SPIK是拟南芥中I亚族的成员,主要在花粉中特异性表达,它在维持花粉管的正常发育中起着重要作用[16]。高粱Shaker蛋白家族中I亚族成员中:SbShaker2在高粱的根中有表达,而且在胚芽、花的分生组织中的表达量最高。由此可预测高粱Shaker成员SbShaker2与拟南芥中AKT1蛋白可能具有类似的功能。KAT1是拟南芥中II亚族中的代表成员,主要表达在叶片保卫细胞,在根和茎维管组织中也有少量表达[33]。本研究结果与该结论相似,从高粱鉴定到Shaker蛋白家族的II亚族成员SbShaker4,主要在花的分生组织中表达,在根中有少量表达。说明SbShaker4与拟南芥AKT1蛋白在功能方面,具有一定的相似性。AKT2是拟南芥中表达最具特点的成员,在整个植物韧皮部组织中都有表达,它是Shaker家族中唯一具有两相激活动力学的弱内向整流功能特点的基因[17]。其中,环境中的pH值是植物中AKT2通道活性的强调节因子,外源pH会显著下调AKT2的活性,即使是很小的细胞质pH变化也会显著影响AKT2的整流特性[34]。本研究中,在该亚族共鉴定到3个成员:SbShaker6、SbShaker7、SbShaker8,根据基因表达以及同源分析可知,SbShaker6是AtAKT2在高粱中同源基因,由此预测SbShaker6具有与AKT2相似的功能特性。与之不同的,高粱中SbShaker7成员在高粱的花中有少量表达。AtKC1存在于细胞内质网中,是异四聚体通道内的调控亚基,能调控其他Shaker通道蛋白的表达,从而提高植物抗逆性[35-36]。在拟南芥中具有外向整流功能的Shaker家族成员有两个,分别是SKOR和GORK。其中SKOR在拟南芥的根茎组织中表达,并将钾离子运输到木质部中;GORK在保衛细胞中表达,并通过保卫细胞的钾离子输出,通道参与植物气孔运动,调节植物蒸腾作用[37-38]。在高粱中鉴定唯一的外向整流通道SbSahekr9,在根与花中的表达量最高。
ABA、PEG能促进保卫细胞内的钾离子快速净外排,从而诱导膨压损失,从而介导气孔关闭[39]。据报道,土壤中盐离子的种类以及含量,对拟南芥中内向整流AKT1基因的转录调控作用不大,但植物为了维持体内稳态,机体会引导KC1基因表达上调,导致AtKC1亚基通过形成异聚的AtKC1-AKT1通道的方式,来调节AKT1的表达量降低,减弱或阻隔对钾离子的吸收,让机体对盐碱等胁迫产生抗性;但该异聚通道对外向整流通道成员的表达无影响[40]。与本研究类似,在高粱中利用植物逆境激素ABA对SbShaker3进行处理,结果显示SbShaker3的表达量增加,而I亚族成员SbShaker2的表达量显著降低,但对SbShaker9无影响。由此可推测,在高粱盐胁迫过程中仍然存在该结论。其次在拟南芥中进行激素控制方面,AKT1对ABA、PEG处理不敏感[41],而且外源施加脱落酸能迅速且强烈地抑制SKOR的表达[42];但ABA会诱导的AKT2、GORK表达上调,说明二者对植物抗干旱起重要作用[43]。在本研究中,对高粱Shaker家族进行胁迫处理发现,对其施加脱落酸会对SbShaker6、SbShaker7、SbShaker8、SbShaker9的表达量有明显的提升。由此预测,SbShaker6、SbShaker7、SbShaker8具有拟南芥中AKT1通道蛋白的相关功能;SbShaker9可能具有AtGORK相似的功能特点。
综上所述,本研究利用生物信息学方法从高粱全基因组鉴定到9个高粱的Shaker蛋白家族成员,它们分布在高粱的2、3、4、9、10号染色体上。该家族成员有4个Shaker通道的特殊蛋白结构域,分别是Ion_trans_2、cNMP、ANK和KHA。SbShakers基因所编码的蛋白序列长度介于601 aa(SbShaker5)与919 aa(SbShaker1)之间,所预测的蛋白质分子质量介于65.80 kDa(SbShaker5)与100.65 kDa(SbShaker1)之间。其次,发现高粱中的Shaker通道具有组织表达特异性,而且部分成员对ABA、PEG的处理有明显的影响。以上结果为进一步研究高粱中Shaker蛋白家族的生物学功能奠定了基础。
参 考 文 献:
[1] YI W,WU W.Regulation of potassium transport and signaling in plants [J].Current Opinion in Plant Biology,2017,39 (39):123-128.
[2] 魏永胜,梁宗锁.钾与提高作物抗旱性的关系[J].植物生理学通讯,2001(6):576-580.
[3] ADAMS E,SHIN R.Transport,signaling,and homeostasis of potassium and sodium in plants [J].Journal of Integrative Plant Biology,2014,56(3):231-249.
[4] NIEVES-CORDONES M,AL SHIBLAWI F R,SENTENAC H.Roles and transport of sodium and potassium in plants [J].Metal Ions in Life Sciences,2016,16:291-324.
[5] AMTMANN A,TROUFFLARD S,ARMENGAUD P.The effect of potassium nutrition on pest and disease resistance in plants [J].Physiologia Plantarum,2008,133(4):682-691.
[6] SCHROEDER J I,WARD J M,GASSMANN W.Perspectives on the physiology and structure of inward rectifying K channels in higher plants:biophysical implications for K uptake [J].Annual Review of Biophysics and Biomolecular Structure,1994,23 (1):441-471.
[7] PILOT G,PRATELLI R,GAYMARD F,et al.Five-group distribution of the Shaker-like K+ channel family in higher plants [J].Journal of Molecular Evolution,2003,56(4):418-434.
[8] GAMBALE F,UOZUMI N.Properties of shaker-type potassium channels in higher plants [J].Journal of Membrane Biology,2006,210(1):1-19.
[9] ANNE L,ANNE-ALINOR V,HERV S.K+ channel activity in plants:Genes,regulations and functions [J].FEBS Letters,2007,581(12):2357-2366.
[10] SHABALA S.Regulation of potassium transport in leaves:from molecular to tissue level.[J].Annals of Botany,2003,92(5):627-634.
[11] SERGEY S.Regulation of potassium transport in leaves:from molecular to tissue level [J].Annals of Botany,2003,92(5):627-634.
[12] BENOIT R.What can be deduced about the structure of Shaker from available data? [J].Novartis Foundation Symposium,2002,245(P):84-100
[13] DARAM P,URBACH S,GAYMARD F,et al.Tetramerization of the AKT1 plant potassium channel involves its C-terminal cytoplasmic domain[J].The EMBO Journal,1997,16(12),3455-3463.
[14] BIRGIT R,ALEXANDER S,NATALYA I,et al.AtKC1,a silent Arabidopsis potassium channel α-subunit modulates root hair K+ influx [J].Proceedings of the National Academy of Sciences of the United States of America,2002,99(6):4079-4084.
[15] DENNISON K L,ROBERTSON W R,LEWIS B D,et al.Functions of AKT1 and AKT2 potassium channels determined by studies of single and double mutants of Arabidopsis [J].Plant Physiology,2001,127(3):1012-1019.
[16] KARINE M,ANNE-ALINOR V,FRDRIC G,et al.Pollen tube development and competitive ability are impaired by disruption of a Shaker K+ channel in Arabidopsis [J].Cold Spring Harbor Laboratory Press,2002,16(3):339-350.
[17] 王偉仁.山西省高粱生产发展现状与对策研究 [D].太古:山西农业大学,2017:12
[18] 季树太,王佐民,郭书刚.酿酒高粱研究刍议 [J].酿酒,2019,46(2):28-30.
[19] CHEN C,CHEN C,ZHANG Y,et al.TBtools:an integrative toolkit developed for interactive analyses of big biological data [J].Molecular Plant,2020,13(8):1194-1202.
[20] KUMAR S,STECHER G,TAMURA K.MEGA7:Molecular evolutionary genetics analysis Version 7.0 for bigger datasets[J].Molecular Biology and Evolution,2016,33(7):1870-1874.
[21] ZHANG A,REN H,TAN Y,et al.S-type anion channels SLAC1 and SLAH3 function as essential negative regulators of inward K+ channels and stomatal opening in Arabidopsis[J].The Plant Cell,2016,28(4):949-965.
[22] FUCHS I,FUCHS S,IVASHIKINA N,et al.Rice K+ uptake channel OsAKT1 is sensitive to salt stress [J].Planta,2005,221(2):212-221.
[23] 周練. GmPIP1:6和GmAKT2基因在大豆抗逆中的功能研究 [D].杭州:浙江大学,2014.
[24] OBATA T,KITAMOTO H K,NAKAMURA A,et al.Rice shaker potassium channel OsKAT1 confers tolerance to salinity stress on yeast and rice cells [J].Plant Physiology,2007,144(4):1978-1985.
[25] NAGA-AMRUTHA R,NATARAJ-SEKHAR P,RAJEEV K,et al.Genome-wide analysis and identification of genes related to potassium transporter families in rice (Oryza sativa L.) [J].Plant Science,2006,172(4):708-721.
[26] JIN R,ZHANG A,SUN J,et al.Identification of Shaker K+ channel family members in sweetpotato and functional exploration of IbAKT1 [J].Gene,2020,768:145311.
[27] CHEN G,CHEN Q,QI K,et al.Identification of Shaker K+ channel family members in rosaceae and a functional exploration of PbrKAT1 [J].Planta,2019,250(6):1911-1925.
[28] 尹欢,卓凤萍,邓可宣,等.马铃薯Shaker基因家族全基因组鉴定和分析 [J].重庆科技学院学报(自然科学版),2019,21(3):104-108,116.
[29] HYUN Y K,EUN H C,MYUNG K M,et al.Differential gene expression of two outward-rectifying shaker-like potassium channels OsSKOR and OsGORK in rice [J].Journal of Plant Biology,2015,58(4):230-235.
[30] ALICE D,JULIEN T,LIMIN W,et al.Functional characterization and physiological roles of the single Shaker outward K+ channel in Medicago truncatula [J].The Plant Journal,2020,102(6):1249-1265.
[31] SENTENAC H,BONNEAUD N,MINET M,et al.Cloning and expression in yeast of a plant potassium ion transport system [J].Science,1992,256(5057):663-665.
[32] LI J,WU W,WU Y.Potassium channel AKT1 is involved in the auxin-mediated root growth inhibition in Arabidopsis response to low K+ stress [J].Journal of Integrative Plant Biology,2017,59(12):895-909.
[33] PILOT G,LACOMBE B,GAYMARD F,et al.Guard cell inward K+ channel activity in Arabidopsis involves expression of the twin channel subunits KAT1 and KAT2 [J].Journal of Biological Chemistry,2001,276(5):3215-3221.
[34] LACOMBE B,PILOT G,MICHARD E,et al.A shaker-like K+ channel with weak rectification is expressed in both source and sink phloem tissues of Arabidopsis [J].The Plant Cell,2000,12(6):837-851.
[35] DUBY G,HOSY ERIC,FIZAMES C,et al.AtKC1,a conditionally targeted Shaker-type subunit,regulates the activity of plant K+ channels [J].The Plant Journal:for Cell and Molecular Biology,2008,53(1):115-123.
[36] WANG Y,H L,LI H,XU,et al.Potassium channel α-subunit AtKC1 negatively regulates AKT1-mediated K+ uptake in Arabidopsis roots under low-K+ stress[J].Cell Research,2010,20(7):826-837.
[37] ANDERSON J A,HUPRIKAR S S,KOCHIAN L V,et al.Functional expression of a probable Arabidopsis thaliana potassium channel in Saccharomyces cerevisiae [J].Proceedings of the National Academy of Sciences of the United States of America,2002,99(6):4079-4084.
[38] NIEVES-CORDONES M,ALEMN F,MARTNEZ V,et al.K+ uptake in plant roots.The systems involved,their regulation and parallels in other organisms [J].Journal of Plant Physiology,2014,171(9):688-695.
[39] JULIAN I S,KLAUS R,ERWIN N.Voltage dependence of K+ channels in guard-cell protoplasts [J].Proceedings of the National Academy of Sciences of the United States of America,1987,84(12):4108-4112.
[40] PONGS O.Molecular biology of voltage-dependent potassium channels [J].Physiological Reviews,1992,72(4):S69-S88.
[41] JEANGUENIN L,ALCON C,DUBY G,et al.AtKC1 is a general modulator of Arabidopsis inward Shaker channel activity[J].The Plant Journal,2011,67(4):570-582.
[42] GAYMARD F,PILOT G,LACOMBE B,et al.Identification and disruption of a plant Shaker-like outward channel involved in K+ release into the xylem sap [J].Cell,1998,94(5),647-655.
[43] CUIN T A,DREYER I,MICHARD E.The role of potassium channels in Arabidopsis thaliana long distance electrical signalling:AKT2 modulates tissue excitability while GORK shapes action potentials [J].International Journal of Molecular Sciences,2018,19(4):926.
通訊作者:谢鑫(1984—),男,博士,副教授,主要从事植物抗病基因功能研究,E-mail:xiexin2097757@163.com.

