苜蓿质膜内在蛋白编码基因MsPIPs家族的密码子偏好性分析
张海霞,王玉道,许雪妮
摘要:利用CodonW软件分析苜蓿质膜内在蛋白编码基因(MsPIPs)的密码子组成,发现它们的密码子在碱基使用中存在偏倚,偏好使用以T/A结尾的密码子,其中T结尾的密码子出现频率为47%。MsPIPs密码子的平均有效密码子数(ENC)为45.47个,偏好性较弱。通过主成分分析(PCA)、对应性分析(COA)、偏倚分析(PR2)、有效密码子数分析(ENC plot)和中性分析等多重分析方法对苜蓿MsPIPs密码子偏好性的成因进行分析,发现苜蓿MsPIPs的偏好性主要受自然选择的影响。
关键词:质膜内在蛋白;密码子偏好性;自然选择;突变
中图分类号:S551+.7文献标识码:A文章编号:1000-4440(2021)06-1393-07
Codon usage bias analysis of MsPIPs gene family which encode plasma membrane intrinsic proteins in alfalfa
ZHANG Hai-xia,WANG Yu-dao,XU Xue-ni
(Forestry Technology Popularization Station of Minqin County in Gansu Province, Minqin 733300, China)
Abstract:By using software CodonW to analyze the codon composition of plasma intrinsic protein encoded by MsPIPs gene in alfalfa, it was found that the codons showed bias in nucleobase use, the codons of MsPIPs preferred T/A endings, and the frequency of occurrence of T-ending codons was 47%. The average effective number of codons (ENC) of MsPIPs codons was 45.47, which had a weak codon bias. Through multivariate analytical methods combined by principal component analysis (PCA), corresponding analysis (COA), parity rule 2 plot mapping analysis (PR2), ENC plot analysis and neutrality plot analysis, the reason of the codon bias of the MsPIPs gene in alfalfa was analyzed, and it was found that the bias of MsPIPs in alfalfa might be driven by natural selection.
Key words:plasma membrane intrinsic proteins;codon usage bias;natural selection;mutation
密码子在遗传信息传递过程中起着至关重要的作用[1]。编码不同氨基酸的密码子的准确识别是保证基因信息准确表达的关键[2]。除了甲硫氨酸(Met)和色氨酸(Trp)由唯一的密码子编码外,大部分氨基酸均由2~6个同义密码子编码,同义密码子在不同植物体内的使用频率有差异,形成了密码子使用偏性[3]。密码子使用偏性受群体中突变压力、选择压力和基因漂变的影响,对研究基因功能、物种进化等具有重要科学意义[4]。
质膜内在蛋白(Plasma membrane intrinsic proteins,PIPs)定位在质膜上,是水通道蛋白(Aquaporins,AQPs)的亚家族成员,属于主要内在膜蛋白(Major intrinsic protein, MIP)家族[5]。目前,研究者们已经从拟南芥、玉米和番茄等中分别鉴定到13个、13个和18个PIPs基因[6]。在植物生长发育过程中,PIPs主要負责植物体内水分的吸收和运输,也参与植物体内营养及元素的运输及细胞生长分化过程[7]。在干旱和盐等非生物胁迫下,通过活性和表达情况的改变,PIPs在植物对非生物胁迫的应答过程中发挥着重要作用[8]。对PIPs基因家族密码子使用偏性的分析可为该家族编码蛋白结构和功能研究提供基础数据。
2020年,苜蓿基因组全序列的公布[9]使得利用生物信息学手段在全基因组水平上研究苜蓿成为可能,但是苜蓿PIPs基因家族尚未见报道,且PIPs的密码子使用偏性规律也不清楚。本研究以从苜蓿基因组中鉴定到的PIPs基因家族为基础,进行密码子偏好性分析,明确该基因家族的密码子使用规律,为进一步展开功能研究和苜蓿抗性改良提供理论依据。
1材料和方法
1.1数据来源
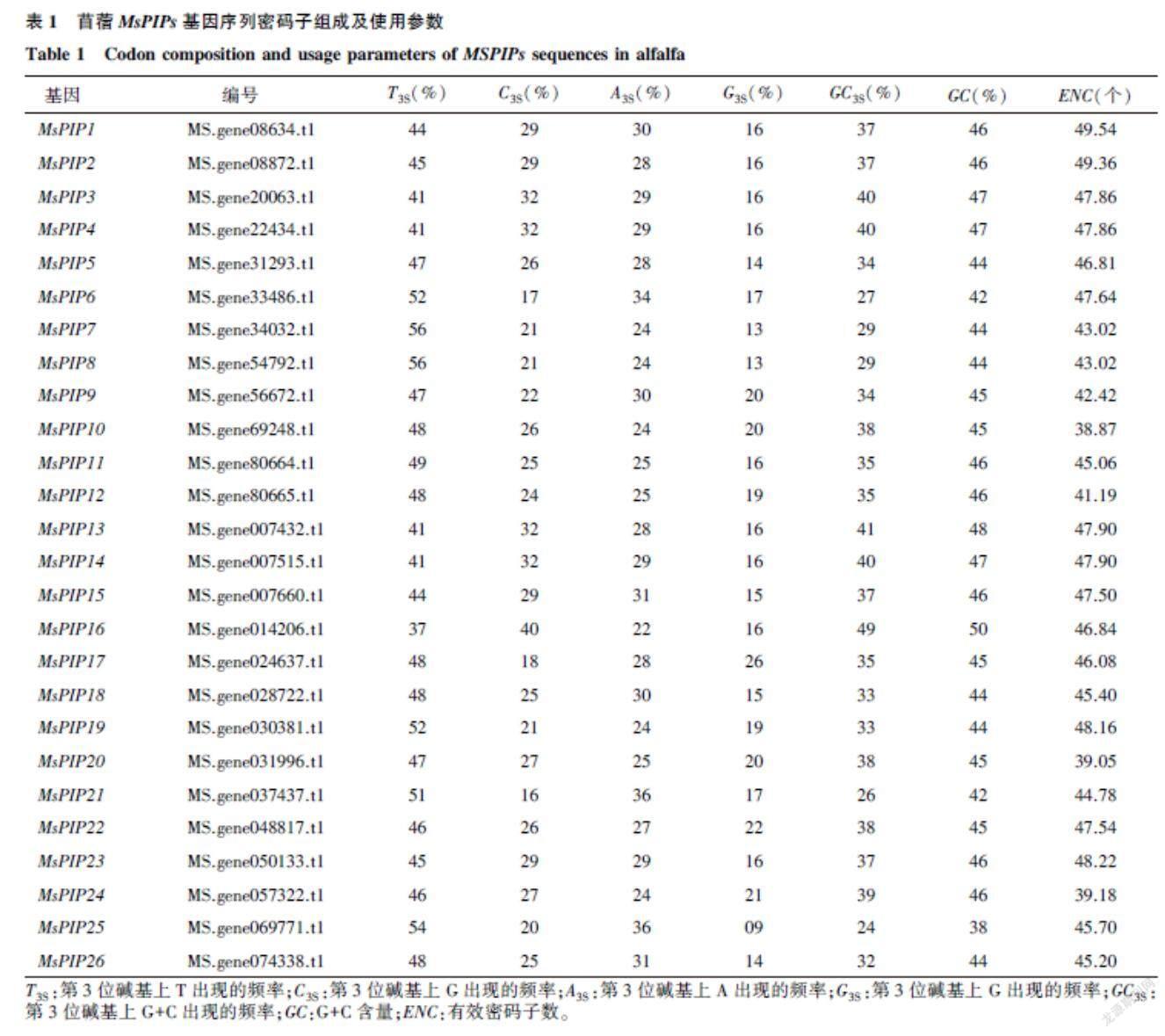
在苜蓿基因组的Swissprot数据库[9]中搜索包含“PIP”和编码序列(CDS)长度大于300 bp的序列,并在NCBI网站上进行这些序列的BLAST搜索,进一步鉴定PIP序列。筛选出的26个MsPIPs基因序列按照MsPIP1~MsPIP26进行命名(表1)。
1.2苜蓿PIPs基因家族密码子组成分析
采用CondonW 1.3软件(https://sourceforge.net/projects/codonw/)分析计算苜蓿PIPs基因家族基因的密码子组成,包括同义密码子相对使用度(Relative synonymous codon usage value,RSCU)值、有效密码子数(Effective number of codons,ENC)、G+C含量、第3位碱基组成(A3S、T3S、G3S和C3S)等指标[10]。第1位碱基的G+C含量(GC1)、第2位碱基的G+C含量(GC2)、第3位碱基的G+C含量(GC3)及第1和2位碱基的G+C含量(GC12)通过EMBOSS (http://imed.med.ucm.es/cgi-bin/emboss.pl?_action=input&_app=cusp)的Cusp功能计算得到[11]。
1.3最优密码子的确定
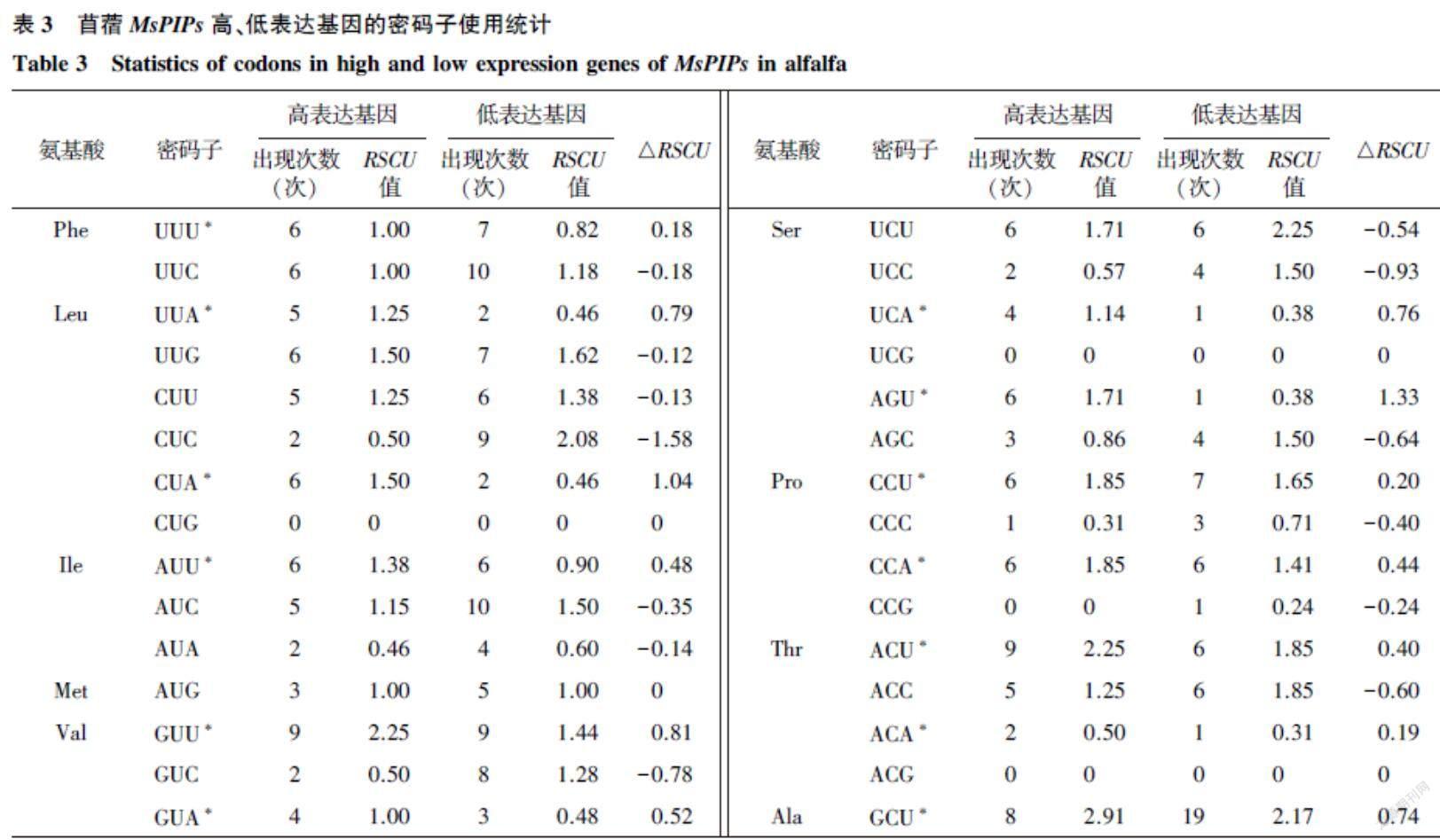
以ENC值为标准对上述筛选出的PIPs序列进行排序,从排序结果的两端各选取5%的基因建立高低偏性库,计算高低偏性库的△RSCU值,計算公式为:△RSCU=RSCU高偏性-RSCU低偏性。具有最高RSCU值的密码子被称为高频密码子,具有最大△RSCU值的密码子被称为高表达密码子,同时满足最高RSCU值和最大△RSCU值的密码子被确定为最优密码子[12]。
1.4苜蓿MsPIPs基因家族密码子的偏好性分析
按照Wei等[13]的方法进行主成分分析(Principal component analysis,PCA),将密码子的使用频率分布在一个59维的向量空间中。利用对应性分析(Corresponding analysis,COA),比较2个或更多类别的变量数据,为密码子使用趋势和基因的主要变化研究提供直观结果。利用偏倚分析(Parity rule 2 plot mapping analysis,PR2)分析各个密码子第3位碱基组成(A3S、T3S、G3S和C3S)的情况。利用ENC-plot分析展示ENC和GC3的函数关系,使用中性分析(Neutrality plot mapping analysis)展示GC12和GC3的函数关系。
1.5数据统计分析
使用Excel 2016制图。
2结果与分析
2.1苜蓿MsPIPs家族基因CDS序列的密码子组成
从苜蓿基因组中筛选到26条MsPIPs基因,总GC含量为38%~50%,平均值为45%(表1)。MsPIPs的密码子第3位碱基上A、T、C、G出现的平均频率分别为28%、47%、26%和17%,表明苜蓿MsPIPs偏好使用以T/A结尾的密码子。MsPIPs的ENC为38.87~49.54个,平均值为45.47个,表明苜蓿MsPIPs对密码子使用的偏好性较弱。
2.2苜蓿MsPIPs的最优密码子
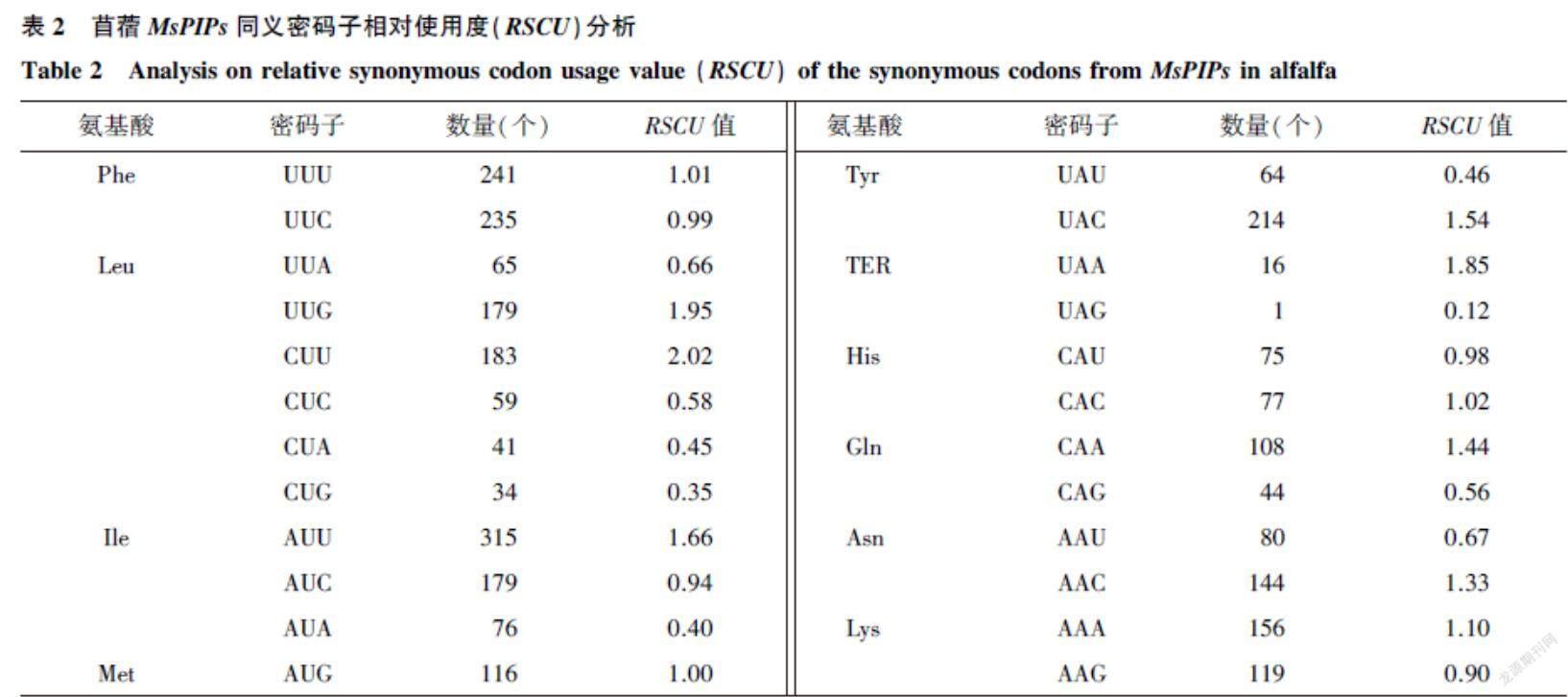
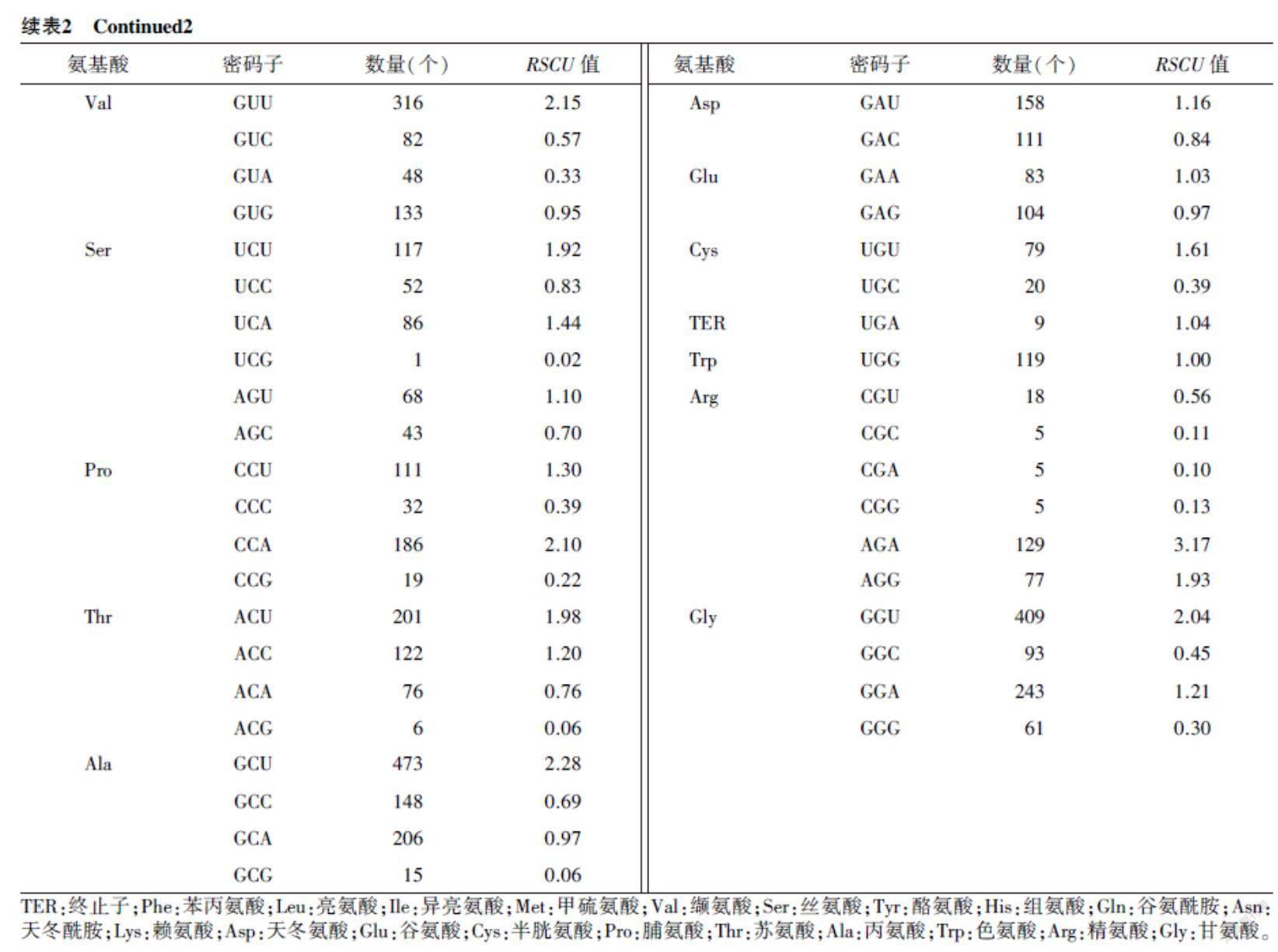
将苜蓿MsPIPs各成员氨基酸对应密码子的RSCU值整理求平均值后,得到MsPIPs家族密码子的RSCU分析结果。各氨基酸中具有最高RSCU值的20个密码子被确定为高频密码子(表2)。27个密码子被确定为高表达密码子(△RSCU>0.08)(表3)。10个密码子被确定为最优密码子,分别是UUU、GUU、CCA、ACU、GCU、UAC、CAA、AAA、UGU和GGU。
2.3苜蓿MsPIPs密码子的偏好性
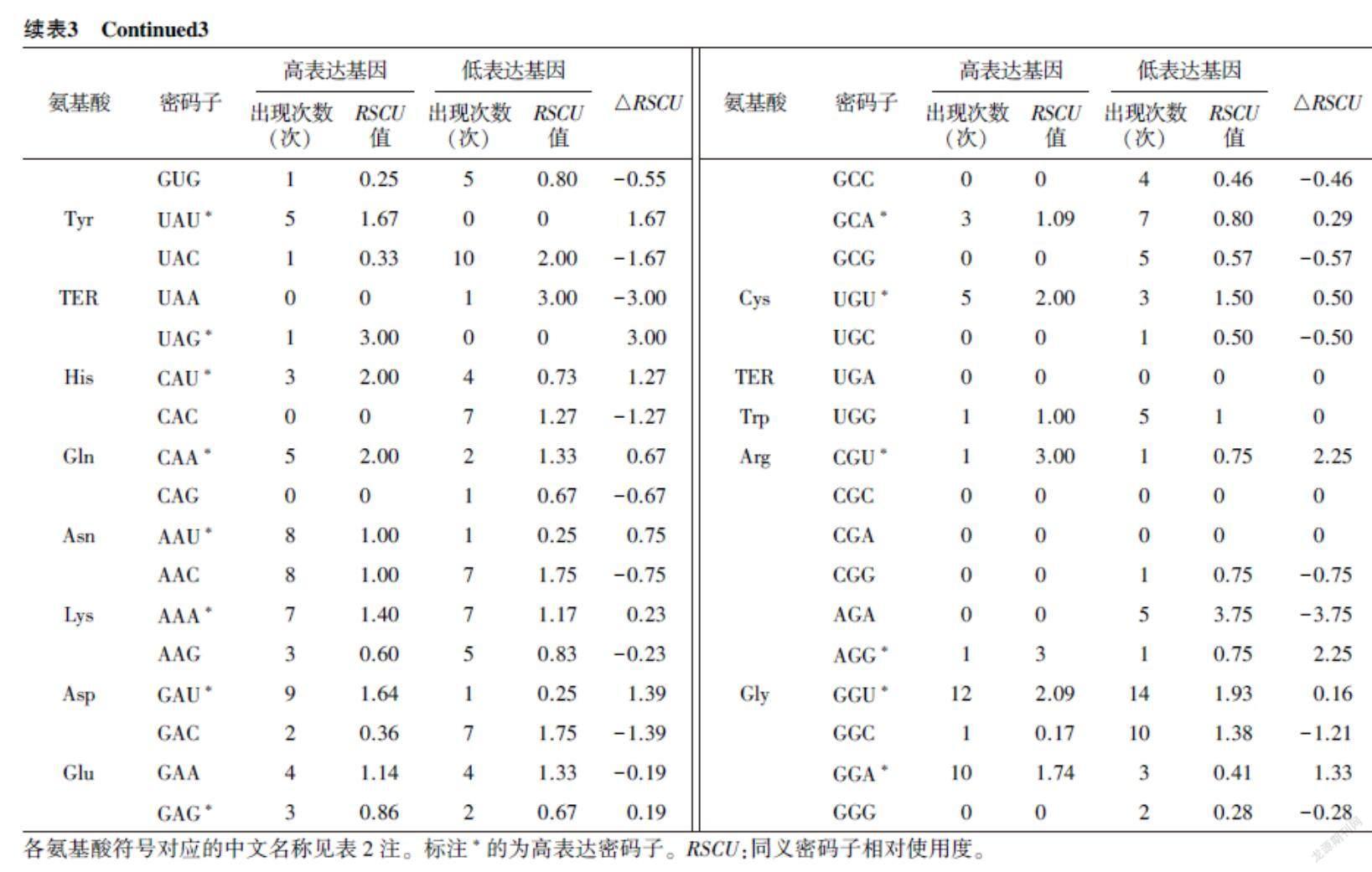
基于RSCU值的PCA结果显示,26个MsPIPs的密码子使用频率分布在25个轴上,向量轴1~4分别解释了20.65%、16.18%、14.57%和13.81%的轴贡献,累计贡献率为65.21%,表明向量轴1~4对密码子偏好性有重要影响(图1)。以第1和第2向量轴将MsPIPs分布在图上,可以看出大部分成员分布在向量轴1的右半轴上(图2)。进一步通过COA分别分析了G+C含量(GC)、ENC值、GC3S和向量轴1~4的相关性。结果表明,GC3S与向量轴1和轴3呈极显著负相关,ENC值与向量轴2和轴4呈显著或极显著正相关,G+C含量与向量轴3也呈显著负相关(表4)。
T3S:第3位碱基上T出现的频率;C3S:第3位碱基上G出现的频率;A3S:第3位碱基上A出现的频率;G3S:第3位碱基上G出现的频率;GC3S:第3位碱基上G+C出现的频率;GC:G+C含量;ENC:有效密码子数。
GCG150.06TER:终止子;Phe:苯丙氨酸;Leu:亮氨酸;Ile:异亮氨酸;Met:甲硫氨酸;Val:缬氨酸;Ser:丝氨酸;Tyr:酪氨酸;His:组氨酸;Gln:谷氨酰胺;Asn:天冬酰胺;Lys:赖氨酸;Asp:天冬氨酸;Glu:谷氨酸;Cys:半胱氨酸;Pro:脯氨酸;Thr:苏氨酸;Ala:丙氨酸;Trp:色氨酸;Arg:精氨酸;Gly:甘氨酸。
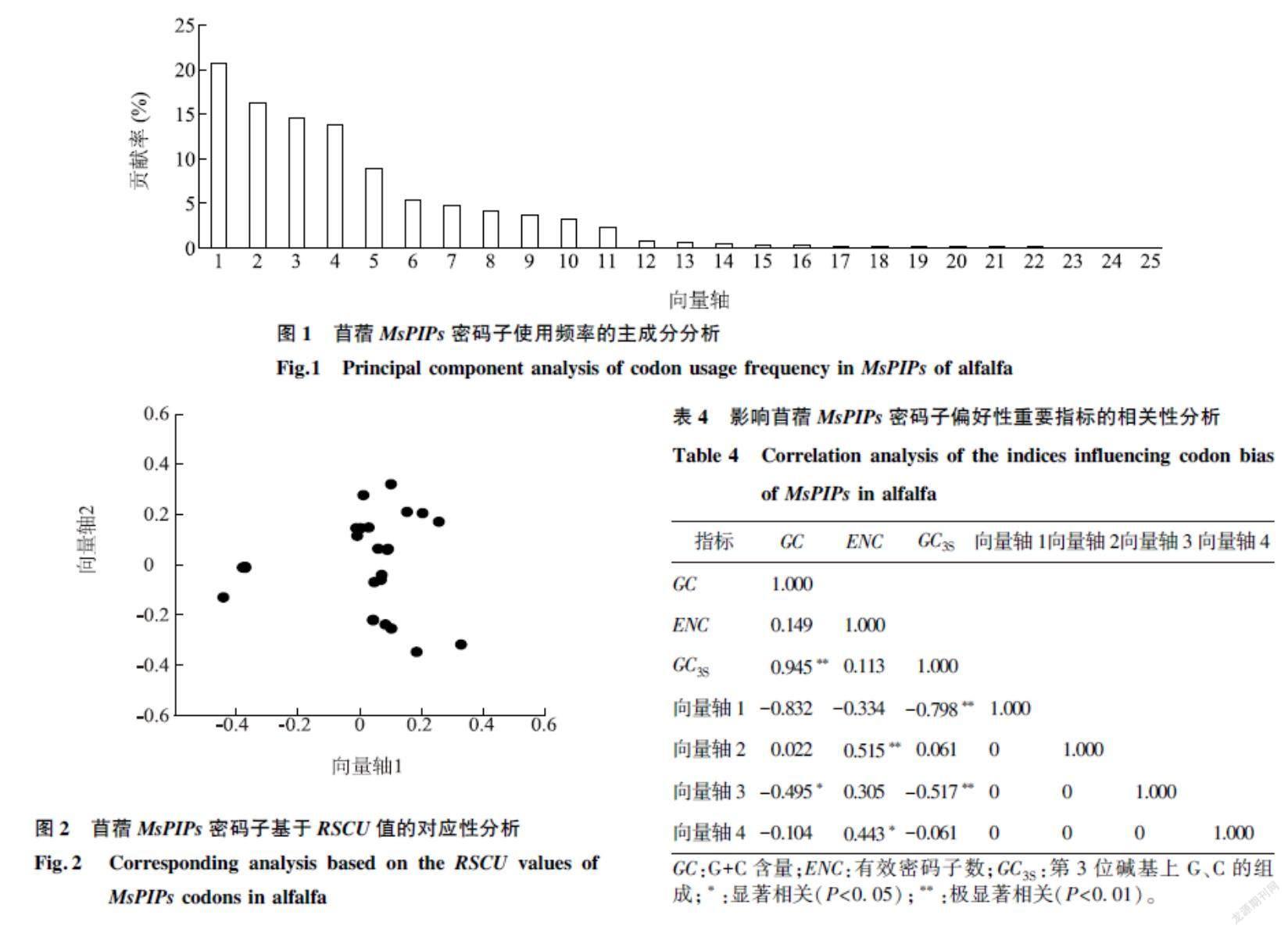
通过PR2分析组成MsPIPs氨基酸的第3位密码子中A/G和T/C的关系(图3)。除MsPIP17和MsPIP21分布在第4象限,MsPIP6分布在中轴线上,其余MsPIPs均分布在第3象限,表明MsPIPs的密码子组成存在偏倚,且大部分MsPIPs结尾T的使用频率高于A,C的使用频率高于G。
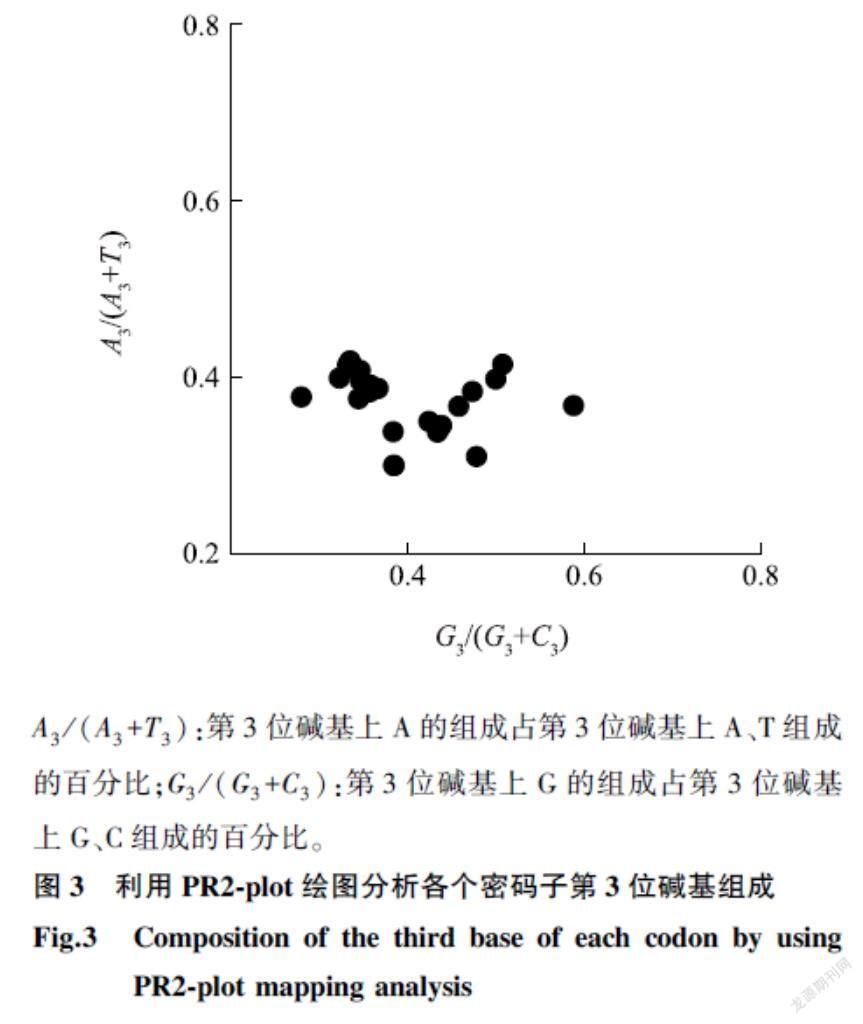
如果基因密码子的组成完全受突变的影响,则ENC的分析图中,所有基因应该落在标准曲线上。但是从ENC的分析结果可见,MsPIPs的所有基因成员均分布在预期ENC值的下方(图4),表明MsPIPs的密码子偏好受其他因素影响。此外,由表4可见,ENC和GC3s之间并无显著相关性(r=0.113),表明第3位碱基上的G/C含量对密码子的使用模式影响较小。
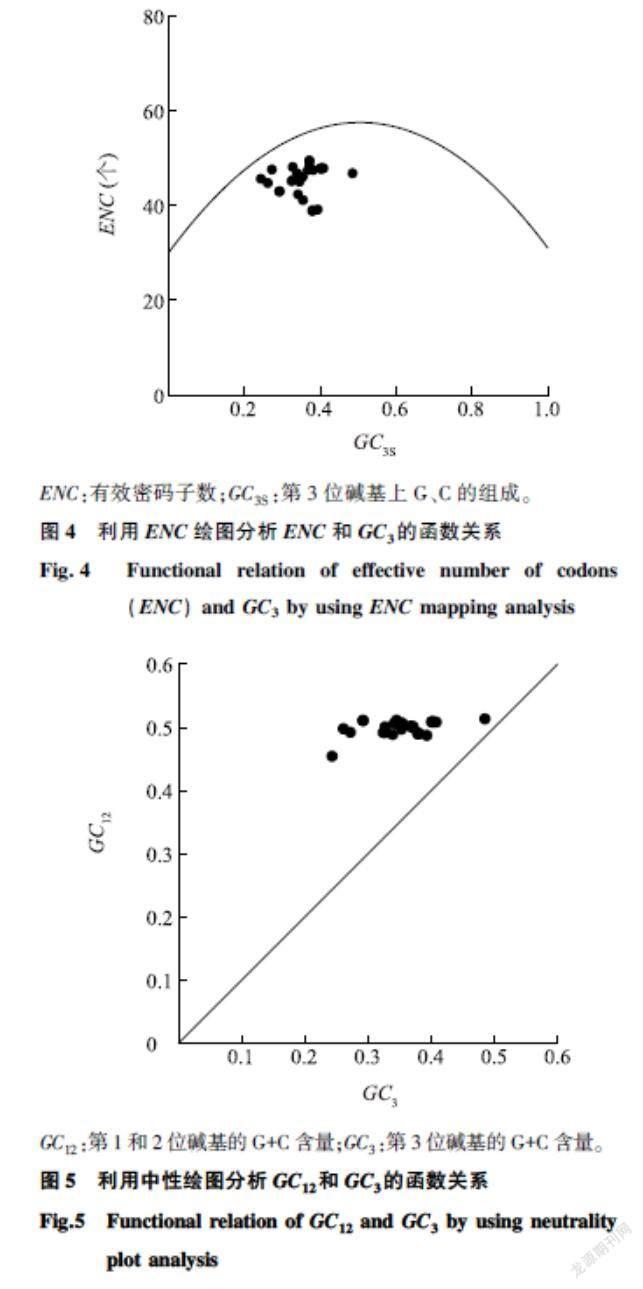
中性分析图将GC12和GC3的关系可视化,并可进一步帮助评估密码子组成受突变和选择压力影响的程度。由图5可见,MsPIPs的所有基因成员均分布在对角线上方。
A3/(A3+T3):第3位碱基上A的组成占第3位碱基上A、T组成的百分比;G3/(G3+C3):第3位碱基上G的组成占第3位碱基上G、C组成的百分比。
ENC:有效密码子数;GC3S:第3位碱基上G、C的组成。
GC12:第1和2位碱基的G+C含量;GC3:第3位碱基的G+C含量。
3讨论
密码子偏好性是多因素共同作用的结果,受自然选择、碱基组成、基因表达水平、蛋白质二级结构等因素的影响[14-16],其中自然选择和突变是主导密码子偏好性的2类主要因素。杨国锋等[17]发现蒺藜苜蓿叶绿体基因组的密码子偏好性主要受到突变的影响。屈蒙蒙等[18]对香蕉质膜内在蛋白(PIPs)的密码子偏好性分析结果表明,该家族基因受自然选择的影响较大。可见,不同物种、不同基因类型的同义密码子偏好性不同。
本研究结果表明,苜蓿MsPIPs家族基因的密码子多以T/A结尾。在得到的10个最优密码子中,有9个也是以T/A结尾。通过PR2分析,进一步证实密码子的第3位碱基組成存在偏倚。本研究结果与其他学者在翠雀[1]、沙枣[12]、蒺藜苜蓿[17]等植物中的研究结果一致。ENC值直接反映密码子偏好性,以35为界,ENC值≤35表示密码子偏好性强,ENC值>35则表示密码子偏好性弱[19]。苜蓿MsPIPs密码子的平均ENC值为45.47,表明MsPIPs存在密码子偏好性,只是程度较弱。
通过PCA、COA、ENC-plot和中性分析,进一步分析造成MsPIPs密码子偏好性的主要原因。从PCA和COA的结果可见,MsPIPs的成员主要分布在轴1的右半轴上,而轴1与GC3s呈显著负相关关系,这表明第3位碱基组成(主要是A/T结尾)对MsPIPs的密码子偏好性有影响。轴1仅解释20.65%的密码子使用频率差异,可见除碱基组成外,还有其他因素对MsPIPs的密码子偏好性产生影响。ENC-plot分析能通过基因和标准曲线的分布关系直接反映自然选择和突变因素中的主导因素,如果基因落在标准曲线上,则表明基因的密码子偏好性完全受突变的影响,反之则主要受自然选择的影响[20]。本研究结果表明,MsPIPs所有成员均落在标准曲线下方,表明它们可能主要受到自然选择的影响。这一推测也被中性分析进一步证实。中性分析能有效地比较自然选择和突变对密码子偏好性的影响程度,当GC12和GC3数值相同时,基因会分布在回归曲线上,且该基因不同位置的碱基组成无差异,密码子使用更多地受突变影响,反之则主要受自然选择的影响[21]。MsPIPs的中性分析中,所有基因均分布在回归曲线的上方,表明所有成员主要受到自然选择的影响。
4结论
苜蓿质膜水通道蛋白家族编码基因(MsPIPs)的密码子偏好性较弱,碱基使用存在偏倚,偏好使用以A/T结尾的密码子,且偏好性主要受自然选择的影响。本研究结果可为通过密码子改造MsPIPs提供理论依据。
参考文献:
[1]DUAN H R, ZHANG Q, WANG C M, et al. Analysis of codon usage patterns of the chloroplast genome in Delphinium grandiflorum L. reveals a preference for AT-ending codons as a result of major selection constraints [J]. PeerJ, 2021, 9(6): e10787.
[2]SAU K, GUPTA S K, SAU S, et al. Factors influencing synonymous codon and amino acid usage biases in Mimivirus [J]. Biosystems, 2006, 85: 107-113.
[3]GUAN D L, MA L B, KHAN M S, et al. Analysis of codon usage patterns in Hirudinaria manillensis reveals a preference for GC-ending codons caused by dominant selection constraints [J]. BMC Genomics, 2018, 19: 542.
[4]BULMER M. The selection mutation drift theory of synonymous codon usage [J]. Genetics, 1991, 129: 897-907.
[5]AYADI M, BRINI F, MASMOUDI K. Overexpression of a wheat aquaporin gene, TdPIP2;1, enhances salt and drought tolerance in transgenic durum wheat cv. Maali [J]. International Journal of Molecular Sciences, 2019, 20(10): 2389.
[6]孙天旭, 李玉花, 张旸. 逆境条件下水孔蛋白PIPs作用的研究进展[J]. 植物生理学报, 2014, 50(6): 749-757.
[7]刘海莉. 苹果质膜水通道蛋白基因PIP2;1的耐旱及耐盐性功能分析[D]. 杨凌:西北农林科技大学, 2019.
[8]王星,张纪龙,冯秀秀,等. 植物质膜水通道蛋白转运及逆境胁迫响应的分子调控机制[J]. 遗传, 2017, 39(4): 293-301.
[9]CHEN H T, ZENG Y, YANG Y Z, et al. Allele-aware chromosome-level genome assembly and efficient transgene-free genome editing for the autotetraploid cultivated alfalfa [J]. Nature Communications, 2020, 11: 2494.
[10]ZHANG Y Y, SHI E, YANG Z P, et al. Development and application of genomic resources in an endangered palaeoendemic tree, Parrotia subaequalis (Hamamelidaceae) from eastern China[J]. Frontiers in Plant Science, 2018, 9: 246.
[11]CUI G X, WANG C M, WEI X X, et al. Analysis of synonymous codon usage of the complete chloroplast genome in Phleum pratense cv. Minshan[J]. International Journal of Agriculture & Biology, 2020, 24(2): 352-358.
[12]王婧,王天翼,王罗云,等. 沙枣叶绿体全基因组序列及其使用密码子偏性分析[J]. 西北植物学报, 2019, 39(9): 1559-1572.
[13]WEI L, HE J, JIA X, et al. Analysis of codon usage bias of mitochondrial genome in Bombyx mori and its relation to evolution [J]. BMC Evolutionary Biology, 2014, 14: 262.
[14]惠小涵,程婷婷,柯卫东,等. 莲藕PPO基因密码子偏好性特征分析[J]. 江苏农业学报,2020,36(2):438-446.
[15]张玉波,周正湘,吴小玉,等. 基于转录组的大头金蝇密码子的偏好性分析[J]. 江苏农业科学,2019,47(11):78-81.
[16]赵森,邓力华,陈芬. 不同植物FERONIA基因密码子使用偏好性分析[J].江苏农业学报,2020,36(5):1073-1081.
[17]杨国锋,苏昆龙,赵怡然,等. 蒺藜苜蓿叶绿体密码子偏好性分析[J]. 草业学报, 2015, 24(12): 171-179.
[18]屈蒙蒙,孙雪丽,郝向阳,等. 香蕉水通道蛋白基因家族的密码子偏好性分析[J]. 园艺与种苗, 2018(5): 4-10.
[19]SONG H, LIU J, CHEN T, et al. Synonymous codon usage pattern in model legume Medicago truncatula[J]. Journal of Integrative Agriculture, 2018, 17: 2074-2081.
[20]WRIGHT F. The effective number of codons used in a gene[J]. Gene, 1990, 87: 23-29.
[21]ZHANG D S, HU P, LIU T G, et al. GC bias lead to increased small amino acids and random coils of proteins in coldwater fishes[J]. BMC Genomics, 2018, 19: 315.
(責任编辑:张震林)
收稿日期:2021-03-29
作者简介:张海霞(1975-),女,甘肃民勤人,本科,高级工程师,研究方向为林草有害生物防治。(E-mail)1098149863@qq.com
通讯作者:王玉道,(E-mail)1602239412@qq.com

