基于多神经网络协作的电子病历命名实体识别方法
张运中 纪 斌 余 杰 刘慧君
1(湖南省电子口岸服务中心 湖南 长沙 410001)2(国防科技大学计算机学院 湖南 长沙 410073)3(中国工程物理研究院计算机应用研究所 四川 绵阳 621999)
0 引 言
随着电子病历的迅速普及和医疗大数据时代的到来,自然语言处理(Natural Language Processing, NLP)技术在医学领域的应用与发展已经成为当前的研究热点。NLP相关技术,如句子的分词、实体识别等,可以从临床医疗记录中提取有科研价值信息,帮助科研人员进行的学术研究,从而可以支持医疗研究和辅助治疗方案决策[1]。
命名实体识别(Named Entity Recognition, NER)是自然语言处理里的一项基础任务。狭义上,NER是识别出人名、地名和组织机构名这三类命名实体[2]。临床医疗命名实体识别是医疗信息抽取最基础的任务,国内诸多有影响力的学术会议将其作为评测任务以推进其研究与发展,如中国知识图谱与语义计算大会(China Conference on Knowledge Graph and Semantic Computing, CCKS)[3]、中国健康信息处理会议(China Health Information Processing Conference, CHIP)等。这些评测任务既推动了医疗命名实体识别的研究与发展,也为后续的研究提供了一批高质量的数据集。
CHIP2018发布中文电子病历临床医疗命名实体识别评测任务[4],此项评测任务来自工业界的真实应用,因此更具有研究价值和挑战性。此次评测任务的目的是从电子病历中抽取出三种恶性肿瘤相关的命名实体,并发布了600份人工标注的病历作为训练数据,200份无标注的病历作为测试数据,在本文中分别用CHIP TR和CHIP TE标识。由于这三种实体的复杂性和特殊性质,单一神经网络模型难以有效地完成本任务。针对此任务,本文提出了一种基于多神经网络协作的复杂医疗命名实体识别方法,通过多种神经网络模型协作的方式实现了复杂医疗命名实体有效识别,并且通过句子级别上的模型迁移应用解决了训练数据集较小及数据分布不一致的问题。本文的贡献可总结如下:
(1) 对于难以通过单一的神经网络模型完成的真实复杂医疗命名实体识别任务,深入分析实体特点,挖掘实体间的依赖关系,提出基于多神经网络协作的复杂医疗命名实体识别方法,有一定的工程实践价值。
(2) 本文方法相对于其他使用规则的方法有更好的泛化能力,在CHIP 2018评测任务中取得了第二名的成绩。
(3) 本文方法的改进版本取得了CCKS2019评测任务一的第一名,为后续的相关研究提供了一个有效的基线成绩。
1 相关研究
医学命名实体识别指的是确定医学领域文本中的专业术语的边界,然后基于领域信息对它们进行分类[5]。目前医学命名实体识别的主要方法分为浅层机器学习和深度学习的方法。浅层机器学习方法主要包括HMM、ME、CRF、SVM,以及上述分类模型的改进等[6]。Wang等[7]验证了基于CRF的Gimli方法,在JNLPBA 2004数据集上取得了72.23%的F1值;于楠等[8]提出了多特征融合的条件随机场方法,可以准确识别中文电子病历中疾病和症状实体,同时也可准确识别未登录词。浅层机器学习方法在很大程度上依赖于人工特征的设计。为减少复杂的人工特征,Tang等[9]采用CRF模型进行生物医学实体识别,在基本人工特征的基础上加入不同的词向量特征,在JNLPBA 2004数据集上取得了71.39%的F1值。Chang等[10]利用少量的人工特征和词向量结合的方式构建CRF模型并添加后处理,在JNLPBA 2004语料上取得了71.77%的F1值。
在使用深层神经网络进行医学命名实体识别的研究中,Yao等[11]首先在无标注的生物医学文本上利用神经网络生成词向量,然后建立多层神经网络,在JNLPBA 2004数据集上取得了71.01%的F1值。Li等[12]采用BiLSTM模型在BioCreative II GM的数据集上取得了88.6%的F1值,同时在JNLPBA 2004语料上取得了72.76%的F1值。李丽双等[13]提出了一种基于CNN-BLSTM-CRF神经网络模型,在Biocreative II GM和JNLPBA 2004数据集上达到了最优的F1值。
此外,基于规则的方法将手工编写的规则与文本进行匹配来识别命名实体,是一种非常有效地命名实体识别的方法[14]。但基于规则方法需要领域专业知识和专业的人员编写规则,并且规则跨领域迁移应用能力较差,基本不具有泛化能力。
2 实体识别方法
2.1 实体定义与分析
CHIP2018评测任务中的肿瘤原发部位、原发肿瘤大小、肿瘤转移部位定义[15-16]如下:
(1) 肿瘤原发部位:肿瘤原发的身体部位,区别于肿瘤转移部位。通常情况下,肿瘤原发部位的下文为“癌”“恶性肿瘤”“MT”“CA”等。
(2) 原发肿瘤大小:描述原发肿瘤长度、面积或体积的量度,包括,常见度量单位有mm、cm等。
(3) 肿瘤转移部位:原发肿瘤的转移部位,理论上除肿瘤原发部位外,肿瘤可向身体任何其他部位转移。
从上述三种实体的定义中可以得出,作为一种描述肿瘤大小的量度,原发肿瘤大小依赖于肿瘤原发部位。一个基于统计得到的事实是原发肿瘤大小与肿瘤原发部位在电子病历中是句子级别共存的,也就是说在绝大多数情况下原发肿瘤大小和肿瘤原发部位出现在同一个句子中。
肿瘤原发部位和肿瘤转移部位都属于身体部位或组织,在电子病历中这两种实体较为稀疏。一般情况下,一份病历中只有一个肿瘤原发部位,数个肿瘤转移部位。但电子病历中包含大量的不属于两类实体的身体部位。并且对于肿瘤转移部位来说,只有“转移”这一特征描述词可以用于辨别一个身体部位是否属于肿瘤转移部位,但这种辨别能力随着句子长度的增加而削弱。现在主流的神经网络模型大多将命名实体识别作为序列标注任务,其基于统计原理的本质决定了当电子病历中包含了大量的与抽取任务无关的身体部位时,肿瘤转移部位的抽取不会有优异的性能。
基于上述分析,将CHIP2018评测任务分解为三个子任务:肿瘤原发部位抽取,原发肿瘤大小抽取和肿瘤转移部位抽取。
2.2 方法设计
图1为基于神经网络模型的复杂临床医疗命名实体抽取方法架构。
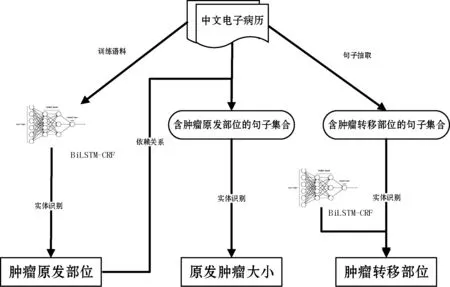
图1 临床医疗命名实体抽取方法架构
(1) 肿瘤原发部位抽取。肿瘤原发部位的抽取是一个典型的命名实体识别过程,采用经典的BiLSTM-CRF模型抽取肿瘤原发部位,模型框架结构如图2所示。

图2 BiLSTM-CRF模型框架结构图
BiLSTM-CRF模型实现句子级别的命名实体识别。模型的第一层是embedding层,其作用是在将句子输入到模型之前,将句子转换为向量表达。从图2中可以看出,本文中的BiLSTM-CRF模型基于字符embedding。具体来说,就是将句子中的每个字符用字符embedding表示,最后得到关于句子的向量表示序列。假设一个句子X含有n个字,则该句的向量表达可表示为X=(x1,x2,…,xn),其中xi∈Rd,d是字符embedding的维度。
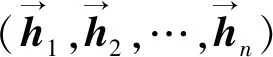
模型的第三层是CRF层,进行句子级的序列标注。CRF层的参数矩阵是一个维度为(k+2)×(k+2)的状态转移矩阵A,其中Aij表示从第i个标签到第j个标签的转移得分,因此在为句子的一个字符进行标注的时候可以利用此前已经标注过的标签信息。假设y=(y1,y2,…,yn)为一个长度等于句子长度的标签序列,那么模型对于句子X的标签序列等于y的计算公式如下:
式中:Pi,yi表示将xi标注为yi的概率,由隐状态Hi计算得到。
模型在预测过程时使用动态规划的Viterbi算法来求解最优路径[8]。
BiLSTM-CRF模型的超参数设置如表1所示,用 BiLSTM-CRF-T标识。训练数据采用BIO[15]的标注模式,依据人工标注信息将CHIP TR处理成适合模型训练的格式。用B-TU、I-TU代表肿瘤原发部位首字和非首字,用O标注不属于命名实体的字符。一个数据标注示例如图3所示。
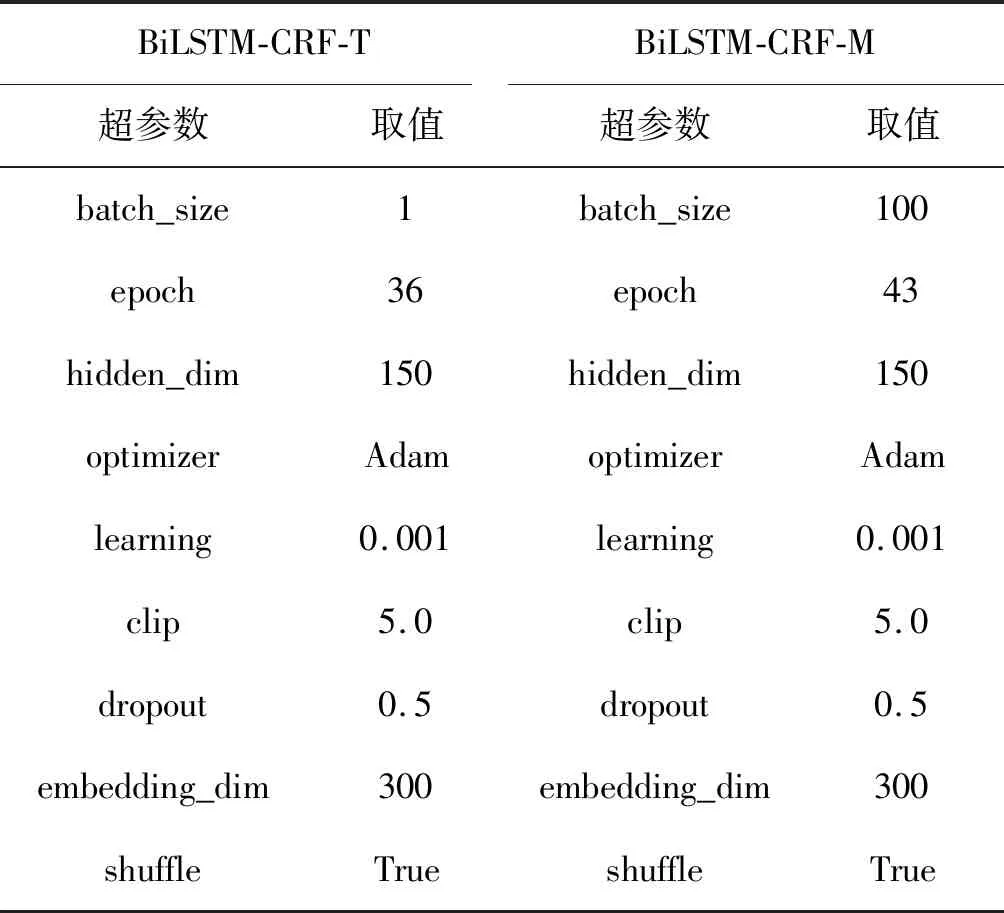
表1 神经网络模型的超参数设置
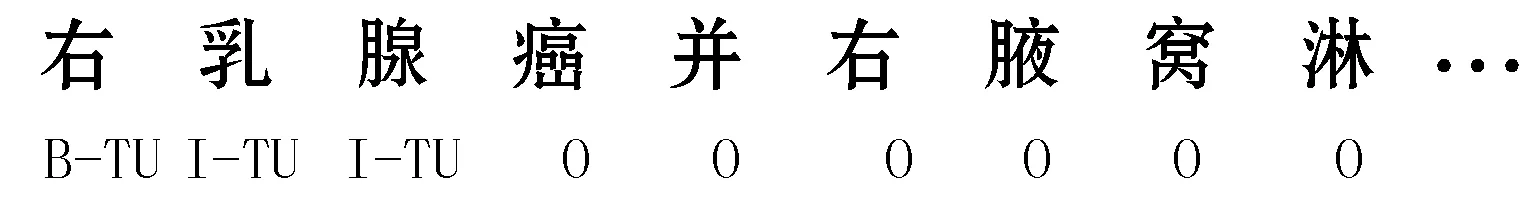
图3 语料标注示例
(2) 原发肿瘤大小抽取。原发肿瘤大小是由数字、长度单位(mm或cm)、表示乘法的二元符号(*、×、X等)组成按照一定的规则构成的描述原发肿瘤的量度。本文采用了基于规则的方法抽取原发肿瘤大小,其抽取流程如下:
① 预处理电子病历。将“?”“?”“;”“;”等标点符号替换为“。”,并依据“。”分割电子病历,得到句子集合。
② 句子筛选。对于第①步得到的句子集合中的每个句子,若其不包含肿瘤原发部位,则将其从句子集合中移除。将句子集合中剩余的句子组合成为短文本。
③ 实体抽取。依据电子病历中的原发肿瘤大小的度量的符号组成规则,编写正则表达式(如式(1)所示),并用其抽取第②步获取的短文本中的度量。
RE=′(d?d?d?.?d?d(([cm][mm]?)|(.?.?[*×
X~].?d?d?d?.?d?))*[cm][mm])′
(1)
原发肿瘤大小的抽取依赖于肿瘤原发部位的抽取结果,因此若肿瘤原发部位的抽取错误,则可能会导致原发肿瘤大小抽取错误,引起错误传播。
(3) 肿瘤转移部位抽取。肿瘤转移部位与其他两种实体无明显的内在关系,并且“转移”作为唯一特征,难以用来抽取长句中的多个肿瘤转移部位。一种启发式的抽取方法如下:
① 电子病历预处理。一个基于统计得到的事实是:包含肿瘤转移部位的句子中绝大多数包含“转移”;在包含其他关键字的情况下,如“考虑转移”、“不除外转移”等,此句的前一句中包含的肿瘤转移部位,统计得到的关键字如下所示。基于关键字列表编写规则筛选包含肿瘤原发部位的句子。
关键字列表={考虑转移,转移,倾向转移,倾向为转移,转移可能,不除外转移,转移不除外,转移待排,疑转移,转移可能,转移不除外,考虑为转移,可疑淋巴结转移,考虑转移性淋巴结,转移性可能,转移瘤可能,考虑多发转移,转移征象可能,转移瘤不除外,转移不能除外,考虑骨转移,转移待除外,考虑为转移瘤,转移可能性大,考虑肺转移,考虑为骨转移,转移?,转移均不除外,均考虑转移不除外,均为骨转移改变,均考虑转移}
② 使用BiLSTM-CRF模型抽取句子中的解剖部位。
③ 处理解剖部位,获取肿瘤转移部位实体。后处理包括实体去重、实体特殊格式处理。其中特殊格式实体主要为补全与淋巴结相关的实体,如:“左侧腮腺、双颈、右侧锁骨上区间隙多发淋巴结,考虑转移”的神经网络模型识别结果为“左侧腮腺”“双颈”“右侧锁骨上区”“淋巴结”,需要将上述实体补全为“左侧腮腺淋巴结”“双颈淋巴结”“右侧锁骨上区淋巴结”。
CHIP TR和CHIP TE中肿瘤原发部位、原发肿瘤大小、肿瘤转移部位的数量统计如表2所示。可以看出,两个数据集中的肿瘤转移部位的实体较为稀疏,因此从解剖部位数量和种类上来看,基于CHIP TR不足以支撑训练性能优异的神经网络模型。在此,引入了CCKS2018评测任务一发布的600份电子病历数据集,该数据集由清华大学知识工程实验室和医渡云(北京)技术有限公司联合提供。本文用CCKS TR标识该数据集,CCKS TR中有8 542个带有标注信息的解剖部位,完全可以满足神经网络模型的训练需求。尽管CCKS TR和CHIP数据集应用场景不同,但是二者在解剖部位的表现形式上是一样的。基于CCKS TR训练的神经网络模型迁移应用于筛选的CHIP数据集的句子上既可以消除数据不一致带来的影响,又解决了CHIP数据集实体稀疏的问题。应用于肿瘤转移部位的抽取BiLSTM-CRF模型结构与图2所示的模型结构一致。模型的参数设置如表1所示,用BiLSTM-CRF-M标识。
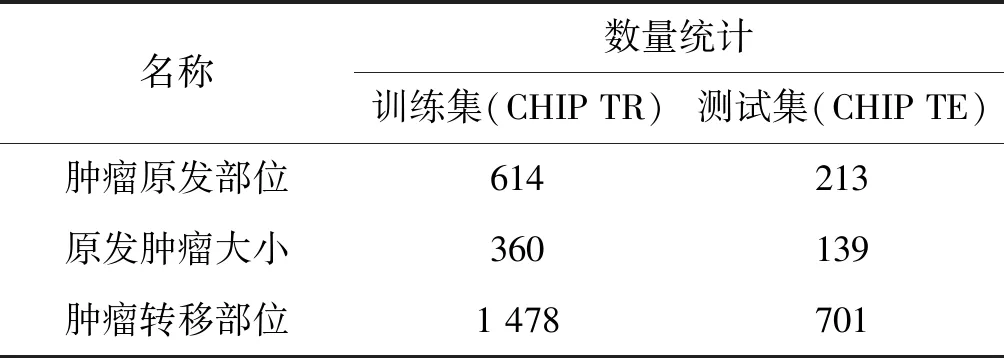
表2 CHIP2018数据集实体统计
3 实 验
3.1 评估标准
本文共使用两种评估标准:标准评估和权重评估。
(1) 标准评估。标准准确率(P)、召回率(R)和F1值(F1)作为标准评估的评估指标,分别使用以下三个公式计算:
(2)
(3)
(4)
(2) 权重评估。权重评估由CHIP2018评测任务定义,用于计算带权重的准确率、召回率和F1值。若用T、S、M分别表示肿瘤原发部位、原发肿瘤大小、肿瘤转移部位,那么在权重评估标准下TP、FP、FN的计算公式如下:
TP=0.2×TPT+0.3×TPS+0.5×TPM
(5)
FP=0.2×FPT+0.3×FPS+0.5×FPM
(6)
FN=0.2×FNT+0.3×FNS+0.5×FNM
(7)
而标准评估标准下三者的计算公式如下:
TP=TPT+TPS+TPM
(8)
FP=FPT+FPS+FPM
(9)
FN=FNT+FNS+FNM
(10)
3.2 实验结果
本文方法在CHIP TE上的测试结果如表3所示,测试结果由CHIP 2018评测平台提供。

表3 命名实体识别方法在CHIP TE数据集上的评估结果%
可以看出,本文方法在CHIP TE上获得了78.38%的权重F1值,在此次评测任务中排名第二。此次评测任务的前四名的成绩统计如表4所示。

表4 CHIP2018评测任务一前四名成绩 %
可以看出,在排名前四的方法中,本文方法是唯一基于神经网络的方法。相比于基于规则的方法,本文方法减少了编写规则的工作量,有更好的泛化能力。
CCKS2019发布了一项与CHIP2018任务形式相同的评测任务,并且提供了900份电子病历作为训练数据集。为验证本文方法的泛化能力,将其迁移应用到CCKS2019数据集上,测试结果如表5所示。

表5 命名实体识别方法在CCKS2019数据集上的评估结果 %
可以看出,本文方法在CCKS2019数据集上取得了带权重的69.09%的F1值,比在CHIP TE上的评估结果低9.29个百分点。深入研究后发现,两次评测任务标准的不完全一致是导致本文方法性能下降较大的原因:在CHIP2018评测任务中,肿瘤原发部位的是不带有方位词的,但在CCKS2019评测任务中要求带有方位词;CCKS2019评测任务不需要对淋巴结相关的实体进行补全。在3.2节给出的例子中,BiLSTM-CRF模型的识别结果就是CCKS2019要求的正确结果。
依据CCKS2019评测任务一的具体任务定义形式对本文方法进行改进,最终在CCKS2019评测任务一中取得了第一名的成绩[17]。简而言之,本文方法的有效性和泛化能力在CCKS2019数据集中得到了验证。
4 结 语
本文提出一种基于神经网络的电子病历命名实体识别方法,探究了融合使用多种神经网络模型实现复杂的、难以通过单一神经网络模型完成的医疗命名实体识别,有一定的工程实践价值。在CHIP2018和CCKS2019评测任务中,本文方法及改进方法分别取得了优异的成绩,验证了本文方法的有效性和泛化能力,为后续相关研究提供了一个有效的基线成绩。
未来仍然还需要许多的工作来完善本文方法。首先,本文方法中仍然使用了基于规则的方法抽取原发肿瘤大小和包含肿瘤转移部位句子,因此未来的工作之一是使用基于神经网络的方法替换基于规则的方法,以进一步提高本文方法的性能和泛化能力。其次,本文方法中使用的两个BiLSTM-CRF模型均是基于随机初始化的字符embeddings,而领域相关的预训练的字符embeddings可以有效提高命名实体识别性能[18-19],因此未来的第二个工作是预训练领域相关的字符embeddings,以进一步提高本文方法的性能。

