基于生信分析ATRA治疗APL后的关键差异基因
罗斯琦,娄世锋
(重庆医科大学附属第二医院血液内科,重庆 400010)
1 研究背景与思路分析
急性早幼粒细白血病(acute promyelocytic leukemia,APL)是一种以骨髓分化在早幼粒细胞阶段停滞导致细胞蓄积为特征的急性髓系细胞白血病(acute myelocytic leukemia,AML),在WHO分型中属M3型急性髓系白血病(AML-M3),APL患者t(15;17)(q22;q21)染色体易位产生PML-RARα融合基因[1],该融合基因是导致APL中骨髓细胞分化停滞的主要机制[2]。全反式维甲酸(all-trans-retinoic acid,ATRA)联合三氧化二砷对APL患者的诱导分化治疗是目前常用的靶向治疗药物,治愈率高,可以将完全缓解(completeremission,CR)率 提 高 到90%[3],ATRA在APL细 胞 中 诱 导 分 化 的 机制包括:(1)ATRA将PML-RARa从转录阻遏物转化为转录激活物[4];(2)ATRA通过半胱氨酸蛋白酶或泛素/蛋白酶体系统降解PML-RARa融合蛋白,同时释放野生型RXR和PML,并诱导PMLNBs重组,PML/RARalpha直接参与赋予APL细胞ATRA敏感性,并 且ATRA诱导的PMLNBs重 组是PML/RARalpha消 失 的 结 果[5];(3)RAF-1/MEK/ERK信 号通路被激活并通过调节C/EBPb,C/EBPe和PU.1的蛋白质水平来调节ATRA诱导的APL细胞分化[6],第四为一个非基因组途径,ATRA激活PKA从而导致ATRA靶基因的转录增加和APL的分化[7]。在ATRA治疗过程中,可能出现分化综合征(Differentiationsyndrome,DS),又称维甲酸综合征(Retinoicacidsyndrome,RAS)[8],同时仍有ATRA治疗后复发的情况发生,减少APL患者的早期死亡率以及减少复发率,是APL治疗的研究热点。因此,本研究旨在通过生物信息学工具分析ATRA处理APLNB4细胞(一种从人急性早幼粒细胞白血病(M3)中分离的具有t(15;17)标记的成熟诱导细胞系,NB4)前后基因表达谱差异,并寻找差异基因富集的信号途径,探讨全反式维甲酸治疗急性早幼粒细白血病的可能分子作用机制,同时筛选出信号通路中高表达提示预后不良的关键基因,为寻找APL治疗的新靶点提供思路。
2 资料和方法
2.1 一般资料
以美国国立生物技术信息中心(NCBI)的高通量基因表达数据库(GEO)为工具,以“all-trans-retinoic acid”为关键词,选取并下载下载ATRA分化的野生型和TG2基因敲除NB4细胞的基因表达谱数据集(GSE23702),其来源于德布勒森大学,采用平台为GPL6244[HuGene-1_0-st] Affymetrix Human Gene 1.0 ST Array [transcript (gene) version]。选取其中ATRA处理前、48h、72h后的野生型APL NB4细胞基因表达数据,每个时间点选取3个样本。
2.2 方法
2.2.1 基因芯片质量验证
使用GEO2R(https://www.ncbi.nlm.nih.gov/geo/geo2r) R boxplot对表达式数据应用分位数生成箱线图,如果若某个芯片的箱线图出现较大偏差则表明该芯片存在问题。使用R limma (plotDensities)生成表达密度图,如果图中曲线相差偏离大,则在差异表达分析之前进行数据归一化。使用limma(qqt) 生成t统计分位线图评估limma测试结果的质量。
2.2.2 差异基因筛选
在GEO2R使 用Bioconductor项 目 中 的GEOquery和limma R包对分析筛选GEO数据集中不同数据组的差异表达水平[9]。在GSE23702数据集中以未经过ATRA处理过的野生型APL NB4细胞基因表达数据(GSM594067、GSM594068、GSM594069)为对照组分别与ATRA处理48h(GSM594073、GSM594074、GSM594075)、72h(GSM594079、GSM594080、GSM594081)后的野生型APL NB4细胞基因表达数据对比,筛选标准为矫正后P<0.05并且l log(Fc)I>2,使用GEO2R在线工具limma(volcanoplot)包生成火山图,并显示统计显着性(-log10P值)与变化幅度(log2倍变化)的对比,使差异基因可视化,最后使用limma(vennDiagram)包生成维恩图,用于探索和下载多个对比之间重要基因的重叠,可联合使用在线工具Draw Venn Diagram(http://bioinformatics.psb.ugent.be/webtools/Venn/)筛选出在ATRA处理48h和72h后同时上调或下调的DEGs用于进一步分析。
2.2.3 基因本体论(gene ontology,GO)富集分析和京都基因与基因组百科全书(Kyotoencyclopedia of genes and genomes,KEGG)通路筛选
用DAVID 6.8在线数据库(https://david.ncifcrf.gov/tools.jsp)对DEGs进行基因本体论(gene ontology,GO)注释和京都基因与基因组百科全书(Kyotoencyclopedia of genes and genomes,KEGG)通路分析[10],联合基于KEGG的注释系统KOBAS(http://kobas.cbi.pku.edu.cn/kobas3/?t=1)寻找差异基因富集的关键信号通路[11]。
2.2.4 PPI网络构建和分析
使 用String在 线 数 据 库(https://string-db.org/)对 上 述DEGs进行分析,构建PPI网络[12]后下载并使用Cytoscape(Version3.8.2)软件进行可视化[13],利用cytoHubba插件分析预测该网络中最高相关度的前10个DEGs作为ATRA诱导细胞分化相关的关键基因,即hub基因[14]。
2.2.5 统计学分析
运用GEO2R工具的Benjamini & Hochberg法分析GSE23702表达谱芯片数据,采用SAM法筛选芯片数据中DEGs。在DAVID数据库中,通过Bonferroni 校正法和Bootstrap 法等进行GO分析的检验,利用Fisher精确概率检验和基因富集分析等方法进行KEGG分析。在Cytoscape中,选取CytoHubba插件中MCC拓扑分析算法进行数据分析。
3 结果
3.1 芯片质量验证
用GEO2R对表达式数据生成箱线图(图1),其中以中位数为中心的值表示数据已标准化且可交叉比较;使用R limma(plotDensities)生成表达密度图(图2),图中9条曲线之间趋势相对平行,使用limma(qqt) 生成t统计分位线图(图3),其中圈基本沿一条直线走行,说明limma测试结果的质量较好,该芯片质量优,检测结果稳定,可为后续分析提供可靠数据。
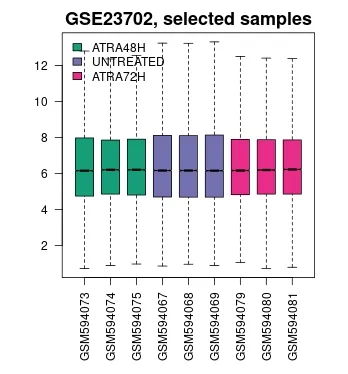
图1 箱线图
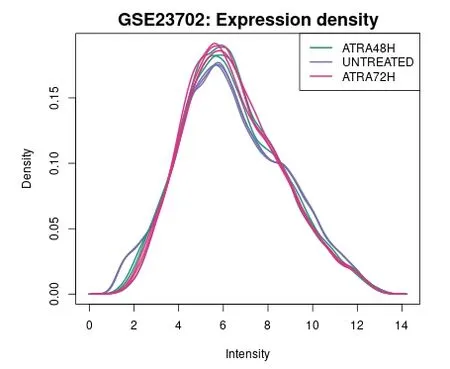
图2 表达密度图

图3 t统计分位线图
3.2 DEGs筛选
用GEO2R使用limma(vennDiagram)对3组数据:ATRA处理 前(UNTREATED)、48h(ATRA48H)、72h(ATRA72H)后 的 野 生型APL NB4细胞基因表达数据进行综合分析(设置P<0.05)并生成韦恩图(图4),可以看出ATRA处理48h后(ATRA48H)与未处理组的差异基因与处理72h(ATRA72H)后与未处理组的差异基因有相同部分,但ATRA处理48h与ATRA处理72h组无差别,因此我们分别选取ATRA48H、ATRA72H与UNTREATED组进行差异分析,用P<0.05并且l log(Fc)I>2进行筛选;与未用ATRA处理相比ATRA处理48h后筛选出上调基因140个,下调基因96个,共计236个 DEGs;ATRA处理72h后筛选出上调基因191个,下调基因131个,共计322个 DEGs,结果分别用火山图(图5和图6)进行展示,上调和下调最显著的前10个DEGs具体信息分别整理为表格(表1和表2),同时两组数据上调和下调最显著的前10个 DEGs取交集得出韦恩图(图7和图8)。
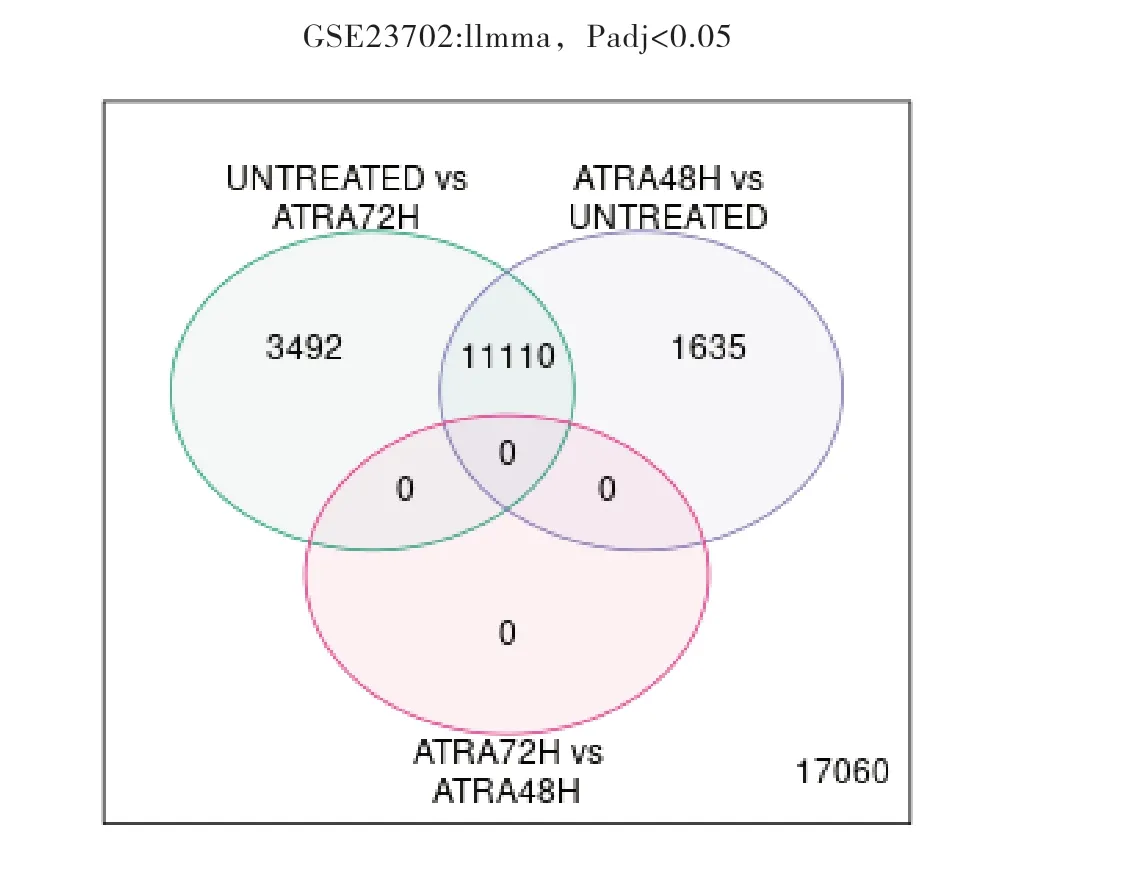
图4 ATRA处理前(UNTREATED)、48h(ATRA48H)、72h(ATRA72H)后的野生型APL NB4细胞基因表达交集数据韦恩图
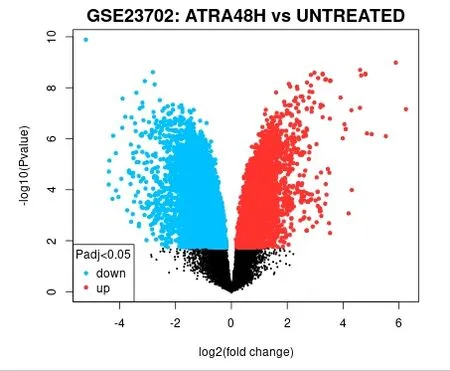
图5 与未用ATRA处理相比ATRA处理48h后基因分析火山图,黑色代表非 DEGs,红色代表上调 DEGs,蓝色代表下调 DEG

图6 与未用ATRA处理相比ATRA处理72h后基因分析火山图,黑色代表非 DEGs,红色代表上调 DEGs,蓝色代表下调 DEG
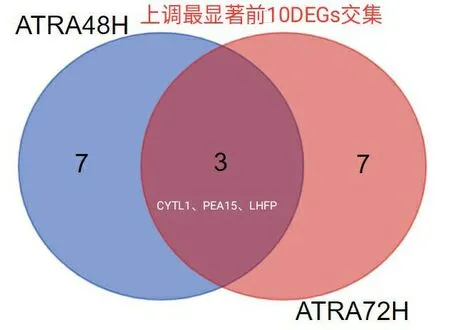
图7 ATRA分别处理48h和72h后上调最显著的前10个 DEGs取交集

图8 ATRA分别处理48h和72h后下调最显著的前10个 DEGs取交集
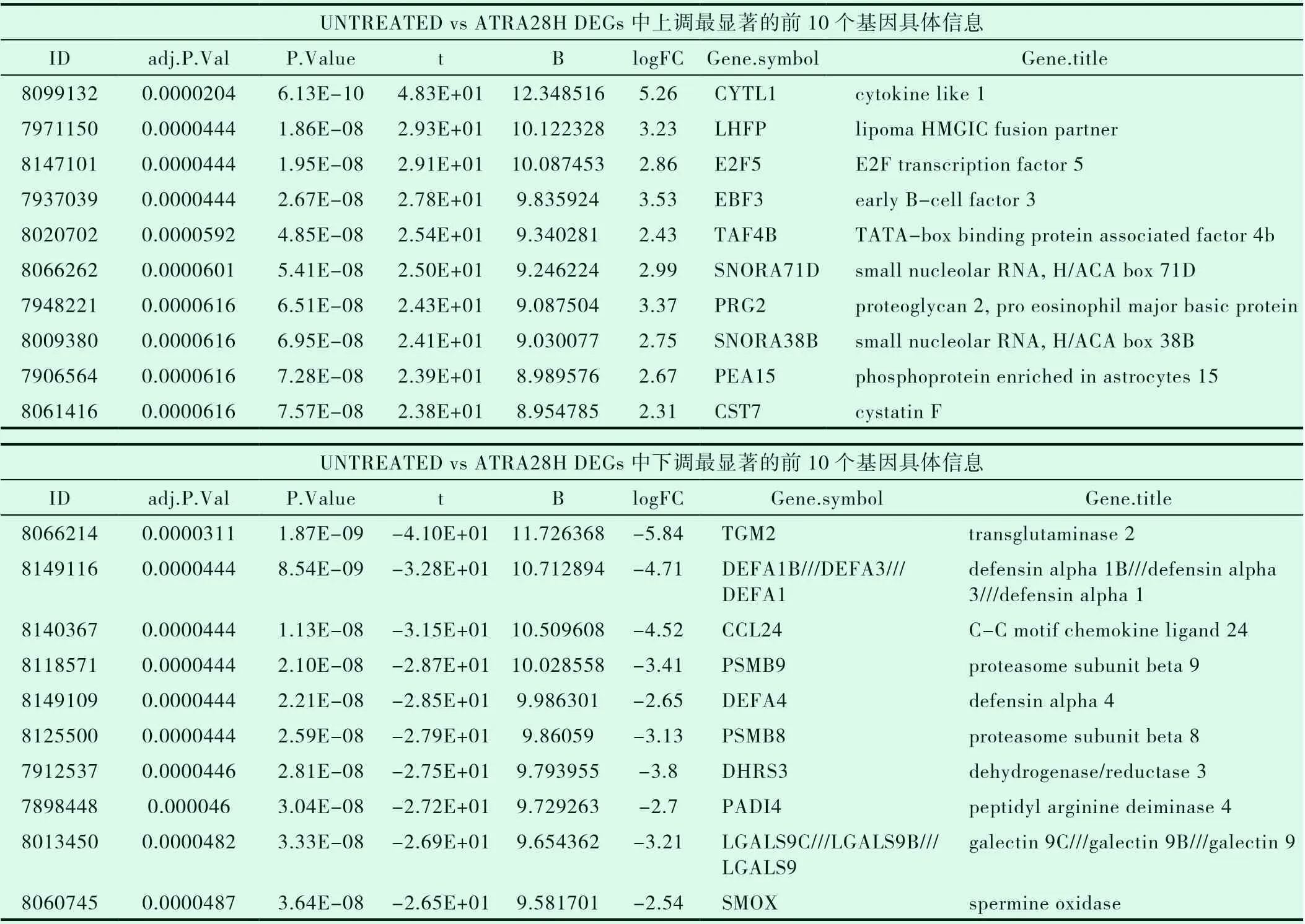
表1 UNTREATED vs ATRA48H DEGs中上、下调最显著的前10个基因具体信息
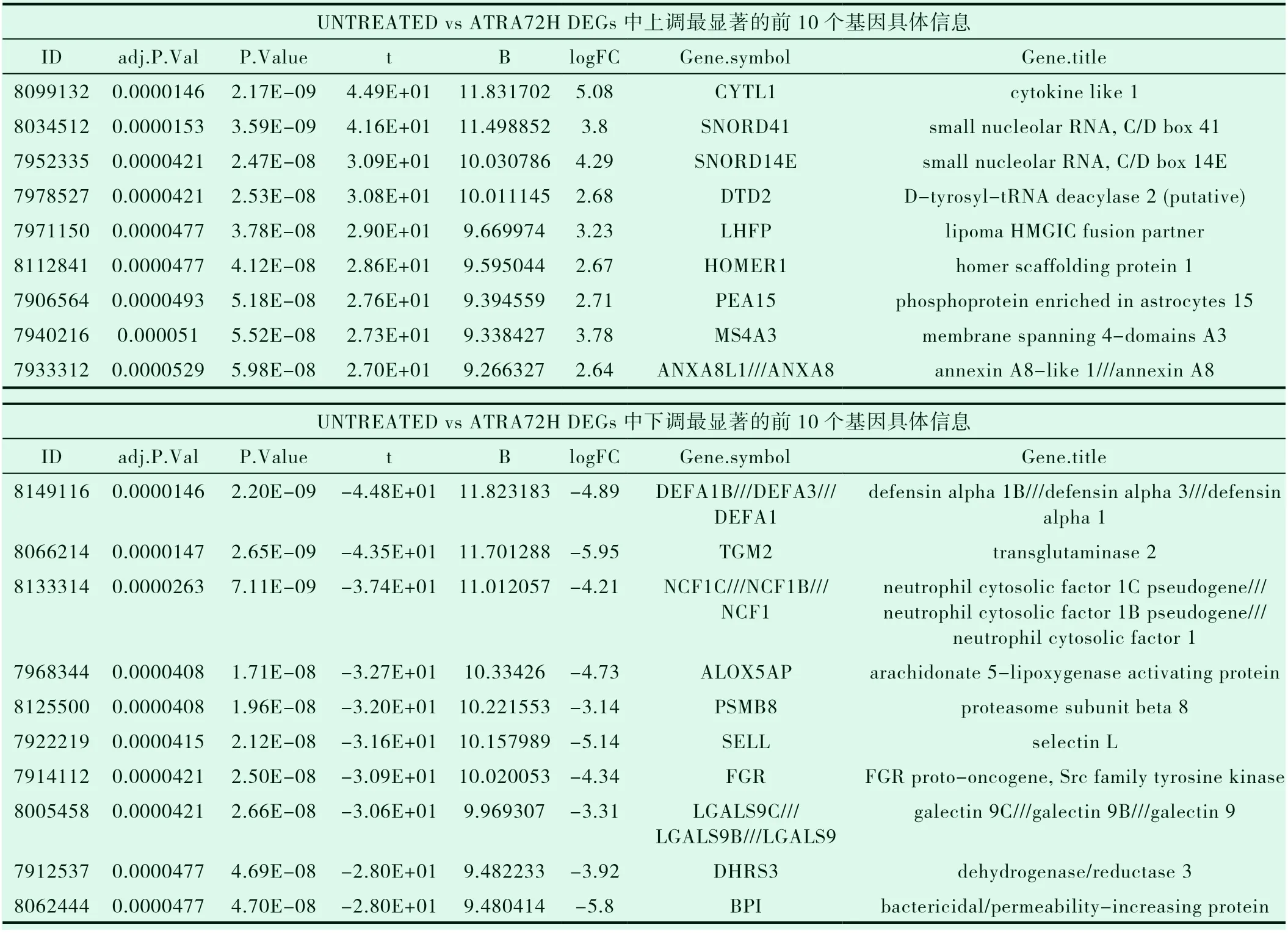
表2 UNTREATED vs ATRA72H DEGs中上、下调最显著的前10个基因具体信息
3.3 DEGs的GO 和KEGG富 集 分 析
DEGs的GO富集分析结果用柱状图(图9和10)表示如下,结果表明ATRA48H后上调DEGs在生物过程(biological process,BP)中主要与一氧化氮的生物合成过程、细胞周期调控、凋亡过程的负调控、葡萄糖进入细胞的负调控、异源代谢过程、RNA聚合酶II转录预起始复合物装配、建立平面极性、宿主共生体生长的负调控、肾脏发育等过程相关;在细胞组成(cellular component,CC)层面主要与细胞纤维网、分泌颗粒、细胞的顶端部分等结构相关;在分子功能(Molecular Function,MF)上主要与苦味受体活性、转录因子活性,RNA聚合酶Ⅱ核心启动子序列特异性结合参与预启动复合物组装、碳酸盐脱水酶活性、味觉受体活性等分子功能相关。ATRA48H后下调DEGs在BP中主要与对抗原刺激的急性炎症反应、激活先天免疫反应、B细胞增殖的负调控、白三烯代谢过程、凋亡细胞清除、干扰素-γ介导和整合素的信号通路、中性粒细胞趋化途径、细胞粘附、调节细胞形状、白细胞迁移、细胞防御反应、细胞对干扰素-β的反应途径、激活先天免疫反应、蛋白质结合的正调控、经由TAP依赖的MHC I类进行抗原加工和呈递外源肽抗原、细胞表面受体信号通路、炎症反应的正调控、血管生成的正调控、Wnt信号通路的正调控、蛋白质瓜氨酸化等过程相关;在CC层面主要与细胞外泌体、胞浆、细胞膜与质膜、蛋白酶体核心复合体、精原蛋白酶体复合物等结构相关;在MF上主要与细胞粘附分子、钙离子、整联蛋白、花生四烯酸结合,蛋白质精氨酸脱亚氨酶、苏氨酸型内肽酶活性等分子功能相关。用同样的方法分析ATRA72H后上调和下调DEGs与ATRA48H差别不大,选取主要富集通路用柱状图(图11和12)显示。结果用KEGG可视化分析(筛选条件为校正后P<0.05)结果表明,ATRA48H上调DEGs主要与流体剪切应力与动脉粥样硬化条信号及代谢通路(hsa05418)密切相关,与下调DEGs相关的通路共筛选出15条,具体信息显示在表3中,其中包括趋化因子信号通路(hsa04062)、细胞粘附分子(hsa04514)、NOD样受体信号转导通路(hsa04621)、白细胞跨内皮迁移(hsa04670)、碳水化合物消化和吸收(hsa04973)、PI3K-Akt信号通路(hsa04151)、造血细胞谱系(hsa04640)、蛋白酶体(hsa03050)等。用同样的方法分析ATRA72H后上调和下调DEGs与ATRA48H差别不大,结果显示在表4中。
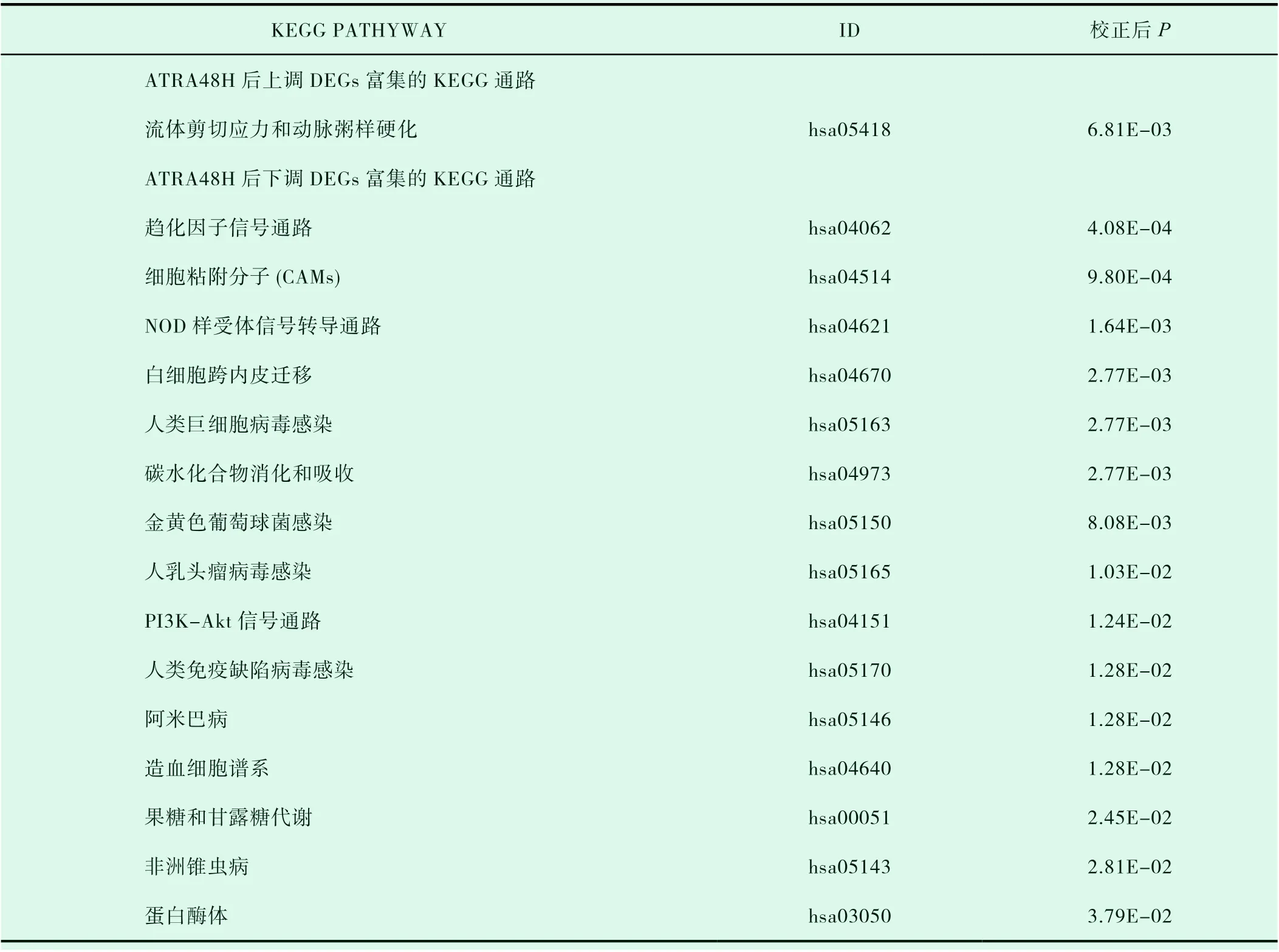
表3 UNTREATED vs ATRA48H 上、下调DEGs通过KEGG分析的主要通路
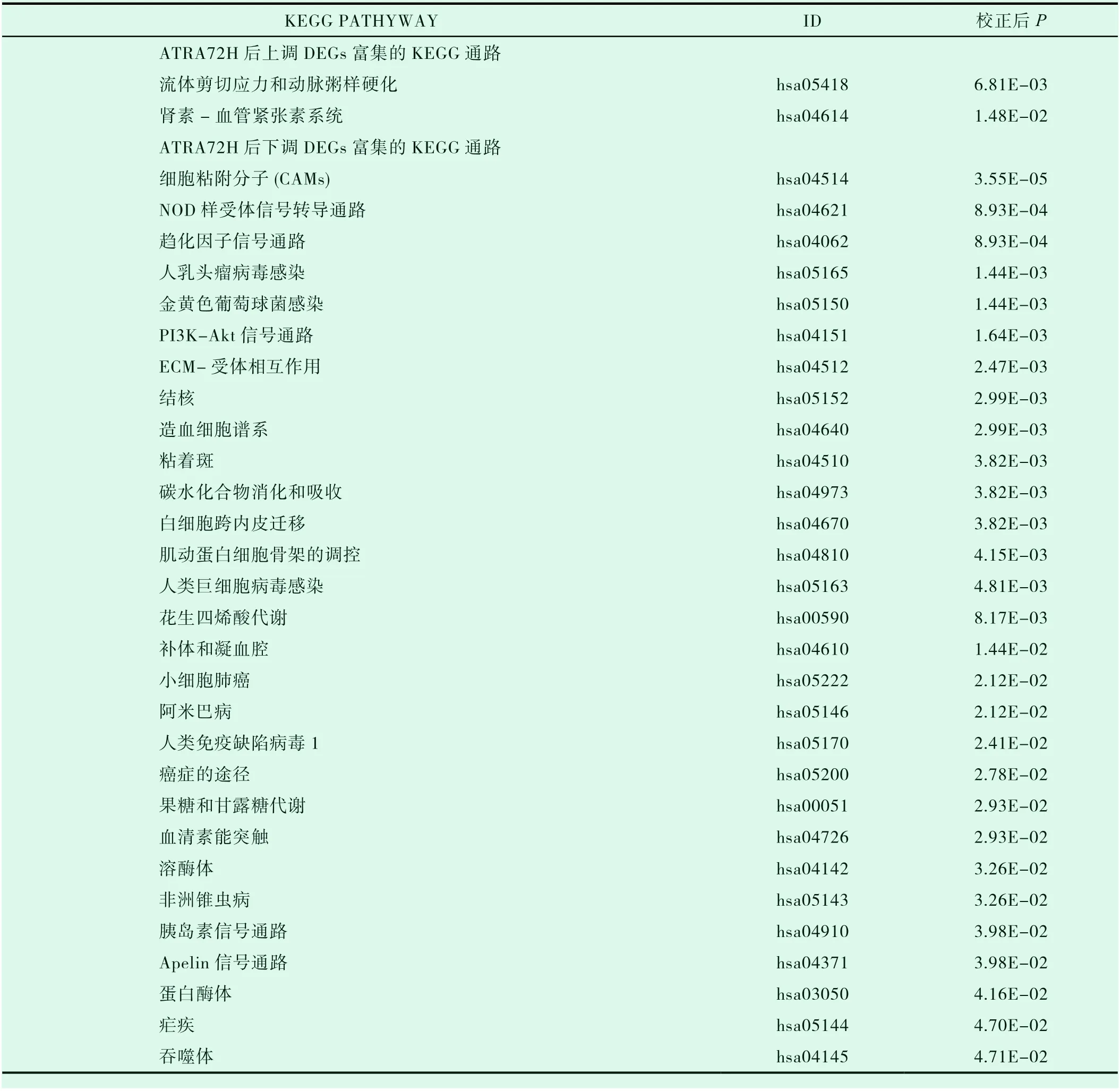
表4 UNTREATED vs ATRA72H 上、下调DEGs通过KEGG分析的主要通路

图9 UNTREATED vs ATRA48H 上调DEGs通过GO富集的主要通路
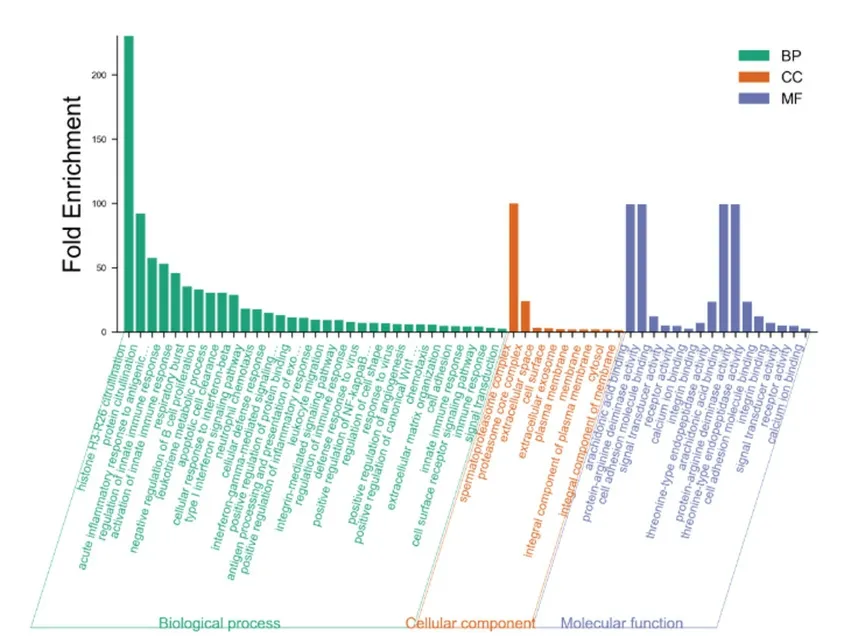
图10 UNTREATED vs ATRA48H 下调DEGs通过GO富集的主要通路

图11 UNTREATED vs ATRA72H 上调DEGs通过GO富集的主要通路
3.4 PPI网络构建及分析
通过 STRING 数据库构建 PPI网络(图13和14)以及Cytoscape软件中的CytoHubba插件进一步明确ATRA48H后上、下调DEGs所涉及的蛋白相互作用及其中参与调控的关键基因(图15和16),结果显示,上调DEGs的PPI网络中包括148个节点蛋白的145种关系,下调DEGs的PPI网络中包括115个节点蛋白的241种关系,其中多数蛋白间都存在相互作用关系,筛选出下调DEGs排名前十的关键基因有:PSMB8、CXCR2、GNG2、CXCL3、PSMB9、CXCL6、P2RY13、TAS2R10、TAS2R7、PSMA3。用同样方法得出ATRA72H后上、下调DEGs所涉及的蛋白相互作用图(图17和18)及其中参与调控的关键基因(图19和20),结果显示,上调DEGs的PPI网络中包括205个节点蛋白的261种关系,下调DEGs的PPI网络中包括166个节点蛋白的508种关系,其中多数蛋白间都存在相互作用关系,筛选出下调DEGs排名前十的关 键 基 因 有:ITGAM、ITGB2、ITGAX、IRF1、OAS1、OAS3、IFI35、LILRB2、IRF2、IFI6。
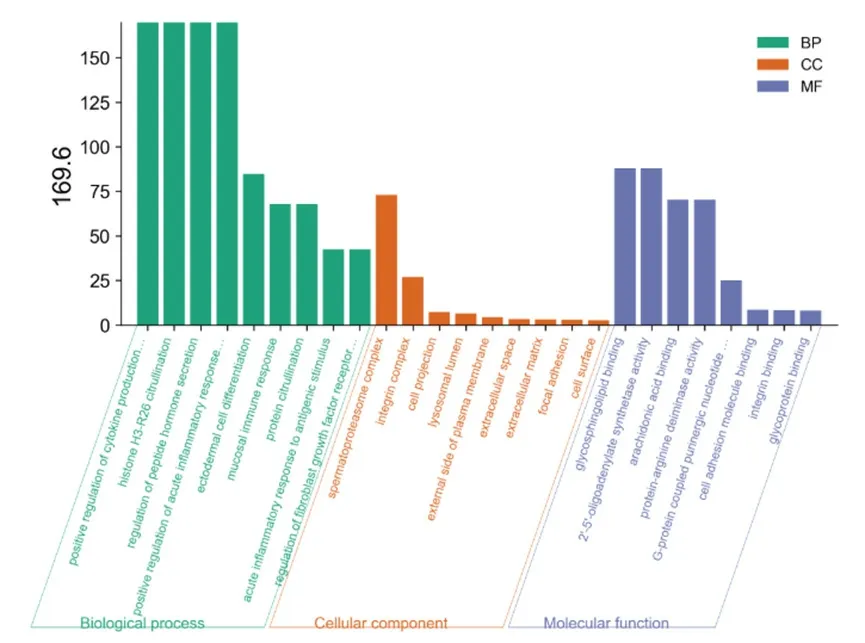
图12 UNTREATED vs ATRA72H 下调DEGs通过GO富集的主要通路
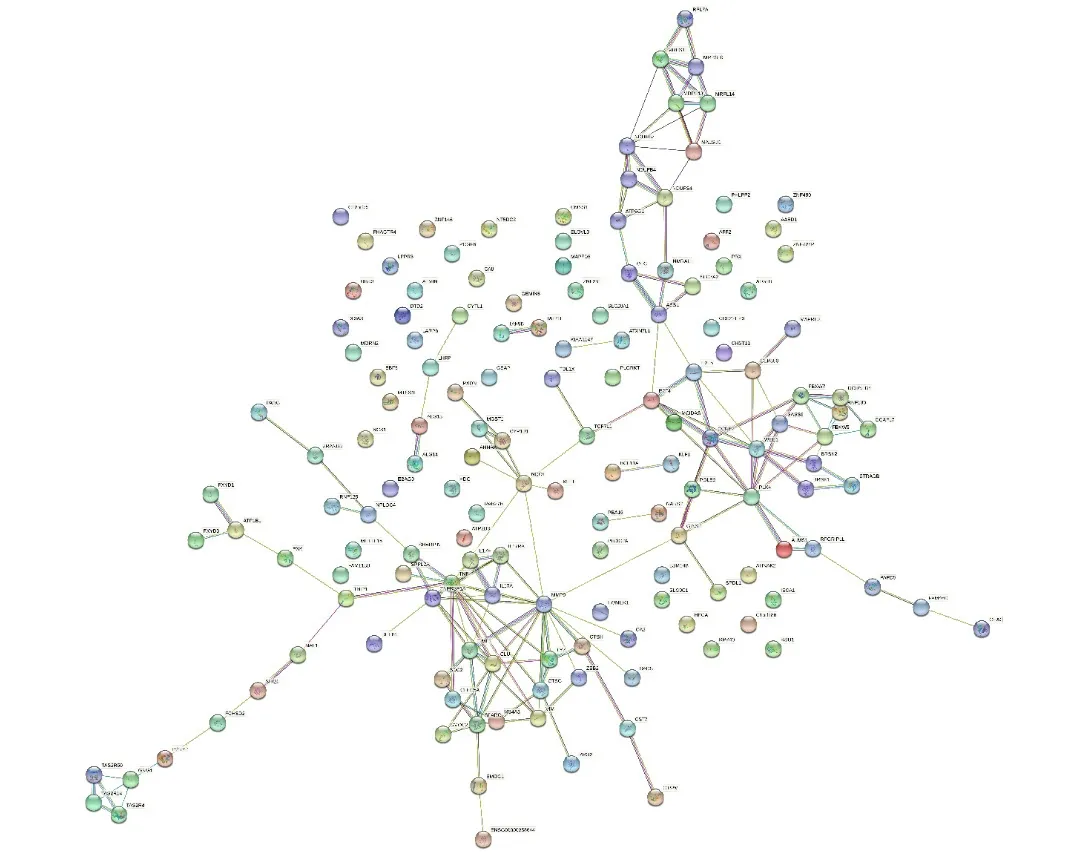
图13 UNTREATED vs ATRA48H 上调DEGs的PPI网络图
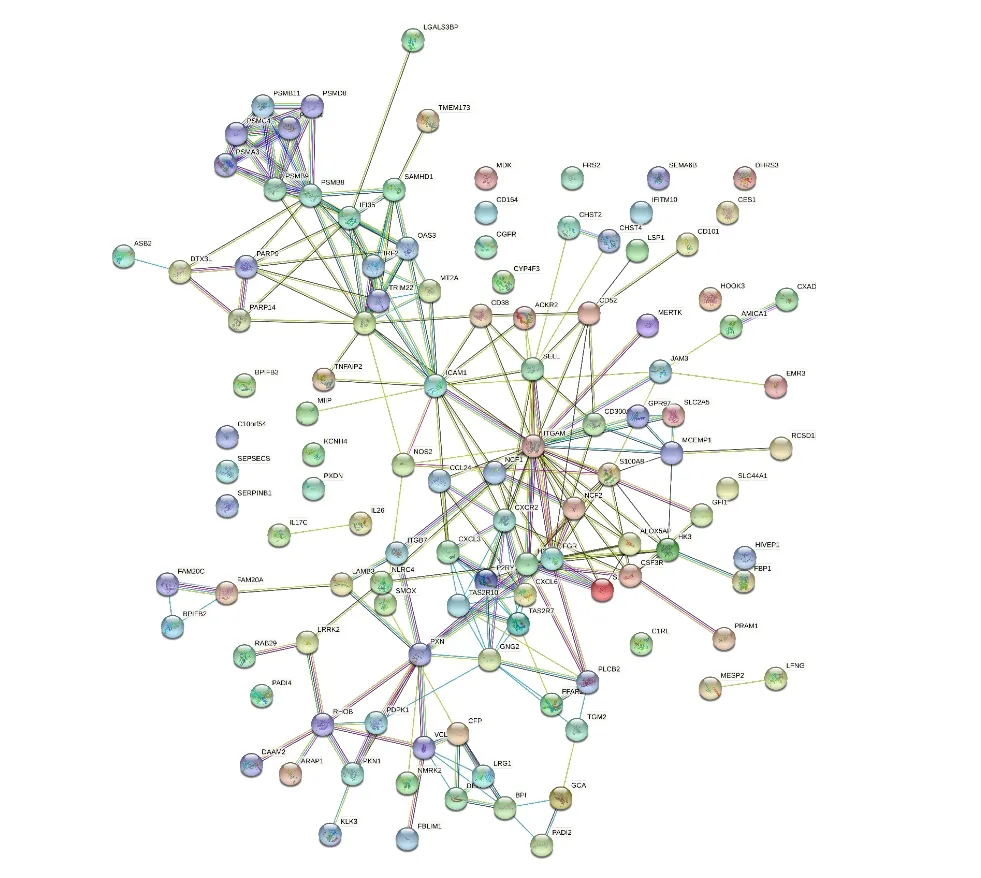
图14 UNTREATED vs ATRA48H 下调DEGs的PPI网络图

图15 UNTREATED vs ATRA48H 上调DEGs的前10个关键基因

图16 UNTREATED vs ATRA48H 下调DEGs的前10个关键基因

图17 UNTREATED vs ATRA72H 上调DEGs的PPI网络图

图18 UNTREATED vs ATRA72H 下调DEGs的PPI网络图

图19 UNTREATED vs ATRA72H 上调DEGs的前10个关键基因
4 讨论
本文分析了ATRA处理NB4细胞后的差异基因,GO分析结果表明上调DEGs主要与一氧化氮的生物合成过程、细胞周期调控、葡萄糖进入细胞的负调控、异源代谢过程、RNA聚合酶II转录预起始复合物装配、建立平面极性、宿主共生体生长的负调控;而下调的DEGs主要与炎症反应、中性粒细胞趋化途径、细胞粘附、细胞凋亡、免疫反应、蛋白质合成相关。同时在 KEGG 分析发现差异基因主要富集在趋化因子信号通路、细胞粘附分子、NOD样受体信号转导通路、白细胞跨内皮迁移、PI3K-Akt信号传导途径。另外,在DEGs形成的PPI网络进行分析,可观察到下调基因参与的通路更加具有相关性,设计的通路和关系节点更加密集,对ATRA48H和ATRA72H后下调DEGs的PPI网络中分别筛选出前10 位与ATRA处理后NB4细胞表达下调的关键基因分别绘制生存曲线(组图23),发现6个高表达提示预后不良,包括:OAS1、OAS3、PSMB9、PSMB8、ITGAM、IFI35可作为AML的危险因素,为AML的治疗提供了新靶点。

图20 UNTREATED vs ATRA72H 下调DEGs的前10个关键基因

图21 KEGG趋化因子通路,红色代表上调DEGs,蓝色代表下调DEGs,

图22 KEGG白细胞跨内皮迁移通路,红色代表上调DEGs,蓝色代表下调DEGs

图23 ATRA处理后NB4细胞表达下调的关键基因在AML中的生存曲线

