基于深度特征融合的癌症病理图像分割网络
黄鸿,王涛,李远,周凡琳,李昱
(1 重庆大学光电技术与系统教育部重点实验室,重庆400044)
(2 重庆大学附属肿瘤医院病理科,重庆400030)
0 引言
在癌症病理图像的临床诊断中,基于病理组织切片的显微分析可用于观察癌组织的发展状态从而判断癌症的变化、转移情况,因此病理图像又被称为癌症临床诊断中的金标准[1]。近年来,随着全视野数字切片技术的发展,针对病理图像的定量分析研究逐渐兴起。病理图像智能分析对癌症患者的诊断和预后具有重要作用,其中病理切片的病灶分割可用于计算癌症病变的相关量化指标、定位癌组织区域以及参与制定手术方案等[2-3]。精确的计算机辅助病理分割不仅能帮助临床医生提高诊断效率,还可以提升诊断的准确性,如何针对癌症病理图像特点设计高效的分割算法已成为医学图像分析领域的研究热点[4]。
传统的图像分割方法主要包括阈值分割法、区域生长法和图割法等。典型的阈值分割法有大津法[5],其主要原理是通过最大化类间方差来计算分割阈值,因其简单和计算量较小而被广泛应用[6]。典型的区域生长法有分水岭算法[7],其主要原理为通过将像素灰度模拟为海拔高度来模拟地理环境,谷底和峰顶的边界即为分水岭,其分割结果具有空间连续性,在分割连续小目标时效果较好,但该算法受噪声影响大且不适用于大区域分割。典型的图割法有Graphcut 法[8],其原理为先建立一张加权图,通过边权值的大小来筛选边,使划分的子图彼此尽量相互分离,从而达到分割的目的[9]。该方法具有较好的泛化性被广泛应用于医学图像分割,但其时间复杂度较高对计算资源的消耗大。上述传统图像分割方法共有的缺点是依赖于手工特征,分割中不能充分利用图像中的语义信息,导致性能受限。
深度学习方法具有特征提取能力强和便于训练优化的特点,开始应用于医用图像分割中。这些方法主要为全卷积模型,代表性方法包括全卷积网络(Fully Convolutional Networks,FCN)[10]、U-Net 网络[11]和UNet++网络[12]等。早期,CIRESAN D C 等[13]基于卷积神经网络(Convolutional Neural Networks,CNN)利用滑窗进行像素级目标分割,但存在计算量大和效率低的问题。LONG J 等提出了FCN 网络作为替代方法,将经典卷积神经网络的全连接层替代为上采样卷积层,从而建立了一个只包含卷积层的网络,取消了输入尺寸固定的限制,可灵活应用于不同大小的图像上。在FCN 网络的基础上,U-Net 网络进一步发展了编码、解码的思想,其上采样和下采样结构呈U 形对称,其解码层增加了相应的上采样层和通过跳跃连接进行特征融合使提取特征更加有效,提高了分割精度。ZHOU Z 等在U-Net 网络基础上提出了U-Net++网络,通过将长连接改用密集连接、在训练时自动剪枝的方式,使网络可以自适应学习合适的下采样层数以获得最优的对应U-Net 网络深度,在提升分割性能的同时没有明显增大计算资源的消耗。黄鸿等[14]基于融合自适应加权聚合策略提出了iU-Net++方法,该方法可以自适应学习各层特征权重,使包含不同信息的特征根据贡献程度聚合起来得到分割结果,在浅层特征融合中突出了关键特征,提升了分割性能。此外,还有学者将病理图像研究拓展到了高光谱病理图像的研究中,在利用空间信息的基础上增加了更丰富的病理组织光谱信息[15]。郑少佳等[16]基于傅里叶变换多频率通道注意力机制结合Inception 网络构建了一种胆管癌高光谱图像分割网络,在一定程度上解决了数据不平衡问题并在胆管癌上取得了较高的分割精度。WEI X等[17]设计了一个端到端网络,通过在原始光谱数据和其主成分之间建立逐像素映射实现了无监督学习,有效解决了监督卷积神经网络模型训练样本有限的问题。
上述方法针对病理图像通过融合不同深度U-Net 网络特征,在CT、MRI 等医学图像上取得了不错的分割效果,但直接在浅层进行预测,在一定程度上削弱了高层语义特征的指导作用,导致没有充分利用高层语义特征在精确定位病灶区域上的重要作用,尤其是病理图像存在切片染色多样、分辨率差异大等问题,限制了病理图像分割的性能。
针对上述问题,本文提出了一种改进的层级特征融合分割方法(Hierarchical Feature-fused U-Net,HUNet)。该方法基于U-Net 网络,利用EfficientNet-B4 网络作为特征编码器,在解码器部分改进了特征融合方法,利用改进的通道注意力模块对融合特征进行筛选,训练时不同深度预测下对应的损失函数都进行反向传播,获得了更有效的深层语义特征,从而提升了模型获取和利用全局语义特征的能力,改善了癌症病理图像病灶分割精度。
1 本文方法
1.1 整体网络结构
U-Net 是一种具有对称性的分割网络,它通过添加长连接的方式在每一层中都进行高层全局特征和局部细节特征的特征融合,有效利用了不同重要性特征进行分割预测[18]。因此采用U-Net 网络作为分割模型的基本网络结构,其整体网络结构如图1。

图1 HU-Net 网络整体结构Fig.1 The overall structure of the proposed HU-Net algorithm
该网络主要包括编码器、解码器和特征融合预测三个部分。图1(a)为基于EfficientNet-B4 网络的特征编码器。图1(b)为解码器,在解码器中改进了U-Net 原有特征融合方式,将不同深度特征重新进行了融合,从深层逐渐到浅层,每一层融合特征都不断上采样到最浅层,每层特征融合时都融合之前深层中的高层语义信息。图1(c)为特征融合预测模块,训练时将不同深度的层融合特征都进行预测输出,都计算损失函数并反向传播。同时,在网络解码器中应用了通道注意力模块(CA1-CA4)对融合后的特征进行通道特征选择,如图1(b)中紫色部分。
1.2 编码器模块
EfficientNets 是Google 利用神经网络架构搜索获得的一系列网络模型,分别为EfficientNet-B0~B7[19]。通过考虑运算量与计算资源,本文网络选择在Imagenet 数据集上预训练的EfficientNet-B4 网络作为特征编码器,通过迁移学习使编码器在病理图像上更快学习到关键特征,在实际使用时去掉了全连接层,具体结构如图2。该网络主要由32 个移动翻转瓶颈卷积模块(Mobile Inverted Bottleneck Convolution,MBCONV)和2 个卷积层构成,各个MBCONV 块中卷积核的大小分别如图2所示。通过将步长设为2,使输出特征图在长和宽两个方向上分别缩小为原来的一半,实现了压缩特征图以进行特征编码,将EfiicientNet-B4 中不同下采样特征通过图1 中的跳跃连接和解码器特征进行融合从而代替了原U-Net 中的编码器进行特征编码。同时,EfficientNet-B4 网络还引入了通道注意力思想,将特征图在特征通道上进行压缩和激发,使得在编码过程中进一步突出了关键特征。
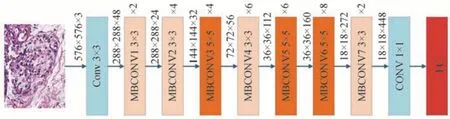
图2 EfficientNet-B4 整体结构Fig.2 The structure of EfficientNet-B4
1.3 解码器模块
U-Net 网络只使用最浅层融合特征进行预测,U-Net++通过增加短连接和剪枝的方式实现了自动学习U-Net 网络的合适层数,丰富了模型表示复杂特征的能力,但这种特征聚合方式并不能充分利用深层特征的全局信息以适用于癌症理图像分割。针对病理图像内容复杂和相似性高的特点,需要进一步增加全局特征的预测作用以增强模型对病灶的区分能力。因此,在保留跳跃连接的基础上设计了如图1所示的解码器,对每一层要上采样的特征都分别进行若干次上采样,直至最浅层。
在每一层融合时,除了融合上一解码层和对应编码层的输出,还包括之前解码层上采样到本层的特征,融合方式均为按特征通道进行叠加。设从网络最深层到最浅层分别为0,1,2,…,当前层为第i层,Conc(·)表示特征叠加融合操作,则每一层中特征融合表示为

通过式(1)中的融合方式,HU-Net 网络增强了深层特征在模型预测中的重要性,最深层的语义特征在浅层进行预测时依然发挥作用,但融合特征数量的增加带来了融合后特征通道数量的增加。为了确定不同通道特征的重要性并筛选有效特征,使用了通道注意力模块对融合特征进行选择。为了使注意力模块更加有效以及方便使用,降低对网络结构的依赖,对通道注意力模块的使用采用如图3所示流程。输入特征在经过通道注意力模块校正后首先使用卷积操作将特征通道压缩为原来的一半,接着对特征进行批归一化和激活,最后将通道压缩的特征进行叠加。这样可以进一步突出经通道注意力校正得到的关键特征,同时使注意力模块在网络各个地方可以灵活使用。具体的融合方式如图1(b)。
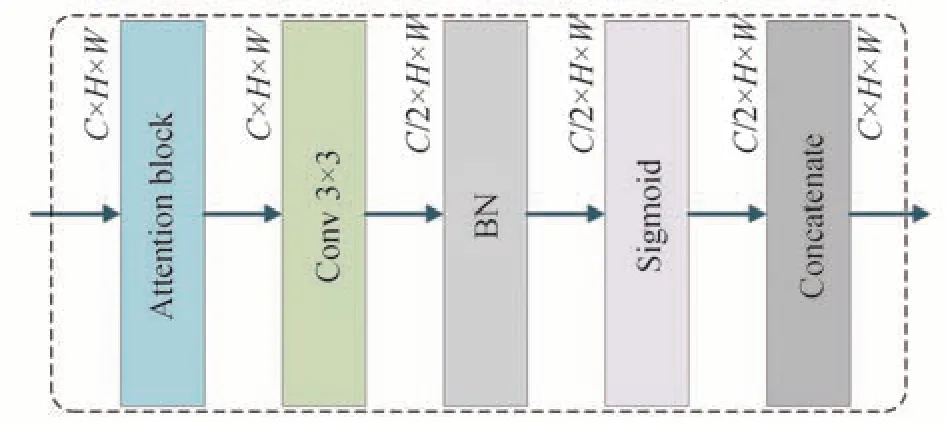
图3 注意力模块的使用方法Fig.3 The method of using attention block
1.4 层级融合模块
为了加速模型训练收敛以及更有效地进行模型微调,同时使网络在训练过程中在不同深度都获得更有效特征,设计了如图1(c)所示的特征融合预测模块。在解码器的不同层,都将该层融合筛选后的特征通过转置卷积直接上采样到图像原尺寸,并利用Sigmoid 函数进行激活,输出之后得到每层对应的分割预测结果。然后,将不同层的预测进行叠加融合,再经过卷积并激活后得到融合的预测结果作为模型最终预测。训练时,模型对最终的融合预测结果和各层单独的预测结果都进行输出,分别依次计算损失函数和进行误差反向传播。在模型验证和测试时,只采用融合预测结果作为模型输出。通过这种方式,可以直接在深层和浅层同时得到更有效的特征,通过观察不同层的预测结果可以分析不同层的训练学习情况以及评估模型的特征表达能力,增强了模型的可解释性。
上述融合和训练方式,使网络在深层中将更加关注病灶位置,可以获得更有效的高层语义信息进行病灶区域定位,利用浅层特征进行边界细节预测,使模型整体分割预测更加精确。
1.5 通道注意力模块

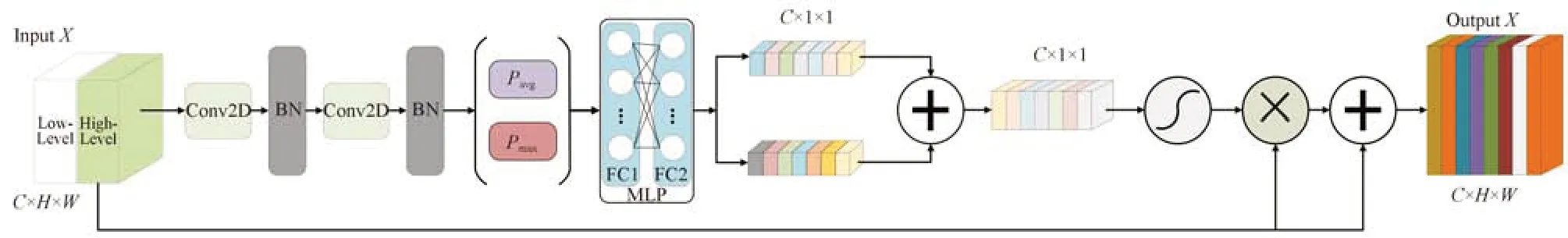
图4 通道注意力模块结构Fig.4 The structure of attention block
进行两次卷积和批归一化操作后,需利用全局池化学习全局参数。目前的压缩激励注意力网络(Squeeze-and-Excitation Networks,SENet)[20]只利用了全局平均池化(Global Average Pooling,GAP)来提取对应通道权重特征,但容易导致细节信息丢失。因此,本文增加了通过全局最大池化(Global Max Pooling,GMP)来提取通道特征,通过保留更多信息从而增强注意力模块的学习和表示能力。

2 实验结果及分析
在整个实验中,实验的硬件环境为PANYAO 7048GR,CPU 为正式版E5-2096 2.3G-3.6G 45M Cache 22NM,内存为256 G,显卡为NVIDIA GeForce GTXTITAN RTX,应用的深度学习框架为基于Ubuntu 系统的Tensorflow2.0 框架。CUDA 以及CUDNN的版本为CUDA10.1.243 和CUDNN7.6.5,采用的编程语言为Python3.8。
2.1 实验数据集
本文使用了两个癌症病理数据集来评估本文算法模型,第一个为2020年“华录杯”江苏大数据开发与应用大赛(“SEED”大赛)—医疗卫生赛道癌症病理数据集[21],第二个数据集为2017年中国大数据人工智能创新创业大赛(Brain Of Things,BOT)—病理切片识别AI 挑战赛中的病理数据集[22]。上述数据集均为胃部癌变组织,为正常、癌变组织分割问题。
1)SEED 病理数据集
该数据集在江苏省人民医院中收集,共包含732 个带胃癌病灶精标注的数字病理切片样本,其中有17个样本病理图像为空白无法使用,1 个样本出现错误标注,在使用中均被剔除,实验中实际使用样本数为714。病理图像以及对应的标注都为RGB 图像。该数据集中图像尺度差异大,如图5(c)所示。不同图像的尺度从372×489 到12 047×18 257 变化,尺度上的巨大变化为病灶精确分割带来了挑战。
2)BOT 病理数据集
BOT 数据集包含560 个样例,每个样本包含病理图像和对应的病灶区域标注,图像大小全部为2 048×2 048。该数据集的病理图像中大部分病灶区域都相对较小,是分割中的难点问题。病理图像和对应专家标注的掩膜如图5(d)~(g)。标注掩膜图像中,白色区域表示癌组织对应区域,黑色区域表示正常组织。


图5 SEED 和BOT 数据集Fig.5 The SEED and BOT data sets used in the experiment
2.2 实验设置
为了弥补训练数据相对不足,实验中对训练集数据进行了数据增广,增广比例设置为1.2 倍,以减小增广数据可能引入的额外干扰。具体增广方式包括旋转、镜像、翻转操作,其中旋转操作固定为30°、60°的顺时针和逆时针旋转,镜像操作为水平镜像,翻转操作为上下翻转。每个训练样本的增广概率为20%,对每次数据增广只随机选取一种变换方式处理图像。每种增广方式下获得的图像数量,其比例相近且在增广数据中均匀分布。
出于尽量保留训练图像细节和减少模型计算量,训练HU-Net 网络时两个数据集中图像均缩放为576×576 大小,训练采用随机梯度下降(Stochastic Gradient Descent,SGD)作为模型优化器。实验采用DICE 系数差异函数作为训练损失函数,训练时利用模型在不同深度下对应的预测结果共同进行训练。预测时,所有预测结果均以0.5 为概率阈值对模型输出进行二值化后得到。训练初始学习率设置为0.01,训练epoch 总数设置为150。随着训练模型逐步拟合,当验证精度超过10 个epoch 没有增加时,则减小学习率,每次学习率减小0.005,最小学习率为0.000 1,当经过连续20 个epoch 验证精度没有提高时结束训练,批次大小(batch size)为5。本实验所有参数为在多次实验条件下确定的最优值。
共选取了DICE 系数、交叉比系数(Intersection Over Union,IOU)、敏感度(Sensitivity,Sen)、准确率(Mean Accuracy,MA)和精确率(Precision,Pre)五个评价指标进行定量评价,具体表示为

式中,Ma表示预测矩阵,Pre表示标签矩阵。TP、FP、TN、FN 分别表示为真阳性、假阳性、真阴性、假阴性的对应像素预测结果,其中最终的预测矩阵为将模型预测结果以0.5 为阈值进行二值化操作后得出。五个评价指标分别从多个角度定量地度量了预测结果的分割效果,使评价结果较为客观和全面。
2.3 BOT 数据集实验结果
2.3.1 HU-Net 网络和经典网络对比
为验证HU-Net 网络有效性,选取了FCN-VGG16[10]、U-Net[11]、SegNet[23]、DeepLabv3+[24]、CA-Net[25]和TransUnet[26]进行对比。训练时,为了减少参数对于各方法分割性能的影响,各个模型的输入大小都设置为576×576 大小,批处理大小(Batch size)设置为5,且采用和HU-Net 训练相同的数据增广方式。初始学习率和学习率设置如下:最小学习率都调整为0.000 1,初始学习率分别设置为0.01、0.005、0.005、0.001、0.005、0.005,每超过10 个epoch 验证精度未提高时触发学习率变化,每次变化学习率下降为原来的0.8 倍,学习率的设置为多次实验取得的最优值。所有算法的实验结果都为五折交叉验证结果的平均值。
实验前,按照4∶1 的比例划分训练集和测试集,不同算法在BOT 数据集上的分割结果如表1。
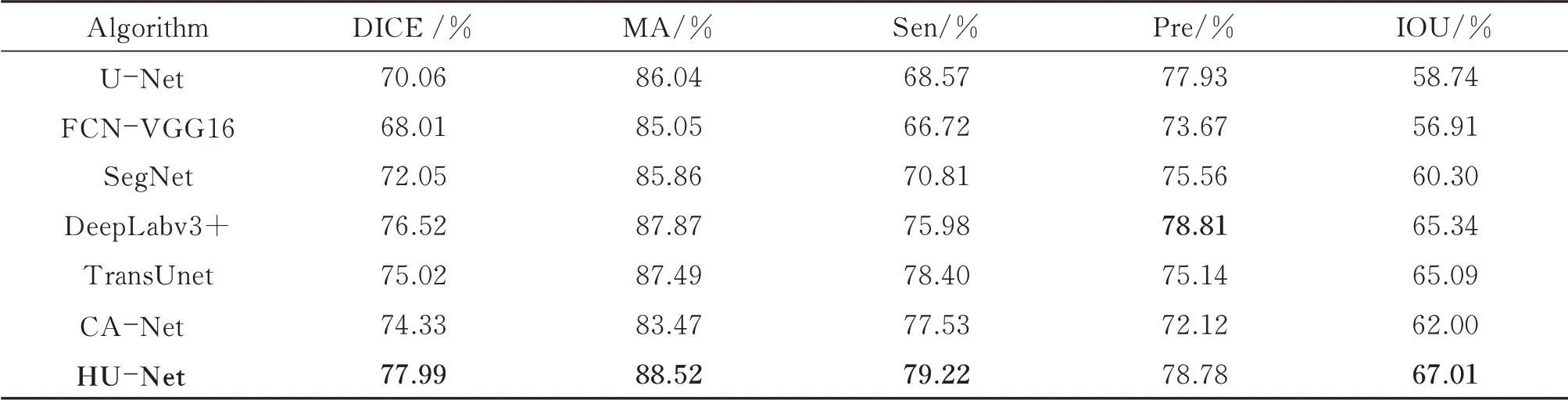
表1 不同算法在BOT 数据集上的结果Table 1 The result of different algorithms on the BOT dataset
由表1 可得知,FCN 网络取得了较差的分割精度,这是因为BOT 病理图像中的病灶区域普遍较小,FCN 网络在进行预测时只利用了最深层语义信息,导致细节信息利用不足,而U-Net 网络因为融合了细节信息因此取得了更高的预测分割精度。TransUnet、CA-Net 和SegNet 在保留细节信息的基础上分别采用了更有效的特征提取器、利用注意力模块进行特征筛选和改进上采样的方式,进一步改善了病灶分割效果,但这些方法共同的缺点是没有突出全局特征在病理图像分割中的作用。本文提出的HU-Net 算法改进了特征融合方式,突出了深层语义信息在预测中的作用,因此在BOT 数据集上取得了最好的分割效果。DeepLabv3+相较于FCN 网络减少了下采样深度,调节了深层特征在预测中作用,在精确度上取得了不错效果,但是减少下采样会导致计算量显著增加。
为了直观对比不同方法的预测,将标注图像(GT)同所有方法在测试集上的预测结果分别进行叠加显示,其中标签图像用红色表示,模型预测结果使用绿色进行表示,预测结果和标注部分重合区域表示为黄色,结果如图6。

图6 不同算法在BOT 数据集上的分割结果Fig.6 Segmentation masks of different algorithms on BOT dataset
从图6 可得知,FCN 因为没有利用细节信息所以出现了严重的过分割,U-Net 和SegNet 网络因为过于强化细节信息在预测中的作用,导致弱化了模型定位病灶的能力,出现欠分割现象。而CA-Net 和TansUnet 等通过筛选有效特征和增强特征提取能力,在一定程度上改善了分割效果。本文提出的HU-Net算法突出了全局特征同时保留了细节信息,使分割结果更加精准。
2.3.2 HU-Net 算法消融实验
在此基础上,进行了消融实验,具体设置为:1)在U-Net 的基础上使用在ImageNet 数据集上预训练的EfficientNet-B4 网络作为特征提取器,实验中称为EU-Net。2)在EU-Net 的基础上使用多深度特征融合策略并使用多输出进行多损失函数共同训练,实验中命名为FU-Net。3)在EU-Net 的基础上增加使用注意力模块,注意力模块的使用位置同样在各解码层对应的特征融合位置,实验中命名为AU-Net。4)本文所提方法HU-Net 网络。消融实验中的模型训练设置与HU-Net 训练相同,取五折交叉验证实验结果均值作为各模型得分,实验结果如表2。
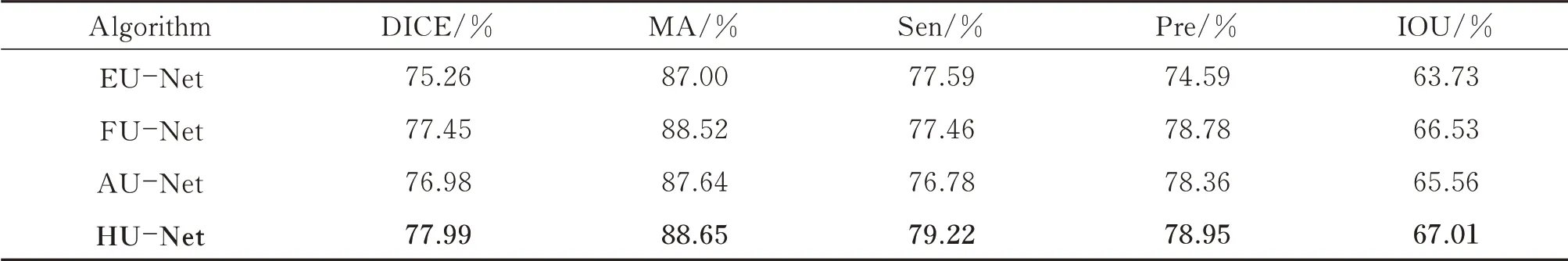
表2 不同模型在BOT 数据集上的结果Table 2 The results of different models on the BOT dataset
从表2 可看出,EU-Net 在BOT 数据集上提升了分割精度,说明更换有效的编码器可以获得更具鉴别力的特征。同时,添加注意力模块(AU-Net)和深度特征融合(FU-Net)两种改进方式相较于EU-Net 又在一定程度上提升了病灶的预测分割精度,FU-Net 得分更高说明深层信息对于预测而言有着重要作用,而HU-Net 模型融合了两种方式的优点获得了最高DICE 系数。
2.4 SEED 数据集实验结果
2.4.1 HU-Net 网络和经典网络对比
为了测试HU-Net 模型在不同数据集上的泛化性,在SEED 数据集上进行了对比实验,实验设置同2.3.1 节,实验结果如表3。
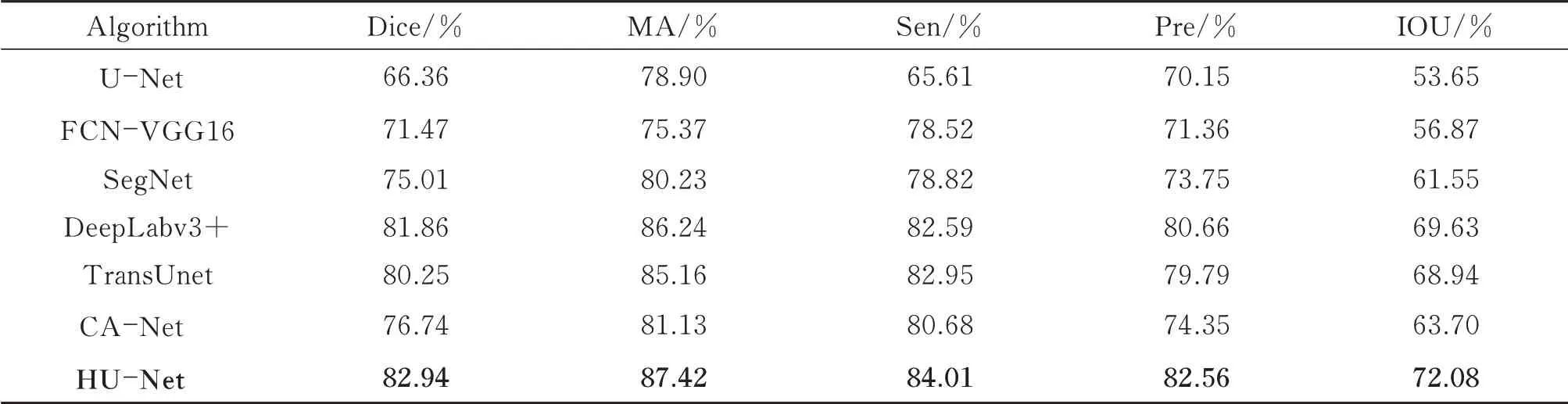
表3 不同方法在SEED 数据集上的结果Table 3 The result of different algorithms on the SEED dataset
在表3 中,FCN 比U-Net 获得了更好的分割效果,主要是因为SEED 病理图像中病灶区域较大,因此突出全局特征的FCN 分割精度更高。SegNet、TransUnet 和CA-Net 分别通过改进上采样方式、替换编码器和利用注意力筛选特征,在不同程度上提升了分割精度。不同于FCN 网络进行了多倍特征下采样和U-Net直接利用浅层特征预测,DeepLabv3+减少了下采样倍数并在深度层中进行预测,调节了深层特征在预测中的作用,提升了分割精度。本文提出的HU-Net 直接在融合结构上突出了深层特征,并利用注意力模块进行筛选,因此在不同评价指标下均取得了最高得分,相较于U-Net 网络DICE 系数提升了16.58%。
各算法在测试集上的预测结果如图7。在图7 中,FCN 能大致分割出病灶区域,但由于没有利用细节特征进行预测,因此在病灶边缘的分割误差较大。U-Net 网络因为弱化了全局特征在预测中的作用,定位病灶区域能力受限,出现欠分割现象。相较于对比算法,HU-Net 取得了最好的分割效果。

图7 不同算法在SEED 数据集上的分割结果Fig.7 Segmentation masks of different algorithms on SEED dataset
2.4.2 算法消融实验
在SEED 数据集上消融实验设置与2.3.2 节一致,实验结果如表4。

表4 不同模型在SEED 数据集上的结果Table 4 The results of different models on the SEED dataset
从表4 可以看出,AU-Net 和FU-Net 网络相较于EU-Net 在DICE 系数上有明显提升,说明采用通道注意力模块和改进的深层特征融合方式都能提高病理图像的病灶分割性能。HU-Net 网络在病灶分割上取得了最高的分割精度,验证了该方法对于提升模型分割性能的有效性。结合表2 中的实验结果,可以发现深层特征融合方式的改进对模型的分割性能提升尤为明显,这说明在癌症病理图像分割任务中,深层语义特征相较于细节特征具有更重要作用。
对实验进行综合对比分析:1)观察图5、7 中所示图像可以看出实验用BOT、SEED 数据集具有明显染色差异,但对比分析表2、4 可以发现在添加注意力模块之后AU-Net 相较于EU-Net 分别提升了1.72%和0.81%,说明本文注意力模块在不同深度层中筛选得到了更有效的通道特征,明显增强了网络应对图像颜色变化的能力。2)FU-Net 相较于EU-Net 在两个数据集上分别提升了2.19%和2.81%,说明改进特征融合方式和增加不同层的反向传播训练能更好地适应病理图像分割需要,而FU-Net 在SEED 数据集上取得了更高分割精度的提升,进一步说明这种改进方法在应对尺度变化的图像上表现出了明显作用。3)同时,分析表1、4 可以看出HU-Net 相较经典网络以及改进对比网络在两个数据集上均取得了最优效果,说明了本文所提方法较好解决了图像染色差异和图像尺度变化的问题。
3 结论
本文提出了一种改进的病理图像语义分割算法HU-Net。该算法首先增强了编码器提取特征的能力,然后利用自然图像到病理图像的迁移学习来缓解病理图像相对不足的问题,在解码器中通过改进深层和浅层特征的融合方式增强了全局特征在预测中的作用,并采用注意力模块筛选了重要特征,最后通过网络多输出训练在各个层中获得了更具鉴别力的特征。在BOT 和SEED 病理图像数据集上的结果表明,本文所提方法获得了更好的分割效果,其DICE 分割精度分别可达77.99%和82.94%,从而为医生临床诊断提供辅助支持。
病理图像往往具有大小不一、尺度变化大的特点,本文所提方法直接将所有图像统一变换为了576×576 大小以方便训练,可能会导致病理图像细节信息丢失,因此下一步研究工作将关注如何更有效利用图像中的细节信息进行网络模型训练,改善分割性能。