普通烟草NAC转录因子家族抗逆基因筛选与表达分析
余世洲,杨志晓,余婧,宋时浩,张孝廉,赵会纳,张长云,林英超,雷波*
普通烟草NAC转录因子家族抗逆基因筛选与表达分析
余世洲1,杨志晓1,余婧1,宋时浩2,张孝廉1,赵会纳1,张长云1,林英超1,雷波1*
1贵州省烟草科学研究院,烟草行业分子遗传重点实验室,贵阳市观山湖区龙滩坝路29号 550081;2青岛颐中科技有限公司,青岛市市北区沾化路3号甲 266011
【】NAC(NAM/ATAF/CUS2)家族是植物特有且最大的转录因子家族之一,广泛参与植物的生长发育调控和逆境胁迫应答。为筛选烟草抗逆目标基因。以公开的普通烟草品种K326的注释蛋白序列为参考,与拟南芥NAC蛋白序列同源比对,结合功能域分析,进行普通烟草的NAC转录因子鉴定,并利用生物信息学软件分析各烟草NAC转录因子的理化性质及系统进化关系。使用普通烟草TobEA表达谱数据对烟草NAC家族成员组织表达模式进行分析,通过qRT-PCR检测候选抗逆基因所在亚组AtNAC3和ATAF各成员在干旱、冷害、盐、ABA处理下的基因表达情况。从普通烟草中共鉴定出190个NtabNAC蛋白,平均氨基酸长度为347,除NtabNAC190外,其余蛋白为亲水性蛋白,大多数定位在细胞核。根据系统进化分析,将拟南芥和烟草NAC家族基因划分为18个亚组,各亚组基因在不同组织中显示表达特异性。AtNAC3和ATAF亚组的9个烟草基因,在受到干旱、冷害、盐和ABA处理后基因表达量上调,且、和表达量变化最为明显。、和参与了烟草植株的逆境胁迫调控,研究结果有助于烟草抗逆调控网络解析和后续抗逆育种。
普通烟草;NAC基因家族;转录因子;系统发育;表达模式;非生物胁迫
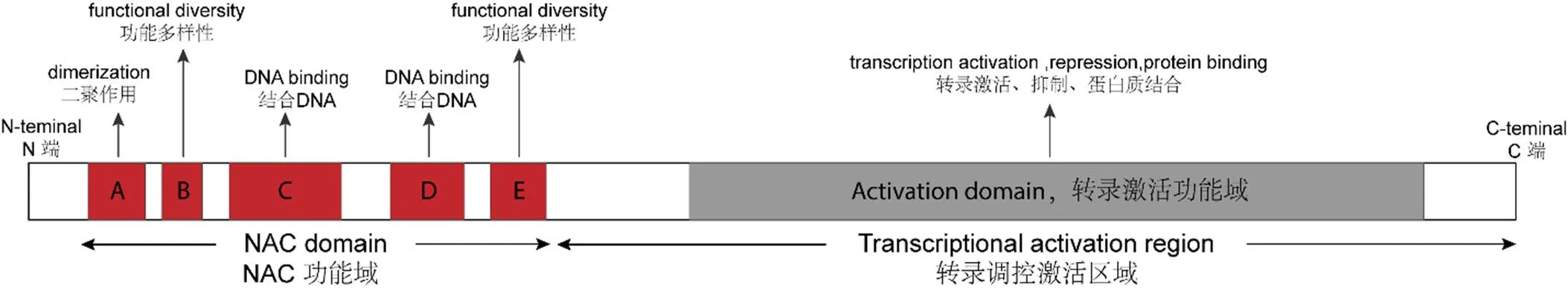
图1 NAC转录因子的典型结构
Ooka等[8]依据基因序列结构和进化关系,将105个拟南芥NAC转录因子和75个水稻NAC转录因子划分为两大类18个亚组。其中NAC1和NAM亚组中的基因多参与植株形态发育过程,如叶片衰老、种子和胚胎发育、细胞分裂、茎尖分生组织形成、纤维发育、花的形态发生及侧根发育等[10-11]。AtNAC3和ATAF亚组基因则多与逆境胁迫应答相关,NAP、NAM和OsNAC3亚组中有少数基因也具有这一功能[8-12]。研究发现在拟南芥植株中过表达AtNAC3亚组中的、和基因,植株的耐旱性显著提高[13]。在水稻中过表达基因可以促进叶片气孔关闭,减少水分损失,从而增强过表达植株的耐旱性和耐盐性[14];在拟南芥中过表达玉米NAC转录因子可提高植株的耐旱性[15]。在小麦中过表达基因或基因能促进根的生长,提高水分利用效率,从而增强植株的耐旱能力[16-17]。
普通烟草(L.)的形态发育对环境因素较为敏感,近年来国内多个烟区受到干旱、低温等不良环境的影响[18]。已有研究报道了[19][20]、[21]等基因的抗旱功能,但这些研究都是从单个基因出发,并未从基因组层面进行系统分析。基于此,本研究利用2017年公开的普通烟草品种K326基因组序列数据[22],通过同源比对的方法对普通烟草的NAC转录因子进行全基因组鉴定,并结合表达模式分析,以期筛选出参与非生物逆境胁迫应答的候选NAC转录因子,为普通烟草抗逆调控网络解析和抗逆品种的选育奠定基础。
1 材料与方法
1.1 试验材料
以普通烟草品种K326为供试材料,种子由贵州省烟草科学研究院提供。植物多酚RNA提取试剂盒(Invitrogen公司);反转录试剂盒和实时荧光定量试剂盒(TaKaRa公司);引物由上海捷瑞生物工程有限公司合成;其它常规试剂均为国产分析纯试剂。
1.2 试验处理
使用漂浮育苗方式育苗,将漂浮盘置于人工气候室中,条件为28 ℃光照16 h,20 ℃黑暗8 h。出苗4周后将幼苗取出,用清水冲洗根部,随后用吸水纸吸干水分后,置于灭菌水培养1周。结合前期预实验及相关文献报道进行胁迫处理:干旱(20% PEG6000 溶液)、盐害(50 mmol/L NaCl溶液)、ABA(100mmol/L)、冷害(置于4 ℃人工气候箱)[15,20-21]。分别在0 h、1 h、3 h、6 h、12 h和24 h对各处理烟株根系进行取样,同时取未经处理的样品作为对照。所取样品立即置于液氮中保存,试验设置3次生物学重复。
1.3 试验方法
1.3.1 普通烟草NAC转录因子家族鉴定
从茄科物种基因组数据库(SGN,https:// solgenomics.net)下载普通烟草品种K326的基因组序列及注释文件,从Pfam数据库(http://pfam.xfam.org)下载隐马尔可夫模型文件(PF02365),利用HMMER3.1b2软件(http://hmmer.org/)进行普通烟草NAC蛋白序列的初步检索,阈值(E-value)设为1.0,将初步获得的蛋白序列作为检索序列,利用Blastp软件(https://blast.ncbi.nlm.nih.gov/Blast.cgi)同K326基因组所有蛋白序列进行比对,参数为默认。所有HMMER和Blastp获得序列结果分别用InterProScan (https://www.ebi.ac.uk/interpro)、SMART (http:// smart.embl-heidelberg.de)和NCBI Conserved Domain Search(https://www.ncbi.nlm.nih.gov/Structure/bwrpsb/ bwrpsb.cgi)进行蛋白保守结构功能域预测,对上述3个工具软件分析结果中含有NAC功能域的蛋白列表求交集,再删除氨基酸序列长度低于110的蛋白序列,剩余序列作为普通烟草NAC转录因子蛋白序列,按各序列对应基因在参考基因组连锁群上的位置顺序依次命名。
1.3.2 序列特征分析
对获得的普通烟草NAC蛋白序列的分子量、等电点(PI)选择使用EXPasy(http://web.expasy.org/ protparam)进行分析;跨膜结构域分析选择使用TMHMM server v.2.0 (https://services.healthtech.dtu.dk/ service.php?TMHMM-2.0);蛋白亚细胞定位预测选择使用Cell-PLoc 2.0(http://www.csbio.sjtu.edu.cn/bioinf/ Cell-PLoc-2)。以上分析工具均为网页在线工具,参数选择默认。
本文中选择的仿真频率为1.5 GHz,该频率是导航卫星信号的主要频率,目标为空客A320和 F-15C型战斗机,材料为金属,采用商用软件CST进行电磁计算,采用的方法为快速多层多极子算法,以1°为间隔对目标模型进行仿真。
1.3.3 系统发育、序列结构和蛋白Motif分析
从拟南芥基因组网站(TAIR 9.0,http://www. arabidopsis.org)下载105个拟南芥NAC蛋白序列,使用Clustal W软件[23]将拟南芥NAC蛋白序列和烟草NAC蛋白序列进行多序列比对,利用MEGA7.0软件[24]的最大似然估计方法构建系统发育树,bootstrap值设为1000。用MEME软件(http://meme-suite.org/tools/ meme)对烟草NAC蛋白进行Motif分析,同时选择K326基因组GFF3注释文件,利用TBtools工具(https://github.com/CJ-Chen/TBtools/releases)对获得的烟草NAC蛋白和基因结构进行可视化展示。
1.3.4 组织表达模式分析
从EMBL-EBI网站(https://www.ebi.ac.uk)下载普通烟草TobEA表达谱数据[25],该数据包含种子、子叶、幼芽、幼根、成熟根、下部茎、上部茎、茎顶端、幼叶、茎生叶、成熟叶、早期衰老叶、中早期衰老叶、中后期衰老叶、后期衰老叶、未开花蕾、开放花蕾、花朵与花顶端共计19个样品组织,贯穿烟草生长发育的整个生命周期。从https://link.springer.com/article/10. 1186/1471-2164-11-142网页下载表达谱对应的CDS序列,利用Blastn程序将NtabNAC家族成员CDS序列比对,max_target_seqs设置为1,其余参数为默认,获得各成员对应的表达谱基因编号。对获得的表达数据进行聚类分析,结合进化关系,分析普通烟草NAC家族成员的表达模式并预测功能。
1.3.5 实时荧光定量PCR验证
根据进化关系和1.3.4中的表达模式分析结果,筛选抗逆候选NAC转录因子基因,选用烟草基因(NTU60495)为内参基因,根据序列特异性分别设计引物(表1),利用TaKaRa公司Taqman探针试剂盒在ABI SteponePlus实时荧光定量PCR仪上进行qPCR实验,采用2-ΔΔCt相对定量法分析候选基因对干旱、盐、ABA和冷害胁迫处理的响应,对照样本各基因表达量作为标准1。
表1 部分普通烟草基因qRT-PCR引物

Tab.1 qRt-PCR primers for part of Nicotiana tabacum L.NAC genes
2 结果与分析
2.1 普通烟草NAC家族序列鉴定及特征分析
利用HMMER和Blastp程序对普通烟草品种K326蛋白序列比对分析,初步获得276个非冗余候选NAC蛋白序列,结构功能域分析和剔除氨基酸长度低于110的蛋白序列后,最终获得190个普通烟草NAC家族基因,按照基因在连锁群上的顺序以依次进行编号。分析序列的理化性质结果显示(附表1,http://ycxb.tobacco.org.cn/ news/video/0f7fbdbf-27af-473e-af81-c7d59bbaf89a.htm),这些蛋白的平均氨基酸长度为347,范围在129~992之间;平均分子量为39537.87 Da,范围在14666.30~113371.89 Da之间;平均等电点为6.82,范围在4.47~9.76之间;平均脂肪族氨基酸指数为66.87,范围在49.75~112.86之间;平均疏水性指数为-0.673,范围在-1.096~0.22 之间,仅NtabNAC190的疏水性指数大于0,为0.22,表明该蛋白为疏水性蛋白,其余NAC家族蛋白全部表现为亲水性。亚细胞定位预测结果发现,基因定位结果为叶绿体和细胞核,基因定位结果为内质网,其余结果均定位到细胞核内;跨膜结构域结果显示17个蛋白含有跨膜结构,其中13个蛋白(NtabNAC014、NtabNAC026、NtabNAC035、NtabNAC041、NtabNAC096、NtabNAC121、NtabNAC123、NtabNAC131、NtabNAC138、NtabNAC139、NtabNAC140、NtabNAC181、NtabNAC184)含有1个跨膜结构,3个蛋白(NtabNAC048、NtabNAC057、NtabNAC120)含有2个跨膜结构,1个蛋白NtabNAC185含有4个跨膜结构。
2.2 普通烟草NAC家族进化分析
将拟南芥全基因组105个NAC家族氨基酸序列和研究获得的190个普通烟草NAC家族氨基酸序列构建系统进化树。参照前人研究结果[8],将拟南芥和普通烟草的NAC蛋白划分为18个亚组,各亚组命名及含有的拟南芥和普通烟草NAC蛋白见图2,其中NtNAC和ANAC104亚组为新命名。在构建的系统进化树上,NtNAC亚组包含16个普通烟草NAC蛋白,这些蛋白组成一个簇,且无拟南芥NAC蛋白存在,在进化关系上显示出一定的特异性,因此将其命名为NtNAC亚组。因ANAC104亚组仅含有1个拟南芥NAC蛋白ANAC104,而ANAC104在Ooka等[8]的研究中,在亚组上未有明确归属,本研究将该亚组命名为ANAC104,同时将8个普通烟草NAC蛋白划分到该亚组。剩余16个亚组中,含有普通烟草NAC蛋白的数目分别为4~26个,其中OsNAC7亚组和NAM亚组含有的普通烟草NAC蛋白数量较多,分别有26和25个,与抗逆胁迫相关的亚组AtNAC3和ATAF分别含有4个和5个普通烟草NAC蛋白。
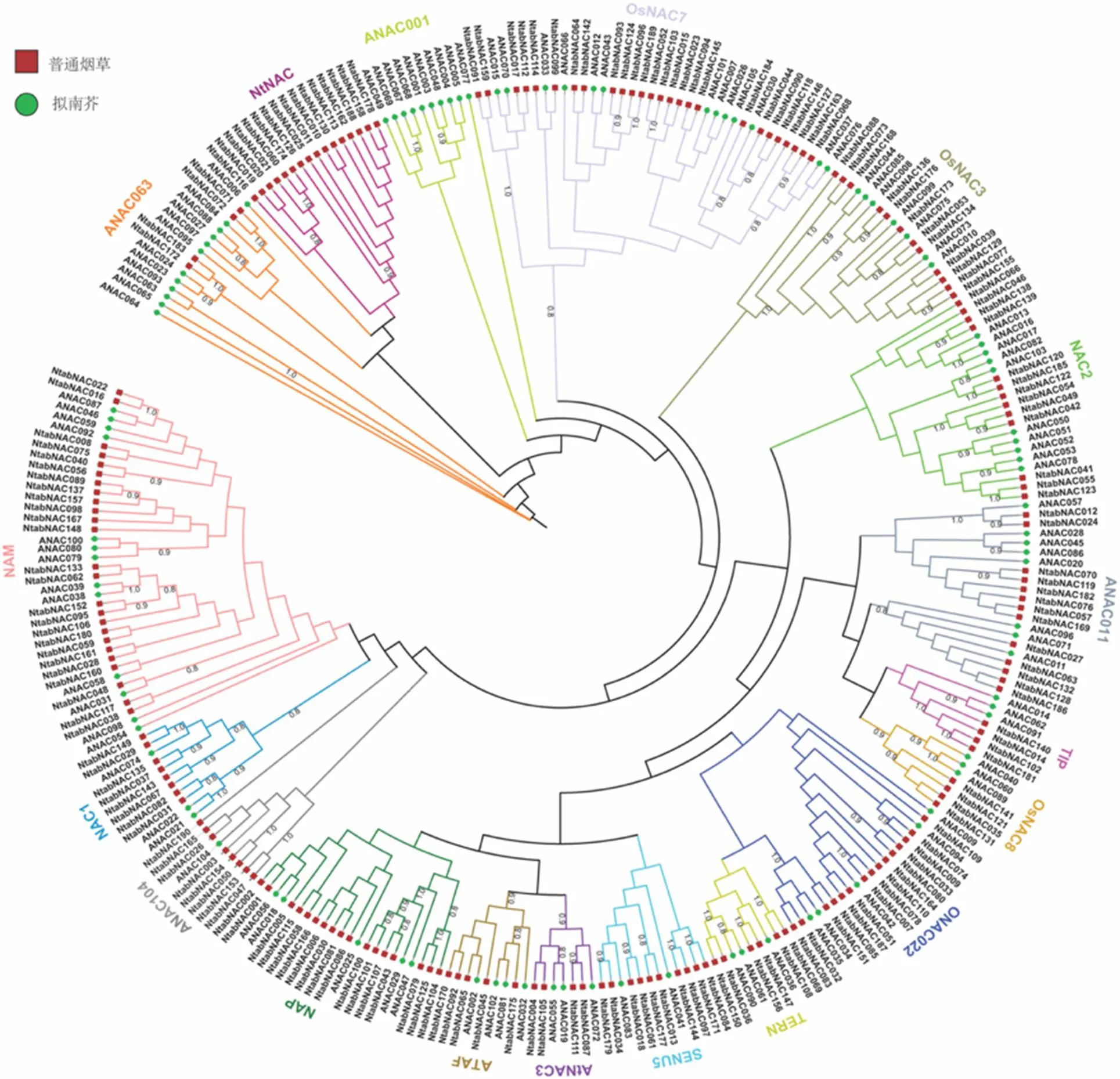
图2 普通烟草与拟南芥NAC 蛋白系统进化树
2.3 普通烟草NAC基因组织表达模式分析
对分析获得的190个普通烟草基因与烟草表达谱(TobEA)包含的序列进行Blastn比对,结果共有80个普通烟草基因在烟草表达谱中检索到表达数据。这80个基因分布在15个亚组(除ANAC063亚组)中,结合表达谱注释文件、进化树信息及拟南芥基因组数据库信息,将检索到的80个基因及其同源表达谱基因ID、拟南芥基因序列号进行汇总整理,同时依据拟南芥数据库注释信息,对每个亚组基因的功能进行预测,结果见表2。
表2 部分普通烟草基因对应表达谱和拟南芥基因序列号及预测基因功能
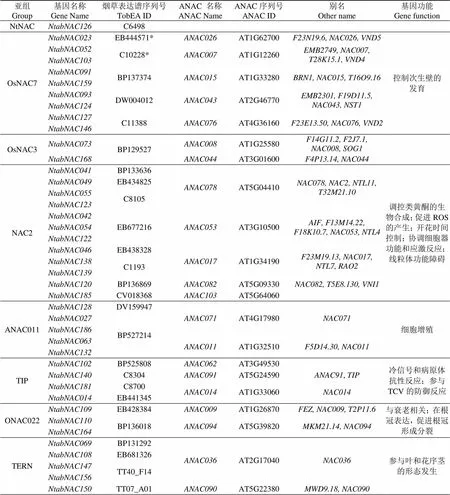
Tab.2 Expression profile ID of some NAC genes in Nicotiana tabacum L., Arabidopsisthaliana ID and prediction of gene function
续表2

亚组Group基因名称Gene Name烟草表达谱序列号TobEA IDANAC 名称ANAC NameANAC序列号ANAC ID别名Other name基因功能Gene function SENU5NtabNAC013CV021715ANAC083AT5G13180NAC083, T19L5.140, VNI2调控木质部导管形成 NtabNAC177 NtabNAC018C4770 NtabNAC061 NtabNAC034C3485 NtabNAC179BP131326 AtNAC3NtabNAC087BP128761ANAC072AT4G27410F27G19.10, NAC072, RD26受干旱、盐害、冷害、脱落酸诱导,参与脱落酸介导的水分调控 NtabNAC111EB434749 ATAFNtabNAC045C5212ANAC102AT5G63790ATAF2,MBK5.27, NAC102参与脱落酸信号转导和合成,受机械损伤、生物逆境和非生物逆境诱导 NtabNAC065EB437400 NtabNAC092C10228ANAC002AT1G01720ATAF1, NAC002, T1N6.12 NtabNAC170 NAPNtabNAC001TT06_M17ANAC056AT3G15510ATNAC2, MJK13.17, NAC056, NAC2, NARS1叶片衰老、芽发育、逆境胁迫 NtabNAC002 NtabNAC005 NtabNAC043EB425871ANAC029AT1G69490ATNAP, F10D13.14, NAC029, NAC29, NAP NtabNAC101 NtabNAC107C10525 NtabNAC100TT16_L09ANAC047AT3G04070NAC047, SHG, SHYG ANAC104NtabNAC050TT10_P18ANAC104AT5G64530MUB3.5, NAC104, XND1 NtabNAC154 NAC1NtabNAC029EB444571ANAC074AT4G28530NAC074调控柱头组织的程序性细胞死亡 NtabNAC067 NtabNAC143 NAMNtabNAC016BP526410ANAC087AT5G18270 正调控衰老;诱导叶片细胞死亡和衰老;在衰老和叶绿素调节中起作用;与植株形态发育相关 NtabNAC022 NtabNAC152 NtabNAC089C4769ANAC100AT5G61430AT5G61430 NtabNAC137ANAC079AT5G07680NAC080, NAC4 NtabNAC148ANAC080 NtabNAC028DW004957ANAC058AT3G18400NAC058 NtabNAC059 NtabNAC161 NtabNAC008TT16_C05ANAC092AT5G39610ATNAC2, ATNAC6, NAC2 NtabNAC040ANAC059AT3G29035ATNAC3, NAC3, ORE1 NtabNAC056ANAC046AT3G04060NAC046 NtabNAC075 NtabNAC062EB683185ANAC039AT2G24430 NtabNAC133
注:含有‘*’表示烟草表达谱序列号出现了2次。
依据表达谱数据,对基因序列进行聚类分析,将19个组织分为3个类群(图3)。组织类群A主要为成熟的叶、花等相关衰老组织,B主要为种子、子叶、茎尖、幼叶等幼嫩组织,C主要为根、茎、花芽等组织。基因聚类结果表明45个基因分为4个类群,其中基因类群I、III的基因表达量结果相对高于II、IV,且不同组织类群间的基因表达差异更显著。4个基因类群两两比较,基因类群I的表达量总体高于其他3个类群,且在成熟衰老组织类群A中的表达量与另外2个组织类群相比呈现相对较高的态势。基因类群II的基因表达量在4个类群中最低,且在3个组织类群中的表达量差异不明显。基因类群III的基因表达量与其他3个基因类群相比,表达量处于中间偏上水平,且在各组织类群间表现明显差异。值得注意的是,在普通烟草NAC基因家族中,主要和逆境胁迫响应相关的AtNAC3和ATAF亚组的基因并没呈现出趋同表达的情况。如AtNAC3亚组中,烟草表达谱序列号为EB434749和BP128761的2个序列,前者基因聚类到基因类群I,而后者则划分到基因类群II,二者的表达模式完全不同,ATAF亚组中基因也表现出类似情形。
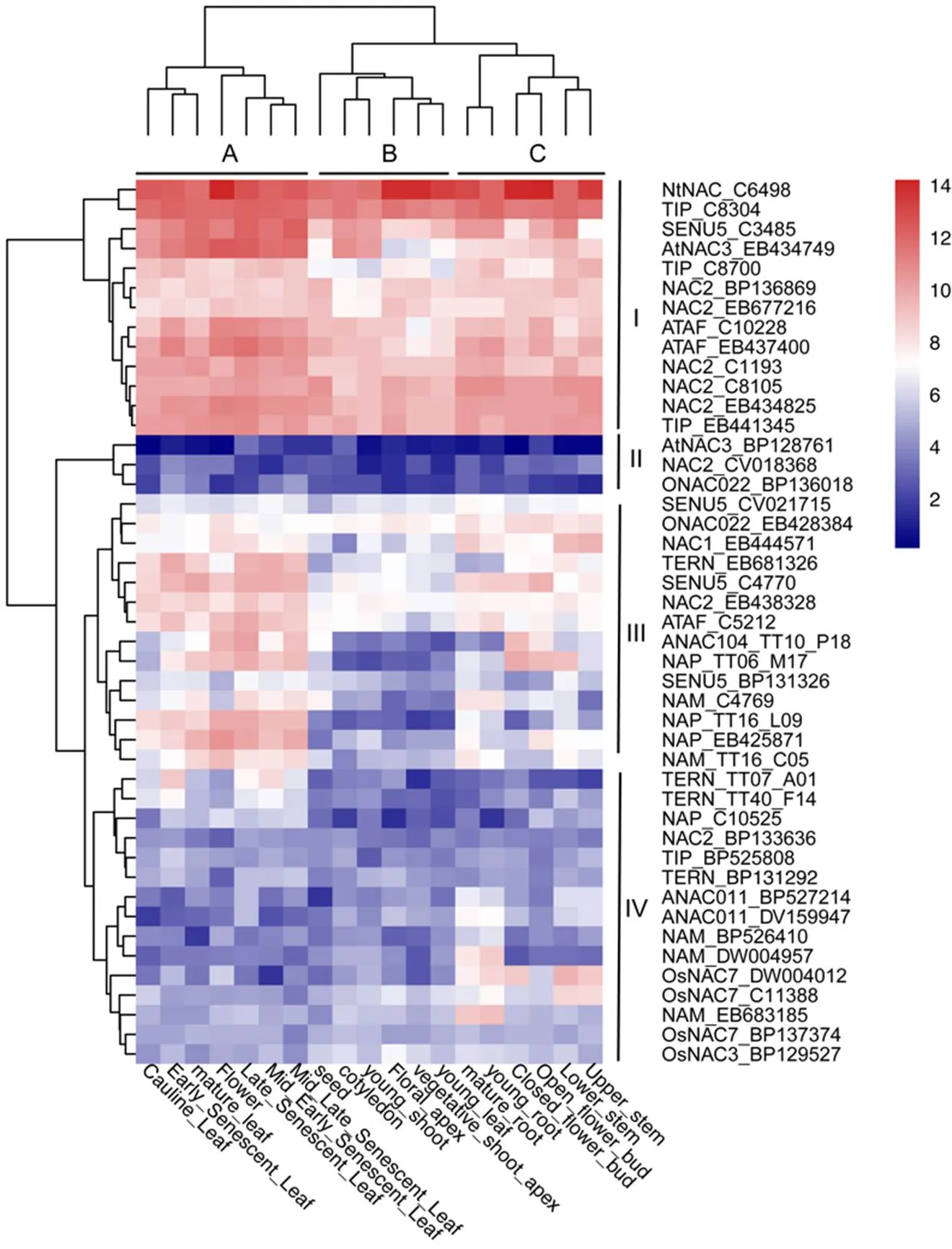
图3 烟草NAC基因家族基因表达热图
2.4 普通烟草NAC基因结构和蛋白Motif分析
利用拟南芥、普通烟草参考基因组注释信息,对2个物种NAC基因家族的基因结构、蛋白功能域模式(Motif)进行分析,其中AtNAC3和ATAF亚组的分析结果见图4。结果表明,本研究划分的亚组内基因结构和功能域模式表现呈一致性,亚组间则差异明显。NAC蛋白功能域中2个保守子域C、D在2个亚组间则差异明显。NAC蛋白功能域中2个保守子域C、D在2个亚组间存在差异特征,其中AtNAC3亚组蛋白保守子域C(motif 3)中,甘氨酸-苏氨酸-天冬氨酸序列(GTD)在ATAF亚组变为甘氨酸-丙氨酸-天冬氨酸序列(GAD)。在保守子域D中,存在于AtNAC3亚组蛋白中甘氨酸-苏氨酸-赖氨酸序列(GTK),在ATAF亚组中变为甘氨酸-谷氨酸-赖氨酸序列(GEK)或甘氨酸-异亮氨酸-赖氨酸序列(GIK)。这些特征分化明显,可作为区分2个亚组成员的序列特征。根据基因组注释信息显示,2个物种的ATAF亚组基因的结构都有3个外显子和2个内含子,蛋白模式中NAC功能域都由motif 1、2、3、8、5组成,表明2个物种的ATAF亚组基因分化程度较小。与之相比,AtNAC3亚组基因则在2个物种中结构和蛋白模式有明显的分化,其中基因的外显子和内含子数目各增加1个,而基因则增加2个,同时发现和基因的内含子序列长度也明显增加。蛋白序列多重比较及蛋白模式结果表明,NtabNAC105蛋白的子域A丢失了motif 1,NtabNAC087和NtabNAC111蛋白的子域B则发生变异,其motif 2 分别变成了motif 6,motif 6 + motif 7。基因的结构、蛋白模式与拟南芥AtNAC3亚组基因相比,表现基本一致。
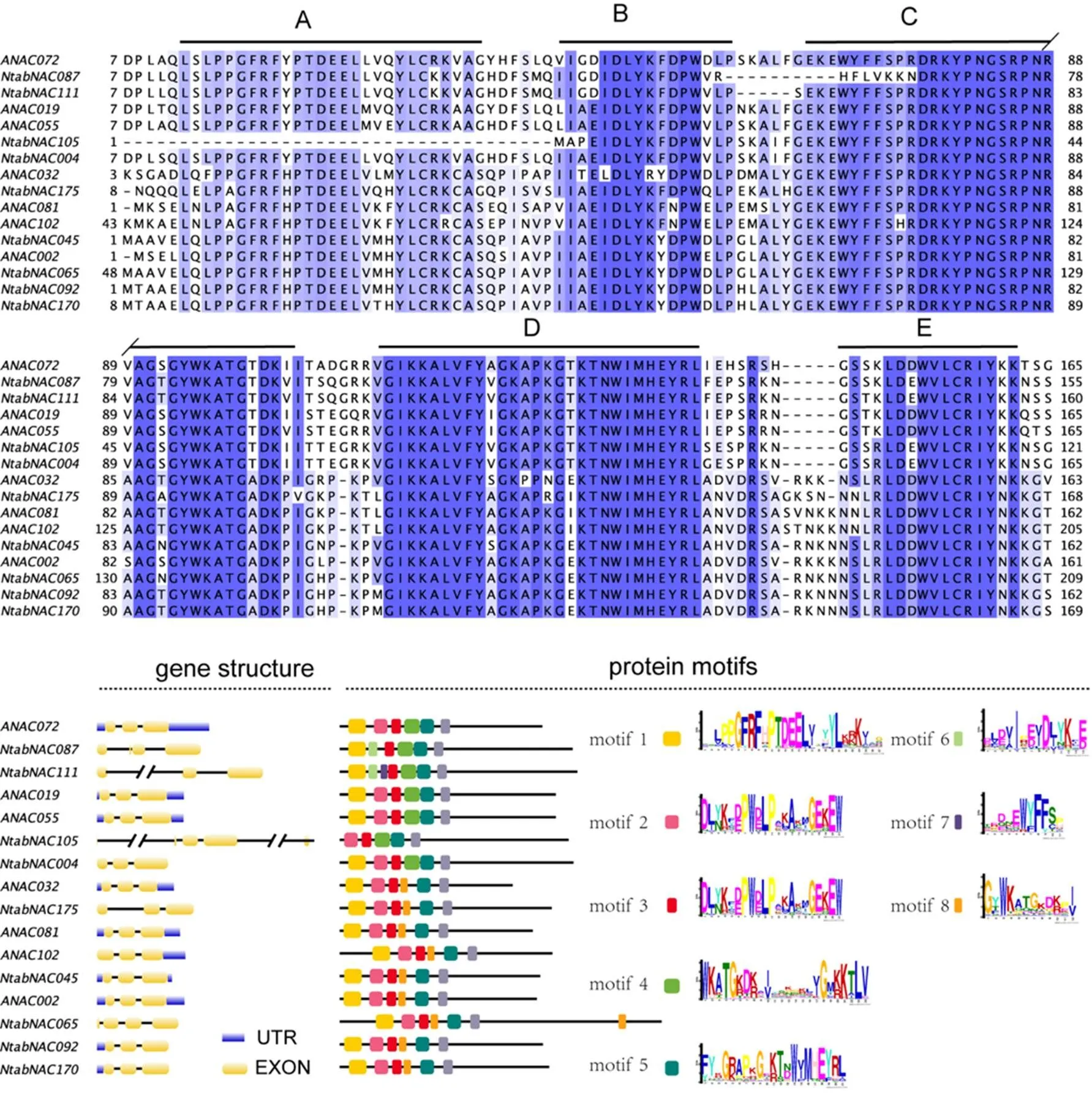
图4 AtNAC和ATAF亚组烟草基因序列结构和蛋白结构域
2.5 普通烟草AtNAC3和ATAF亚组基因逆境处理下表达特征
qRT-PCR试验结果表明烟草幼苗在遭受逆境胁迫处理后,AtNAC3和ATAF亚组的9个基因在根部的表达量均呈现出不同程度的上调,但不同基因上调程度差异显著,其中、和上调最为明显。从时间梯度分析,除外,其余基因受胁迫处理后,基因表达量多在1~3 h内达到最高。2个亚组内基因相比,和在AtNAC3亚组,二者的表达变化趋势基本相同,但二者对应的直系同源基因与之相比,在遭受逆境胁迫处理后,表达量变异范围显著变小,同时ATAF亚组的基因与同亚组基因相比表现出相同趋势。另外,值得注意的是和在受ABA处理后,表达量显著增加,而变化不显著。
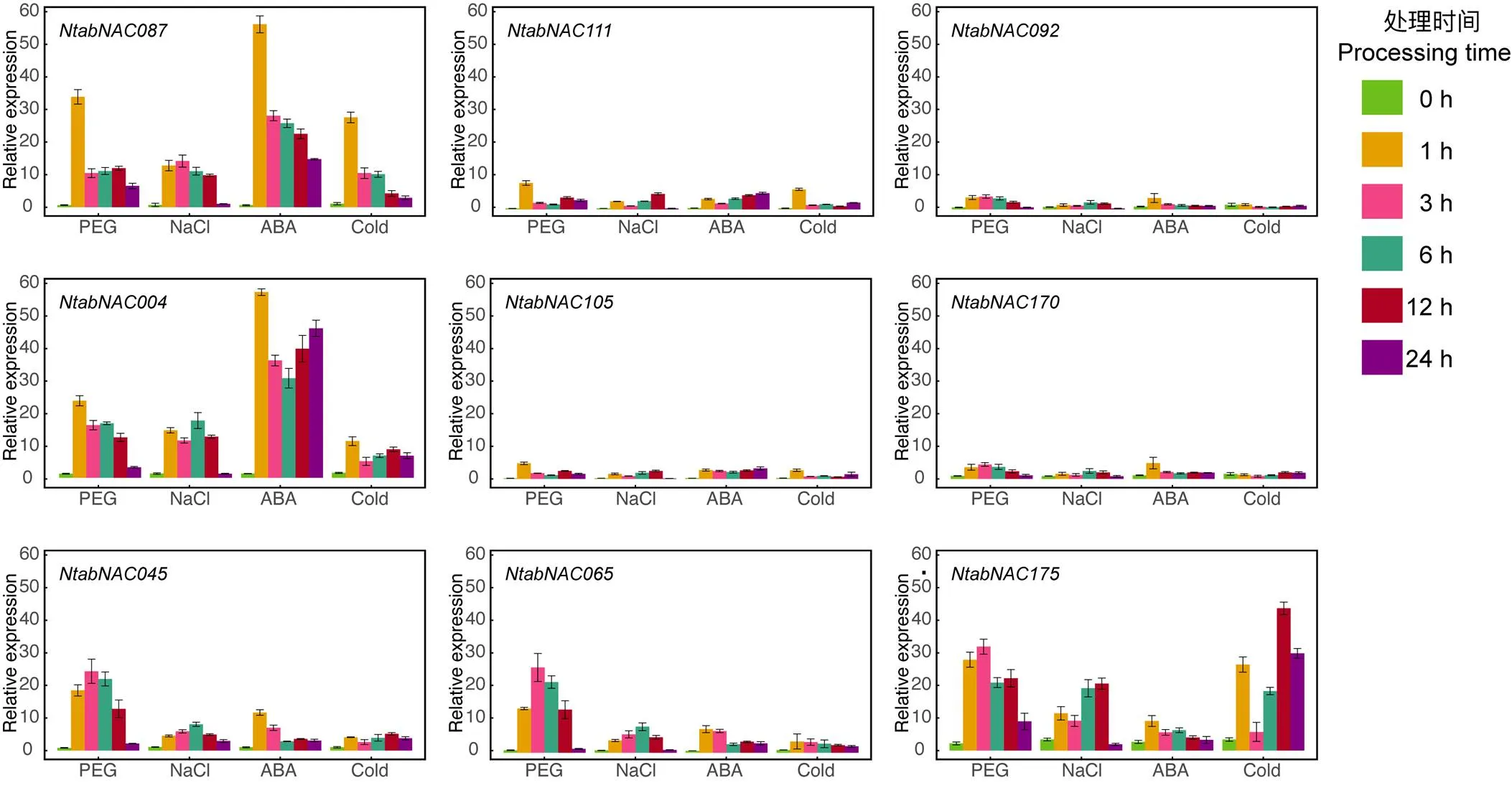
图5 普通烟草AtNAC3和ATAF亚组NAC基因在逆境胁迫下基因表达模式
3 讨论
基因是植物特有的转录因子,参与植物生长发育和生物胁迫应答[26]。普通烟草作为一个典型的模式生物,具有易转化、生物量大和对环境敏感等特点,系统研究和鉴定烟草中的NAC转录因子家族,对烟草及其他作物生物学研究都具有重要意义。早期的普通烟草NAC基因家族研究分别鉴定出152和154个基因[27-28],而植物转录因子数据库(PlantTFDB V5.0,http://planttfdb.gao-lab.org/index.php)则收录有280个预测的普通烟草NAC转录因子序列,以上结果对系统研究普通烟草NAC转录因子造成一定困扰。本研究利用2017年公开的K326基因组序列[22]及相关数据共鉴定出190个普通烟草NAC蛋白,多于前期研究发现的152个和154个,这是普通烟草参考基因组不断完善带来的结果。本研究利用HMMER和Blastp程序获得的276个原始基因列表,同PlantTFDB数据库收录的烟草NAC转录因子列表结果相当,但其中部分序列不含有NAC功能域。
序列比对、结构分析和进化分析结果表明,同一个亚组上的蛋白序列和结构相似程度很高,不同亚组间则有一定区别,这可能和NAC功能分化相关。在划分的18个亚组中,AtNAC3和ATAF亚组是拟南芥中被报道和参与逆境胁迫最多的2个亚组[9],水稻[14]、玉米[15]和[16]等基因与这2个亚组的拟南芥基因高度同源。本研究共有9个烟草基因划分到这2个亚组,与拟南芥基因相比,普通烟草基因存在多倍化造成的基因加倍现象。如基因,在烟草中具有和两个直系同源基因,这2个基因就是由普通烟草的2个祖先种林烟草和绒毛状烟草基因组融合导致。同时这2个亚组中烟草基因结构与拟南芥的结构上发生一些变化,如拟南芥及其他物种基因在这2个亚组中多为3个外显子和2个内含子[29-30],但在本研究中,这2个亚组中的、、和等基因的外显子和内含子都发生增多,造成基因结构多样性的变化,这种变化可能会造成基因功能的改变,影响植物对环境的适应性[31]。
基因表达模式分析结果表明,AtNAC3和ATAF亚组中基因,在各组织间以及遭受逆境胁迫后,基因表达量变化存在显著差异,原因可能是基因序列结构变异导致基因功能产生变化,2个直系同源基因中的一个丧失了逆境胁迫调控应答功能,但更可能是普通烟草在进化过程中,由于染色体加倍造成2个直系同源基因仅一个基因发挥功能,简称基因功能冗余[32]。本研究中,AtNAC3亚组和ATAF亚组分别包含4个和5个基因,其中、和在受胁迫处理后,相较其基因的表达量,变化更为显著,且值得注意的是AtNAC3亚组中的、两个基因受ABA处理处理后的表达量变化与相比存在差异。前人研究表明AtNAC3亚组基因,对干旱、盐害、低温等多个非生物逆境皆有响应[33],ATAF亚组中等基因除和非生物胁迫相关外,还参与生物胁迫的调控[34]。综合表2和前人研究结果,推测AtNAC3亚组基因可能参与植物体内ABA代谢途径,而ATAF亚组基因则可能和茉莉酸(JAs)、水杨酸(SA)等代谢途径更为密切[12,15,35]。
4 结论
本研究利用公开的普通烟草参考基因组数据,系统鉴定了普通烟草NAC家族基因,并对这些基因的理化性质、结构变异、系统发育及表达模式进行分析,主要发现:(1)鉴定的190个基因具有典型的转录因子特征,主要在细胞核中发挥作用。(2)根据基因结构和进化关系,将普通烟草和拟南芥NAC蛋白划分为18个亚组,不同亚组之间的结构和功能存在差异,其中AtNAC3和ATAF亚组是普通烟草基因响应胁迫的重点关注对象。(3)、和可作为NAC转录因子家族中,研究烟草抗逆的基因调控网络和后续抗逆育种的重点关注基因,且可能涉及不同代谢调控途径。
[1] Godoy F, Olivos-Hernández K, Stange C, et al. Abiotic stress in crop species: improving tolerance by applying alant metabolites[J]. Plants, 2021, 10(2): 186-205.
[2] Baillo E H, Kimotho R N, Zhang Zhengbin, et al. Transcription factors associated with abiotic and biotic stress tolerance and their potential for crops improvement[J]. Genes, 2019, 10(10): 771-794.
[3] Lan Thi Hoang X, Du Nhi N H, Binh Anh Thu N, et al. Transcription factors and their roles in signal transduction in plants under abiotic stresses[J]. Current Genomics, 2017, 18(6): 483-497.
[4] Yoon Y, Seo D H, Shin H, et al. The role of stress-responsive transcription factors in modulating abiotic stress tolerance in plants[J]. Agronomy, 2020, 10(6): 788-811.
[5] Aida M, Ishida T, Fukaki H, et al. Genes involved in organ separation in: an analysis of the cup-shaped cotyledon mutant[J]. The Plant Cell, 1997, 9(6): 841-857.
[6] Aida M, Ishida T, Tasaka M. Shoot apical meristem and cotyledon formation duringembryogenesis: interaction among the CUP-SHAPED COTYLEDON and SHOOT MERISTEMLESS genes[J]. Development, 1999, 126(8): 1563-1570.
[7] Souer E, van Houwelingen A, Kloos D, et al. The no apical meristem gene of Petunia is required for pattern formation in embryos and flowers and is expressed at meristem and primordia boundaries[J]. Cell, 1996, 85(2): 159-170.
[8] Ooka H, Satoh K, Doi K, et al. Comprehensive analysis of NAC family genes inand[J]. DNA Research, 2003, 10(6): 239-247.
[9] Dudhate A, Shinde H, Yu P, et al. Comprehensive analysis of NAC transcription factor family uncovers drought and salinity stress response in pearl millet ()[J]. BMC Genomics, 2021, 22(1): 1-15.
[10] Olsen A N, Ernst H A, Leggio L L, et al. NAC transcription factors: structurally distinct, functionally diverse[J]. Trends in Plant Science, 2005, 10(2): 79-87.
[11] Fujita M, Fujita Y, Maruyama K, et al. A dehydration-induced NAC protein, RD26, is involved in a novel ABA-dependent stress- signaling pathway[J]. The Plant Journal, 2004, 39(6): 863-876.
[12] Puranik S, Sahu P P, Srivastava P S, et al. NAC proteins: regulation and role in stress tolerance[J]. Trends in Plant Science, 2012, 17(6): 369-381.
[13] Tran L S P, Nakashima K, Sakuma Y, et al. Isolation and functional analysis ofstress-inducible NAC transcription factors that bind to a drought-responsive-element in the early responsive to dehydration stress 1 promoter[J]. The Plant Cell, 2004, 16(9): 2481-2498.
[14] Hu Honghong, Dai Mingqiu, Yao Jialing, et al. Overexpressing a NAM, ATAF, and CUC (NAC) transcription factor enhances drought resistance and salt tolerance in rice[J]. Proceedings of the National Academy of Sciences, 2006, 103(35): 12987-12992.
[15] Mao Hude, Yu Lijuan, Han Ran, et al. ZmNAC55, a maize stress-responsive NAC transcription factor, confers drought resistance in transgenic[J]. Plant Physiology and Biochemistry, 2016, 105: 55-66.
[16] Chen Dandan, Richardson T, Chai Shoucheng, et al. Drought- up-regulated TaNAC69-1 is a transcriptional repressor ofand, and enhances root length and biomass in wheat[J]. Plant and Cell Physiology, 2016, 57(10): 2076-2090.
[17] Mao Hude, Li Shumin, Wang Zhongxue, et al. Regulatory changes inare associated with drought tolerance in wheat seedlings[J]. Plant Biotechnology Journal, 2020, 18(4): 1078-1092.
[18] Zhao Qiang, Hu Risheng, Liu Dan, et al. The AP2 transcription factorconfers drought resistance by modifying[J]. Plant Biotechnology Journal, 2020, 18(12): 2444-2455.
[19] 徐小艳,姚新转,吕立堂,等. 烟草基因的克隆及其在烟草中的抗旱功能分析[J]. 植物生理学报,2018, 54(6): 1085-1094.
XU Xiaoyan, YAO Xinzhuan, LV Litang, et al. Cloning ofgene fromand its analysis of drought-resistant function[J]. Plant Physiology Journal, 2018, 54(6): 1085-1094.
[20] 代婷婷,姚新转,吕立堂,等. 烟草基因的克隆及其抗旱功能分析[J]. 农业生物技术学报,2018, 26(5): 764-773.
DAI Tingting, YAO Xinzhuan, LV Litang, et al. Cloning and drought-resistant function analysis ofGene in tobacco () [J]. Journal of Agricultural Biotechnology, 2018, 26(5): 764-773.
[21] 李晓旭,郭存,刘成,等. 普通烟草基因的克隆,鉴定及表达模式分析[J]. 烟草科技,2019, 52(3): 10-17.
LI Xiaoxu, GUO Cun, LIU Cheng, et al. Cloning, characterization and expression pattern analysis ofgene in[J]. Tobacco Science & Technology, 2019, 52(3):10-17.
[22] Edwards K D, Fernandez-Pozo N, Drake-Stowe K, et al. A reference genome forenables map-based cloning of homeologous loci implicated in nitrogen utilization efficiency[J]. BMC Genomics, 2017, 18(1): 1-14.
[23] Larkin MA, Blackshields G, Brown NP, et al. Clustal W and Clustal X version 2.0[J]. Bioinformatics, 2007:23, 2947- 2948.
[24] Kumar S, Stecher G, Tamura K. MEGA7: Molecular Evolutionary Genetics Analysis version 7.0 for bigger datasets [J] Molecular Biology and Evolution, 2016:33,1870-1874
[25] Edwards KD, Bombarely A, Story GW, et al. TobEA: an atlas of tobacco gene expression from seed to senescence[J]. Bmc Genomics, 2010, 11(1):142.
[26] Gao Yujiao, An Kexin, Guo Weiwei, et al. The endosperm- specific transcription factor TaNAC019 regulates glutenin and starch accumulation and its elite allele improves wheat grain quality[J]. The Plant Cell, 2021, 33(3): 603-622.
[27] Li Wei, Li Xiaoxu, Chao Jiangtao, et al. NAC family transcription factors in tobacco and their potential role in regulating leaf senescence[J]. Frontiers in Plant Science, 2018, 9: 1900-1915.
[28] Rushton P J, Bokowiec M T, Han S, et al. Tobacco transcription factors: novel insights into transcriptional regulation in the[J]. Plant Physiology, 2008, 147(1): 280-295.
[29] Li Feng, Guo Xuhu, Liu Jianxia, et al. Genome-wide identification, characterization, and expression analysis of the NAC transcription factor in[J]. Genes, 2019, 10(7): 500-513.
[30] Liu Xingwang, Bartholomew E, Black K, et al. Comprehensive analysis of NAC transcription factors and their expression during fruit spine development in cucumber (L.)[J]. Horticulture Research, 2018, 5(1): 1-14.
[31] Fan K, Bibi N, Gan S, et al. A novel NAP member GhNAP is involved in leaf senescence in[J]. Journal of Experimental Botany, 2015, 66(15): 4669-4682.
[32] Takasaki H, Maruyama K, Takahashi F, et al. SNAC-As, stress- responsive NAC transcription factors, mediate ABA-inducible leaf senescence[J]. The Plant Journal, 2015, 84(6): 1114-1123.
[33] Ye Huaxun, Liu Sanzhen, Tang Buyun, et al. RD26 mediates crosstalk between drought and brassinosteroid signalling pathways[J]. Nature Communications, 2017, 8(1): 1-13.
[34] Trishla V S, Marriboina S, Boyidi P, et al. GUS-reporter based analysis of the promoter activity ofNAC transcription factor, GhNAC4 that is induced by phytohormones and environmental stresses[J]. Plant cell, tissue and organ culture, 2020, 141(3): 643-654.
[35] Li Mengya, Yu Guanghui, Cao Congli, et al. Metabolism, signaling and transport of jasmonates[J]. Plant Communications, 2021: 100231.
Identification and expression analyses of the NAC transcription factor family inL.
YU Shizhou1, YANG Zhixiao1, YU Jing1, SONG Shihao2, ZHANG Xiaolian1, ZHAO Huina1, ZHANG Changyun1, LIN Yingchao1, LEI Bo1*
1 Molecular Genetics Key Laboratory of China Tobacco, Guizhou Academy of Tobacco Science, Guiyang 550081, China;2 Qingdao Etsong Technology Co.,Ltd., Qingdao 266011, China
NAC (NAM/ATAF/CUS2) gene family, as one of the unique and largest transcription factor families of plants, is widely involved in development regulation and stress response of plants.Based on the available reference genome of(K326), we tried to identify NAC family by motif analysis and homolog search with. Moreover, the tissue expression patterns, physicochemical properties and phylogenetic relationships for these NACs were predicted using bioinformatics strategy. Moreover, qRT-PCR assays were performed to examine expression patterns of thesegenes in AtNAC3 and ATAF subgroups under drought, cold, salt and ABA treatments.A total of 190 NACs were identified in tobacco, and the average length of amino acids was 347. The proteins were hydrophilic except NtabNAC190, and most of them were located in the nucleus according to Subcellular localization. Further, the proteins we classified into 18 subgroups, and expression pattern analysis showed that the expression specificity of different subgroups was different. 9 tobaccogenes in AtNAC3 and ATAF subgroupwere significantly up-regulated under abiotic stress treatment, whereas,andwere the most significant candidates.,andare involved in the regulation of adversity stress response, which may be good regulators to improve tobacco breeding in stress resistant.
L.; NAC gene family; transcription factors; phylogeny; expression pattern; abiotic stress
. Email:leibo_1981@163.com
余世洲,杨志晓,余婧,等. 普通烟草NAC转录因子家族抗逆基因筛选与表达分析[J]. 中国烟草学报,2022,28(2).YU Shizhou, YANG Zhixiao, YU Jing, et al. Identification and expression analyses of the NAC transcription factor family in Nicotiana tabacum L. [J]. Acta Tabacaria Sinica, 2022,28(2).doi:10.16472/j.chinatobacco. 2021.T0158
烟草基因组计划重大专项(No. 110202001027(JY-10));中国烟草总公司贵州省公司项目(2021XM05);贵州省科学技术基金项目(黔科合基础[2020]1Y106)
余世洲(1986—),博士,副研究员,主要从事数量遗传学与生物信息学研究,Tel:0851-84116909,Email:yusz@nwafu.edu.cn
雷波(1981—),博士,研究员,主要从事烟草分子生物学研究,Tel:0851-84116909,Email:leibo_1981@163.com
2021-09-08;
2021-12-29

