多色单分子定位显微技术研究进展(特邀)
赵悦晗,郝翔
(浙江大学 光电科学与工程学院现代光学仪器国家重点实验室,杭州310027)
0 引言
光学显微镜因具有非接触、高特异性等技术优势,已被广泛应用于生物、医学、化学等领域。然而,由于衍射的存在,标准光学显微镜技术的空间分辨率被限制在大约一半的光波长,横向尺寸约为200~300 nm,轴向尺寸约为500~700 nm。这限制了光学显微镜技术解析单个分子或分子复合物的亚细胞组织的能力,例如,由数百种单独的蛋白质组成的核孔复合物的结构,直径仅为约120 nm,无法由常规的光学显微镜观察。为了克服衍射极限带来的光学显微镜的分辨率屏障,在过去15年中诞生了一系列以受激发射损耗显微成像术(Stimulated Emission Depletion,STED)[1-2]、结构光照明显微术(Structured Illumination Microscopy,SIM)[3-4]和单分子定位成像技术(Single-Molecule Localization Microscopy,SMLM)[5-7]为基础的超分辨荧光显微成像技术。荧光显微镜的分辨率也从几百纳米提高至几纳米[8-9],其中,单分子定位显微镜也因为其天然的分子级别的定位方式,较高的分辨率,可较为便捷地对细胞结构进行定量分析,成为科研人员研究细胞在分子水平功能的重要工具。自1995年被BETZIG E 等提出后[10],就一直迅速地发展,2017年,BALZAROTTI F 等将SMLM 与STED 结合,提出了MINFLUX 概念[11],实现了约1 nm 的定位精度和6 nm 的成像空间分辨率。
在SMLM 技术的一系列应用中,标记不同的蛋白质以探测其空间关系和相互作用的多色SMLM 成像可以大大增加从样品中提取的信息含量,帮助我们更好地研究和理解不同细胞结构间的相互作用。2013年,XU Ke 等[12]使用多色SMLM 首次揭示了神经细胞轴突部分的周期性结构,这一发现证实了多色单分子定位显微镜在研究亚细胞尺度生命结构与相互作用方面的竞争力。目前已发展出的一系列多色SMLM 技术在可视化真核细胞中的细胞结构[12],研究复杂蛋白质的结构和组成[13],研究病原体与宿主细胞的相互作用[14-15]等方面已经得到了广泛应用。2017年,潘雷霆等[16]详尽地总结归纳了当时主流的六种多色SMLM,并从分色能力、光谱窜扰、数据采集方式等角度分析了各方法的优缺点。5年来,多色单分子技术发展迅速,本文在前文的基础上,从生物样品制备和光学系统改进两个角度分析归纳这六类多色SMLM 在5年来的发展现状与生物应用,以便研究人员根据自身需求选择合适的多色SMLM 开展相应的研究。
1 单分子定位技术的分辨率
若单个荧光分子的点扩散函数(Point-Spread-Function,PSF)不与其它分子重叠,则可以高精度地确定其空间坐标。基于这一条件,SMLM 通过使用一系列可在亮暗态之间切换的染料或荧光蛋白,将衍射极限内的两个相邻分子发出的荧光从时间上分离,从而可以分别定位单个分子,通过计算多个成像周期的数据并叠加重建,最终得到超分辨图像。
单分子定位显微镜图像的分辨率主要有几种评估方式,一种常用的方法是利用定位精度估算图像的分辨率,2002年,THOMPSON R E 等[17]利用最小二乘法对单个艾里斑进行高斯拟合,推导了单个荧光分子的横向定位精度公式为

式中,σloc为定位的精度;a为像素大小;b为背景噪声,N为分子发出的光子数,s是点扩散函数的标准差。
另一种计算定位精度σloc的方法是计算CRLB,CRLB 为任何无偏算法的方差提供了下限,如果点扩散函数为标准差为σ0的高斯函数,忽略了背景噪声和读出噪声,在这种理想情况下,定位精度可以表述[18]为

可以看出,不论使用哪一种方法计算,当单个分子的光子数降低时,定位精度都会相应下降。
目前另外一种常见的计算方法是对连续的生物样品(例如微管)成像,并计算其半高全宽。傅立叶环相关分析[19]也可以用于度量图像的空间分辨率但这种方法需要依赖用户定义的阈值。目前最令人信服的表明图像具有分辨率R或更高的分辨率的证据是在距离≤R时清楚区分不同的结构。如在生物样品中,可以区分已知距离的复合物(例如核孔)上的分子,则表明系统具有相应的分辨率。
2 基于样品制备的多色单分子定位技术
根据样品制备时选取的荧光探针种类及其添加顺序,可以将一些多色SMLM 归纳为基于多种合成染料的多色SMLM 技术,基于激活子和报告子组合的荧光探针对的多色SMLM 技术,基于反复淬灭-标记的多色SMLM 技术三大类。
2.1 基于多种合成染料的多色SMLM 技术
最常用的多色单分子样品制备方法是选用多种不同发光谱段的荧光染料对生物结构分别进行标记,在实验过程中依次使用不同波长的激发光进行特异性激发,从而分别探测各颜色通道的荧光来实现多色成像。使用这种方法,2012年,LÖSCHBERGER A 等[20]使用Atto 520 和AF647 的染料组合,实现了核孔复合物(Nuclear Pore Complex,NPC)及其中央通道的双色图像,观测到了高度对称的NPC 结构。
这种方法原理简单,各颜色通道分时成像,窜扰较小,但需要进行通道间的漂移校正,但为了保证染料分子的特异性激发,无法全部选用红外波段的闪烁性能较好的染料,同时不同的荧光染料需要不同的化学缓冲液条件来保持其闪烁性能最优,将多种荧光染料放在同一样品的化学环境中,限制了荧光团的发光效率,导致部分通道探测到的光子数降低,所获得的多色图像的空间分辨率受到较大影响。
使用光活化荧光蛋白的多色SMLM 受到荧光蛋白在不同光谱区域的亮度和光稳定性的影响。2011年,ENDESFELDER U 等[21]发现mEos2 荧光蛋白在各种成像缓冲条件下均有较好的成像表现,得益于这些耐受成像缓冲液的荧光蛋白的发光特性,将不同发射光谱的荧光蛋白和荧光探针结合使用,可以同时解决成像缓冲液对荧光染料性能的影响及荧光蛋白的选择的问题。通过将mEos2[12]和AF647 结合,实现了细胞中肌动蛋白和微管的双色成像,同年,LEHMANN M[22]将Dronpa 和AF647 相结合,实现了HIV 病毒与宿主之间的定量分析,研究了二者之间的相互作用。2015年,SPAHN C 等[23]以相同的原理,实现了细菌中蛋白质、膜和染色体结构的多色相关成像。
基于多种合成染料的多色SMLM 方法需要多个与荧光染料波段相匹配的激光器作为激发光,同时不同的颜色通道间需要分时依次成像,这导致系统的复杂度增高,同时实验时间长,各通道之间需要进行漂移校正[24]。
2.2 基于荧光探针对的多色SMLM 技术
使用激活子和报告子相结合的荧光分子对的标记方式克服了上一方法的劣势。这项技术的关键在于合成一对由激活子和报告子组成的探针对,其中报告子可在亮态和暗态之间进行多次切换,激活子则可对报告子实现光谱选择性激活,通过选择具有不同激活波长的激活子和报告子进行组合配对可合成具有不同激活光谱的荧光探针。在实验过程中依次切换不同波段的激活光,再由相同的激发光探测同一个报告子发出的荧光,从而实现多色成像。
基于不同荧光探针对的多色SMLM 方法的关键优势在于检测的为相同报告子发出的同波段的荧光,可以将不同颜色通道间的色差最小化,且一次成像就可获得所有颜色通道的数据,不需要专门校正通道间的位移。同时,可以选择Cy5、AF647 等亮度高,闪烁性能更好的报告子,保证单分子系统的空间分辨率。
这种方法在2007年由BATES M 等[25]提出,使用AF405-Cy5,Cy2-Cy5 和Cy3-Cy5 三种荧光探针对,结合了三个不同的激活波长对DNA 模型样品进行三色成像,同时使用Cy2-AF647 和Cy3-AF647 探针对实现了20~30 nm 分辨率的哺乳动物细胞双色成像。2010年,DANI A 等[26]使用相同的激活子搭配AF647 作为报告子实现了对突触蛋白分布的纳米精度测量。2012年,LUBECK E 等[27]使用AF405,AF488 和Cy3 用作激活子,Cy5,AF680 和AF750 作报告子的荧光探针对标记了酵母细胞中的mRNA,测量了32 个基因的mRNA 水平,同时实现了7 色成像。
但在这个方法中,激发光本身也会导致报告子的非特异性激活,这致使不同颜色通道之间的窜扰较高(约30%)[25],导致在处理数据时需要舍弃一些定位数据,不利于其在定量多色单分子显微镜中的进一步应用。
2.3 基于反复淬灭-标记的多色SMLM 技术
为在简化光学系统的同时降低通道间的窜扰,2014年,TAM J 等[28]从样品制备的角度出发,提出了基于荧光淬灭技术的多色SMLM,通过在不同目标蛋白标记上不同的一抗,并分批次对其进行二抗标记成像及淬灭,实现了五色单分子成像。这种方法仅需一个激发光源和一个激活光源即可实现多色成像,克服了不同荧光团的成像缓冲液的不兼容以及各颜色通道间的色差影响空间分辨率的问题,同时颜色通道数仅与一抗种类有关,可较简单地实现4 色以上的多色成像。但由于需要在成像的同时对生物样品进行操作,实验过程复杂,且成像时间过长,需要对各通道间的颜色通道进行漂移校正,限制了这种方法的适用范围。
基于与上述方法相似的原理,同年,JUNGMANN R 等[29]开发了一种基于DNA-PAINT 的顺序多路复用方法Exchange-PAINT,其原理如图1 所示,该方法利用具有同种颜色但不同核苷酸链序列的成像链与不同对接链结合进行多色成像。在具体的实验过程中先使用具有不同核苷酸序列的对接链标记靶蛋白,再加入成像链P1*,该成像链与对接链P1 互补,可仅获得用P1 标记的靶蛋白的DNA-PAINT 图像,采集完成后洗涤去取成像链P1*,并引入成像链P2*,获得另一组DNA-PAINT 图像,重复洗涤和成像步骤,将采集到的图像对齐并组合从而实现多色成像。使用这种方法,在细胞样品中实现了对微管、线粒体、高尔基体和过氧化物酶的四色成像及DNA 折纸结构上获得了第一个十色的体外超分辨率图像。与TAM J 等[27]的方法相比,Exchange-PAINT 同样具有仅需单个激光源,使用单个染料各通道间无色差影响的优势。同时利用DNA-PAINT 成像链与对接链仅短时间结合的特点,Exchange-PAINT 在洗涤时不涉及标记和擦除的步骤,因此,各成像周期之间仅间隔1~2 min,这种实验流程的简化带来了更快的图像采集速度,这使得其拥有更大的应用范围。这种方法的多色能力取决于对接链DNA 序列的数量,这也使得其可以研究更为复杂的生物分子系统,2019年,WADE O K 等[30]结合此方法中顺序发光的各颜色通道的闪烁频率,实现了DNA 折纸结构上的124 色成像。
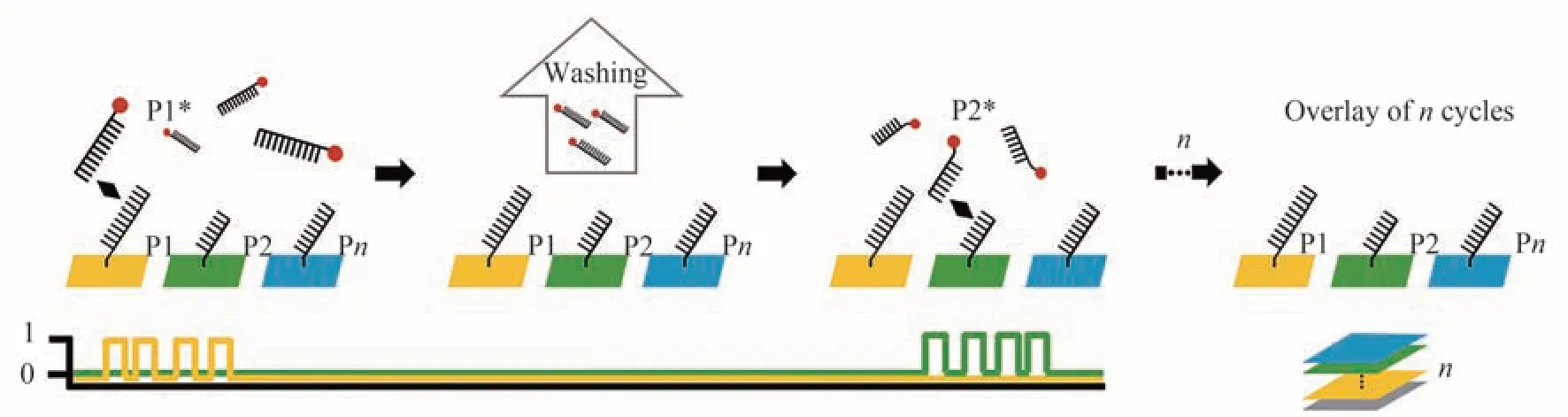
图1 Exchange-PAINT 的成像原理示意图[29]Fig.1 Schematic diagram of the imaging principle of Exchange-PAINT[29]
3 基于光谱信息处理的多色单分子定位技术
除了上述在样品方面实施的方法外,还可以通过在光学系统中添加一系列光学元件,根据荧光团的光谱信息针对性的对不同波长的颜色通道进行处理,并通过后期数据计算实现多色成像。基于光学方法实现的多色SMLM 主要分为基于分光技术的比例成像方法、基于色散的光谱分析方法和基于点扩散函数工程技术的方法三大类。
3.1 基于分光技术的比例成像方法
比例成像的具体原理是具有相近发射光谱的荧光染料在经过同一个激发光照射后,所发出的荧光被后续光路中的二向色镜分为透射光和反射光两路,这两路光再分别被透镜聚焦到同一个像面的不同位置成像。依据每个分子在两个数据通道间的光强比对每个荧光点进行颜色识别,从而获得多色图像。
由于比例成像方法可以使用光谱相近的荧光染料,避免了不同荧光团对成像缓冲液的需求冲突,同时可以自主选择闪烁能力最好的红外波段的荧光染料,仅使用一个激发光源就可以同时获得多个颜色通道的数据。但这也导致所选用的荧光探针间的发射谱发生交叠,由于染料受激发射荧光时会引入一定的泊松噪声,从而导致一些单分子荧光点的两通道间光强比处于无法被识别的中间区域,这导致在后期数据处理时丢掉一些数据点,并且造成了各颜色通道间的窜扰。除此之外,由于二色镜分光导致不同通道间的光谱组成存在差异,这使得各通道间存在随横向位置变化的倍率色差,无法将两路通道的数据进行叠加用于定位,这导致每种荧光染料都仅能选择一个通道的光子用于定位,而光子数直接影响了单分子系统的定位精度[17],这是基于比例成像的多色SMLM 方法的一大缺陷。
基于比例成像的多色SMLM 方法于2008年由BOSSI M 等[32]提出,使用SRA552 和SRA577 两种染料,对哺乳动物的PtK2 细胞中的微管和角蛋白网络进行成像,实现了定位精度为15 nm 的多色成像。2010年,TESTA I 等[33]使用比例成像的方法首次在固定的PtK2 细胞中实现了四色成像,并在活细胞中实现了双色单分子成像。2012年,LAMPE A 等[34]基于相同的原理,使用AF647 和AF700 两种近红外波段的荧光染料实现了双色成像,克服了以往使用的荧光染料所需的最佳缓冲液条件不同的影响。2015年,WINTERFLOOD C. M 等[35]将比例成像的方法与双平面的3D 成像方法相结合,对海马神经元的轴突结构实现了3D 双色成像,观测到了其周期性结构。
在这一研究的基础上,ZHANG Yongdeng 等[36]在2020年对4pi-SMLM 系统进行了改进,提出了一种“拯救荧光”的4π 单点切换超分辨率显微镜,通过在4π 系统的一路探测光路中额外添加一片二向色镜并使用另一台相机收集此二色镜反射的荧光,通过将此通道的荧光光强与原本4π 探测路中的相机接收到的光强做比,可以确定单分子荧光团的颜色。这种方法通道间的窜扰较低,同时通过二向色镜反射收集的通道光强较低,分光对光子数的影响较小,降低了分光对定位精度的影响,通过这种方法实现了对高尔基体,内质网等生物样品的5~10 nm 精度的多色成像。
2020年,VISSA A 等[37]用可调节角度的薄膜滤光片代替传统方法中的二向色镜在光谱上分离远红外波段的荧光团,在不同角度下,薄膜滤光片具有不同波长的通带,由于这种调整具有连续的梯度,可以实现精细的调节,可以针对当前实验所使用的荧光染料选择最佳窗口,使通道间具有最低的窜扰。通过对线粒体蛋白TOM20 和过氧化物酶体蛋白PMP70 之间相互作用的双色超分辨成像证明了这种方法的实用性。
2021年,WANG Yujie 等[38]将分色这一思路扩展到了CCD 像面上,通过在CCD 前添加按特定规则排列的滤光片阵列,一次成像得到成像不同发射光谱的像素,再通过不同滤光片下像素的光强占比确定分子颜色,从而得到多色图像。这一方法简化了实验系统,在现有的单色单分子定位显微系统中将相机替换为上述特制的CCD,即可实现多色成像。但同时由于只有未经过滤的像素所采集到的数据被用来定位,即仅有一半的光子用于定位成像,所以这一方法的定位精度也会相应下降。
2022年,CHEN Jianwei等[39]将4π 系统原本的四个探测光路分为具有π 相位差的两组通道,并插入滤光片在两个通道间创造光强差,通过荧光团在两个通道的光子数的强度差来确定其颜色信息。这种方法利用了4π系统原本的光路设置,不添加额外的检测通道,具有较高的光子收集效率,因此可实现更高的定位精度。
LI Yiming 等于2022年提出了globLoc 算法[40],解决了比例成像应用于单分子定位显微镜时,由于分光致使的定位精度降低的问题,通过计算确定多通道间的映射关系,进一步生成多通道的PSF 模型,并对多通道的单分子数据进行处理。使用AF647、DY634、CF660C 和CF680 四种染料,对单个NPC 中的NUP96,NUP62,ELYS 和WGA 进行标记并成像,同时对突触复合物进行了三色成像,见图2,可以在很低窜扰的情况下清晰分辨,三个组件的空间布置与先前的研究非常吻合,证实了该方法的应用前景。

图2 应用globLoc 算法在比例成像中对NPC 进行成像,使用AF647、DY634、CF660C 和CF680 分别对NUP96,NUP62,ELYS 和WGA 进行标记[40]Fig.2 Performance of globLoc on ratiometric multi-color data. 4 color 3D imaging of Nup62-DY634,Nup96-AF647,ELYS-CF660C and WGA-CF680 in the NPC[40]
3.2 基于色散的光谱分析方法
基于光谱技术的多色随机光学重建显微技术(Spectrally Resolved STORM,SR-STORM)主要是通过在探测通道中添加三棱镜[41]、光栅[42]等色散元件,将荧光分为两路,其中聚焦的一路光强较强,用于分子三维位置的计算,另一路荧光信号则由色散元件进行空间光谱展开,用于获取荧光分子的光谱信息,进行颜色识别,两路荧光信息相结合实现多色超分辨成像,这种单分子多色成像方法同时也广泛应用于单分子动力学和单分子极性研究领域[43]。
基于光谱技术的多色SMLM 因为选用的荧光染料之间的光谱较为接近,可使用同一套激发光源与激活光源,通过一次成像就可同时获得多色超分辨图像。同时由于可获得荧光分子的光谱信息,对荧光分子的种类进行精确识别,不同颜色通道间的窜扰很低。但同样由于分光减少了用于定位的荧光光子数,一定程度上影响了该方法的空间分辨率。除此之外,单个分子的PSF 在光谱展开后,在像面上会占据更大的面积,在成像过程中更容易在单帧图像中发生重叠,限制了其在高密度生物样品中的应用。
根据使用的色散元件的不同,基于光谱的SMLM 方法可大致分为使用棱镜进行分光和使用光栅进行分光两大类。
使用棱镜分光的多色SMLM 方法在2015年由ZHANG Zhengyang 等[41]首次提出,该系统通过在双物镜系统的一路中添加三棱镜进行分光,选用Dy634,DL650,CF660C 和CF680 共4种荧光染料,对PtK2 细胞中过氧化物酶体,波形蛋白丝,微管和线粒体外膜进行成像,实现了10 nm 光谱分辨率的低窜扰3D 四色成像。2017年,同组研究人员[44]通过分束镜将收集到的荧光信号拆分为两个光路,证明了这一原理在单物镜系统中实现活细胞实时成像的可行性。MLODZIANOSKI M J 等[45]将棱镜应用于PALM 中,同时测量单个荧光蛋白分子的空间位置和发射光谱,量化了单个荧光蛋白分子的发射光谱随时间的波动变化,见图3。2018年,HUANG Tao 等[46]将这种方法应用在单分子追踪中,使用单个激光激发,实现了实时低窜扰的三色单分子追踪,在获得10~15 nm 的光谱分辨率的同时,实现了20~40 nm 的空间分辨率。

图3 使用光栅进行色散对微管和线粒体进行双色成像[42]Fig.3 Dual-color imaging of microtubules and mitochondria using grating[42]
使用衍射光栅代替棱镜对探测荧光的PSF 进行色散的光谱光子定位显微镜(spectroscopic photon localization microscopy,SPLM)在2016年由DONG Biqin 等[42]首先实现,如图4(a)所示,该系统利用反射光栅进行分光,区分出了光谱上相距15 nm 的两种荧光分子,在Cos7 细胞中实现了对微管和线粒体进行了双色同步成像。随后,ZHANG Yang 等[47]利用透射光栅将探测荧光分成零级和一级光,使用AF647、CF660C和CF680 共三种染料分别对COS-7 细胞中的ATPB、微管蛋白和TOM20 蛋白进行标记,实现了低误差识别率(5%)的三色单分子成像。在随后的研究中,同一研究小组于2021年[48]使用基于衍射光栅的SRSTROM 系统,实现了角膜内皮细胞中的β-微管蛋白和组蛋白-H4 的双色成像,空间分辨率为20 nm,和基于棱镜的SR-STROM 相当。
在对分子进行3D 定位时,对于使用光栅分光的SR-STORM 系统,引入像散会使荧光分子的PSF 形状同时受到光谱信息和分子轴向位置的影响,所以无法像使用棱镜的SR-STROM 一样通过在定位光路中简单添加柱透镜的方式来实现3D 成像。为解决这一问题,SONG K H 等[49]使用双平面的方法从已经建立的空间和光谱成像通道中获得了轴向位置信息,使用AF647 和CF660C 对COS-7 细胞中的线粒体和微管进行标记,实现了三维双色单分子成像。
2021年,KIM G 等[50]将基于光栅的SR-STROM 与基于棱镜的SR-STROM 进行了比较。基于棱镜的SR-STROM 由于需要两路光进行严格的光学对准,在装置上更为复杂,同时使用棱镜会使荧光需要通过多个光学元件,导致荧光信号的衰减,从而导致系统空间分辨率的下降。KIM G 等还研究了这种光学损耗及光栅本身相对较高的传输损耗对定位精度的影响,发现光栅(26 nm)可提供稍高于棱镜(30 nm)的空间分辨能力。针对上述光栅的高传输损耗以及对光学对准的严格要求带来的限制,2022年,SONG K H 等[51]通过设计特殊的双楔棱镜制作了单片成像光谱仪,如图4(b)所示,并将其应用于光谱展开的单分子定位显微系统中,实现了对微管和线粒体的3D 双色成像。
除此之外,在基于光栅的SR-STROM 和基于棱镜的单目标的SR-STROM 方法中,空间和光谱分析都仅使用总光子的一部分,从而导致两个通道中的精度降低。研究人员通过开发更明亮的荧光团或者提高系统的光子检测效率的方法来解决这一问题。DANYLCHUK D I 等[52]提出了一种基于尼罗红分子的新型探针,通过引入低亲和力的膜粘合剂来提高亮度和改善其特异性。CHUNG J 等[53]通过优化成像缓冲液来增加荧光团的光子数。根据其最新研究,可以通过改变成像缓冲液的组成如硫醇试剂的类型和浓度,折射率和pH 值等来改变荧光团的光子数量,实现增加探测到的光子数的目的。
为了提高光子检测效率,SONG K H 等[54]提出了一种对称色散的SR-STROM 方法(Symmetrically Dispersed sSMLM,SDsSMLM),其原理图如图4(c)所示,同时收集荧光在通过衍射光栅后的-1 级和+1 级衍射光,用这两个对称分散的光谱通道代替传统的一个空间通道和一个光谱通道的分光方案,从而完全利用来自单个分子的所有光子,实现了对固定细胞的多色成像,将空间和光谱精度提高了42%和10%,在1 000个光子预算的条件下实现了10 nm 的空间分辨率和0.8 nm 的光谱精度。
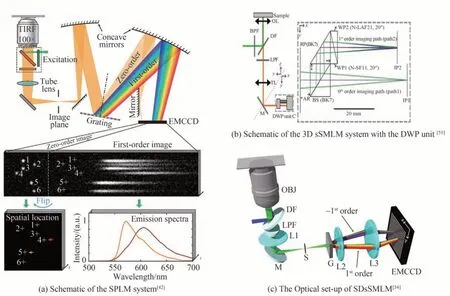
图4 SR-STROM 的光学装置示意图Fi g.4 The optical set-up of SR-STROMs
3.3 基于点扩散函数工程技术的方法
点扩散函数工程[55](Point-Spread-Function Engineering ,PSFE)是指使用柱透镜、空间光调制器等器件对探测荧光进行编码的方法,将PSFE 与SMLM 相结合可以使得PSF 形状随分子的轴向位置变化,从而携带深度信息。通过PSF 的形状,可以得到荧光分子的三维位置信息。
这种方法适合于低密度的荧光分子的跟踪,同时通道间的窜扰较低,但受限于单分子定位显微镜的成像原理,要精确定位荧光分子的位置,需要每帧图像中的荧光分子保持稀疏性,而在荧光分子密度较高时,编码后形状较大的PSF 会在成像面发生交叠,因此在生物样品的成像方面应用受限。
2016年,SHECHTMAN Y 等[56]提出利用一种特殊设计的相位板,在对深度信息进行编码的同时直接对光谱信息进行编码,同时为不同的波长产生不同的PSF,从而在不分光的条件下实现了同时对双色荧光微球进行单分子追踪及对生物细胞的双色超分辨成像。2017年,利用上述的超分辨显微系统,实现了对酿酒酵母的三维双色活细胞单分子追踪[57],分析了围绕GAL 位点的两个荧光标记的DNA 位点的动力学原理,证明了此方法在研究生物学环境中染色质结构的构象和动态方面的潜力。为进一步提高基于点扩展函数工程技术的多色SMLM 的成像效果,SIEMONS M 等[58]使用空间光调制器对光学系统的像差进行了校正,实现了远低于衍射极限的高精度波前控制,为更好地应用复杂的PSFE 创造了条件。
近年来,神经网络已被广泛应用于超分辨显微镜领域[59],而单分子定位显微镜因为具有发射器相对稀疏的特性,降低了机器学习通常需要的大量训练数据的要求,得到了迅速发展,目前已经提出了多种机器学习与PSFE 相结合的多色单分子成像方法。2019年,KIM T 等[60]利用神经网络从单个分子的PSF 图像中提取了单分子颜色,在常规定位显微镜的同一通道中对微管和线粒体实现了同时双色成像,这种方法不必修改PSF 形状,降低了对生物样品的要求。同年,HERSHKO E 等[61]将深度学习应用于单分子定位显微镜,利用神经网络将PSF 的调制过程与单分子图像的颜色识别相结合,可同时区分单分子图像中的四个颜色通道。NEHME E 等通过神经网络为高密度发射器的情况优化了最佳的编码PSF[62]及对双通道光学系统的多个PSF 进行了端到端学习的方法[63],分别实现了4 μm 深度的单分子成像和活细胞内的快速单分子追踪,这两种方法都在一定程度上降低了PSF 编码导致的扩大的PSF 在荧光分子密度较高时发生交叠所带来的影响。2021年,OPATOVSKI N 等[64]利用二色镜将荧光分为三个光谱通道,分别在瞳面上放置相位板,从而产生通道特异性的PSF,再将它们重新组合到摄像机的同一区域中,实现了在110×70×4 μm3的大视场情况下荧光纳米颗粒的五色单分子追踪及活细胞中荧光标记DNA 的3D 追踪,见图5。

图5 基于PSFE 的三色SMLM 的光学系统和瞳孔平面上的相位掩模[64]Fig.5 The optical system and Representation of the phase masks placed at the pupil plane[64]
以上介绍了目前流行的几种多色SMLM 技术,这几种多色SMLM 的实现原理各异,在空间分辨率、光谱分辨率、成像时间、通道间窜扰和适用的样品范围等方面各有优缺点,都在生物医学领域有所应用,对目前已发展出的多色单分子定位技术的优势做总结,并将其进行了比较,如图6 所示。

图6 多色单分子定位技术的对比Fig.6 Comparison of different Multicolor SMLM
这些方法尤其是基于光学改进的方法,所使用的染料对多为常用的几种性能较好的近红外染料,表1 列举了每种多色SMLM 方法中具有代表性的染料组合及其相关使用场景。
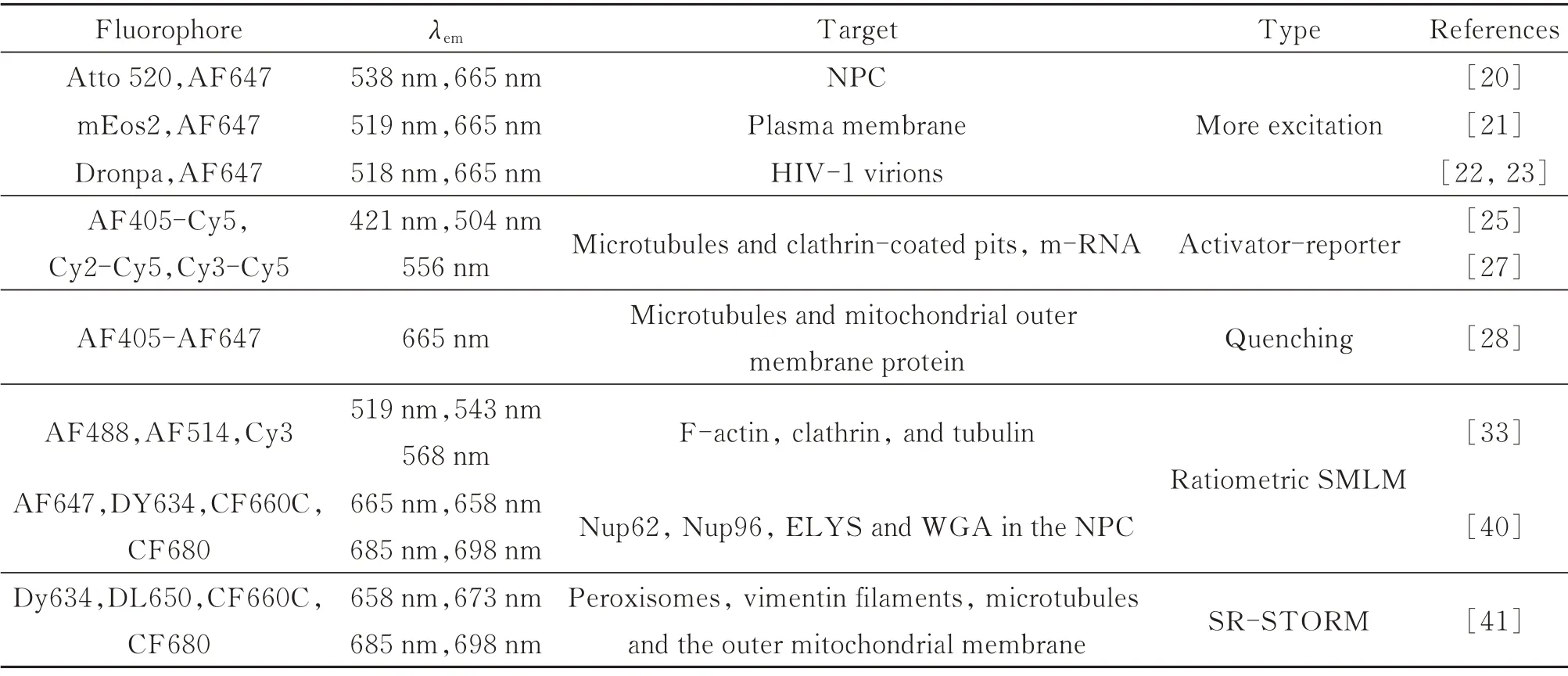
表1 不同多色SMLM 方法所使用的染料组合Table 1 Dye combinations used in different multicolor SMLM
4 结论
本文从生物样品制备和光学系统改进两个角度,追踪了近5年来提升多色单分子定位显微技术性能的各类方法的最新进展,比较了其适用的生物场景。多色单分子定位显微镜是研究细胞间相互作用的重要工具,其未来的发展可以聚焦于以下几方面:1)进一步提高染料分子的抗漂白特性,能否通过对荧光探针和缓冲液的研究,如改变氧气或硫醇的浓度等缓冲液的配比来获得更长的状态寿命和更高的光子产量,进而在不增加成像时长的同时提高定位精度。2)快速成像,多色SMLM 的基本原理是通过时间换取空间的策略分离PSF,这也导致了SMLM 需要较长的成像时间,在配合高帧率的sCMOS 相机和高激发光光强(>50 kW·cm-2)的情况下对超过10 000 个的哺乳动物细胞进行双色三维成像需要26 h[65],提高成像速度的一个方法是提高激活概率,但这同时也会带来PSF 重叠进而影响定位结果的问题。目前已经开发了一系列针对这一问题的算法[66],通过连续图像之间的计算差异或者计算荧光波动的时间和空间相关性[67]来产生超分辨率图像,但无论数据分析的改进如何,PSF 重叠导致的分辨率下降仍普遍存在,进一步开发适用于重叠PSF 定位的算法,提高成像速度,对扩展多色SMLM 的生物应用范围十分重要。3)活细胞成像,在研究活细胞中结构动力学方面,多色SMLM 收到较大的限制,快速成像所需的高激光功率带来的光漂白和光毒性使目前的研究主要局限于相对缓慢移动的结构,而选取合适的透膜染料或荧光蛋白又进一步的限制了多色SMLM 成像对生物样品的空间关系和相互作用的研究。

