鸭蛋质量相关遗传变异及功能基因筛选与鉴定
陶志云 朱春红 刘宏祥 施祖灏 章双杰 徐文娟 宋卫涛 王志成 李慧芳


摘要: 本研究根据金定鸭个体蛋质量情况,选择2种极端表型,分为高蛋质量组(WH)和低蛋质量组(WL)。基于混池全基因组重测序和选择清除分析技术筛选组间差异显著基因组区域内的单核苷酸多态性(SNP)位点及相关功能基因,并通过单个样本重测序对筛选的蛋质量相关SNP进行验证,分析各SNP位点不同基因型之间的蛋质量大小差异。研究发现,在低蛋质量组和高蛋质量组获得的高质量reads数量分别为194 115 424和228 089 084,共定位到SNP差异显著区间178个,共富集到受选择候选基因40个,而且这些区间和基因均位于Z号染色体。鉴定出候选基因ARSB上Z-22908831、Z-22966695突变位点显著影响300 d、450 d蛋质量,RORB上Z-35251072、Z-35256947突变位点显著影响450 d蛋质量,RORB上Z-35278196突变位点显著影响300 d、450 d蛋质量,RASEF上Z-39588570突变位点显著影响450 d蛋质量。本研究结果为通过分子育种技术提高蛋鸭产蛋性能选育提供了依据,加快了选育进程。
关键词: 鸭;蛋质量;遗傳变异;功能基因
中图分类号: S834 文献标识码: A 文章编号: 1000-4440(2023)02-0461-10
Screening and identification of genetic variation and functional genes related to egg weight in ducks
TAO Zhi-yun1, ZHU Chun-hong1, LIU Hong-xiang1, SHI Zu-hao2, ZHANG Shuang-jie1, XU Wen-juan1, SONG Wei-tao1, WANG Zhi-cheng1, LI Hui-fang1
(1.Jiangsu Institute of Poultry Sciences, Yangzhou 225125, China;2.Pony Testing Group Jiangsu Co., Ltd., Suzhou 215123, China)
Abstract: In the study, two extreme phenotypes of Jinding ducks were selected according to individual egg weight: a high-egg-weight group (WH) and a low-egg-weight group (WL). Single nucleotide polymorphism (SNP) sites and related functional genes that located in regions with significant genomic differences between the groups were screened based on mixed pool whole-genome re-sequencing and selective clearance analysis technology. The screened SNPs related to egg weight were verified by re-sequencing of each sample, and differences in egg weight between different genotypes of each SNP site were analyzed. Totals of 194 115 424 and 228 089 084 high-quality reads were obtained in the WL and WH groups, respectively. There were 178 significantly different SNP fragment regions, and 40 candidate genes were selected. These intervals and genes were located on chromosome Z. Z-22908831 and Z-22966695 mutation sites in the candidate gene ARSB significantly affected the egg weight of 300-day-old and 450-day-old ducks. Z-35251072 and Z-35256947 mutation sites in RORB significantly affected the egg weight of 450-day-old ducks. Z-35278196 mutation site in RORB significantly affected the egg weight of 300-day-old and 450-day-old ducks, and Z-39588570 mutation site in RASEF significantly affected the egg weight of 450-day-old ducks. These results provide a molecular basis for improving egg-laying performance and accelerating the breeding process in ducks.
Key words: duck;egg weight;genetic variation;functional gene
蛋鸭的产蛋性能,包括开产日龄、平均蛋质量、产蛋数、蛋品质等,是衡量其经济价值的主要指标[1-2]。通过生物学方法,筛选影响蛋鸭产蛋性能的候选基因,将分子育种相关技术用于蛋鸭育种,是提高蛋鸭产蛋性能的有效手段[1-2]。全基因组重测序技术是对基因组序列已知的个体进行全基因组测序,并在个体或群体水平上进行差异性分析的方法,可挖掘出大量的遗传变异位点[3],进而对挖掘的变异位点进行定位和鉴定,发现影响性状的重要基因及其功能,从而阐释性状差异的原因[4]。随着测序成本的不断降低,该技术已成为育种研究领域中快捷、有效的方法之一,在畜禽育种研究中也得到了广泛应用[5]。目前,利用全基因组重测序技术对鸡产蛋性能进行研究已获得较大进展[6]。如,Liu等[7]发现13号染色体ODZ2基因上1个单核苷酸多态性(SNP)与开产日龄显著相关,7号染色体GRB14基因上1个SNP与产蛋数相关;Shen等[8]发现5号染色体GARNL1基因上5个SNP与开产日龄相关。Fan等[9]发现9个与开产日龄及体质量显著相关的SNP,4个与产蛋数显著相关的SNP,5个与蛋质量相关的SNP。全基因组重测序技术用于监测鸭产蛋性能的相关研究较少,在绍兴鸭中发现了10个与产蛋性能显著关联的SNP,其中4个位于2号染色体的SNP与开产日龄相关,4个位于2号染色体和2个位于29号染色体的SNP与66周龄产蛋数显著关联[1]。蛋质量相关SNP的研究更是非常有限。本研究拟利用全基因组混池重测序和选择清除分析技术,分析蛋质量差异大的鸭群体,以期筛选、鉴定出鸭蛋质量相关差异基因组区域内的遗传变异位点和功能基因,为通过分子育种技术提高蛋鸭产蛋性能提供依据,加快育种进程。
1 材料与方法
1.1 试验材料
试验鸭为金定鸭,来源于国家家养动物种质资源库,共500只,按照蛋鸭饲养标准饲养,育成期结束后上笼饲养,在299 d、300 d、301 d、449 d、450 d和451 d时分别称量蛋质量,统计个体300 d和450 d的平均蛋质量。
1.2 个体选择和样品采集
根据个体300日龄时蛋质量情况分别选取蛋质量高、蛋质量低2种极端表型个体各30只,经统计,高蛋质量组(WH)的平均蛋质量为(86.09±2.22) g,低蛋质量组(WL)的平均蛋质量为(64.43±1.68) g,差异极显著(P<0.01)。分别采集2组个体的抗凝血,置于-20 ℃冰箱,用于DNA提取。
1.3 DNA提取、文库构建及测序
基因组DNA的提取采用十六烷基三甲基溴化铵(CTAB)法,提取后用NanoDrop2000分光光度计检测其纯度和质量浓度。采用NEB建库试剂盒进行建库,再用仪器qPCR-ABI7500、Agilent2100对文库进行定量和检测。库检合格后,在Hiseq X10 PE150平台进行双末端(PE)测序。最后去除片段低于10 bp的低质量reads以及一些接头被污染的reads,获得clean reads。
1.4 序列比对、基因变异的检测
利用比对软件BWA将获得的clean reads数据比对到参考基因组,利用软件Picard对结果进行排序并标记重复序列。用Samtols软件将比对结果转换成Mpileup格式,转换过程中,将碱基质量值小于20和质量值小于20的碱基去除。转换成Mpileup格式后,为避免插入/缺失(InDel)引起的SNP簇对计算结果的影响,将InDel及InDel附近5 bp内的变异位点去除。
1.5 选择清除分析
基于过滤后的Mpileup文件,使用Popoolation软件计算单个混池内的π值,将2个池的π值相除,获得π-ratio指标(piRatio)。将2个混池的Mpileup格式转换为Sync格式(Popoolation2专用格式)后,用Popoolation2软件进行混池间的固定指数(Fst)计算。
1.6 SNP准确性检验
基于Illumina X-10测序平台对筛选获得的遗传变异进行单样本的个体测序验证。在差异显著的基因组区间内随机选择12个差异显著的SNP,扩增出2个混池中60个个体的目标位点片段,统计每个个体12个位点的基因分型情况。
1.7 不同基因型鸭蛋质量差异分析
采用SPSS20.0软件对在WH、WL组间基因组差异显著区域内随机选择的12个多态位点基因型鸭300 d、450 d時蛋质量进行One-way ANOVA分析,比较各位点不同基因型间的蛋质量差异。
2 结果与分析
2.1 测序结果概述
2.1.1 全基因重测序和组装 表1显示,WL、WH 2个混池得到的reads数分别为195 579 134和229 696 180,过滤后得到的高质量reads数分别为194 115 424和228 089 084,过滤率均超过99%。2个池比对到基因组上的reads数分别为186 063 404和218 730 593,比对率超过95%,2个池分别产生6 652 356个和6 731 941个SNP。
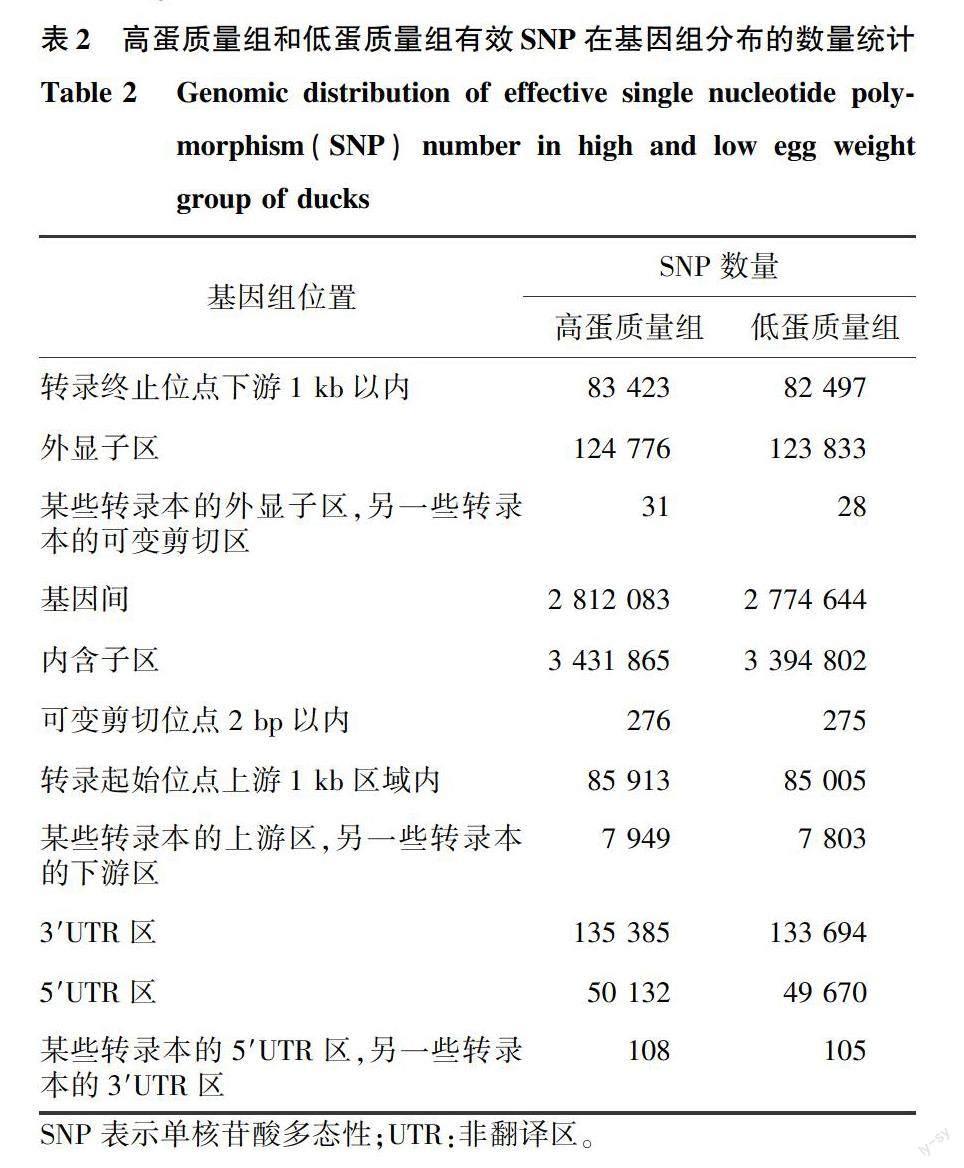
2.1.2 SNP在基因组中的分布 表2显示,在WH和WL中均获得了大量的SNP,其中在外显子区的SNP数量分别为124 776和123 833,内含子区的SNP数量分别为3 431 865和3 394 802。
2.1.3 SNP编码信息统计 对WH和WL的鸭进行DNA测序,统计分析比对后获得的SNP编码信息情况,结果(表3)表明,2组获得的非同义突变数分别为29 237和29 089,同义突变数分别为87 178和86 418。
2.2 鸭高蛋质量组和低蛋质量组的选择清除分析
通过群体多样性差异指标分析WL和WH 2个群体SNP在染色体上的多样性差异情况,2个群体多样性差异主要分布在1号、2号、3号、4号、5号及Z号染色体(图1A)。由WL和WH 2个群体固定指数值分布的曼哈顿图(图1B)可知,2个群体遗传分化相对严重的区域集中位于Z号染色体。
结合固定指数、多样性差异倍数的结果,各自按0.01水平筛选显著区域,交集部分为可靠的候选区间,共定位到178个SNP显著差异区间(图1C),这些区间均位于Z号染色体,对2个指标筛选出的显著区间,提取出各自区间的基因,并用韦恩图将2个区间基因的交集和并集情况进行展示,共富集到40个受选择候选基因(图1D)。
2.3 鸭高蛋质量组和低蛋质量组的受选择基因
通过Fst和π-Ratio指标确定的40个受选择基因情况见表4,其中包括与跨膜转运、物质转运和代谢相关的Slc49a3、TRPM6、Sema4d、RASEF;与发育相关的NIPBL、ZFAND5等,有多个基因功能未知。
2.4 测序结果准确性验证
使用Illumina X-10测序平台对60个样本12个SNP位点进行单样本的个体重测序,以验证等位基因频率准确性,结果(表5)表明,全基因组混池重测序和单个样本重测序所得等位基因频率的一致性为0.833。
2.5 候选基因GO富集分析
将获得的差异基因向GO数据库的各条目映射,计算每个条目的基因数并分类统计,图2显示,在0.01水平,这些差异基因在生物过程、细胞组分、分子功能中均有涉及。在生物过程方面,以细胞过程、单生物体过程、生物过程调节、生物调节、代谢过程几个条目中涉及基因较多,数量分别为14、13、12、12、11;在细胞组分方面,以细胞器、细胞、细胞部分几个条目中涉及基因较多,数量分别为12、11、11;在分子功能方面,以结合、催化活性2个条目中涉及基因较多,数量分别为14和4。
2.6 候选基因KEGG分析
进行通路显著性富集分析,前20个显著富集的KEGG通路见图3,其中代谢通路信号转导途径中富集的基因最多,甘氨酸、丝氨酸和苏氨酸代谢途径次之,富集到代谢通路的基因包括Gda、ACO1、Bhmt、DMGDH、MAN2A1、ARSB。
2.7 12个突变位点不同基因型鸭蛋质量差异分析
将筛选获得的12个突变位点不同基因型鸭个体蛋质量数据进行差异分析,结果(表6)表明,ARSB基因上突变位點Z-22908831的G→A的突变引起300 d、450 d蛋质量显著下降(P<0.05);ARSB基因上突变位点Z-22966695的A→G的突变和RORB基因上突变位点Z-35278196的T→C的突变引起鸭300 d和450 d蛋质量显著增加(P<0.05);RORB基因上突变位点Z-35251072的C→T突变以及Z-35256947的T→C的突变引起鸭450 d蛋质量显著增加;RASEF基因上突变位点Z-39588570的A→G突变引起450 d蛋质量显著增加(P<0.05)。
3 讨论
全基因组关联(GWAS)技术被广泛用于遗传变异的发现和新基因的挖掘,且取得了较大进展。本研究利用全基因组重测序技术对金定鸭WL和WH 2个不同蛋质量组进行混池重测序,获得的高质量reads数分别为194 115 424和228 089 084,比对到基因组的比对率均高于95%,获得的SNP数量分别为6 652 356和6 731 941,说明本研究的测序质量较高,可用于后续差异分析。采用选择清除分析共定位到SNP差异显著的基因组区间为178个,受选择候选基因为40个,这些差异基因组区间和基因均位于Z号染色体,说明Z号染色体与鸭蛋质量密切相关。在受选择区间内选择12个突变位点进行个体测序验证,结果与全基因组混池重测序的一致性为83.33%,说明全基因组混池结果可靠,可用于进一步数据分析。
有研究结果表明,影响蛋质量的因素很多,其中遗传因素是重要的影响因素之一[10],蛋质量的遗传力较高,可达到0.450~0.550[11],山麻鸭的300日龄蛋质量遗传力高达0.614[12],因此,可通过选育改变蛋质量。蛋质量大小受到多个基因控制[11],禽类中与蛋质量相关的基因有PRL[13]、PRLR[14]、GHR[15]、GnIH[16]、OVR[17]、VIPR-1[18]等。最近在鸡的研究中发现了一些新的候选基因,包括PRKAR2B、HMGA2、LEMD3、GRIP1、EHBP1、MAP3K7和MYH[19],在鸭的研究中也陆续发现一些新的与蛋质量相关的基因,如COLX[20]、PNRC[21]、CA2[22]。本研究在鸭蛋质量差异较大的2个群体中,发现40个与300 d、450 d蛋质量相关的受选择基因,主要包括参与跨膜转运的Slc49a3、RASEF、TRPM6,参与DNA损伤修复的GADD45G,尿镁钙排泄相关的NMRK1,具有催化活性的Gda、Bhmt和DMGDH,具有水解作用的MAN2A1、LPL、ARSB,与发育调节相关的NIPBL、ZFAND5,与微管蛋白结合活性相关的MAP1B、TPPP2,以及与昼夜节律相关的RORB等,这些基因均是在鸭上新发现的与蛋质量相关的候选基因。
对获得的受选择基因进行GO富集分析和KEGG分析,GO分析结果表明,在生物进程分类中,受选择基因主要涉及单生物体过程、细胞过程、生物调节、生物过程调节;细胞组分分类中,受选择基因主要涉及细胞、细胞部分和细胞器;在分子功能分类中,受选择基因主要涉及结合、催化活性。KEGG分析结果表明,受选择基因以代谢通路信号途径中参与的基因最多,包括Gda、ACO1、Bhmt、DMGDH、MAN2A1和ARSB,推测这些基因可能是通过参与物质代谢过程调节蛋质量大小。
RORB是一种孤儿核受体,与维甲酸、甲状腺激素受体相关,主要在大脑皮质、丘脑中表达,在视交叉上核、松果体和视网膜中也有表达,而且在松果体和视网膜中mRNA的丰度随昼夜节律振荡,在夜间达到峰值[23]。RORB-/-小鼠表现出鸭子般的步态、视网膜发育缺陷、雄性生育能力延迟等[24]。小鼠RORβ缺乏,还表现出昼夜节律异常[25],研究结果表明,RORs可被BMAL1、CLOCK和CRY1等几个时钟基因识别,从而调节昼夜节律[26-29]。除此之外,RORs还调节下游靶基因的节律表达,因此,RORs可作为耦合昼夜节律振荡与各种生理过程的循环控制的中间产物,包括能量平衡、脂质代谢等[30-31]。本研究发现,RORB基因上3个位点突变显著影响300 d或450 d的鸭蛋质量,推测鸭RORB可能是通过调控鸭昼夜节律基因的表达而影响其能量代谢过程,从而引起蛋质量的变化。
ARSB的2个位点Z-22908831、Z-22966695突变显著影响300 d、450 d蛋质量。ARSB是一种溶酶体酶,这种酶活性的缺乏会导致未降解底物的积累和溶酶体储存障碍,即粘多糖病VI型。已经发现ARSB的多个突变均与人的VI型粘多糖病相关[32-34],尚未见ARSB在禽类中的相关报道,本研究首次发现ARSB的位点突变与蛋质量相关,提示鸭的ARSB基因可能通过影响机体代谢参与鸭产蛋过程。
RASEF(含RAS和EF域的蛋白质)属Rab GTPase蛋白家族成员,在C端区域包含1个Rab GTPase结构域,内部区域包含1个卷曲的线圈基序和2个EF结构域,它们对结合N端的钙离子非常重要[35-36]。研究结果表明,Rab45/RASEF可在人的心、肝、肺、肾脏、前列腺和睾丸等中检测到[35],而且与结直肠癌[37-38]、肺癌[35]、葡萄膜黑色素瘤[39]、乳腺癌[40-42]等肿瘤相关,RASEF在肿瘤中的作用机制可能是通过抑制肿瘤细胞的生长从而抑制肿瘤[38-39]。本研究首次发现RASEF点突变与鸭蛋质量相关,但具体调节机制有待进一步挖掘。
4 结论
本研究通过高通量混池重测序结合选择清除分析技术筛选鸭蛋高质量组、低质量组组间基因组差异显著区域内的SNP和功能基因,筛选到178个定位于Z号染色体与鸭蛋质量相关的SNP显著差异区间及40个受选择候选基因。KEGG分析结果表明,这些受选择SNP位点和基因主要涉及代谢通路。通过比较12个突变位点不同基因型间鸭300 d、450 d蛋质量差异,鉴定出一些蛋质量相关候选突变位点及基因,这些基因可作为目标候选基因做进一步研究。本研究结果可为通过分子育种技术提高蛋鸭产蛋性能选育提供依據。
参考文献:
[1] 王珍珍. 不同蛋鸭品种产蛋性能的比较分析及绍兴鸭产蛋性能的全基因组关联分析[D]. 金华:浙江师范大学,2020.
[2] 刘 杰. 利用高通量测序技术挖掘影响鸡剩余采食量的遗传变异和功能基因[D]. 北京:中国农业科学院,2017.
[3] BENTLEY D R. Whole-genome re-sequencing[J]. Current Opinon in Genetics & Development, 2006, 16(6):545-552.
[4] 岳桂东,高 强,罗龙海,等. 高通量测序技术在动植物研究领域中的应用[J]. 中国科学, 2012, 42(2):107-124.
[5] 杨德智,侯冠彧,施力光,等. 全基因组测序在畜禽中应用的研究进展[J]. 中国畜牧兽医, 2021,48(9):3403-3414.
[6] 杜彦丽,王 坤,葛长荣. 鸡产蛋性能遗传因子的研究进展[J]. 中国畜牧杂志, 2020, 56(8):32-38.
[7] LIU W B, LI D F, LIU J F, et al. A genome-wide SNP scan reveals novel loci for egg production and quality traits in white leghorn and brown-egg dwarf[J]. PLoS One, 2011, 6(12):e28600.
[8] SHEN X, ZENG H, XIE L, et al. The GTPase activating Rap/Ran GAP domain-like 1 gene is associated with chicken reproductive traits[J]. PLoS One, 2012, 7(4):e33851.
[9] FAN Q C, WU P F, DAI G J, et al. Identification of 19 loci for reproductive traits in a local Chinese chicken by genome-wide study[J]. Genetics and Molecular Research, 2017, 16(1):1-8.
[10]张海林. 蛋鸡蛋重的影响因素和控制措施[J]. 现代畜牧科技, 2020(5):29-30.
[11]陆 本,译. 蛋重的遗传[J]. 国外畜牧学(猪与禽),1992(4):38-39.
[12]林如龙. 山麻鸭体重、产蛋量和蛋重的遗传参数研究[J]. 中国家禽, 2016,38(11):62-64.
[13]CHANG M T, CHENG Y S, HUANG M C. Association of prolactin haplotypes with reproductive traits in Tsaiya ducks[J]. Animal Reproduction Science, 2012, 135(1/4):91-96.
[14]李凤宁,徐桂云,张俊楠,等. 广西麻鸡产蛋相关基因的SNPs检测及其与蛋用性能关联分析[J]. 中国畜牧杂志, 2020,56(5):78-82.
[15]李丛艳. 鸡PRL-R基因多态性与就巢和产蛋性状的关联研究[D]. 成都: 四川农业大学, 2009.
[16]FENG X P, KUHNLEIN U, AGGREY S E, et al. Trait association of genetic markers in the growth hormone and the growth hormone receptor gene in a White Leghorn strain[J]. Poultry Science, 1997, 76(12):1770-1775.
[17]詹慧琴. 雞产蛋相关基因的SNPs检测及其与蛋用性能关系的研究[D]. 武汉:华中农业大学,2006.
[18]PU Y J, WU Y, XU X J, et al. Association of VIPR-1 gene polymorphisms and haplotypes with egg production in laying quails[J]. Journal of Zhejiang University-Science B (Biomedicine & Biotechnology), 2016, 17(8):591-596.
[19]KHALTABADI FARAHANI A H, MOHAMMADI H, MORADI M H, et al. Identification of potential genomic regions for egg weight by a haplotype-based genome-wide association study using Bayesian methods[J]. British Poultry Science, 2020, 61(3):251-257.
[20]CHANG M T, CHENG Y S, HUANG M C. A novel non-synonymous SNP of the COLX gene and its association with duck reproductive traits[J]. Molecular and Cellular Probes, 2012, 26(5):204-207.
[21]CHANG M T, CHENG Y S, HUANG M C. A novel SNP of the PNRC1 gene and its association with reproductive traits in Tsaiya ducks[J]. Theriogenology, 2012, 78(1): 140-146.
[22]CHANG M T, CHENG Y S, HUANG M C. Novel genetic markers of the carbonic anhydrase II gene associated with egg production and reproduction traits in Tsaiya ducks[J]. Reproduction in Domestic Animals, 2013, 48(1):98-104.
[23]SCHAEREN-WIEMERS N, ANDRE E, KAPFHAMMER J P, et al. The ExDression pattern of the orphan nuclear receptor ROR β in the developing and adult rat nervous system suggests a role in the processing of sensory information and in circadian rhythm[J]. European Journal of Neuroscience, 1997, 9(12):2687-2701.
[24]ANDR? E, CONQUET F, STEINMAYR M, et al. Disruption of retinoid-related orphan receptor beta changes circadian behavior, causes retinal degeneration and leads to vacillans phenotype in mice[J].The EMBO Journal, 1998, 17(14):3867-3877.
[25]MASANA M I, SUMAYA I C, BECKER-ANDRE M, et al. Behavioral characterization and modulation of circadian rhythms by light and melatonin in C3H/HeN mice homozygous for the ROR β knockout[J]. American Journal of Physiology Regulatory, Integrative and Comparative Physiology, 2007, 292(6): R2357- R2367.
[26]GUILLAUMOND F, DARDENTE H, GIGU?RE V, et al. Differential control of Bmal1 circadian transcription by REV-ERB and ROR nuclear receptors[J]. Journal of Biological Rhythms, 2005, 20(5):391-403.
[27]KUMAKI Y, UKAI-TADENUMA M, UNO K D, et al. Analysis and synthesis of high-amplitude Cis-elements in the mammalian circadian clock[J]. Proceeding of the National Academy of Sciences of the United States of America, 2008, 105(39):14946-14951.
[28]PREITNER N, DAMIOLA F, LOPEZ-MOLINA L, et al. The orphan nuclear receptor REV-ERB α controls circadian transcription within the positive limb of the mammalian circadian oscillator[J]. Cell, 2002, 110(2):251-260.
[29]SATO T K, PANDA S, MIRAGLIA L J, et al. A functional genomics strategy reveals Rora as a component of the mammalian circadian clock[J]. Neuron, 2004, 43(4): 527-537.
[30]KANG H S, ANGERS M, BEAK J Y, et al. Gene expression profiling reveals a regulatory role for RORα and RORγ in phase I and phase II metabolism[J]. Physiological Genomics, 2007, 31(2):281-294.
[31]JETTEN A M. Retinoid-related orphan receptors (RORs): critical roles in development, immunity, circadian rhythm, and cellular metabolism[J]. Nuclear Receptor Signaling, 2009, 7:e003.
[32]UTTARILLI A, RANGANATH P, JAIN S J, et al. Novel mutations of the arylsulphatase B (ARSB) gene in Indian patients with mucopolysaccharidosis type VI[J]. Indian Journal of Medical Research, 2015, 142(4):414-425.
[33]WANG P, MARGOLIS C, LIN G, et al. Mucopolysaccharidosis type VI in a great Dane caused by a nonsense mutation in the ARSB gene[J]. Veternary Pathology, 2017, 55(2):286-293.
[34]MALEKPOUR N, VAKILI R, HAMZEHLOIE T. Mutational analysis of ARSB gene in mucopolysaccharidosis type VI: identification of three novel mutations in Iranian patients[J]. Iranian Journal of Basic Medical Sciences, 2018, 21(9): 950-956.
[35]OSHITA H, NISHINO R, TAKANO A, et al. RASEF is a novel diagnostic biomarker and a therapeutic target for lung cancer[J]. Molecular Cancer Research, 2013, 11(8):937-951.
[36]MITRA S, FEDERICO L, ZHAO W, et al. Rab25 acts as an oncogene in luminal B breast cancer and is causally associated with Snail driven EMT[J]. Oncotarget, 2016, 7(26):40252-40265.
[37]YU X, FANG Z, LI G, et al. High RASEF expression is associated with a significantly better prognosis in colorectal cancer[J]. International Journal of Clinical and Experimental Pathology, 2018, 11(9):4276-4282.
[38]YU X, FANG Z, LI G, et al. High RASEF expression is associated with a significantly better prognosis in colorectal cancer[J]. International Journal of Clinical and Experimental Pathology, 2018, 11(9):4276-4282.
[39]MAAT W, BEIBOER S H, JAGER M J, et al. Epigenetic regulation identifies RASEF as a tumor-suppressor gene in uveal melanoma[J]. Investigative Ophthalmology & Visual Science, 2008, 49(4):1291-1298.
[40]SHIBATA M, KANDA M, SHIMIZU D, et al. RASEF expression correlates with hormone receptor status in breast cancer[J]. Oncology Letters, 2018, 16(6):7223-7230.
[41]XIAO B, HANG J, LEI T, et al. Identification of key genes relevant to the prognosis of ER-positive and ER-negative breast cancer based on a prognostic prediction system[J]. Molecular Biology Reports, 2019, 46(2): 2111-2119.
[42]CAI M J, LIANG X, SUN X, et al. Nuclear receptor coactivator 2 promotes human breast cancer cell growth by positively regulating the MAPK/ERK pathway[J]. Frontiers in Oncology, 2019, 9:164.
(責任编辑:王 妮)

