黍稷高基元EST-SSR标记开发及200份核心种质资源遗传多样性分析
王 倩 张立媛 许 月 李 海 刘少雄 薛亚鹏 陆 平 王瑞云 刘敏轩,*
研究简报
黍稷高基元EST-SSR标记开发及200份核心种质资源遗传多样性分析
王 倩1,2张立媛3许 月4李 海5刘少雄2薛亚鹏1,2陆 平2王瑞云1,*刘敏轩2,*
1山西农业大学农学院, 山西太谷 030801;2中国农业科学院作物科学研究所, 北京 100081;3赤峰市农牧科学研究所, 内蒙古赤峰 024031;4吉林大学生命科学学院, 吉林长春 130012;5山西农业大学高寒区作物研究所, 山西大同 037008
为搞清黍稷核心种质资源的遗传背景, 开发新的高基元EST-SSR标记用以评估国内不同生态区资源的遗传多样性, 为加快黍稷育种进程和挖掘优异种质提供依据。本研究基于转录组测序开发了200个高基元EST-SSR标记并进行多态性筛选, 利用筛选到的多态性标记评价200份黍稷核心种质资源的遗传多样性。结果表明, 200个标记在6份地理来源差异显著的黍稷材料中有52个呈多态性, 开发效率为26%, 其中四、五和六碱基重复多态性标记分别为17个(32.7%)、17个(32.7%)和18个(34.6%)。利用52个高基元EST-SSR多态性标记对200份核心资源进行检测, 共得到129个观测等位变异, 每个位点检测到2~3个; Shannon多样性指数为0.3093~1.0910, 平均为0.7277; 多态性信息含量为0.1948~0.8211, 平均为0.5104。基于UPGMA将200份资源划分为6个群组。基于Structure的遗传结构(=7)将资源划分为7个类群, 黍稷资源主要来自4个(东北地区、华北地区、黄土高原和北方地区)基因库。基于主成分分析将材料聚为6个类群, 与其地理来源一致。构建了52个四–六碱基重复EST-SSR标记, 开发率为26%; 用其分析材料的遗传差异发现黍稷地方核心种质遗传多样性较为丰富。
黍稷; 核心种质; 高基元EST-SSR; 遗传多样性; 群体结构
黍稷(L.), 又叫糜子、糜黍和粢, 是起源于中国的一年生暖季型作物, 栽培历史超过10,000年[1-3]。黍稷生长周期短, 具有耐旱、耐瘠、耐盐碱和水资源利用效率高的特性, 适应不良环境的能力强, 在世界范围内广泛种植[4-6]。黍稷富含蛋白质、矿物质、维生素和微量元素, 包括铜、锌、铁、锰[7], 其籽粒中的必需氨基酸(亮氨酸、异亮氨酸、蛋氨酸)含量高于小麦[8]。黍稷脱壳后称黄米, 可制作粘糕、酿制黄酒。目前中国保存黍稷资源9800余份[9], 对其进行遗传多样性研究, 有助于探究其起源与进化关系, 同时为良种选育、资源保护等提供材料基础。
评估种质资源遗传多样性可以通过表型性状或者分子标记等方法[10]。表型性状具有不稳定性, 容易受到外界环境的影响, 从而导致评价结果不准确[11]; 而分子标记技术是从DNA水平上揭示遗传变异, 受外界环境影响小, 结果稳定可靠[12]。目前广泛应用于黍稷遗传差异分析的分子标记有SSR。根据SSR的来源可将其分为基因组SSR和EST-SSR (表达序列标签, expressed sequence tag, EST) 2个类型[13]。基因组SSR已广泛用于黍稷遗传多样性分析、遗传图谱构建等领域[1,6,14-15]。连帅等[16]用63个基因组SSR分析国内外192份黍稷地方品种和野生种质遗传多样性, 检测出161个等位变异, 发现不同地理来源群体间表现出显著的遗传多样性差异, 其中内蒙地区、东北地区和黄土高原地区种质资源遗传多样性最丰富。Liu等[1]用67个基因组SSR分析88份黍稷种质, 检测到179个等位基因, 多态性信息含量平均值为0.376, 不同生态区内遗传变异丰富, 而且不同群体间遗传关系复杂。薛延桃等[17]用103个基因组SSR评估216份甘肃和新疆黍稷种质, 共检测到299个等位基因, 发现甘肃资源遗传多样性较高。EST-SSR (表达序列标签, expressed sequence tag, EST)标记是基于转录组测序技术开发得出的, 具有重复性好、多态性高、稳定性强、信息量大等特点[18]。目前, 基于转录组测序技术挖掘了大量的黍稷EST-SSR标记。刘笑瑜等[19]从一个抗旱和抗盐品种转录组测序结果中开发了151个高基元SSR标记, 检测发现85个标记具有多态性(四、五和六碱基重复标记分别为71个、10个和4个), 开发效率为56.29%。王璐琳等[20]根据实验室前期黍稷转录组测序结果设计了70个三碱基重复SSR标记, 筛选出17个多态性标记, 开发效率为25.37%。Jiang等[21]从雁黍5号中发掘了8139个EST-SSR, 随机验证24个标记发现, 3个具有多态性, 开发效率为12.5%。陈小红等[22]基于黄糜子和镇原大糜子转录组测序结果构建了200个低基元SSR, 验证发现80个标记具有多态性(一、二和三碱基重复SSR标记分别为10个、15个和55个), 开发效率为40%。高基元SSR引物分辨率高, 对不同基因型分辨力强, 可以检测到低基元SSR扩增不出的变异[19]。何杰丽等[23]用80个低基元EST-SSR标记对来自国内外的144份黍稷种质进行遗传差异评估, 检测到206个等位变异, PIC平均值为0.4573, 基于主成分分析将材料分为7个类群, 该结果与材料的地理来源一致。王舒婷等[24]用27个高基元SSR扩增57份山西黍稷种质, 检测到71个等位变异, 发现山西省黍稷资源的遗传多样性丰富。王瑞云等[25]用85个高基元SSR评估国内6个生态栽培区的96份黍稷资源, 检测到232个等位变异, 基于UPGMA和遗传结构均将材料划分为4个类群。陈小红等[26]用30个高基元SSR标记评估国内4个生态栽培区的133份种质, 检测到90个等位变异, 基于UPGMA将试材聚为3个类群, 主成分分析将材料划归4类。
核心种质是以较小的种质资源最大限度地代表整个种质资源的遗传多样性, 因此对于核心种质遗传多样性的评估极为重要[27]。随着分子生物学的发展, 分子标记在核心种质遗传多样性检测方面应用也较为广泛。姜俊烨等[27]用24个SSR分析了国内外1075份蚕豆核心种质的遗传多样性。车卓等[28]用13个SRAP标记分析了100份黑芝麻核心种质遗传多样性。徐彦刚等[29]用45个SSR对不同来源的78份西瓜核心种质进行了遗传多样性分析。种质资源是创造新性状、培育新品种的材料基础, 课题组前期构建了516份国内外黍稷核心种质, 含有国内外地方品种、育成品种和野生材料, 从中筛选出来自18个省份的200份具有代表性的优良性状的黍稷地方核心资源, 以促进黍稷种质资源的进一步鉴定和有效利用。
黍稷虽然是一种小宗作物, 但其栽培历史悠久、种质资源丰富, 且目前已完成了全基因组测序工作[30-31], 并广泛开发了SSR等分子标记, 从分子水平开展了较多遗传多样性和群体遗传结构研究。但所用遗传标记多为低基元SSR, 对研究材料的基因型分辨能力不足, 而开发适用于黍稷研究的高基元多态性SSR标记并利用其进行遗传多样性分析的相关研究较少。课题组前期对抗旱和干旱敏感品种进行了转录组测序, 发现存在大量高基元EST-SSR标记, 其中五、六碱基重复标记较多, 因此, 本研究开发了200个新的高基元SSR标记。并且前期构建了一批黍稷核心种质资源, 可以大大提高黍稷种质资源利用效率。基于此, 本研究拟首先利用6份地理来源差异显著的材料对本试验开发的200个高基元SSR标记进行多态性初筛, 随后利用筛选到的高基元SSR标记对构建的200份黍稷核心种质进行遗传多样性分析, 为鉴定黍稷优异种质、育种亲本选配提供理论依据。
1 材料与方法
1.1 试验材料
参试材料为中国农业科学院作物科学研究所资源中心小宗作物创新组所构建的黍稷地方品种核心种质200份, 来源于国内6个生态栽培区(表1和附表1)。

表1 参试黍稷资源的生态区分布
其他糜子区: 南方秋冬糜子区和青藏高原春糜子区。
Other sowing ecotope: the Southern autumn & winter-sowing ecotope and the Qinghai-Tibet plateau spring-sowing ecotope.
1.2 试验方法
1.2.1 DNA提取与检测 选取15 d左右的幼叶, 用DNA提取试剂盒(北京鼎国昌盛生物有限公司)提取全基因组DNA。用1%的琼脂糖凝胶电泳检测DNA的质量, 用超微量分光光度计测定其浓度。调试其终浓度至30~60 ng μL–1, 保存于–20℃备用。
1.2.2 EST-SSR标记分析 用地理来源差异较大的6份材料(表2)对前期通过转录组测序获得的200对高基元EST-SSR引物进行多态性筛选, 其中52对引物(附表2, 由英潍捷基(上海)贸易有限公司合成)的扩增条带清晰且多态性稳定, 可用于下一步评估200份黍稷核心种质资源的遗传多样性。

表2 用于筛选EST-SSR引物的黍稷材料
在MY-CYCLER PCR仪上进行PCR扩增反应。PCR反应体系(20 μL)包括PCR Master Mix (北京擎科生物技术有限公司) 10 μL、正向引物和反向引物各0.8 μL、基因组DNA 1 μL和ddH2O 7.4 μL。PCR反应程序为98℃ 5 min; 98℃ 10 s,m值10 s, 72℃ 15 s, 36个循环; 72℃ 2 min, 4℃保存。用8%聚丙烯酰胺凝胶电泳分离PCR产物, 硝酸银染色检测扩增产物。
1.3 数据统计与分析
读取电泳结果, 同一位置有扩增条带的记为1, 否则记为0。用PopGen 1.32计算每对引物的遗传多样性参数, 包括观测等位基因()、有效等位基因()、Shannon多样性指数()等。用PowerMarker 3.25计算多态性信息含量(PIC)和遗传距离。用MEGA 6.06构建聚类图。分别用NTSYSpc 2.11和Structure 2.3.4进行主成分和群体遗传结构分析。
2 结果与分析
2.1 高基元EST-SSR引物筛选
利用6份地理来源差异显著的黍稷对200个高基元EST-SSR标记进行多态性扩增发现, 14对引物无扩增条带, 186对引物有扩增条带, 其中52对在6份材料间呈多态性,开发效率为26%。52对多态性引物中四、五和六碱基重复引物分别有17对(32.7%)、17对(32.7%)和18对(34.6%)。
2.2 基于52对高基元EST-SSR标记的200份黍稷资源遗传多样性分析
利用52对高基元EST-SSR引物评估200份黍稷核心种质资源的遗传多样性(附表3)发现, 52个位点共检测出129个观测等位变异, 每个位点检测到2~3个, 平均为2.4804个; 有效等位变异变化范围为1.1893 (BMGJY80)~ 2.9565 (BMGJY141), 平均为1.9729; Shannon多样性指数为0.3093 (BMGJY155)~1.0910 (BMGJY141), 平均为0.7277; 观测杂合度为0 (BMGJY5、BMGJY12和BMGJY19等)~0.9282 (BMGJY177), 平均为0.2695; 期望杂合度为0.1597 (BMGJY80)~0.6636 (BMGJY141), 平均为0.4609; Nei’s期望杂合度为0.1592 (BMGJY80)~0.6618 (BMGJY141), 平均为0.4593; 多态性信息含量为0.1948 (BMGJY86)~0.8211 (BMGJY46), 平均为0.5104。
2.3 不同生态区黍稷资源的遗传多样性和相似性分析
分析不同生态区黍稷资源的遗传多样性参数(表3)发现, 各生态区观测等位基因变化范围为2.1346~2.4808, 最高的是北方春糜子区和黄土高原春夏糜子区, 最低的是其他糜子区。有效等位基因变化范围为1.7636~1.9570, 最高的是东北春糜子区, 最低的是华北夏糜子区。从Nei’s基因杂合度分析, 观测杂合度最高的是黄土高原春夏糜子区(0.2803), 最低的是华北夏糜子区(0.2272)。期望杂合度最高的是东北春糜子区(0.4558), 最低的是华北夏糜子区(0.3715)。就Shannon多样性指数和PIC值而言, 黄土高原春夏糜子区材料最高, 其他糜子区材料最低, 说明前者遗传多样性比较丰富。
分析不同生态区黍稷资源的遗传距离与遗传一致度参数(表4)发现, 遗传距离变化范围为0.0177~0.1238, 平均值为0.0670; 遗传一致度变化范围为0.8835~0.9824, 平均值为0.9357。北方春糜子区和黄土高原春夏糜子区间的遗传距离最小(0.0177), 遗传一致度最高(0.9824), 东北春糜子区和其他糜子区间的遗传距离最大(0.1238), 遗传一致度最低(0.8835)。由此表明地理位置越近, 遗传距离越小, 遗传相似性越高, 亲缘关系越近; 地理位置越远, 遗传距离越大, 遗传相似性越低, 亲缘关系越远。
2.4 基于UPGMA的黍稷资源聚类分析
基于UPGMA对200份黍稷核心种质进行聚类分析(图1)发现, 200份黍稷核心种质分为6个组群(表5)。组群I有6份材料, 主要来自青海, 属于黄土高原春夏糜子区。组群II有8份材料, 主要来自新疆, 属于西北春夏糜子区。组群III有18份, 主要来自内蒙古, 属于北方春糜子区。组群IV有33份, 主要来自甘肃, 属于黄土高原春夏糜子区。组群V有112份, 主要来自山西, 属于北方春糜子区。组群VI有23份, 主要来自山东, 属于华北夏糜子区。

表3 6个生态区黍稷的遗传多样性参数
其他糜子区: 南方秋冬糜子区和青藏高原春糜子区。
Other sowing ecotope: the Southern autumn & winter-sowing ecotope and the Qinghai-Tibet plateau spring-sowing ecotope. NES: Northeast spring-sowing ecotope; NSU: Northern summer-sowing ecotope; NSP: Northern spring-sowing ecotope; LPSS: Loess plateau spring & summer-sowing ecotope; NWSS: Northwest spring & summer-sowing ecotope; OSP: other sowing ecotope.
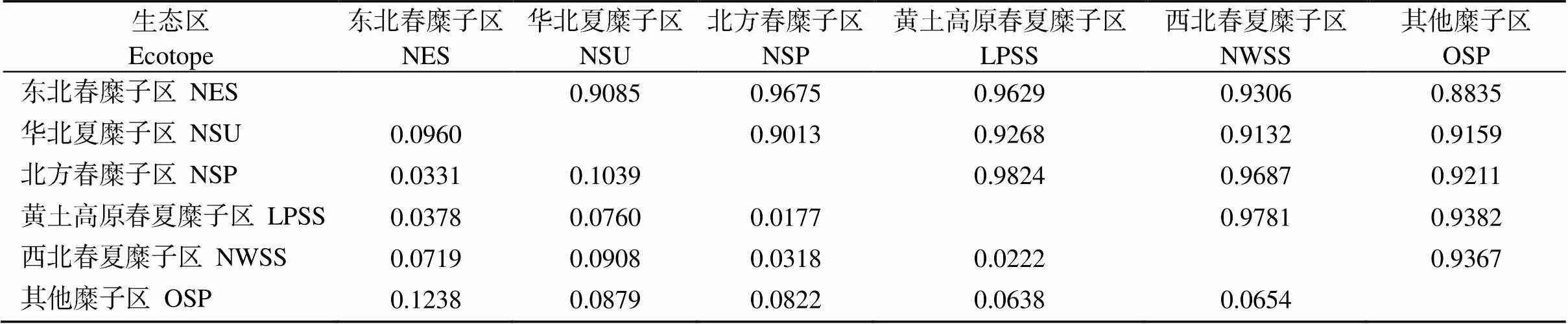
表4 不同黍稷群体间的遗传距离与遗传一致度
Nei氏遗传一致度(对角线以上)和遗传距离(对角线以下)。其他糜子区: 南方秋冬糜子区和青藏高原春糜子区。
Nei’s genetic identity (above diagonal) and genetic distance (below diagonal). NES: Northeast spring-sowing ecotope; NSU: Northern summer-sowing ecotope; NSP: Northern spring-sowing ecotope; LPSS: Loess plateau spring & summer-sowing ecotope; NWSS: Northwest spring & summer-sowing ecotope; OSP: other sowing ecotope. Other sowing ecotope: the Southern autumn & winter-sowing ecotope and the Qinghai-Tibet plateau spring-sowing ecotope.

表5 基于UPGMA的200份黍稷资源组群分布

图1 基于UPGMA的200份黍稷资源聚类分析
NES: 东北春糜子区; NSU: 华北夏糜子区; NSP: 北方春糜子区; LPSS: 黄土高原春夏糜子区; NWSS: 西北春夏糜子区; OSP: 其他糜子区。其他糜子区: 南方秋冬糜子区和青藏高原春糜子区。
NES: Northeast spring-sowing ecotope; NSU: Northern summer-sowing ecotope; NSP: Northern spring-sowing ecotope; LPSS: Loess plateau spring & summer-sowing ecotope; NWSS: Northwest spring & summer-sowing ecotope; OSP: other sowing ecotope. Other sowing ecotope:the Southern autumn & winter-sowing ecotope and the Qinghai-Tibet plateau spring-sowing ecotope.
2.5 基于Structure的群体遗传结构分析
用Structure软件对200份供试材料基因库数目进行建模(图2)发现, ∆在=2和=7处存在峰值。由图3可知,=2时, 将试材分为2个类群, 红色类群(121份), 主要为山西和陕西种质, 属于北方春和黄土高原春夏糜子区; 绿色类群(79份), 主要为甘肃和河北种质, 属于北方春和黄土高原春夏糜子区。=7时, 划分为7个类群, 红色类群(32份), 主要来自山西和陕西种质, 代表黄土高原基因库; 绿色类群(26份), 主要来自青海种质, 代表黄土高原基因库; 深蓝色类群(31份), 主要来自黑龙江和吉林种质, 代表东北基因库; 黄色类群(19份), 主要来自山东种质, 代表华北基因库; 紫色类群(25份), 主要来自山西种质, 代表北方基因库; 浅蓝色类群(34份), 主要来自山西种质, 代表北方基因库; 橙色类群(33份), 主要来自甘肃种质, 代表黄土高原基因库。

图2 基于Structure对200份黍稷资源群体建模

图3 基于Structure的黍稷核心种质资源群体遗传结构图
颜色代表类群。横坐标数字分别代表黍稷材料序号和生态区类型。NES: 东北春糜子区; NSU: 华北夏糜子区; NSP: 北方春糜子区; LPSS: 黄土高原春夏糜子区; NWSS: 西北春夏糜子区; OSP: 其他糜子区。其他糜子区: 南方秋冬糜子区和青藏高原春糜子区。
Different color represents the different group. Numbers in the horizontal axis represent accession number and type of ecotope area, respectively.NES: Northeast spring-sowing ecotope; NSU: Northern summer-sowing ecotope; NSP: Northern spring-sowing ecotope; LPSS: Loess plateau spring & summer-sowing ecotope; NWSS: Northwest spring & summer-sowing ecotope; OSP: other sowing ecotope. Other sowing ecotope:the Southern autumn & winter-sowing ecotope and the Qinghai-Tibet plateau spring-sowing ecotope.
2.6 200份黍稷资源遗传差异的主成分分析
对200份供试材料进行三维PCA分析(图4)发现, 前3个主成分PC1、PC2和PC3分别解释总变异的49.7%、5.4%和3.5%, 累积解释变异58.6%。200份黍稷资源可以划分为6个类群(东北春糜子区28份、华北夏糜子区23份、北方春糜子区63份、黄土高原春夏糜子区67份、西北春夏糜子区12份、其他糜子区7份), 与材料的地理来源一致。

图4 200份黍稷资源遗传多样性的主成分分析
NES: 东北春糜子区; NSU: 华北夏糜子区; NSP: 北方春糜子区; LPSS: 黄土高原春夏糜子区; NWSS: 西北春夏糜子区; OSP: 其他糜子区。其他糜子区: 南方秋冬糜子区和青藏高原春糜子区。
NES: Northeast spring-sowing ecotope; NSU: Northern summer-sowing ecotope; NSP: Northern spring-sowing ecotope; LPSS:Loess plateau spring & summer-sowing ecotope; NWSS: Northwest spring & summer-sowing ecotope; OSP: other sowing ecotope. Other sowing ecotope: the Southern autumn & winter-sowing ecotope and the Qinghai-Tibet plateau spring-sowing ecotope.
3 讨论
3.1 黍稷高基元SSR标记的开发利用
黍稷是世界上最早驯化的谷类作物之一, 具有重要的农业、历史和进化意义。黍稷种质资源丰富, 因此开发新的分子标记对于促进其遗传多样性的研究意义重大。高基元SSR标记的引物分辨率高, 对不同基因型的分辨能力强, 2017年, 刘笑瑜[19]用151个高基元SSR扩增6份黍稷材料, 发现85个标记呈多态性, 多态率为56.3%。本研究从200对通过转录组测序得到的高基元SSR引物中筛选得到52对多态性引物, 多态率为26%, 低于刘笑瑜的结果, 这可能与引物筛选标准不同以及获得引物渠道有关。本研究开发的四、五和六碱基重复多态性引物分别为17对、17对和18对, 而刘笑瑜的研究结果分别为71对、10对和4对, 表明转录组测序材料不同, 获得的SSR标记存在差异, 本研究开发的五、六碱基SSR标记数量占比显著高于刘笑瑜的结果。一般来说, PIC<0.25, 为低度多态性引物, 0.25≤PIC<0.5为中度多态性引物, PIC≥0.5, 为高度多态性引物[32]。王舒婷等[24]利用27对高基元SSR检测57份黍稷种质的PIC平均值为0.5667, 陈小红等[26]用30对高基元SSR扩增130份黍稷材料发现PIC平均值为0.6966。本研究与前人结果一致, PIC均大于0.5, 所选引物为高度多态性引物, 说明都可用于黍稷遗传多样性的研究。
3.2 黍稷种质资源的遗传差异
本研究参试200份黍稷地方核心种质来源于国内6个生态区的18个省、市、自治区, 具有很强的代表性。因此, 利用新开发的高基元SSR分子标记对200份黍稷核心种质进行遗传多样性分析, 将为黍稷种质资源的收集保存以及育种研究提供理论依据。
本研究评估200份黍稷核心种质资源遗传差异发现Shannon多样性指数()变化范围为0.3093~1.0910, 平均为0.7277, 低于王舒婷等[24](0.7686)和王瑞云等[25](0.7708)结果, 这与选取试验材料差异有关, 前人所选试材中含有农家种和育成品种, 材料来源更广, 而本研究材料均为地方品种; 高于连帅等[16](0.6275)结果, 这可能与我们所用高基元EST-SSR标记有关, 该标记可以检测到的变异更多。
有效等位基因数、Shannon多样性指数()和多态信息含量(PIC)可以综合的反映群体遗传多样性水平[33]。从生态区角度对黍稷资源进行遗传多样性分析, 结果表明黄土高原春夏糜子区材料的有效等位变异、基因多样性和PIC指标均高于其他6个生态区材料, 显现出较丰富的遗传多样性, 这与王瑞云等[25]的结果一致。何杰丽等[23]、石甜甜等[34]和寇淑君等[35]研究均发现北方春糜子区与黄土高原春夏糜子区黍稷资源间的遗传距离最小, 遗传一致度最高, 亲缘关系较近, 本研究也发现类似结果, 表明不同生态区黍稷种质资源间遗传距离的远近与其栽培环境及地理分布密切相关。
3.3 黍稷种质资源的聚类分析与遗传结构
聚类分析与群体结构分析是研究作物种质资源遗传多样性和遗传背景的有效手段, 是作物种质资源有效保护和利用的前提[36-37]。董俊丽等[38]对96份黍稷资源的表型性状进行遗传结构和聚类分析结果表明, 类群划分与地理来源有一定的相关性。王瑞云等[25]利用高基元微卫星标记对96份中国糜子遗传多样性研究发现, 聚类分析和遗传结构分析结果均与地理起源相关。本研究基于遗传距离的聚类分析和遗传结构分析发现, 地理来源相近的资源基本聚在同一类群, 与前人结果一致。因此, 本研究采用高基元EST-SSR标记方法对200份黍稷核心种质资源的遗传关系和群体结构进行分析, 明确了黍稷核心种质资源间遗传背景和群体间的遗传多样性情况, 对今后育种材料的选用具有指导意义。
3.4 核心种质的构建
作物种质资源是作物新品种的选育、特异种质材料的利用以及种质创新的物质基础[39]。随着科学技术的发展和育种水平的不断提高, 国内外已构建了包括四大粮食作物水稻[40]、小麦[41]、大麦[42]、玉米[43]等多种作物的核心种质。中国是黍稷的起源中心, 之前也有报道构建了黍稷核心种质, 包括胡兴雨等[44]以国家作物种质库中保存的8016份国内外黍稷资源为材料, 在11个表型性状聚类的基础上, 按比例法取样, 并依各组的遗传多样性指数进行适当调整, 构建了780份国内外黍稷核心种质; 董孔军等[45]以国内黍稷地方种质资源566份为基础, 按照20%取样比例确定相应数量类群, 根据各基因型在类群中的平均离差度确定取样样本, 构建了甘肃省黍稷地方核心种质122份。课题组前期构建了516份国内外黍稷核心种质资源, 其中含有国内外地方品种、育成品种及野生材料, 本试验材料200份是从中选出的具有优良性状、代表性强的国内地方品种。因此, 本研究利用新开发的高基元SSR标记技术评估200份黍稷核心种质资源的遗传多样性, 对于了解黍稷亲缘关系、挖掘优异种质、提高育种效率和种质资源利用率意义重大。
附表 请见网络版: 1) 本刊网站http://zwxb. chinacrops.org/; 2) 中国知网http://www.cnki.net/; 3) 万方数据http://c.wanfangdata.com.cn/Periodical-zuowxb.aspx。
[1] Liu M X, Xu Y, He J H, Zhang S, Wang Y Y, Lu P. Genetic diversity and population structure of broomcorn millet (L.) cultivars and landraces in China based on microsatellite markers., 2016, 17: 370.
[2] Lu H, Zhang J, Liu K B, Wu N, LI Y, Zhou K, Ye M, Zhang T, Zhang H, Yang X, Shen L, Xu D, Li Q. Earliest domestication of common millet (L.) in East Asia extended to 10,000 years ago., 2009, 106: 7367–7372.
[3] Wang R Y, Hunt H V, Qiao Z J, Wang L, Han Y H. Diversity and cultivation of broomcorn millet (L.) in China: a review., 2016, 70: 332–342.
[4] 董明, 降彦苗, 李海权, 耿玲玲, 刘建烨, 乔志红, 刘国庆. 光周期变化对糜子形态建成及幼穗发育进程的影响. 中国农业科学, 2020, 53: 1118–1125. Dong M, Jiang Y M, Li H Q, Geng L L, Liu J Y, Qiao Z H, Liu G Q. Effects of photoperiod changes on morphological characters and young panicle development in proso millet (L.)., 2020, 53: 1118–1125 (in Chinese with English abstract).
[5] Amadoubr I, Le M. Millets: nutritional composition, some health benefits and processing: a review., 2013, 25: 501.
[6] 薛延桃, 陆平, 乔治军, 刘敏轩, 王瑞云. 基于SSR标记的黍稷种质资源遗传多样性及亲缘关系研究. 中国农业科学, 2018, 51: 2846–2859. Xue Y T, Lu P, Qiao Z J, Liu M X, Wang R Y. Genetic diversity and genetic relationship of broomcorn millet (L.) germplasm based on SSR markers., 2018, 51: 2846–2859 (in Chinese with English abstract).
[7] Boukail S, Macharia M, Miculan M, Masoni A, Calamai A, Palchetti E, Dell’Acqua M. Genome wide association study of agronomic and seed traits in a world collection of proso millet (L.)., 2021, 21: 330.
[8] Kalinova J, Moudry J. Content and quality of protein in proso millet (L.) varieties., 2006, 61: 43–47.
[9] 王瑞云. 糜子遗传多样性及进化研究进展. 北京: 中国农业出版社, 2017. pp 2–3. Wang R Y. Genetic Diversity and Evolution Advancement in Common Millet (L.). Beijing: China Agriculture Press, 2017. pp 2–3 (in Chinese with English abstract).
[10] 宋芸, 张鑫瑞, 李政, 孙哲, 李澳旋, 杜晓蓉, 乔永刚. 基于SSR分子标记的柴胡遗传多样性与遗传结构分析. 药学学报, 2022, 57: 1193–1202. Song Y, Zhang X R, Li Z, Sun Z, Li A X, Du X R, Qiao Y G. Genetic diversity and genetic structure analysis ofDC. based on SSR molecular marker., 2022, 57: 1193–1202 (in Chinese with English abstract).
[11] 葛淑俊, 孟义江, 李广敏, 马峙英. 我国药用植物遗传多样性研究进展. 中草药, 2006, (10): 1584–1589. Ge S J, Meng Y J, Li G M, Ma S Y. Research progress on genetic diversity in Chinese medicinal plants., 2006, (10): 1584–1589 (in Chinese with English abstract).
[12] Grover A, Sharma P C. Development and use of molecular markers: past and present., 2016, 36: 290–302.
[13] 程小毛, 黄晓霞. SSR标记开发及其在植物中的应用. 中国农学通报, 2011, 27(5): 304–307. Cheng X M, Huang X X. Development and application of SSR markers in plants., 2011, 27(5): 304–307 (in Chinese with English abstract).
[14] 王银月, 刘敏轩, 陆平, 乔治军, 杨天育, 李海, 崔喜艳. 构建黍稷分子遗传图谱SSR引物的筛选. 作物杂志, 2014, (4): 32–38. Wang Y Y, Liu M X, Lu P, Qiao Z J, Yang T Y, Li H, Cui X Y. The SSR marker selection of broomcorn millet (L.) for construction of genetic linkage map., 2014, (4): 32–38 (in Chinese with English abstract).
[15] 王银月. 黍稷种质遗传资源多样性及遗传图谱构建研究. 吉林农业大学硕士学位论文, 吉林长春, 2015. Wang Y Y. Assessment of Genetic Diversity and Construction of Genetic Linkage Map of Broomcorn Millet (L.). MS Thesis of Jilin Agricultural University, Changchun, Jilin, China, 2015 (in Chinese with English abstract).
[16] 连帅, 陆平, 乔治军, 张琦, 张茜, 刘敏轩, 王瑞云. 利用SSR分子标记研究国内外黍稷地方品种和野生资源的遗传多样性. 中国农业科学, 2016, 49: 3264–3275. Lian S, Lu P, Qiao Z J, Zhang Q, Zhang Q, Liu M X, Wang R Y. Genetic diversity in broomcorn millet (L.) from China and abroad by using SSR markers., 2016, 49: 3264–3275 (in Chinese with English abstract).
[17] 薛延桃, 陆平, 史梦莎, 孙昊月, 刘敏轩, 王瑞云. 新疆、甘肃黍稷资源的遗传多样性与群体遗传结构研究. 作物学报, 2019, 45: 1511–1521. Xue Y T, Lu P, Shi M S, Sun H Y, Liu M X, Wang R Y. Genetic diversity and population genetic structure of broomcorn millet accessions in Xinjiang and Gansu., 2019, 45: 1511–1521 (in Chinese with English abstract).
[18] 马杰, 屈雯, 陈春艳, 王磊, 马俊, 刘针杉, 马维, 周平, 何远宽, 孙勃. 基于转录组序列的叶用芥菜奶奶青菜EST-SSR标记开发与遗传多样性分析. 浙江农业学报, 2021, 33: 1640–1649. Ma J, Qu W, Chen C Y, Wang L, Ma J, Liu Z S, Ma W, Zhou P, He Y K, Sun B. Development of SSR markers based on transcriptome sequencing and genetic diversity analysis of Nainaiqingcai leaf mustard., 2021, 33: 1640–1649 (in Chinese with English abstract).
[19] 刘笑瑜. 利用高基元SSR分析中国糜子资源的遗传多样性. 山西农业大学硕士学位论文, 山西太谷, 2017. Liu X Y. Analysis of Genetic Diversity of Chinese Proso Millet Resources by High Motif SSR. MS Thesis of Shanxi Agricultural University, Taigu, Shanxi, China, 2017 (in Chinese with English abstract).
[20] 王璐琳, 王瑞云, 何杰丽, 薛延桃, 陈凌, 王海岗, 乔治军. 糜子特异性 SSR 标记的开发. 山西农业科学, 2018, 46(1): 1–4. Wang L L, Wang R Y, He J L, Xue Y T, Chen L, Wang H G, Qiao Z J. Development of specific SSR marker in common millet., 2018, 46(1): 1–4 (in Chinese with English abstract).
[21] Jiang Y M, Li H Q, Zhang J N, Xiang J Y, Cheng R H, Liu G Q. Whole genomic EST-SSR development based on high-throughput transcript sequencing in proso millet (L.)., 2018, 20: 617–620.
[22] 陈小红, 何杰丽, 石甜甜, 邵欢欢, 王海岗, 陈凌, 高志军, 王瑞云, 乔治军. 基于转录组测序开发糜子SSR标记. 中国农业科学, 2020, 53: 1940–1949. Chen X H, He J L, Shi T T, Shao H H, Wang H G, Chen L, Gao Z J, Wang R Y, Qiao Z J. Developing SSR markers of proso millet based on transcriptome sequencing., 2020, 53: 1940–1949 (in Chinese with English abstract).
[23] 何杰丽, 石甜甜, 陈凌, 王海岗, 高志军, 杨美红, 王瑞云, 乔治军. 糜子EST-SSR的开发及种质资源遗传多样性分析. 植物学报, 2019, 54: 723–732. He J L, Shi T T, Chen L, Wang H G, Gao Z J, Yang M H, Wang R Y, Qiao Z J. The genetic diversity of common millet () germplasm resources based on the EST-SSR markers., 2019, 54: 723–732 (in Chinese with English abstract).
[24] 王舒婷, 何杰丽, 石甜甜, 陈凌, 王海岗, 王瑞云, 乔治军. 利用微卫星标记分析山西糜子的遗传多样性. 植物遗传资源学报, 2019, 20: 69–78. Wang S T, He J L, Shi T T, Chen L, Wang H G, Wang R Y, Qiao Z J. Genetic diversity analysis of broomcorn millet (L.) of Shanxi province using microsatellite markers., 2019, 20: 69–78 (in Chinese with English abstract).
[25] 王瑞云, 刘笑瑜, 王海岗, 陆平, 刘敏轩, 陈凌, 乔治军. 用高基元微卫星标记分析中国糜子遗传多样性. 中国农业科学, 2017, 50: 3848–3859. Wang R Y, Liu X Y, Wang H G, Lu P, Liu M X, Chen L, Qiao Z J. Evaluation of genetic diversity of common millet () germplasm available in China using high motif nucleotide repeat SSR markers., 2017, 50: 3848–3859 (in Chinese with English abstract).
[26] 陈小红, 林元香, 王倩, 丁敏, 王海岗, 陈凌, 高志军, 王瑞云,乔治军. 基于高基元SSR构建黍稷种质资源的分子身份证. 作物学报, 2022, 48: 908–919. Chen X H, Lin Y X, Wang Q, Ding M, Wang H G, Chen L, Gao Z J, Wang R Y, Qiao Z J. Development of DNA molecular ID card in hog millet germplasm based on high motif SSR., 2022, 48: 908–919 (in Chinese with English abstract).
[27] 姜俊烨, 杨涛, 王芳, 方俐, 仲伟文, 关建平, 宗绪晓. 国内外蚕豆核心种质SSR遗传多样性对比及微核心种质构建. 作物学报, 2014, 40: 1311–1319. Jiang J Y, Yang T, Wang F, Fang L, Zhong W W, Guan J P, Zong X X. Genetic diversity analysis of germplasm resources and construction of mini-core collections forL. at home and abroad., 2014, 40: 1311–1319 (in Chinese with English abstract).
[28] 车卓, 张艳欣, 孙建, 张秀荣, 尚勋武, 王化俊. 应用SRAP标记分析黑芝麻核心种质遗传多样性. 作物学报, 2009, 35: 1936–1941. Che Z, Zhang Y X, Sun J, Zhang X R, Shang X W, Wang H J. Genetic diversity analysis of black sesame (DC) core collection of China using SRAP markers., 2009, 35: 1936–1941 (in Chinese with English abstract).
[29] 徐彦刚, 姚协丰, 李良俊, 徐锦华, 任润生, 张曼, 刘广, 娄丽娜, 朱凌丽, 侯茜, 徐建, 羊杏平. 西瓜核心种质遗传多样性分析及指纹图谱构建. 分子植物育种, 2021, 19: 305–317. Xu Y G, Yao X F, Li L J, Xu J H, Ren R S, Zhang M, Liu G, Lou L N, Zhu L L, Hou Q, Xu J,Yang X P. Analysis of genetic diversity and construction of DNA fingerprints for watermelon core germplasms., 2021, 19: 305–317 (in Chinese with English abstract).
[30] Shi J, Ma X, Zhang J, Zhou Y, Liu M, Huang L, Sun S, Zhang X, Gao X, Zhan W, Li P, Wang L, Lu P, Zhao H, Song W, Lai J. Chromosome conformation capture resolved near complete genome assembly of broomcorn millet., 2019, 10: 464.
[31] Zou C, Li L, Miki D, Li D, Tang Q, Xiao L, Rajput S, Deng P, Peng L, Jia W, Huang R, Zhang M, Sun Y, Hu J, Fu X, Schnable P S, Chang Y, Li F, Zhang H, Feng B, Zhu X, Liu R, Schnable J C, Zhu J K, Zhang H. The genome of broomcorn millet., 2019, 10: 436.
[32] Sharma P, Mehta G, Shefail, Muthusamy S K, Singh S K, Singh G P. Development and validation of heat-responsive candidate gene and miRNA gene based SSR markers to analysis genetic diversity in wheat for heat tolerance breeding., 2021, 48: 381–393.
[33] 何瑞超. 绿豆遗传多样性研究及种子萌发期耐盐性评价. 内蒙古农业大学硕士学位论文, 内蒙古呼和浩特, 2021.He R C. Study on Genetic Diversity of Mung Bean and Evaluation of Salt Tolerance During Seed Germination. MS Thesis of Inner Mongolia Agricultural University, Hohhot, Inner Mongolia, China, 2021 (in Chinese with English abstract).
[34] 石甜甜, 何杰丽, 高志军, 陈凌, 王海岗, 乔治军, 王瑞云. 利用EST-SSR评估糜子资源遗传差异. 中国农业科学, 2019, 52: 4100–4109. Shi T T, He J L, Gao Z J, Chen L, Wang H G, Qiao Z J, Wang R Y. Genetic diversity of common millet resources assessed with EST-SSR markers., 2019, 52: 4100–4109 (in Chinese with English abstract).
[35] 寇淑君, 霍阿红, 付国庆, 纪军建, 王瑶, 左振兴, 刘敏轩, 陆平. 利用荧光SSR分析中国糜子的遗传多样性和群体遗传结构. 中国农业科学, 2019, 52: 1475–1487. Kou S J, Huo A H, Fu G Q, Ji J J, Wang Y, Zuo Z X, Liu M X, Lu P. Genetic diversity and population structure of broomcorn millet in China based on fluorescently labeled SSR., 2019, 52: 1475–1487 (in Chinese with English abstract).
[36] 陈志祥, 罗小燕, 李拴林, 吴如月, 王文强, 丁西朋. 基于SSR标记的木豆种质资源遗传多样性与群体结构分析. 草地学报, 2021, 29: 904–911. Chen Z X, Luo X Y, Li S L, Wu R Y, Wang W Q, Ding X P. Genetic diversity and population structure analysis of the pigeonpea (L.) germplasm resources based on SSR markers., 2021, 29: 904–911 (in Chinese with English abstract).
[37] 谢立峰, 李宁, 李烨, 姚明华. 茄子种质遗传多样性及群体结构的SRAP分析. 植物学报, 2019, 54: 58–63. Xie L F, Li N, Li Y, Yao M H. Genetic diversity and population structure of eggplant () germplasm resources based on SRAP method., 2019, 54: 58–63 (in Chinese with English abstract).
[38] 董俊丽, 王海岗, 陈凌, 王君杰, 曹晓宁, 王纶, 乔治军. 糜子骨干种质遗传多样性和遗传结构分析. 中国农业科学, 2015, 48: 3121–3131. Dong J L, Wang H G, Chen L, Wang J J, Cao X N, Wang L, Qiao Z J. Analysis of genetic diversity and genetic structure of broomcorn millet of the Chinese., 2015, 48: 3121–3131 (in Chinese with English abstract).
[39] 韦祖生, 付海天, 田益农. 作物核心种质评价与应用研究进展.现代农业科技, 2015, (21): 9–11. Wei Z S, Fu H T, Tian Y N. Overview of evaluation and application for crops core collection., 2015, (21): 9–11 (in Chinese with English abstract).
[40] 李自超, 张洪亮, 曾亚文, 杨忠义, 申时全, 孙传清, 王象坤. 云南地方稻种资源核心种质取样方案研究. 中国农业科学, 2000, 33(5): 1–7. Li Z C, Zhang H L, Zeng Y W, Yang Z Y, Shen S Q, Sun C Q, Wang X K. Study on sampling schemes of core collection of local varieties of rice in Yunnan, China., 2000, 33(5): 1–7 (in Chinese with English abstract).
[41] 董玉琛, 曹永生, 张学勇, 刘三才, 王兰芬, 游光霞, 庞斌双, 李立会, 贾继增. 中国普通小麦初选核心种质的产生. 植物遗传资源学报, 2003, 4: 1–8. Dong Y C, Cao Y S, Zhang X Y, Liu S C, Wang L F, You G X, Pang B S, Li L H, Jia J Z. Establishment of candidate core collections in Chinese common wheat germplasm., 2003, 4: 1–8 (in Chinese with English abstract).
[42] 胥婷婷, 林峰, 华为, 汪军妹, 朱靖环, 贾巧君, 尚毅, 杨建明.我国大麦选育品种核心种质的初步构建. 浙江农业学报, 2011, 23: 483–488. Xu T T, Lin F, Hua W, Wang J M, Zhu J H, Jia Q J, Shang Y, Yang J M. Establishment of core collection on bred cultivars of Chinese barley., 2011, 23: 483–488 (in Chinese with English abstract).
[43] 李健英, 解睿, 李正莉, 曹宇宾, 郭俊宏. 玉米核心种质及构建方法研究进展. 山西农业科学, 2010, 38(9): 81–85. Li J Y, Xie R, Li Z L, Cao Y B, Guo J H. Maize core collection and its establishment., 2010, 38(9): 81–85 (in Chinese with English abstract).
[44] 胡兴雨, 王纶, 张宗文, 陆平, 张红生. 中国黍稷核心种质的构建. 中国农业科学, 2008, 41: 3489–3502. Hu X Y, Wang L, Zhang Z W, Lu P, Zhang H S. Establishment of broomcorn millet core collection in China., 2008, 41: 3489–3502 (in Chinese with English abstract).
[45] 董孔军, 杨天育, 何继红, 任瑞玉, 张磊. 甘肃省糜子地方品种资源核心种质的构建. 甘肃农业科技, 2012, (7): 7–11. Dong K J, Yang T Y, He J H, Ren R Y, Zhang L. Construction of core germplasm of local variety resources of millet in Gansu province., 2012, (7): 7–11 (in Chinese with English abstract).
High motif EST-SSR markers development and genetic diversity evaluation for 200 core germplasms in proso millet
WANG Qian1,2, ZHANG Li-Yuan3, XU Yue4, LI Hai5, LIU Shao-Xiong2, XUE Ya-Peng1,2,LU Ping2, WANG Rui-Yun1,*, and LIU Min-Xuan2,*
1College of Agronomy, Shanxi Agricultural University, Taigu 030801, Shanxi, China;2Institute of Crop Sciences, Chinese Academy of Agricultural Sciences, Beijing 100081, China;3Chifeng Academy of Agricultural and Animal Husbandry Sciences, Chifeng 024031, Inner Mongolia, China;4College of Life Sciences, Jilin University, Changchun 130012, Jilin, China;5Institute of High Latitude Crops, Shanxi Agricultural University, Datong 037008, Shanxi, China
The objective of this study is to clarify the genetic background for the core germplasm resources of proso millet and to evaluate the genetic diversity from different ecological regions in China. To provide a basis for accelerating proso millet breeding process and mining excellent germplasm, a new high motif EST-SSR markers were developed. In this study, 200 high motif EST-SSR markers were developed based on transcriptome sequencing results, and used to evaluate the genetic diversity for 200 core germplasm accessions of proso millet. Results showed that 52 of the 200 markers were polymorphic in 6 proso millet accessions with significant geographical differences, with the development efficiency of 26%, including 17 tetra-nucleotide repeat SSRs (32.7%), 17 penta-nucleotide repeat ones (32.7%), and 18 hexa-nucleotide repeat ones (34.6%). Among the examined 200 accessions, a total of 129 observed alleles were detected in 52 loci, changed 2–3 alleles at each locus. The Shannon diversity index was 0.3093–1.0910, with an average of 0.7277. The polymorphism information content was 0.1948–0.8211, with an average of 0.5104. Based on UPGMA, the 200 proso millet accessions were divided into six groups. Based on Structure (=7), proso millet were mainly from four gene pools in Northeast, Northern, Loess Plateau, and North China. Based on principal component analysis, the accessions were classified into six clusters, consistent with the geographic region. This study constructed a set of 52 tetra-, penta- and hexa-nucleotide repeat EST-SSR markers, with the efficiency of 26%. Based on the above markers, genetic diversity of proso millet for landrace core germplasm was abundant.
proso millet; core germplasm; high motif EST-SSR; genetic diversity; population structure
2023-02-13;
2023-02-28.
10.3724/SP.J.1006.2023.24201
通信作者(Corresponding authors):王瑞云, E-mail: wry925@126.com; 刘敏轩, E-mail: liuminxuan@caas.cn
E-mail: 457836483@qq.com
2022-08-31;
本研究由财政部和农业农村部国家现代农业产业技术体系建设专项(CARS-06-14.5-A2), 农作物种质资源保护与利用专项(2022NWB036-06), 中国农业科学院科技创新工程, 吉林省自然科学基金(20200201153JC)和山西省现代农业产业技术体系建设专项资金(2023CYJSTX03-12)资助。
This study was supported by the China Agriculture Research System of MOF and MARA (CARS-06-14.5-A2), the Protection and Utilization for Crop Germplasm Resources (2022NWB036-06), the Agricultural Science and Technology Innovation Program of the Chinese Academy of Agricultural Sciences, the Natural Science Foundation of Jilin Province (20200201153JC), and the Earmarked Fund for Modern Agro-industry Technology Research System (2023CYJSTX03-12).
URL: https://kns.cnki.net/kcms/detail//11.1809.S.20230227.1702.014.html
This is an open access article under the CC BY-NC-ND license (http://creativecommons.org/licenses/by-nc-nd/4.0/).

