基于核基因和微卫星的叶城沙蜥遗传多样性分析
谢慧 蒋罗雪 赵伟 祁玥 段嘉宝 李孟纯 刘筱烨 李铀





摘要 为了研究我国特有物种叶城沙蜥(Phrynocephalus axillaris)的遗传多样性,采用微卫星和核基因分子标记方法,对新疆7个地区所采集的96只叶城沙蜥进行了遗传多样性分析。基于对6个微卫星位点和核基因BDNF的PCR扩增及琼脂糖凝胶电泳检测,利用软件进行分析,结果表明群体单倍型多样性为0.707和平均核酸多样性为0.003 14,观察到的杂合度平均值和预期杂合度平均值分别为0.52和0.65,总Fst值为0.175,发现试验研究所采集的叶城沙蜥群体存在明显的遗传差异及种群间存在较大的遗传分化。综合分析说明采集的叶城沙蜥群群体间遗传交流水平低,存在较高程度的遗传分化,但由于样本较少仍需对叶城沙蜥种群遗传特征进一步研究。补充和丰富了新疆部分地区叶城沙蜥的基础遗传数据,为后续合理利用及保护该地区内其他物种的种质资源奠定了基础。
关键词 叶城沙蜥;微卫星; 脑源性神经营养因子(BDNF);遗传;种群
中图分类号 Q953 文献标识码 A
文章编号 0517-6611(2023)06-0081-07
doi:10.3969/j.issn.0517-6611.2023.06.021
Genetic Diversity Analysis of Phrynocephalus axillaris Based on Nuclear Gene and Microsatellite
XIE Hui1,JIANG Luo-xue1,ZHAO Wei2 et al
(1.Life Science and Engineering College,Northwest University for Nationalities,Lanzhou,Gansu 730100;2.College of Life Sciences,Lanzhou University,Gansu Provincial Key Laboratory of Environmental Biomonitoring and Restoration,Lanzhou,Gansu 730030)
Abstract In order to study the genetic diversity of the endemic species Phrynocephalus axillaris in China,the genetic diversity of 96 P.axillaris collected from seven regions of Xinjiang was analyzed by microsatellite and nuclear gene molecular markers.Based on the PCR amplification and agarose gel electrophoresis detection of six microsatellite loci and nuclear gene BDNF,the software was used for analysis.The results showed that the haplotype diversity of the population was 0.707 and the average nucleic acid diversity was 0.003 14.The average observed heterozygosity and expected heterozygosity were 0.52 and 0.65,respectively,and the total Fst value was 0.175.It was found that there were obvious genetic differences and large genetic differentiation among the populations collected in the experiment.Comprehensive analysis showed that the level of genetic communication among the collected populations was low,and there was a high degree of genetic differentiation.However,due to the lack of samples,it is still necessary to further study the genetic characteristics of the population.This study supplemented and enriched the basic genetic data of P.axillaris in some regions of Xinjiang,and laid the foundation for the subsequent rational utilization and protection of the germplasm resources of other species in the region.
Key words Phrynocephalus axillaris;SSR;BDNF;Heredity;Populations
葉城沙蜥(Phrynocephalus axillaris)是鬣蜥科(Agamidae)沙蜥属(Phrynocephalus)的爬行动物[1],属于我国特有的一种小型爬行动物,已被列入《世界自然保护联盟》(IUCN)2010年濒危物种红色名录。广泛分布于我国甘肃、西藏、新疆天山山脉南部地区,包括塔里木盆地及周边的吐鲁番—哈密盆地和敦煌盆地[2]。栖息在戈壁荒漠或沙漠边缘地带以及固定沙丘的丘间平地,有少数植被覆盖,垂直分布的海拔高度为65~1 500 m,在阿尔金山南麓甚至可达3 000 m左右[3]。利用线粒体基因对于叶城沙蜥种群遗传结构的研究未得到很好的解析,有可能与其分化时间太短或不完全谱系分选有关[4-5]。而基于微卫星位点变异速率快、多态性高等特点,有望更全面地解析叶城沙蜥的种群遗传结构[6]。
微卫星标记,又称简短串联重复(short tandem repeats,STRs)或简单序列重复(simple sequence repeats,SSRs),是一种基于DNA长度多态性的分子标记技术。微卫星DNA由于具有特异性的PCR扩增、多态信息容量(PIC)高、引物通用性好、突变率高、共显性、符合孟德尔遗传、易于检测等优势,已被广泛应用于种群遗传、谱系地理和亲子鉴定等领域[7-11]。微卫星标记较线粒体基因更具丰富的多态性,也是野生动物遗传学分析常用的分子标记之一,研究者可以利用分子标记研究种群的扩散模式,从而更有效地保护和管理野生动物。仲光启等[12]筛选了在黑鹳上可扩增的18个微卫星位点并进行了遗传特点分析,王萌等[13]探讨了微卫星标记方法在大型猫科动物保护群遗传学研究中的应用与挑战,刘旭等[14]概述了微卫星技术在珍稀濒危野生动物中的相关应用以及分子生物学手段在珍稀濒危野生动物扩散生态学的研究情况。对于沙蜥属的研究主要与环境气候变化有关,有红尾沙蜥超氧化物歧化酶适应高原的分子机制[15]、青海沙蜥遗传和表型分化的环境相关性[16]、增温对青海沙蜥繁殖生活史及后代适合度的影响[17-18]等。目前关于叶城沙蜥中的研究相对较少,主要在两性异形和形态特征差异、线粒体和基于高通量测序微卫星特征分析[19],而关于微卫星分析研究较少。该研究利用高质量的SSR标记评估96只叶城沙蜥的遗传多样性,解析不同群体遗传结构,为叶城沙蜥及新疆地区的物种保护提供理论依据。该研究利用6个微卫星位点和1对核基因引物,分析了7个地理种群96个样本的遗传多样性、遗传分化和系统地理结构,对叶城沙蜥进行系统发育分析,补充和丰富新疆地区叶城沙蜥的基础遗传数据,为保护该地区内其他物种提供参考。
1 材料与方法
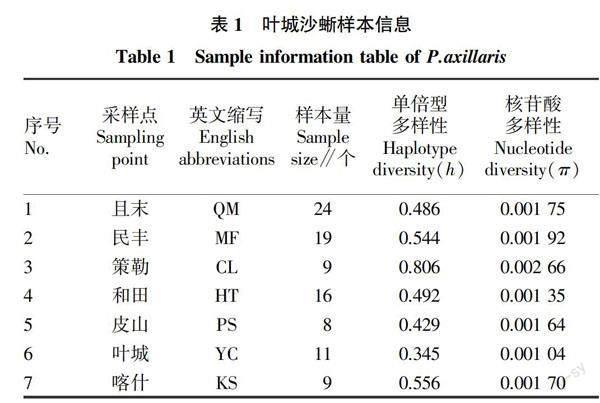
1.1 试验材料 试验研究对象为新疆7个地区(且末、民丰、策勒、和田、皮山、叶城、喀什)采集的96只叶城沙蜥,将组织样本均置于无水乙醇与生理盐水(1∶1配比)组织保存液中,-20 ℃保存备用(表1)。
1.2 基因组DNA的提取
使用通用型基因组DNA提取试剂盒进行基因组DNA的提取,并用红色荧光核酸染料染色的1.4%琼脂糖凝胶电泳进行DNA的完整性和浓度检测,并于放置于-20 ℃冰柜保存备用。
1.3 引物筛选及PCR扩增
试验随机选取4只叶城沙蜥组织作为筛选阶段的材料,根据已发表的沙蜥文献中引物信息及重新设计的叶城引物进行筛选[6],选出3对南疆沙蜥(Pvms33、Pvms39、Phr51)[20-21]和新设计的3对叶城沙蜥(PA11、PA36、PA37)微卫星引物(表2)。PCR反应体系为(12 μL):5×buffer(Mg2+ free) 2.4 μL、5 U/μL Immolase DNA聚合酶0.06 μL、10 μmol/L荧光标记上游引物tag F(Fam、Ned、Vic、Pet)0.09 μL、10 μmol/L荧光标记下游引物tag R 0.09 μL、0.4 μmol/L Primers 2.4 μL、DNA模板2 μL、其余由dd H2O补足12 μL。PCR扩增反应条件:95 ℃预变性10 min;92 ℃变性60 s,50 ℃退火90 s,72 ℃延伸60 s,5个循环;92 ℃变性30 s,63 ℃退火90 s,72 ℃延伸60 s,20个循环;92 ℃变性15 s,54 ℃退火60 s,72 ℃延伸60 s,40个循环;最后72 ℃延伸10 min。扩增产物进行琼脂糖凝胶电泳,并在凝胶成像仪上观察结果。将成功扩增的合格PCR产物送至苏州金唯智生物科技有限公司进行毛细管电泳分析。
根据GenBank公布的叶城沙蜥核基因(脑源性神经营养因子,brain-derived neurotrophic factor,BDNF)部分序列(序列号为KJ363410),使用Geneious Prime 2022.0.1(www.geneious.com)进行引物设计,上游引物:BDNF-11F(5′-TGCATGAAAGCTGCCCCAAT-3′),下游引物BDNF-600R(5′-CTGGGTAGTTCGGCACTGAT-3′)。引物由艾科瑞生物(湖南)公司合成。使用设计的引物对叶城沙蜥样本进行扩增。通过试验,建立最佳PCR扩增体系(25 μL):2×Pro Taq Master Mix(dye plus)12.5 μL、10 μmol/L上下游引物各0.5 μL、模板DNA 2.0 μL、dd H2O 9.5 μL。PCR反应条件:94 ℃预变性3 min;94 ℃变性45 s,58 ℃退火45 s,72 ℃延伸1 min,共循环 35 次;72 ℃延伸10 min,4 ℃保存。扩增产物经1.4%琼脂糖凝胶电泳进行纯度检测。检测合格的扩增产物由兰州天启基因生物科技有限公司进行测序。
1.4 数据分析
试验中所得到微卫星数据用GeneMarker[22]对测序仪得到的原始数据进行基因分型分析。用GenAlex V 6.5[23]和Popgene32[24]软件分析等位基因数(number of allele,Na)、有效等位基因数(effective number of allele,Ne)、观测杂合度(observed heterozygosity,Ho)、期望杂合度(expected heterozygosity,He)、群體间Nei’s遗传距离和遗传相似度、群体内近交系数(population inbreeding coefficient,Fis)、群体间的遗传分化指数(genetic differentiation index,Fst)、基因流(gene flow,Nm)、Shannon多样性指数(I)及方差分析(analysis of molecular variance,AMOVA)。用Cervus3.0软件[25]计算各位点的多态信息含量(Poly-morphism information content,PIC),并进行哈迪-温伯格平衡(Hardy-Weinberg equilibrium,HWE)检验。用MEGA X[26]软件绘制基于群体间的Nei’s遗传距离的UPGMA(unweighted pair-group method with arithmetic mean,UPGMA)聚类树。
试验中所选取的核基因BDNF片段长度在550~650 bp,PCR产物测序方式为正反向。采用Geneious 2020.2.5(http://www.geneious.com)处理测序结果原始ABI文件,经过人工校对后获得96条叶城沙蜥BDNF基因样本,序列长度为524 bp。基于核基因BDNF分析叶城沙蜥7个种群的遗传多样性,主要包括单倍型多样性(haplotype diversity,h)和核苷酸多样性(nucleotide diversity,π)。下载Genbank上已发表的红原沙蜥(P.hongyuanensis)的BDNF全基因(序列号为AF497714.1)作为外群的物种序列。使用MEGA-X软件[26]构建NJ(neighbor-joining)和ML(maximum likelihood)系统发育树,进化树Bootstrap置信值为重复抽样1 000次。
2 结果与分析
2.1 PCR扩增结果
试验筛选出6对多态性高且稳定的微卫星及1对核基因引物,进行后续遗传多样性分析。对选取微卫星引物和核基因引物(其目的条带在500~750 bp)在96只样本上进行PCR扩增(核基因有1样本未成功扩增),采用凝胶电泳检测,目的条带符合预期大小且清晰,核基因序列未提交至Genbank,将成功扩增的产物送公司进行测序。以部分样品在引物PA37和BDNF的电泳结果为例(图1),电泳图和测序结果一致,并且能准确读取具体的片段大小。
2.2 遗传多样性分析
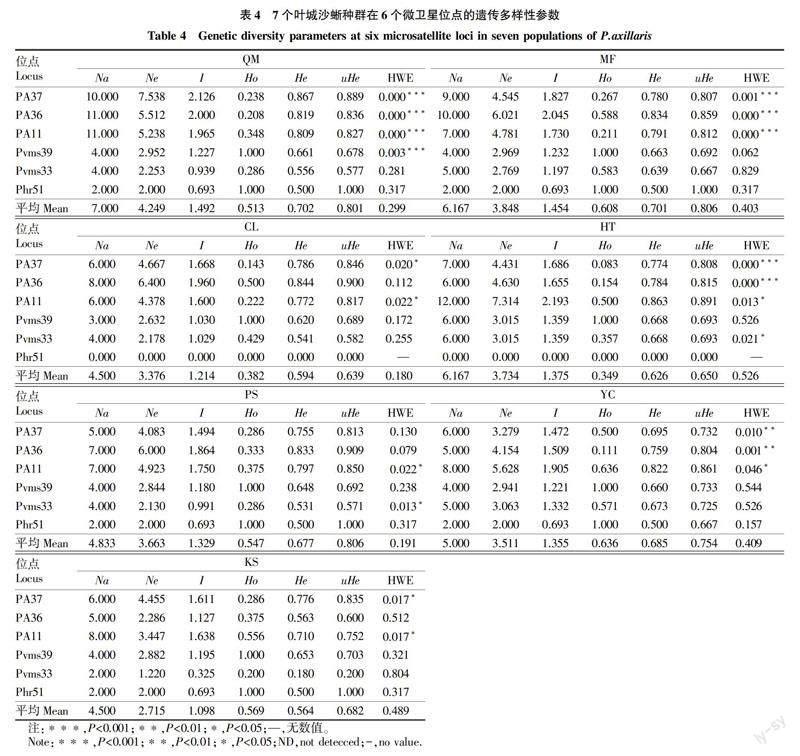
6对SSR引物对7个叶城沙蜥群体进行扩增,各位点的平均等位基因数(Na)和有效等位基因数(Ne)分别为5.452和3.585个,平均观测杂合度(Ho)、期望杂合度(He)、无偏预期杂合度(uHe)、群内近郊系数(Fis)、群体间分化系数(Fst)和基因流(Nm)分别为0.515、0.650、0.734、0.082、0.175和3.473(表3)。7个叶城沙蜥群体的等位基因数存在一定差异,其中Na最多的为QM群体(7.000个),CL和KS群体较少(表4)。分析叶城沙蜥种群在微卫星位点的遗传多样性参数,就位点而言,可知PA11位点上的平均Na、Ne、I、He较高,最低的是在Phr51(表3);就种群而言,QM最高,KS最低,在杂合度方面,QM表现出较高的杂合性且有最为丰富的遗传多样性(表4)。
基于核基因BDNF对叶城沙蜥7个种群的遗传多样性分析,主要包括单倍型多样性和核苷酸多样性(表1)。首先分析单倍型多样性,95只蜥蜴中共定义14个单倍型,群体平均单倍型多样性为0.523,群体单倍型多样性范围为0.345~0.806,其中策勒(CL)单倍型多样性最高,为0.806;叶城(YC)最低,为0.345。在核酸多样性方面,叶城沙蜥平均核苷酸多样性为0.001 72,核苷酸多样性范围为0.001 04~0.002 66,种群内平均单倍型以策勒(CL)最高,为0.002 66,叶城(YC)最低,为0.001 04,表明叶城沙蜥整体遗传多样性为高单倍型多样性和低核苷酸多样性模式。
2.2.1 基于微卫星标记的遗传分化。
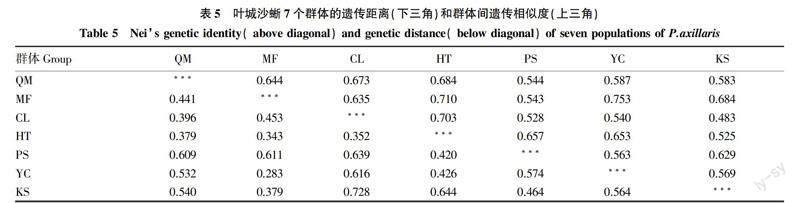
叶城沙蜥7个群体间的Nei’s遗传相似度和遗传距离见表5。7个群体间的遗传距离介于0.283~0.728,Nei’s遗传相似度介于0.483~0.753。YC和MF之间遗传距离最短,KS和CL之间的遗传距离最长。
基于群体间Nei’s遗传距离的聚类树结果(图2),7个叶城沙蜥种群分为两大类,第一大类包括PS和KS 2个种群,其他种群为第二大类。第二大类再分为3个亚类,第Ⅰ类包括QM,第Ⅱ类包括CL和HT,第Ⅲ类包括MF和YC。AMOVA分析结果显示,7个群体内的遗传变异占总体变异的62%。群体间的遗传变异占38%,分子方差AMOVA数据表明的变异大部分存在于群体内,说明这7个群体之间遗传分化较小。
2.2.2 BDNF基因叶城沙蜥的系统发育分析。
基于核基因NJ系统发育分析,由图3a可知,95个叶城沙蜥群体样本均在同一大分支,同时KS、YC和PS聚在同一枝上。红原沙蜥作为外群,独立于另一分支上。基于核基因建成的ML进化树,通过计算找到此次最大似然法构建进化树的最佳模型为Kimura 2-paprameter model+Gamma Distributed,由图3b可知,95个叶城沙蜥群体样本均在同一大分支,同时KS、YC和PS聚在同一枝上,而MF、CL、HT和QM分別聚在两支上。红原沙蜥作为外群,独立于另一分支上,结果基本一致。
3 讨论
3.1 7个叶城沙蜥群体遗传多样性
遗传多样性研究是生物多样性研究的重要组成部分,是生物进化和物种分化的基础,研究物种遗传多样性对于评估物种资源状况及在制定野生物种资源保护策略等方面具有重要意义[27]。等位基因数、观测杂合度、期望杂合度和多态信息含量等参数均反映群体遗传基础多样性水平,其值越大,说明基因丰富度越高、生物对环境的适应能力越强。赵丹等[28]利用微卫星标记对我国胎生蜥蜴的3个种群的18只母本和67只子代进行遗传多样性分析,检测到各种群母本和子代的观测等位基因数在3.90~8.13。骆爽[29]基于微卫星对西藏高原的80个红尾沙蜥和34个西藏沙蜥样本进行多样性分析,分别检测出44和42个等位基因,平均观测杂合度分别为0.360 7和0.399 6。该研究利用6对微卫星分子标记对7个叶城沙蜥群体进行遗传多样性分析,发现平均观测杂合度、平均期望杂合度分别为0.515和0.650。哈迪-温伯格平衡检验可以作为衡量群体遗传结构的标准,该研究中,各个群体偏离HWE平衡,杂合体缺失和无效等位基因的存在是各个群体显著偏离HWE平衡的原因。
该研究的7个叶城沙蜥种群在6个所用的微卫星位点上检测到的平均Na/Ne值大于1,检测到的平均等位基因数高于平均有效等位基因数,说明等位基因分布不匀,比值较大可能是由近交的原因及样本数量不够导致的。衡量群体遗传变异的一个重要参数就是基因杂合度,一般期望杂合度(He)越高说明群体的遗传变异越大;观测杂合度(Ho)越接近期望杂合度(He),说明群体受外来因素影响越小,处于遗传平衡状态。而基于杂合度计算的近交系数(Fis)可以反映出群体内部的近交程度,它的数值大小反映出了群体中杂合子的缺失或者过剩程度,该研究中表明叶城沙蜥群体间存在一定程度的基因流,这也可以解释基于BDNF构建的NJ和ML进化树中,叶城沙蜥的各种群没有完全独立于一支的原因。
核基因在系统发育、生物地理等方面研究的巨大潜力逐渐被发现,由于核基因进化速率相对较慢,能够反映更丰富的生物学信息,因此选择BDNF基因作为研究叶城沙蜥种群进化的分子标记,能够得到更丰富的结果。基于BDNF基因采用邻接法(NJ)和最大似然法(ML)构建的2种进化树结果几乎一致,通过分析可知,在叶城沙蜥种群中,7个地区共计95个样本均在1大分支上,喀什地区与同市的叶城和皮山的叶城沙蜥亲缘关系最近,且末、民丰、策勒和部分和田地区的叶城沙蜥群体在同一大支,与基于遗传距离构建的UPGMA图大致一致,但其之间是否存在基因交流的个例无法确定,有待进一步研究。
3.2 叶城沙蜥群体遗传分化和遗传结构
遗传分化参数Fst最大值为1表明两个群体完全分化,最小值为0表明群体间无分化,在实际研究中,Fst在0~0.05时,表明物种群体间的遗传分化水平很低,可以不做考虑;在0.05~0.15时,表明群体间的遗传分化水平处于中等程度;在0.15~0.25时群体间存在较大的遗传分化;0.25以上时群体间就存在很大的遗传分化[30]。该研究揭示的7个叶城沙蜥群体间遗传分化指数Fst为0.175,分子方差AMOVA数据表明38%的变异存在于群体间,62%的变异存在于群体内,表明群体间存在中等偏高程度的遗传分化。
4 结论
研究使用多重荧光PCR及琼脂糖凝胶电泳的方法,使用6对微卫星引物和BDNF基因对新疆地区7个叶城沙蜥群体进行遗传多样性分析。研究发现7个叶城沙蜥群体在6个微卫星位点上的近交系数均值为0.082,Shannon多样性指数为1.336;遗传分化指数均值为0.175,基因流均值为3.473,表明群体间遗传交流水平低,存在较高程度的遗传分化;群体单倍型多样性范围为0.345~0.806及核苷酸多样性范围为0.001 04~0.002 66。目前我国叶城沙蜥群体的遗传多样性处于中等水平,群体间的遗传分化受自身迁移能力和地理隔离等因素影响,亟需对我国叶城沙蜥进行种质资源保护,防止其遗传多样性继续下降。
哈迪-温伯格平衡是评价种群遗传平衡的参数,处于遗传平衡的种群不容易受到近交及外来种群的干扰[31],种群相对稳定。濒危物种常常表现HWE平衡偏离,造成偏离的原因很多,如近亲交配、种群迁移和遗传漂变等。该研究中有6个位点偏离HWE平衡,均为杂合不足,且表现近亲繁殖。对新疆7个地区叶城沙蜥的微卫星和核基因进行扩增测序进行分析,研究其遗传多样性,但目前的数据无法证明叶城沙蜥种群表现的遗传特征及原因,有待进一步研究,该研究补充和丰富了新疆部分地区叶城沙蜥的基础遗传数据,可为后续合理利用及保护该地区内其他物种的种质资源奠定基础。
参考文献
[1] LI J,GUO X G,WANG Y Z.Population genetic differentiation of Phrynocephalus axillaris in east of Xinjiang Uygur Autonomous Region based on sequence variation of mitochondrial ND4-tRNALeu gene[J].Zool Res,2010,31(4):361-369.
[2] 赵尔宓,赵肯堂,周开亚,等.中国动物志:爬行纲 第二卷 有鳞目 蜥蜴亚目[M].北京:科学出版社,1999.
[3] 张鹏,袁国映.新疆两栖爬行动物[M].乌鲁木齐:新疆科学技术出版社,2005.
[4] ZHANG Q,XIA L,HE J B,et al.Comparison of phylogeographic structure and population history of two Phrynocephalus species in the Tarim Basin and adjacent areas[J].Mol Phylogenet Evol,2010,57(3):1091-1104.
[5] 丁新華.基于线粒体和核基因分子标记的黑鳍鳈亲缘地理研究[D].上海:上海海洋大学,2020.
[6] 宋琪,刘金龙,郭宪光.基于Roche 454 GS FLX高通量测序的叶城沙蜥基因组微卫星特征分析[J].四川动物,2019,38(1):62-67.
[7] 张敏莹,方弟安,周彦锋,等.基于微卫星标记的钱塘江中下游三角鲂亲本群体和自然捕捞群体遗传多样性及遗传结构[J].大连海洋大学学报,2022,37(5):775-783.
[8] 赵蒙蒙,滕晖,吴海花,等.用于番鸭亲子鉴定的微卫星分子标记初步筛选[J].黑龙江畜牧兽医,2021(5):1-7,156.
[9] 陈政昆,韦聪聪,朱雯,等.安徽白山羊保种群微卫星标记遗传多样性分析[J].黑龙江畜牧兽医,2020(9):54-56,63.
[10] 张代臻,唐伯平,孙红英,等.DNA分子标记在虾蟹类系统地理学研究中的应用[J].水产科学,2008,27(5):262-265.
[11] VASHISTHA G,DEEPIKA S,DHAKATE P M,et al.The effectiveness of microsatellite DNA as a genetic tool in crocodilian conservation[J].Conserv Genet Resour,2020,12(4):733-744.
[12] 仲光启,由玉岩,徐艳春,等.黑鹳微卫星位点的筛选及遗传特点分析[J].野生动物学报,2019,40(3):729-36.
[13] 王萌,朱思雨,薛茂盛,等.微卫星标记方法在大型猫科动物保护群遗传学研究中的应用与挑战[J].野生动物学报,2019,40(3):780-786.
[14] 刘旭,丁由中.分子生物学技术应用于野生动物保护及扩散研究概况[J].野生动物学报,2019,40(2):497-501.
[15] 鲁松松,黄宇哲,张梦洋.紅尾沙蜥超氧化物歧化酶适应高原的分子机制[J].兰州大学学报(自然科学版),2022,58(1):69-78,88.
[16] 谢凡.青海沙蜥遗传和表型分化的环境相关性[D].南京:南京师范大学,2021.
[17] 王坤.增温对青海沙蜥繁殖生活史及后代适合度的影响[D].保定: 河北大学,2020.
[18] 祁玥.南疆沙蜥分子系统地理学[D].兰州:兰州大学,2019.
[19] 阿尔孜古·衣米提,徐峰,李俊.塔里木盆地西南部叶城沙蜥的两性异形研究[J].干旱区资源与环境,2021,35(7):190-195.
[20] URQUHART J,BI K,GOZDZIK A,et al.Isolation and characterization of microsatellite DNA loci in the toad-headed lizards,Phrynocephalus przewalskii complex[J].Mol Ecol Notes,2005,5(4):928-930.
[21] ZHAN A B,FU J Z.Microsatellite DNA markers for three toad-headed lizard species (Phrynocephalus vlangalii,P.przewalskii and P.guttatus)[J].Mol Ecol Resour,2009,9(2):535-538.
[22] HOLLAND M M,PARSON W.GeneMarker HID:A reliable software tool for the analysis of forensic STR data[J].J Forensic Sci,2011,56(1):29-35.
[23] PEAKALL R,SMOUSE P E.GenAIEx 6.5:Genetic analysis in Excel.Population genetic software for teaching and research:An update[J].Bioinformatics,2012,28(19):2537-2539.
[24] ROUSSET F.GENEPOP’ 007:A complete re-implementation of the genepop software for Windows and Linux[J].Mol Ecol Resour,2008,8(1):103-106.
[25] KALINOWSKI S T,TAPER M L,MARSHALL T C.Revising how the computer program CERVUS accommodates genotyping error increases success in paternity assignment[J].Mol Ecol,2007,16(5):1099-1106.
[26] KUMAR S,STECHER G,LI M,et al.MEGA X:Molecular evolutionary genetics analysis across computing platforms[J].Mol Biol Evol,2018,35(6):1547-1549.
[27] 魏辅文,马天笑,胡义波.中国濒危兽类保护遗传学研究进展与展望[J].兽类学报,2021,41(5):571-580.
[28] 赵丹,刘玉芬,赵文阁,等.基于微卫星标记的胎生蜥蜴母本与子代的遗传多样性研究[J].哈尔滨师范大学自然科学学报,2017,33(4):50-53.
[29] 骆爽.西藏高原两种沙蜥遗传结构及三种沙晰亲子鉴定[D].兰州:兰州大学,2009.
[30] 冯一宁,李因刚,祁铭,等.基于SSR标记的福建省闽楠代表性群体遗传多样性分析[J].南京林业大学学报(自然科学版),2022,46(4):102-108.
[31] 杨建宝,张艳萍,苏军虎,等.引进美洲红点鲑群体遗传多样性微卫星的分析[J].四川动物,2012,31(3):348-352.
基金项目 国家自然科学基金地区科学基金项目(32060311);甘肃省自然科学基金项目(20JR10RA124);西北民族大学中央高校基本科研业务费项目(31920210143,31920200004,31920190083);西北民族大学研究生科研创新项目(Yxm2021076)。
作者简介 谢慧(1997—),女,河南商丘人,硕士研究生,研究方向:动物遗传育种与繁殖。*通信作者,副教授,博士,硕士生导师,从事分子生态与进化生物学研究。
收稿日期 2022-07-27

