基于SRAP分子标记的147份睡莲属植物遗传多样性分析
毛立彦 龙凌云 黄秋伟 丁丽琼 李慧敏 池昭锦 唐毓玮 苏群 农晓慧 朱天龙

摘要:【目的】对147份睡莲属植物的遗传多样性及亲缘关系进行分析,为睡莲属种质资源保护、开发利用及新品种选育的亲本选择提供科学参考。【方法】筛选获得扩增条带清晰、多态性好的SRAP引物,对112份睡莲属植物种质和35份杂交后代进行多态性扩增,基于扩增结果构建0/1矩阵,利用Popgene 1.32计算遗传多样性相关参数。最后采用NTSYS 2.1的非加权组平均法(UPGMA)计算遗传相似系数和遗传距离并构建聚类图。【结果】利用筛选的10个引物对从147份睡莲属植物材料中扩增出207个条带,其中多态性条带207条,多态性比率100%,平均每对引物扩增20.7条。147份睡莲属植物的观测等位基因数(Na)为2.0000,有效等位基因数(Ne)为1.1777~1.3339,平均为1.2535,Shannon信息指数(I)为0.2459~0.3703,平均为0.3155,Neis遗传多样性指数(H')为0.1345~0.2230,平均为0.1835。在遗传相似系数0.65和遗传距离为1.12时,均可将147份睡莲属植物材料划分为六大类,并依据10个引物对扩增的0/1矩阵构建了147份睡莲属植物材料的DNA分子身份证。【结论】睡莲属植物具有丰富的遗传多样性。采用SRAP分子标记可有效鉴定睡莲属植物材料的亲缘关系远近,有助于提高亲本选择率和育种进程。筛选出的10个引物对能有效地鉴定35份杂交后代。
关键词: 睡莲;种质资源;遗传多样性;SRAP分子标记
中圖分类号:S682.320.24 文献标志码:A 文章编号:2095-1191(2023)02-0454-13
Abstract:【Objective】To analyze genetic diversity and genetic relationships among 147 Nymphaea Linn. plants, and to provide scientific reference for conservation,development and utilization of Nymphaea Linn. germplasm resources and parents selection for new varieties breeding. 【Method】Ten SRAP primer pairs with clear amplification bands,stable structure and abundant polymorphism were screened to perform polymorphic amplification on 112 Nymphaea Linn. germplasm resources and 35 hybrid progenies. Based on amplification results,a 0/1 matrix was established and genetic diversity indexes were calculated using POPGENE 1.32. Genetic similarity coefficient and genetic distance were calculated through unweighted group average (UPGMA) method of NTSYS 2.1 and cluster diagram was constructed. 【Result】Using the 10 screened primer pairs,207 polymorphic bands were detected from the 207 amplified bands, indicating a polymorphic rate of 100% and on average every primer pair produced 20.7 polymorphic loci. For 147 Nymphaea Linn. plants, observed number of alleles (Na) was 2.0000 and effective number of alleles (Ne) was from 1.1777 to 1.3339,with an average of 1.2535; Shannons information index (I) was from 0.2459 to 0.3703,with an average of 0.3155,and the Neis genetic diversity index (H') was from 0.1345 to 0.2230,with an average of 0.1835. The 147 Nymphaea Linn. plants were divi-ded into 6 groups when similarity coefficient was 0.65 and genetic distance was 1.12. DNA molecular ID of 147 Nymphaea Linn. plants were established according to the 0/1 matrix based on 10 primer pairs. 【Conclusion】Nymphaea Linn. plants enjoy rich genetic diversity. With SRAP molecular markers, genetic relationship can be effectively identified, which is helpful for increasing parental selection rate and improving breeding process. And the 10 screened primer pairs can effectively identify 35 hybrid progenies.
Key words: water lily; germplasm resource; genetic diversity; SRAP molecular marker
Foundation items: Guangxi Key Research and Development Plan Project (Guike AB18221054); Basic Scientific Research Project of Guangxi Academy of Agricultural Sciences (Guinongke 2021YT152)
0 引言
【研究意义】睡莲是睡莲科(Nymphaeaceae)睡莲属(Nymphaea Linn.)多年生宿根浮叶植物的统称,常用于园林景观建设、湿地水体绿化和净化,还可作为日化产品、食品和医药生产的重要原料用于生产化妆品、香皂等(Agnihotri et al.,2008;赵军等,2014;宋敏等,2016;黄秋伟等,2020;宋杰等,2021)。全球现有睡莲属的种(含变种)和园艺品种1000多个,主要分布于热带、亚热带和温带地区,我国现保存的种(变种)和品种400多个,在国内多数省份广泛栽培(黄国振等,2008;李淑娟等,2019;程哲等,2022)。分析睡莲属内种质之间的亲缘关系和遗传多样性,不仅为充分挖掘利用睡莲种质和新品种选育提供参考,还可为研究被子植物系统的起源、进化、发育提供重要数据支持,对睡莲新品种快速鉴定具有重要意义。【前人研究进展】睡莲属种质资源传统分类主要依据心皮、雄蕊形态等植物学特征、生态学特征及地理来源划分为5个亚属,分别为白天开花类型的澳大利亚睡莲亚属(Anecphya)、广热带睡莲亚属(Brachyceras)和广温带睡莲亚属(Nymphaea),以及晚上开花类型的古热带亚属(Lotos)和新热带亚属(Hydrocallis)(黄国振等,2008;李淑娟等,2019;潘庆龙等,2021;毛立彦等,2022)。但随着睡莲育种研究工作的深入开展,产生了由广热带睡莲亚属(Brachyceras)与广温带睡莲亚属(Nymphaea)、广热带睡莲亚属(Brachyceras)与澳大利亚睡莲亚属(Anecphya)杂交而成的跨亚属(Intersubgeneric)类型(李淑娟等,2019;苏群等,2019;黄祥等,2022),睡莲属传统分类方式无法准确有效的劃分新选育种质的进化归属。分子标记技术是研究物种遗传多样性的重要手段,其能反映生物个体或种群间DNA某些特征片段的差异,进而快速评价物种的遗传多样性。SRAP分子标记是采用特定引物对基因的开放阅读框(ORF)进行扩增,其上游引物对外显子进行特异扩增,下游引物对内含子区域和启动子区域进行特异扩增,因个体和物种的不同,内含子和启动子的间隔长度不等而产生多态性(Li and Quiros,2001)。依据SRAP分子标记扩增的多态性条带构建0/1矩阵,并计算遗传相似系数或者遗传距离,以此进行聚类分析,遗传相似系数越大说明亲缘关系越近,反之则亲缘关系越远,遗传距离越大说明亲缘关系越远,反之则亲缘关系越近(周丽霞和曹红星,2020;林榕燕等,2021)。与RAPD、ISSR、DNA条形码等标记相比,SRAP分子标记具有DNA需要量少、多态性强、高共显性、标记分布均匀、简便且花费少等特点(叶兴状等,2021;Danial et al.,2021;李萍萍等,2022)。在玉兰(张妹等,2019)、国兰(袁媛等,2020)、菊花(沈瑶等,2020)、百合(王子康等,2022)等花卉,猕猴桃(张坤等,2021)、油梨(甘霖等,2021)、荔枝(胡福初等,2021)等果树,南瓜(卢丽芳等,2015)、香椿(陈倩倩等,2018)、苦瓜(吴立东等,2019)、黄椒(邱胤晖等,2021)等蔬菜及水稻(林增顺等,2019)等粮食作物上得到广泛应用,已成为分析物种亲缘关系和构建遗传图谱可靠而高效的方法。目前关于睡莲的遗传多样性分析主要采用表型、ISSR分子标记和DNA条形码等手段,如Péter等(2011)采用ISSR分子标记分析了6个原生种睡莲的进化关系,结果发现Nymphaea‘Panama Pacific和N. caerulea为杂交起源,并将6份种质分别归入了Brachyceras亚属、Nymphaea亚属和Lotos亚属;Jeremy等(2013)基于花粉颜色和根茎形态、RAPD分子标记和DNA条形码(ITS、trnK、matK、rbcL)对分布于印度不同地区的7个睡莲种质12份材料进行聚类分析,结果发现部分原生种的起源可能是远缘杂交而来,并根据根茎形态和花粉颜色将其聚为3个分支;Qian等(2022)采用核基因内转录间隔区(ITS)、叶绿体非编码区trnT-trnF和编码区rpl16对非洲大陆7个国家的158个野生睡莲属植物进行分子鉴定,结果发现同时利用ITS、trnT-trnF和rpl16可有效鉴定睡莲属植物,通过聚类分析发现这些种质属于Brachyceras亚属和Lotos亚属,但无法更进一步分类;苏群等(2020)采用ISSR分子标记分析了睡莲种质的亲缘关系,分析结果与黄国振等(2009)的睡莲传统分类结果基本一致。【本研究切入点】当前我国睡莲产业发展迅速,各地涌现了多家睡莲新品种选育的科研机构和企业,但随着众多新种质投入生产使用,由于原引进品种的管理不规范,导致新种质的遗传背景未能有效辨析清晰,出现了命名混乱、图片与种苗实物不对版等现象,严重制约了睡莲种质资源的创新利用。采用分子标记技术了解睡莲种质资源之间的亲缘关系远近是杂交育种的基础,快速鉴定杂交后代有助于加快种质创新进程。虽然在睡莲种质资源方面已有表型和分子标记的相关研究,但未见利用SRAP分子标记分析睡莲属植物遗传多样性方面的研究报道。【拟解决的关键问题】采用SRAP分子标记技术对从国内外引进的112份睡莲种质资源及自主杂交选育的35份杂交后代进行遗传多样性分析,并对原生种和自主选育杂交后代进行亲缘关系鉴定,同时采用10对SRAP引物构建睡莲指纹图谱,为后期杂交育种和种质创新利用提供科学依据和技术支持。
1 材料与方法
1. 1 试验材料
供试的147份睡莲属植物材料采集于广西亚热带作物研究所睡莲种质资源圃和广西平果华莲科技研究所,样品编号及名称详见表1。于2020年8月晴朗天气下选取无病虫害、生长旺盛的植株,取其内圈或水下的幼嫩叶片2~3片,用吸水纸擦干叶片表面水分,至于装有1/3硅胶的样品袋中,室温保存3 d,去除硅胶,封好封口,置于干燥器内室温保存。
1. 2 试验方法
1. 2. 1 DNA提取 采用传统CTAB法提取睡莲叶片DNA,采用超微量紫外光度计NanoDrop 2000检测DNA浓度,并逐个稀释至25 ng/μL,-20 ℃冰箱保存备用。
1. 2. 2 SRAP-PCR扩增 采用反应体系20.0 μL:10 μL 2×EasyTaq? PCR SuperMix(+dye),10 μmol/L正、反向引物各0.5 μL,DNA模板2.0 μL,ddH2O补足至20.0 μL。PCR扩增程序:94 ℃ 3 min;94 ℃ 30 s,36 ℃ 50 s,72 ℃ 1.5 min,进行40个循环;72 ℃延伸10 min。PCR产物4 ℃保存备用。采用2%琼脂糖凝胶电泳进行检测,电泳液采用1×TAE buffer。
1. 2. 3 SRAP引物筛选 以植物形态学分类为依据,分别从5个亚属各选1个原生种DNA为模板DNA,5个原生种分别为粉巨睡莲(Nymphaea gigantea)、雪白睡莲(Nymphaea candida)、埃及白睡莲(Nymphaea lotus var. pubescens)、小雪夜(Nymphaea potamophila)、美洲白睡莲(Nymphaea ampla)。根据文献查阅大蒜(周静,2011)、荷花(孙祖霞,2014)、野生皂荚(田红红,2022)等其他作物SRAP引物序列,分别从25条上游引物和15条下游引物中随机选取5条正向引物(Me1~Me5)和5条反向引物(Em1~Em5),两两组合共获得25个引物对,从中筛选出10个扩增条带清晰可辨、多态性高的引物(表2),用于后续147份睡莲属植物材料的遗传多样性分析。
1. 2. 4 遗传多样性和聚类分析 以PCR扩增产物在相同迁移率位置上的条带有无进行计数,有条带的记为1,无条带的记为0,制作成0/1矩阵。采用Excel 2010对总位点数、多态性位点数及多态性比率进行统计分析,并利用Popgene 1.32计算观测等位基因数(Na)、有效等位基因数(Ne)、Neis遗传多样性指数(H′)和Shannon信息指数(I)等4个遗传多样性参数。利用0/1矩阵,采用NTSYS 2.1计算遗传相似系数(GS)和遗传距离(GD),进一步采用非加权组平均法(UPGMA)进行聚类分析,并构建聚类图。
1. 2. 5 DNA分子身份证构建 基于10对引物对147份睡莲属植物材料的扩增结果,获得每个睡莲样品扩增的条带数据,将条带数据按照统一的扩增引物顺序进行串联排列,即可得到每个睡莲样品的以0/1扩增条带表示的字符串DNA分子身份證(白晓倩等,2022)。结合种质的基本信息,利用二维码生成技术(http://cli.im/)将对应字符串生成直观的可扫描的二维码分子身份证。
2 结果与分析
2. 1 SRAP引物筛选及多态性扩增结果
采用CTAB法提取的147份睡莲属植物材料叶片DNA,部分材料的叶片DNA电泳结果如图1所示。DNA条带清晰完整。NanoDrop 2000检测结果显示,DNA浓度均在50 ng/μL以上;OD260/OD230值为1.8~2.0,OD260/OD280值为1.80~1.95,DNA的完整性、浓度和纯度均符合后续试验要求。
筛选获得10个扩增条带数目多、条带清晰的引物对。10个引物对共扩增出207条条带,均具有多态性,多态性比率为100%(表3)。每个引物对扩增的清晰条带数为18~26,其中引物对Me1-Em3扩增出的条带最多,达26条,平均每对引物扩增的条带数为20.7条,其中引物对Me1-Em4对部分样品的扩增结果如图2所示。
由表4可知,147个份睡莲属植物材料的Na为2.0000,Ne为1.1777~1.3339,平均为1.2535,H'为0.1345~0.2230,平均为0.1835,I为0.2459~0.3703,平均为0.3155。整体来说,引物对Me1-Em1扩增获得的遗传多样性参数最低,引物对Me1-Em4扩增获得的遗传多样性参数最高。
2. 2 147份睡莲种质的SRAP聚类分析
基于10个引物对扩增结果计算供试147份睡莲属植物材料的遗传相似系数和遗传距离,结果显示,147份睡莲属植物材料的遗传相似系数为0.52~0.94,平均为0.70,其中87号材料(玛丽安)与88号材料(黑美人)的遗传相似系数最大,为0.94,说明二者的亲缘关系最近,132号材料(变色澳洲)与105号材料(山莓)的遗传相似系数最小0.52,二者亲缘关系最远。147份睡莲样品遗传距离为0.15~3.52,平均为1.17,其中94号材料(红IM)与95号材料(紫IM)的遗传距离最小为0.15,说明二者的亲缘关系最近,140号材料(蓝星睡莲)与11号材料(昼开型杂交后代9)的遗传距离最大为3.52,说明二者的亲缘关系最远。
在遗传相似系数为0.65将147份睡莲属植物材料聚为六大类群(图3-A)。第Ⅰ大类群由111份园艺杂交种和18份原生种组成,且在遗传相似系数为0.69时进一步聚为5个亚群,其中第ⅰ亚群由Nymphaea亚属的2个杂交种克罗拉多、霍兰迪亚和1个原生种小白子午莲,Anecphya亚属的2个原生种白巨和卡奔,Lotos亚属1个原生种喀麦隆,跨亚属(Nymphaea亚属×Brachyceras亚属)的1个杂交种暹罗紫1和Brachyceras亚属的全部杂交种和杂交后代组成,第ii亚群由Nymphaea亚属的9个杂交种(100~107号材料、111号材料)和跨亚属的2个(Nymphaea亚属×Brachyceras亚属)杂交种诗丽吉皇后和暹罗粉1组成,第ⅲ亚群由除喀麦隆以外的夜开型Lotos亚属的3个原生种(印度红、泰国粉、埃及白)、3个杂交种(英国红、红色闪耀、安塔利亚)和6个杂交后代组成;第ⅳ亚群由Anecphya亚属2个原生种(紫IM、白IM)、2个杂交种(流苏、红IM)和跨亚属(Anecphya亚属×Brachyceras)的2个杂交种(Tuonta、凯斯隆)组成;第ⅴ类由Nymphaea亚属2个原生种(白睡莲、雪白睡莲)和1个杂交种(佛琴娜莉斯)组成。第ⅰ类在遗传相似系数0.75时又分为5个分支,其中a分支由Brachyceras亚属的83个杂交种和杂交后代及6个原生种(卵叶睡莲、蓝蟹爪、米努它、粉色好望角、蓝色好望角、仲克尼、小花)组成;b分支由Anecphya亚属的2个原生种(白巨、卡奔)、Nymphaea亚属的1个杂交种霍兰迪亚、跨亚属(Nymphaea亚属×Brachy-ceras)的1个杂交种暹罗紫1和Lotos亚属的1个原生种喀麦隆组成;93号材料(杂交后代27)、67号材料(科罗拉多)和109号材料(小白子午莲)分别单独聚在c、d和e分支。第Ⅱ类群仅由Brachyceras亚属的134号材料(越南延药)组成,第Ⅲ类群由Nymphaea亚属的2个杂交种(黄乔伊、科曼契)和1个原生种(墨西哥黄睡莲)组成,第Ⅳ类群由Anecphya亚属的2个原生种(变色澳洲、粉巨)组成,第Ⅴ类群主要由Brachyceras亚属的9个原生种组成,第Ⅵ类群主要由夜开型Hydrocallis亚属的3个原生种(小雪夜、秘鲁睡莲、增殖睡莲)组成。在遗传相似系数0.80时,多数睡莲单独聚为一支,且多以原生种为主。
在遗传距离为1.12时将147份睡莲属植物材料聚为六大类群(图3-B),其中Brachyceras亚属的81个杂交种和杂交后代及6个原生种(卵叶睡莲、黄蟹爪、蓝蟹爪、米努它、粉色好望角、蓝色好望角)聚为第Ⅰ类群,杂交种热带风情和Brachyceras亞属的杂交后代9分别单独聚为第Ⅱ和Ⅵ类群;Nymphaea亚属的杂交种科罗拉多原生种小白子午莲聚为第Ⅲ类群;Anecphya亚属、Lotos亚属、Hydrocallis亚属的全部原生种(小雪夜睡莲、秘鲁睡莲、增殖睡莲)、Nymphaea亚属的3个原生种(雪白睡莲、白睡莲、墨西哥黄睡莲)和4个杂交种(佛琴娜莉斯、霍兰迪亚、科曼契、黄乔伊)、跨亚属(Anecphya亚属×Brachyceras亚属)的2个杂交种(Tuonta、凯斯隆)、Brachyceras亚属的10个原生种(白蓝星、蓝星、越南延药、印度延药、海南延药、非洲延药、优雅睡莲、埃及蓝、美洲白睡莲、美洲白睡莲红叶)聚为第Ⅳ类群;剩余Nymphaea亚属的1个原生种(小白子午莲)、10个杂交种和跨亚属(Nymphaea亚属×Brachyceras亚属)的3个杂交种聚为第Ⅴ类。其中第Ⅴ类群的进一步分类将Nymphaea亚属的1个原生种(小白子午莲)、10个杂交种聚在一起,跨亚属(Nymphaea亚属×Brachyceras亚属)的3个杂交种聚在一起,与实际形态学分类一致。比较复杂的第Ⅳ类群进一步在遗传距离0.85聚为5个亚群:第ⅰ亚群除93号以外的91~99号材料组成,包含Anecphya亚属的4个原生种(白巨、卡奔、紫IM、白IM)、2个杂交种(红IM、流苏)以及跨亚属(Anecphya亚属×Brachyceras亚属)的2个杂交种(Tuonta、凯斯隆);第ⅱ亚群由Nymphaea亚属的2个原生种(白睡莲、雪白睡莲)和2个杂交种(佛琴娜莉斯、霍兰迪亚)组成;第ⅲ亚群由130~147号材料组成,包含了Hydrocallis亚属的3个原生种(小雪夜睡莲、秘鲁睡莲、增殖睡莲)、Brachyceras亚属的10个原生种(白蓝星、蓝星、越南延药、印度延药、海南延药、非洲延药、优雅睡莲、埃及蓝、美洲白睡莲、美洲白睡莲红叶)、Anecphya亚属的2个原生种(变色澳洲、粉巨)、Nymphaea亚属的1个原生种(墨西哥黄)共计16份原生种和Nymphaea亚属的2个杂交种(科曼契、黄乔伊);Lotos亚属的原生种(喀麦隆)单独聚为第ⅳ亚群,第ⅴ亚群由除喀麦隆以外的Lotos亚属的材料(117~129号)组成,包含3个原生种(印度红、泰国粉、埃及白)、3个杂交种(英国红、红色闪耀、安塔利亚)及6个杂交后代。材料数量最多的第Ⅰ类群在遗传距离0.97时聚为4个分支,均为Brachyceras亚属睡莲种质材料,其中a分支由除11号杂交后代9以外的1~24号杂交种和杂交后代组成,b分支由除34号卵叶睡莲和84号泰王以外的25~89号杂交种组成,34号材料(卵叶睡莲)和84号材料(泰王)分别单独聚为在c和d分支。
2. 3 34份原生种和35份杂交后代的聚类分析结果
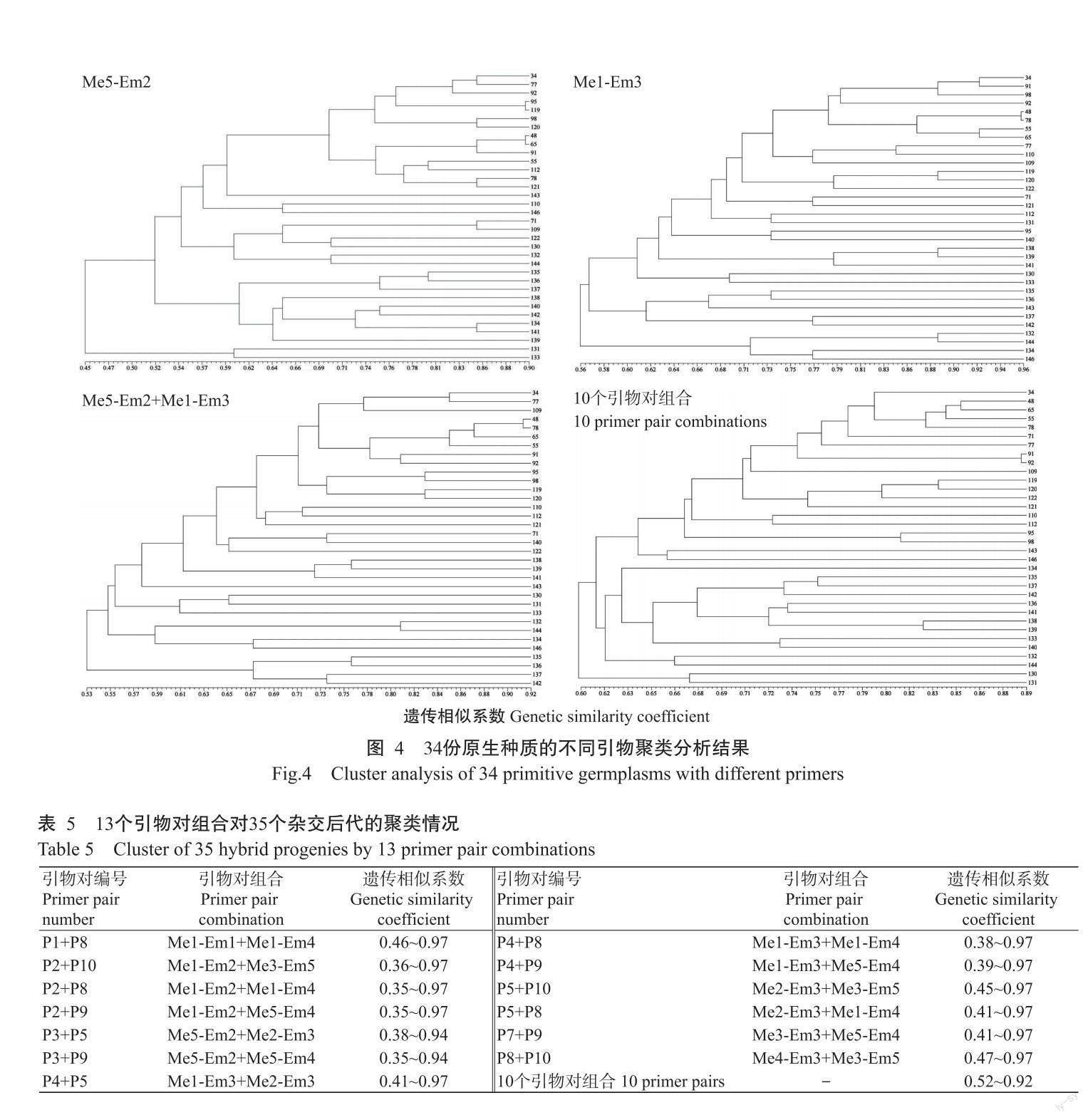
利用筛选出的10个引物对对34份原生种进行聚类分析,其中引物对Me5-Em2和Me1-Em3能够将34份原生种质完全分开。分别采用4种引物组合方式(Me5-Em2、Me1-Em3、Me5-Em2+Me1-Em3、10个引物对组合)对34个原生种进行聚类分析结果如图4所示,4种引物组合方式得到的遗传相似系数分别为0.20~0.91、0.35~0.97、0.35~0.91和0.51~0.90,分别在遗传相似系数0.59、0.62、0.58、0.64将34份原生种聚为五大类,其中4种引物组合聚类结果中均被聚为一类的材料为34号(卵叶睡莲)、48号(蓝蟹爪)、55号(米努它)、65号(粉色好望角)、77号(仲克尼)、78号(小花)、91号(卡本)、92号(白巨)、95号(紫IM)、98号(白IM)、110号(雪白睡莲)、112号(白睡莲)、119号(印度红)、120号(泰国粉)、121号(喀麦隆)、135号(印度延药)、136号(优雅睡莲)、132号(变色澳洲)、144号(粉巨)。4种引物组合方式聚类分析结果均与形态学分类具有较大的差异,如119号(印度红)、120号(泰国粉)、121号(喀麦隆)、122号(埃及白)材料在形态学上均属于热带夜开型热带睡莲,91号(卡奔)、92号(白巨)、95号(紫IM)、98号(白IM)、132号(变色澳洲)、144号(粉巨)在形态学上均属于澳洲睡莲亚属,109号(小白子午莲)、110号(雪白睡莲)、112号(白睡莲)在形态学上均属于广温带睡莲,实际的分子标记并没有将其很理想地聚在一支。
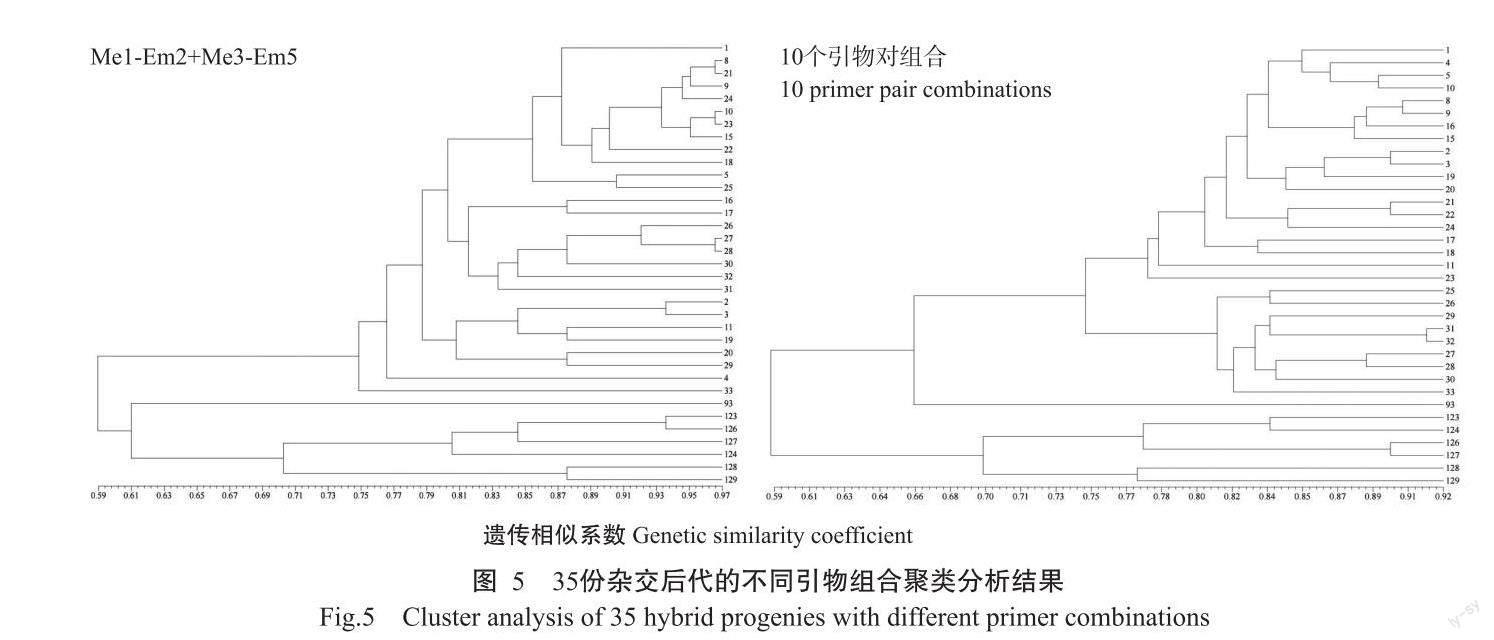
采用10个引物对对35份杂交后代进行聚类分析,结果显示单独的每个引物对均不能将35份杂交后代进行完全分开。进一步将10个引物对进行两两组合,得到13个引物组合,可完全将35个杂交后代完全分开,遗传相似系数为0.35~0.98(表5),单独的13个引物组合与10个引物对组合共同聚类的结果(图5)相似,整体上除Me1-Em2+Me3-Em5组合外,所有的夜开型Lotos亚属聚在一起,其他白天开花类型聚在一起,93号材料是以Anecphya亚属为父本,Brachyceras亚属为母本进行杂交,在所有的聚类分析结果中,最终均单独列为一支,与实际情况符合。而在Me1-Em2+Me3-Em5组合的聚类分析结果中,93号材料在遗传相似系数0.59时与夜开型的材料聚为一支,随后在遗传相似系数0.61时,单独聚为一支(图5)。
2. 4 睡莲属植物材料的DNA分子身份证构建
将147份睡莲的扩增条带结果按照P1~P10先后顺序进行串联排列,获得每个种质唯
一的237位字符串,即每个样品的字符串分子身份证,以原生种蓝星睡莲为例,生成的码如下:P1-01000010000010010000,P2-00001000100100000 1000,P3-00110100101001001000,P4-0000011100110 1011001101000,P5-01011001101000010000,P6-101 010001000000000,P7-11011010101001100010,P8-0 100110001100000100100,P9-0000001010001010000, P10-001100010101010000000;生成的二维码和扫码结果如图6。
3 讨论
SRAP分子标记最早应用于芸薹属种质资源的鉴定与评价,与ISSR和SSR等分子标记相比,具有多态性丰富、高共显性、操作简便和费用低,可省去了引物开发过程等优点(苏群等,2019;李萍萍等,2022),是植物遗传育种研究中鉴定种质亲缘关系的高效手段之一。本研究基于前人已发表的25个SRAP引物对,筛选出10个引物对即可从147份睡莲属植物中扩增出207条多态性条带,多态性比率达100%。同类物种遗传多样性研究中,苏群等(2020)利用ISSR分子标记技术对46份睡莲种质的亲缘关系进行鉴定,从100条ISSR引物中筛选的10条引物共扩增出281个多态性条带,多态性比率达100%。Parveen等(2022)基于睡莲基因组测序数据,开发了217对SSR引物并从中筛选出57对SSR引物用于97份睡莲种质的鉴定,扩增产物的多态性比率79.79%。从以上研究结果可知,相较于ISSR和SSR分子标记,将SRAP分子标记应用于睡莲种质资源遗传多样性分析可简化了引物开发步骤,提高睡莲种质鉴定效率。
Na、Ne、H和I是衡量植物物种间种质资源遗传多样性丰富度的常用指标(周丽霞和曹红星,2020)。本研究中147份睡莲属种质材料的I平均值为0.3155;Ne平均值為1.2535;H′平均值为0.1835,Na平均值为2.0000,遗传相似系数为0.52~0.94,遗传距离为0.15~3.52,说明供试的147份睡莲种质的遗传多样性丰富,具有高度广泛的遗传变异。这与油棕(周丽霞和曹红星,2020)、棕榈(毛跃雄等,2020)、黄椒(邱胤晖等,2021)、百合(王子康等,2022)等研究结果相似。研究发现,睡莲属植物普遍存在雌蕊先熟,外圈雄蕊与雌蕊同步成熟的特点(黄国振等,2009),雌蕊和雄蕊性成熟时间不同步的习性,可促进了该属植物的自身繁衍,并通过外界介质加强了种间的基因交流。这可能是造成睡莲属植物遗传多样性水平较高的重要原因。
本研究对147份睡莲属植物材料进行聚类分析,结果显示在遗传相似系数为0.65和遗传距离1.12,可将这些材料聚为六大类群,其中大部分的Brachyceras亚属杂交种聚在一起,在进一步的细化聚类种,大部分依据形态学的不同亚属能较好的聚在一起。不论是依据遗传相似系数还是依据遗传距离,跨亚属的3个Nymphaea亚属×Brachyceras亚属和2个Anecphya亚属×Brachyceras亚属杂交种均与母本所在亚属种质聚类在一起,形态学特征也与母本所在亚属种质相近,杂交新种质在遗传上更偏向于母本;而广温带睡莲许多经典品种如科罗拉多、佛琴娜莉斯等均为早期杂交种,遗传背景相对后期的杂交种更为复杂,因此两种依据的聚类方式均未将所有Nymphaea亚属完全聚在一起。本研究中多数原生种的遗传距离较远,遗传系数较小,依据远缘杂交优势,同属物种亲缘关系越远,杂交后代更易获得比母系和父系亲本更优良的性状,研究中选择的原生种可作为睡莲属种质创新中优良的育种材料,在今后的研究中应加强原生种质的收集保存,并将其应用于杂交育种中。
本研究筛选到10个引物对组合才能将147份种质有效区分,2个SRAP引物对(Me5-Em2和Me1-Em3)均能独立地将34份原生种区分,且采用单一引物对、Me5-Em2和Me1-Em3双引物对组合以及10个引物对组合的聚类结果均可将34份原生种划分为五大类群,但部分原生种聚类结果的类群归属划分有差别,且与形态学分类存在差异。这些现象可能是由于单一分子标记局限性和引物丰富度不够、覆盖面不足,或原生种之间在早期进化过程中存在着基因交流导致。因此,在今后的研究中应结合形态表型数据进行多种分子标记联合分析。
本研究35份杂交后代的鉴定结果显示,至少需要2个以上的SRAP引物对组合才能将35份杂交后代完全区分,且不同引物对组合的聚类结果均存在差异,故在杂交后代鉴定过程中需利用多对引物组合的互补作用,提高杂交后代鉴定结果的可靠性。DNA分子身份证将不同睡莲属植物DNA水平上的差异转变为字符串和二维码,可更加快捷直观地反映样品的相关信息,对于睡莲属植物的快速识别和推广具有重要意义。本研究采用多对引物组合的方式鉴定了34份原生种质和35份杂交后代,有效地克服了单一引物扩增存在的特征条带少、鉴定效率低的问题。因此,应用筛选得到的10个引物对构建的DNA指纹图谱,可为睡莲属原生种和杂交种的分子鉴定提供参考。
4 结论
147份睡莲属植物的遗传多样性丰富,原生种之间的遗传距离较大,具有极大的育种潜力。SRAP分子标记可辅助睡莲新品种选育,用于亲本的选择和杂交后代的快速鉴定。
参考文献:
白晓倩,陈于,张仕杰,赵玉强,王武,朱灿灿. 2022. 基于表型性状和SSR标记的板栗品种遗传多样性分析及分子身份证构建[J]. 植物遗传资源学报,23(4):972-984. [Bai X Q,Chen Y,Zhang S J,Zhao Y Q,Wang W,Zhu C C. 2022. Genetic diversity analysis and identification of chestnut cultivars based on phenotypic traits and SSR markers[J]. Journal of Plant Genetic Resources,23(4):972-984.] doi:10.13430/j.cnki.jpgr.20220113001.
陈倩倩,荣丽媛,邵紫君,刘甜,魏蕾,宋振巧. 2018. 利用SRAP和EST-SSR分析香椿资源的遗传多样性[J]. 园艺学报,45(5):967-976. [Chen Q Q,Rong L Y,Shao Z J,Liu T,Wei L,Song Z Q. 2018. Genetic diversity analysis of Toona sinensis germplasms based on SRAP and EST-SSR markers[J]. Acta Horticulturae Sinica,45(5):967-976.] doi:10.16420/j.issn.0513-353x.2018-0044.
程哲,王彦杰,吕存红,金奇江,杨梅花,王雪莲,叶春秀,徐迎春. 2022. 自然低温下7种热带睡莲的耐寒性评价[J]. 江西农业学报,34(5):46-52. [Cheng Z,Wang Y J,Lü C H,Jin Q J,Yang M H,Wang X L,Ye C X,Xu Y C. 2022. Evaluation of cold tolerance of seven tropical waterlily varieties under natural low temperature[J]. Acta Agriculturae Jiangxi,34(5):46-52.] doi:10.19386/j.cnki.jxnyxb.2022.05.009.
甘霖,高乐,陈丽娜,何鹏. 2021. 基于SRAP分子标记的油梨种质资源遗传多样性分析[J]. 南方农业学报,52(7):1798-1805. [Gan L,Gao L,Chen L N,He P. 2021. Genetic diversity analysis of avocado germplasm resources based on SRAP molecular markers[J]. Journal of Sou-thern Agriculture,52(7):1798-1805.] doi:10.3969/j.issn. 2095-1191.2021.07.008.
胡福初,吴小波,陈哲,吴凤芝,周文静,冯学杰,范鸿雁,周瑞云,王祥和. 2021. 基于SRAP分子标记的特早熟荔枝种质资源遗传多样性分析[J]. 热带作物学报,42(4):920-926. [Hu F C,Wu X B,Chen Z,Wu F Z,Zhou W J,Feng X J,Fan H Y,Zhou R Y,Wang X H. 2021. Gene-tic diversity analysis of very early maturing litchi germplasm resources based on SRAP molecular markers[J]. Chinese Journal of Tropical Crops,42(4):920-926.] doi: 10.3969/j.issn.1000-2561.2021.04.002.
黄国振,邓惠勤,李祖修,李刚. 2009. 睡莲[M]. 北京:中国林业出版社. [Huang G Z,Deng H Q,Li Z X,Li G. 2009. Water lily[M]. Beijing:China Forestry Publishing House.]
黄秋伟,毛立彦,龙凌云,唐毓玮. 2020. 热带睡莲精油的超临界CO2萃取优化及其成分GC-MS分析[J]. 食品研究与开发,41(7):188-195. [Huang Q W,Mao L Y,Long L Y,Tang Y W. 2020. Optimization of essential oil from tropical water lily extracted by supercritical CO2 and volatile components of essential oil analysis by GC-MS[J]. Food Research and Development,41(7):188-195.] doi:10.121 61/j.issn.1005-6521.2020.07.032.
黄祥,杨梅花,楚光明,钟敏,徐迎春,王彦杰. 2022. 耐寒睡莲种质资源分析及观赏性评价[J]. 分子植物育种,20(4):1348-1357. [Huang X,Yang M H,Chu G M,Zhong M,Xu Y C, Wang Y J. 2022. Analysis of variety resources and evaluation of ornamental value in hardy waterlily[J]. Molecular Plant Breeding,20(4):1348-1357.] doi:10.13271/j.mpb.020.001348.
李萍萍,王泽榕,王丁,莫晶晶,陈美霞,叶祖云. 2022. 基于DNA条形码和SRAP的太子参种质遗传多样性分析[J]. 热带作物学报,43(10):2037-2046. [Li P P,Wang Z R,Wang D,Mo J J,Chen M X,Ye Z Y. 2022. Genetic diversity analysis of Pseudostellaria heterophylla germplasms based on DNA barcoding and SRAP[J]. Chinese Journal of Tropical Crops,43(10):2037-2046.] doi:10. 3969/j.issn.1000-2561.2022.10.009.
李淑娟,尉倩,陈尘,张燕,吴永朋,余刚. 2019. 中国睡莲属植物育种研究进展[J]. 植物遗传资源学报, 20(4):829-835. [Li S J,Wei Q,Chen C,Zhang Y,Wu Y P,Yu G. 2019. Breeding progress of waterlilies in China[J]. Journal of Plant Genetic Resources,20(4):829-835.] doi:10. 13430/j.cnki.jpgr.20181209001.
林榕燕,葉秀仙,钟淮钦,林兵,黄敏玲. 2018. 基于SRAP分子标记的石斛兰种质资源遗传多样性分析[J]. 福建农业学报,33(5):469-473. [Lin R Y,Ye X X,Zhong H Q,Lin B,Huang M L. 2018. Genetic diversity of Dendrobium germplasms accessed by SRAP makers[J]. Fujian Journal of Agricultural Sciences,33(5):469-473.] doi:10.19303/j. issn.1008-0384.2018.05.004.
林增顺,刘冠明,徐庆国. 2019. 利用SRAP标记分析水稻新品系遗传多样性[J]. 基因组学与应用生物学,38(4):1683-1688. [Lin Z S,Liu G M,Xu Q G. 2019. Analysis of genetic diversity of new rice lines by SRAP markers[J]. Genomics and Applied Biology,38(4):1683-1688.] doi:10.13417/j.gab.038.001683.
卢丽芳,朱海生,温庆放,林义章. 2015. 南瓜种质资源遗传多样性的SRAP分析[J]. 热带作物学报,36(12):2142-2148. [Lu L F,Zhu H S,Wen Q F,Lin Y Z. 2015. Ana-lysis of genetic diversity in squash by SRAP markers[J]. Chinese Journal of Tropical Crops,36(12):2142-2148.]doi:10.3969/j.issn.1000-2561.2015.12.006.
毛立彦,黄秋伟,龙凌云,檀小辉,谢红辉,唐毓玮,丁丽琼. 2022. 7种睡莲属植物叶绿体基因组密码子偏好性分析[J]. 西北林学院学报,37(2):98-107. [Mao L Y,Huang Q W,Long L Y,Tan X H,Xie H H,Tang Y W,Ding L Q. 2022. Comparative analysis of codon usage bias in chloroplast genomes of seven Nymphaea species[J]. Journal of Northwest Forestry University,37(2):98-107.] doi:10.3969/j.issn.1001-7461.2022.02.13.
毛跃雄,陆跃堂,胡志姣,赵杨. 2020. 基于SRAP分子标记的贵州省棕榈种质资源遗传多样性分析[J]. 南方农业学报,51(1):27-35. [Mao Y X,Lu Y T,Hu Z J,Zhao Y. 2020. Genetic diversity of Trachycarpus fortunei(Hook.) H. Wendl germplasm resources in Guizhou by SRAP mole-cular markers analysis[J]. Journal of Southern Agriculture,51(1):27-35.] doi:10.3969/j.issn.2095-1191.2020.01.004.
潘庆龙,付瑛格,谷佳,盛玉辉,李清雪,饶英,朱天龙,周扬,史佑海,趙莹,王健. 2021. 海南引种睡莲表型多样性分析及评价[J]. 热带作物学报,42(10):2777-2788. [Pan Q L,Fu Y G,Gu J,Sheng Y H,Li Q X,Rao Y,Zhu T L,Zhou Y,Shi Y H,Zhao Y,Wang J. 2021. Analysis of phenotypic diversity of Nymphaea L. in Hainan,China[J]. Chinese Journal of Tropical Crops,42(10):2777-2788.] doi:10.3969/j.issn.1000-2561.2021.10.005.
邱胤晖,林淑婷,曾绍贵,罗英,朱邦彤,李永清. 2021. 41份黄椒种质资源表型及SRAP遗传多样性分析[J]. 南方农业学报,52(8):2165-2173. [Qiu Y H,Lin S T,Zeng S G,Luo Y,Zhu B T,Li Y Q. 2021. Phenotypic and SRAP genetic diversity analysis of 41 yellow pepper germplasm resources[J]. Journal of Southern Agriculture,52(8):2165-2173.] doi:10.3969/j.issn.2095-1191.2021.08.015.
沈瑶,王晗璇,侯海娴,吴智明,周厚高. 2020. 基于表型和SRAP标记的盆栽菊遗传多样性分析[J]. 热带作物学报,41(11):2156-2164. [Shen Y,Wang H X,Hou H X,Wu Z M,Zhou H G. 2020. Genetic diversity analysis of the potted chrysanthemum based on phenotype and SRAP marker[J]. Chinese Journal of Tropical Crops,41(11):2156-2164.] doi:10.3969/j.issn.1000-2561.2020. 11.003.
宋杰,郭子雨,姚雨含,李晨阳,赵军. 2021. 睡莲多酚提取工艺优化及其抗油脂氧化作用[J]. 中国粮油学报,36(10):149-153. [Song J,Guo Z Y,Yao Y H,Li C Y,Zhao J. 2021. Extraction optimization of total polyphenols from Nymphaea candida and its antioxidant activity on oil[J]. Journal of the Chinese Cereals and Oils Association,36(10):149-153.] doi:10.3969/j.issn.1003-0174. 2021.10.024.
宋敏,李晨阳,谭为,赵军,徐芳,敬思群. 2016. 雪白睡莲花中烟花苷的提取工艺研究[J]. 食品研究与开发,37(9):128-131. [Song M,Li C Y,Tan W,Zhao J,Xu F,Jing S Q. 2016. Optimization of extraction technology of nicotiflorin from Nymphaea candida Presl.[J]. Food Research and Development,37(9):128-131.] doi:10.3969/j.issn. 1005-6521.2016.09.029.
苏群,杨亚涵,田敏,卜朝阳,毛立彦,张进忠,潘介春,卢家仕. 2020. 睡莲种质资源遗传多样性分析及DNA指纹图谱构建[J]. 热带作物学报,41(2):258-266. [Su Q,Yang Y H,Tian M,Bu C Y,Mao L Y,Zhang J Z,Pan J C,Lu J S. 2020. Genetic diversity analysis and DNA fingerprinting construction of waterlily germplasm resources[J]. Chinese Journal of Tropical Crops,41(2):258-266.] doi:10.3969/j.issn.1000-2561.2020.02.008.
苏群,杨亚涵,田敏,张进忠,毛立彦,唐毓玮,卜朝阳,卢家仕. 2019. 49份睡莲资源表型多样性分析及综合评价[J]. 西南农业学报,32(11):2670-2681. [Su Q,Yang Y H,Tian M,Zhang J Z,Mao L Y,Tang Y W,Bu C Y,Lu J S. 2019. Phenotypic diversity analysis and comprehensive evaluation of 49 waterlily resources[J]. Southwest China Journal of Agricultural Sciences,32(11):2670-2681.] doi:10.16213/j.cnki.scjas.2019.11.027.
孙祖霞. 2014. 荷花种质资源遗传多样性的SRAP和ISSR研究[D]. 南京:南京农业大学. [Sun Z X. 2014. Study on genetic diversity and phylogenetic relationship of Nelumbo nucifera by SRAP and ISSR markers[D]. Nanjing:Nanjing Agricultural University.]
田紅红. 2022. 贵州省野生皂荚资源遗传多样性研究[D]. 贵阳:贵州大学. [Tian H H. 2022. Genetic diversity of wild Gleditsia sinensis resources in Guizhou Province[D]. Guiyang:Guizhou University.]
王子康,苏江硕,张雪峰,宁心怡,周迎雪,房伟民,陈发棣,张飞. 2022. 32个百合品种遗传多样性分析和DNA指纹图谱构建[J]. 植物资源与环境学报,31(5):58-65. [Wang Z K,Su J S,Zhang X F,Ning X Y,Zhou Y X,Fang W M,Chen F D,Zhang F. 2022. Analysis on genetic diversity and construction of DNA fingerprinting of 32 Lilium spp. cultivars[J]. Journal of Plant Resources and Environment,31(5):58-65.] doi:10.3969/j.issn.1674-7895. 2022.05.07.
吴立东,林淑婷,刘亚婷,钟金仙,罗英. 2019. 基于SRAP标记和主要表型性状的苦瓜优株选择与遗传分析[J]. 江苏农业学报,35(5):1175-1183. [Wu L D,Lin S T,Liu Y T,Zhong J X,Luo Y. 2019. Genetic analysis and superior plant selection of bitter gourd based on SRAP markers and main phenotypic traits[J]. Jiangsu Journal of Agricultural Sciences,35(5):1175-1183.] doi:10.3969/j.issn. 1000-4440.2019.05.025.
叶兴状,张明珠,刘益鹏,文国卫,赖文峰,范辉华,张国防,刘宝. 2021. 基于SRAP标记半枫荷天然种群的遗传多样性分析[J]. 植物资源与环境学报,30(4):60-68. [Ye X Z,Zhang M Z,Liu Y P,Wen G W,Lai W F,Fan H H,Zhang G F,Liu B. 2021. Analysis on genetic diversity of natural populations of Semiliquidambar cathayensis based on SRAP marker[J]. Journal of Plant Resources and Environment,30(4):60-68.] doi:10.3969/j.issn.1674-7895. 2021.04.07.
袁媛,曹彬,張咏琪,陈清西,陈南川. 2020. 基于SRAP标记的国兰种质资源遗传多样性分析[J]. 热带作物学报,41(5):929-938. [Yuan Y,Cao B,Zhang Y Q,Chen Q X,Chen N C. 2020. Genetic diversity analysis of Cymbidium germplasms based on SRAP markers[J]. Chinese Journal of Tropical Crops,41(5):929-938.] doi:10.3969/j.issn. 1000-2561.2020.05.012.
张坤,周源洁,李尧,刘芯伶,郭玉琪,夏惠,梁东. 2021. 基于SRAP和SCoT标记的猕猴桃种质遗传多样性分析及变异材料鉴定[J]. 果树学报,38(12):2059-2071. [Zhang K,Zhou Y J,Li Y,Liu X L,Guo Y Q,Xia H,Liang D. 2021. Genetic diversity analysis of kiwifruit germplasm and identification of variant based on SRAP and SCoT markers[J]. Journal of Fruit Science,38(12):2059-2071.] doi:10.13925/j.cnki.gsxb.20210264.
张妹,何正权,马江,桑子阳,朱仲龙,张德春,马履一,陈发菊. 2019. 基于SSR和SRAP标记的红花玉兰品种遗传关系分析及分子鉴定[J]. 北京林业大学学报,41(9): 69-80. [Zhang M,He Z Q,Ma J,Sang Z Y,Zhu Z L,Zhang D C,Ma L Y,Chen F J. 2019. Genetic relationship analysis and molecular identification of Magnolia wufengensis cultivars based on SSR and SRAP markers[J]. Journal of Beijing Forestry University,41(9):69-80.] doi:10.133 32/j.1000-1522.20190204.
赵军,徐芳,吉腾飞,顾政一,李晨阳. 2014. 睡莲属植物化学成分及生物活性研究进展[J]. 天然产物研究与开发,26(1):142-147. [Zhao J,Xu F,Ji T F,Gu Z Y,Li C Y. 2014. Advances in the study on chemical constituents and biological activities in Nymphaea genus[J]. Natural Product Research and Development,26(1):142-147.] doi:10.16333/j.1001-6880.2014.01.027.
周静. 2011. 大蒜种质亲缘关系的形态学和分子标记研究[D]. 杨凌:西北农林科技大学. [Zhou J. 2011. Study on genetic relationship of garlic (Allium sativum) germplasm resources by morphological and molecular techniques[D]. Yangling:Northwest A & F University.]
周丽霞,曹红星. 2020. 基于SRAP分子标记的不同种壳厚度油棕种质资源遗传多样性分析[J]. 南方农业学报,51(9):2097-2103. [Zhou L X,Cao H X. 2022. Genetic diversity analysis of oil palm germplasm resources with different seed shell thicknesses based on SRAP molecular marker[J]. Journal of Southern Agriculture,51(9):2097-2103.] doi:10.3969/j.issn.2095-1191.2020.09.006.
Danial S,Mehdi R,Ghodratollah S. 2021. Polyphenolic and molecular variation in Thymus species using HPLC and SRAP analyses[J]. Scientific Reports,11(1):5019. doi:10.1038/s41598-021-84449-6.
Jeremy D,Suman K,Satyawada R R,Pramod T. 2013. New insights into character evolution,hybridization and diversity of Indian Nymphaea (Nymphaeaceae):evidence from molecular and morphological data[J]. Systematics and Biodiversity,11(1):77-86. doi:10.1080/14772000.2013. 773949.
Li G,Quiros C F. 2001. Sequence-related amplified polymorphism (SRAP),a new marker system based on a simple PCR reaction:Its application to mapping and gene tagging in Brassica[J]. Theoretical and Applied Genetics,103:455-461. doi:10.1007/s001220100570.
Parveen S,Singh N,Adit A,Kumaria S,Tandon R,Agarwal M,Jagannath A,Goel S. 2022. Contrasting reproductive strategies of two Nymphaea species affect existing natural genetic diversity as assessed by microsatellite mar-kers:Implications for conservation and wetlands restoration[J]. Frontiers in Plant Science,13:773572. doi:10. 3389/fpls.2022.773572.
Péter P,Kinga K M,István S,Ildikó V,Jaakko H,István C,Ahmad M G, Kincs? D,János T. 2011. Genetic variability of thermal Nymphaea (Nymphaeaceae) populations based on ISSR markers:Implications on relationships,hybridization,and conservation[J]. Plant Molecular Biology Reporter,29:906-918. doi:10.1007/s11105-011-0302-9.
Qian Z Z,Justus M M,Wang Q F,Itambo M,Li Z Z,Chen J M. 2022. Molecular identification of African Nymphaea species (Water Lily) based on ITS,trnT-trnF and rpl16[J]. Plants,11(18):2431. doi:10.3390/plants11182431.
Agnihotri V,ElSohly H N,Khan S I,Jacob M,Tekwani B J,Khan I A,Walker L A. 2008. Antioxidant constituents of Nymphaea caerulea flowers[J]. Phytochemistry,69(10):2061-2066. doi:10.1016/j.phytochem.2008.04.009.
(責任编辑 陈 燕)

