基于分子生成模型的SOS1抑制剂衍生物设计
成凯阳 胡晨骏 胡孔法


关键词:数据增强;强化学习;虚拟筛选;多样性
中图分类号:TP399 文献标识码A 文章编号:1006-8228(2023)11-94-06
0 引言
在传统的药物设计中,药化专家通过实验及高通量筛选(High Throughput Screening,HTS)[1]从而发现具有潜在活性的化合物。然而仅通过实验方法筛选药物,耗时耗力,以计算机辅助药物设计(Computer-Aided Drug Design,CADD)[2]或通过AI 算法设计更多类药化合物已成为药物研究的重要内容。
近年来,以分子生成模型为代表的药物设计方法为先导化合物发现和优化提供了新途径。如AMABILINO等人提出的递归神经网络(Recurrent Neural Network,RNN)运用迁移学习方法扩展化合物库[3],但也存在一些问题,即当时间步长较大或循环网络层数较深时,RNN 在采样分子时可能会出现梯度消失或梯度爆炸的情况,导致模型生成有效分子数量较少。此外,Bagal 等人提出的Transformer 模型[4]使用了自注意力机制,具有处理长序列的能力和并行计算的优势。但在生成过程中,通常使用固定的采样策略生成序列,这可能导致生成的分子缺乏多样性。
由于激酶靶点的成药性高,本文选择SOS1 靶点进行设计与研究[5]。通过设计SOS1 抑制剂的衍生物希望发现潜在能抑制SOS1 活性的药物,从而抑制肿瘤细胞的生长和扩散。因此,本文提出一种结合RNN与Transformer 的R-T 算法来设计分子,该方法避免了单独使用RNN 所存在的梯度消失的问题。通过减少模型的复杂度,可以实现更快的训练收敛速度,相较于Transformer 可以生成更多样化的分子序列。此外,进一步运用SMILES 枚举的数据增强方法扩充数据集以生成更多样化的分子结构。最后用SOS1 抑制剂对R-T 模型进行微调,并以MRTX0902 为例来设计其衍生物,从而为激酶抑制剂的研究和开发提供新的思路和方法。
1 生成模型相关工作
在分子生成领域中,Blaschke 等人提出的变分自编码器(Variational Autoencoders,VAE)架构[6],该方法通过对编码器的隐空间进行优化,并加入正态分布的噪声以及惩罚,以减少重构误差的方式生成与多巴胺受体(DDR2)化合物属性分布近似的化合物。实验证明,基于隐空间优化的方法在指导化合物优化目标属性方面具有显著效果。
此外,Segler 等人[7]首次利用RNN 模型运用于分子生成并证明了该方法的可靠性,先是利用大规模的生物活性数据对RNN 模型进行预训练,然后根据PPARγ 靶标的活性化合物对模型做进一步的微调从而设计靶向PPARγ 的化合物。Kim 等人提出的Transformer 模型[8]在生成分子的有效性及新颖性上相较于RNN 更有优势,由于其多头注意力机制及并行计算的能力使得在少量训练轮次即可达到收敛状态。同时,相关研究发现,通过对SMILES 枚举的方法可以较好地实现生成分子的多样性[9]。因此本文运用数据增强方法,先对Transformer 模型做预训练,并将其采样的分子通过调整RNN 模型参数来生成更多样化的化学结构。
2 数据收集及预处理
首先从ChEMBL 数据库[10]中下载70 万条SMILES格式的分子,数据预处理流程如图1A 所示,首先过滤掉同位素及相同结构的分子,并进一步移除PAINS 结构以及不满足于类药五原则的分子,最终得到50112个分子。其中类药五原则为Lipinski[11]等人提出的筛选类药分子的五条基本法则。此外,微调数据集为ChEMBL 中现有对SOS1 靶标有pIC50 值的1329 个分子,并从中筛选掉多羟基、重原子数量大于40 及pIC50小于4.5,最终得到222 个分子(图1B)。
3 模型改进方法
3.1 R-T 算法流程
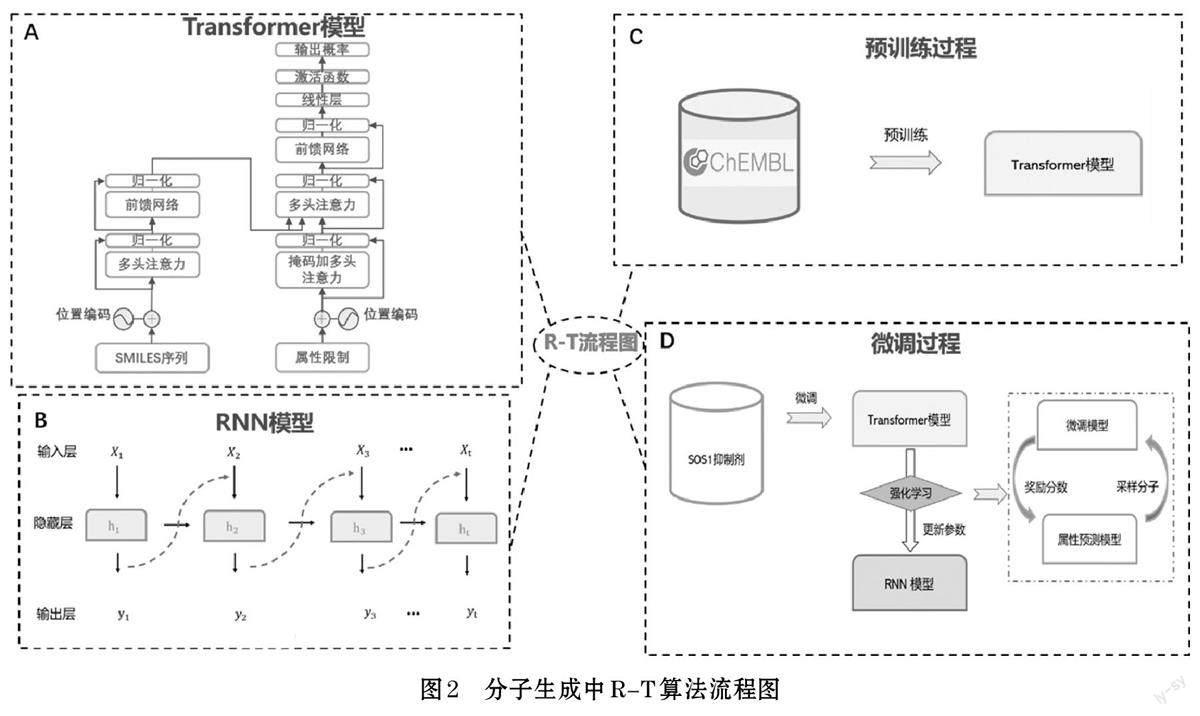
本文所用的R-T算法框架如图2所示,Transformer模型及RNN 模型如图2A 和图2B 所示,其中RNN 模型由输入层,隐藏层及输出层所组成。Transformer 模块是由编码块以及解码块堆叠而成。每个编码块包含一个多头注意力层和一个前馈网络(feed forwardnetwork,FFN)。在解码块中有三个子层,分别是掩码加自注意力子层、多头编码器-解码器注意力子层和前馈网络子层。其中多头自注意力可以捕捉到全局的依赖关系,通过位置编码使得模型能够关注输入SMILES 序列中每个字符的位置信息,并将相关信息传递给后续的层。
首先,本文用预处理后的数据集对Transformer 模型进行预训练(图2C),并用SOS1抑制剂来对Transformer模型进行微调,从而使R-T 学习这批分子的属性分布。其次,根据强化学习方法将Transformer 对RNN模型进行蒸馏,从而指导RNN 模型采样分子,并不断迭代来更新模型参数,使得在較少的训练轮次下采样出期望属性的分子(图2D)。
分子相似性表示为基于拓扑指纹和谷本相似性方法指纹计算分子之间的相似性,即将分子转化(编码)为比特位串,若存在该子结构则该位为1,否则为0。本文选用两个分子表示向量之间的Tanimoto 距离来表示两个分子间的相似性分数。
此外,本实验选择了MOSES 提供的评价指标[15]来评价生成分子的属性分布。包括logP(the octanolwaterpartition coefficient, 脂水分配系数),SAscore(Synthetic Accessibility Score,合成可及性),用来评价合成的难易,该指标范围在1 到10 区间内,越低越易于合成。
4.2 实验设置
本文采用Colab pro Tesla V100(32G),为了防止训练时产生过拟合,引入了早停机制,即当验证集的损失在20 个epoch 不下降就停止训练,具体参数如表1 所示。
4.3 具体实验过程
4.3.1 模型训练的损失曲线
本文设置预训练轮数为120 次,由图3 可知,我们可视化了R-T 模型在训练集和验证集的损失曲线。第100 个轮次损失值降低到0.06,并保持20 个轮次不变,至此预训练过程结束。
4.3.2 预训练模型比较
为了比较R-T 方法在分子生成中的优势,分别对RNN、Transformer 及Blaschke 等人提出的VAE 模型进行预训练,在训练完毕后,对上述三种方法随机采样10000 个分子来比较生成分子的质量。如表2 所示,可以发现,R-T 模型的四个指标都在0.9 以上,且在多样性、唯一性、新颖性指标中,R-T 相较于仅使用单一模型上效果更好。具体而言,在多样性指标中,相较于排名第二的Transformer 增长了0.053,而在合法性指标中也仅次于最优值0.016。由此可见,R-T 模型生成的分子质量相较于上述方法有竞争性的优势。
4.3.3 数据增强方法比较
对于阿司匹林(Aspirin)药物分子而言,虽然以不同的原子作为起点出发遍历分子图得到不一样的SMILES,但是最终的分子图是一样的(图4)。
为了验证不同的数据增强方法是否可以改善R-T模型生成分子的多样性,首先根据SMILES 枚举算法将预训练数据集中的每个分子都分别枚举五次和十次,并与不采用数据增强方法构成的训练集来分别训练R-T 模型。预训练结束后,再从三个模型中分别采样了1000 个分子。首先为每个分子提取了其1024 位的分子指纹向量。随后,使用t-SNE 算法对分子表征进行降维,并通过可视化展示来展现他们的化学空间分布。如图5 所示,基于枚举十次的方法训练出的RT模型采样得到的新颖结构基本上覆盖了前两种采样方法得到的化学空间,且覆盖面积更广,可见该方法生成的分子较上述方法更多样化。
4.3.4 MRTX0902 分子衍生物设计
MRTX0902 是目前已报道的口服SOS1 抑制剂,有助于阻断异常的细胞增殖和转移过程[16]。本研究以它为例通过强化学习方法来设计其衍生物[17]。具体而言,首先设置生成分子与MRTX0902 相似性分数之间的阈值为0.5,模型在生成的过程中会将相似性评分值大于0.5 的给予一个较高的奖励值,并将满足条件的分子保留下来,直到数量达到设定值则停止迭代。与之类比,从Specs 数据库[18]中搜索与MRTX0902 相似度大于0.5 的分子。并且根据搜索时间、搜索数量、分子多样性及可合成性来比较R-T 方法相较于搜索化合物库的显著优势[19]。本研究首先从Specs 中随机采样50000、100000、150000 个分子来作为基准比较。Specs 库的搜索时间为遍历整个化合物库所需时间,R-T 的搜索时间为生成满足数量的分子所需时间,在这里设定生成分子的数量为10000。结果如表3 所示,R-T 方法在时间略逊色于上述三种筛选方法的情况下,能够生成数量、多样性远超基于Specs 库的搜索方法,且可合成性分数更低,可见该方法的优势。此外,如图6 所示,还可视化了R-T 方法生成的四个与MRTX0902 相似度较高的化合物,其中红框标记的化合物出现于Reaxy 数据库中,可见该方法生成分子的合理性及可靠性,有助于扩展现有的化合物库,弥补虚拟筛选化合物库[20]多样性不足的问题。
4.4 实验总结
改进的R-T 算法使用强化学习方法来设计SOS1 抑制剂的衍生物,改善了传统虚拟筛选方法中筛选与SOS1抑制剂结构相似且新颖的化合物数量不足的问题。实验结果表明,相较于仅使用单一模型的情况下,R-T算法在生成分子的唯一性、合法性、新颖性和多样性方面具有明显的优势。此外,相较于未使用SMILES枚举的数据增强方法,通过对预训练集进行数据增强可以使得生成的化合物具有更高的多样性。同时,在设计MRTX0902 小分子衍生物时,R-T 模型可以快速生成数量更多、更多样化且易于合成的结构,从而更好地填补现有化合物库所涉及的化学空间不足的问题。
5 結束语
本文采用强化学习方法,来设计SOS1 抑制剂的衍生物。首先通过海量分子对Transformer 模型进行训练,再调整RNN 模型的参数从而生成更多样的化合物。实验数据显示,改进的R-T 算法性能相较于单独使用一种模型生成分子的质量明显更优。此外还比较了使用数据增强扩充训练集的方法设计分子的优势,表明该增强方法的有效性。进一步,与虚拟筛选的相似性搜索策略相比,发现改进的R-T 算法在生成分子的多样性和数量方面较有优势。总之,该方法为药物化学专家进行先导化合物的优化提供了便利。未来的研究,将继续探索在不同的激酶数据集上设计和优化小分子衍生物的能力。

