基于生物信息学的医学基础课程案例设计
王 哲,祁 冉,刘媛媛,高 路△
1.中国人民解放军海军军医大学基础医学院生理学教研室,上海 200433;2.中国人民解放军海军航空大学青岛校区航空工程基础教研室,山东青岛 264001
随着高通量测序技术的不断发展,其在医学临床上的应用日益广泛,例如,高通量测序和生物信息学分析可为出生缺陷三级预防、遗传病辅助诊断和肿瘤靶向治疗等提供依据[1]。医学院校的学生不论将来从事临床工作还是相关领域的基础研究工作,都需要掌握一定的生物信息学知识和数据分析技能,以配合临床检测报告解读和研发新的诊断、治疗方法等。这些生物信息学知识与医学基础主干课程具有天然的联系,因此,在医学基础课程教学中适当增加生物信息学分析示例教学对学生深入理解基础知识、提高综合素质具有重要意义。但是,生物信息学是宏观概念,其研究范围广、相关方法繁杂,涉及多种计算机语言编程和数学模型算法,与医学知识结构存在较大差异。因此,对医学生进行生物信息学教学需要从学生的专业背景、知识结构和目标需求等角度出发,科学设立教学目标、规划教学内容、探索教学方法,在促进学生综合素质的同时,不过多增加学生的课程负担。因此,本研究尝试将生物信息学的方法、实际操作与学生的专业学习相结合。在理论课程中寻找一个知识点,结合权威文献介绍相关研究的最新进展,对文献涉及的主要生物信息学方法进行理论讲解、分析示范和结果解读,让学生初步了解分析步骤,掌握分析结果的解读方法,有兴趣的学生可以进一步进行实战分析练习和分组讨论。以期让学生在扩展专业知识、了解国际前沿进展的同时学习生物信息学分析方法。相对于知识灌输型的传统课堂,在课程设计过程中结合“以学生为中心”的教学理念[2-4],最大程度发掘学生的自主学习能力和自我能动性。本研究结合《生理学》内分泌章节的相关知识点[5],设计了一次课程的基本教学内容,现报道如下。
1 材料与方法
1.1知识点引入 在《生理学》内分泌章节中,下丘脑-垂体系统是其重点内容。首先对该知识点进行简要回顾[5]:(1)下丘脑神经细胞分泌促释放激素和释放抑制激素,腺垂体释放促靶腺激素和直接作用激素,包括生长素、催乳素、促黑激素、促甲状腺激素、促肾上腺皮质激素、促卵泡激素和促黄体激素等[2]。(2)生长素在促进生长过程中具有重要作用,幼年时期生长素分泌不足,可导致侏儒症;幼年时期生长素分泌过多,可导致巨人症,若成年时期生长素分泌过多,可导致肢端肥大症,其中垂体瘤是造成生长素分泌过多的重要原因之一。
1.2知识点延伸
1.2.1对垂体神经内分泌瘤进行延伸介绍 (1)垂体神经内分泌瘤是最常见的神经内分泌肿瘤,发病率在脑肿瘤中居第2位。(2)临床表现:占位效应和激素过度分泌,导致视力下降、停经、溢乳、肢端肥大等,累及全身多个重要脏器[6]。(3)分类:根据激素分泌和免疫组化分为10种亚型[7-8]。在临床治疗上,明确肿瘤起源和分化程度有助于预测患者预后情况。然而现有分类方式不能明确不同肿瘤亚型的起源。因此,需要基于肿瘤起源和分化程度进行垂体瘤分子分型,从而预测肿瘤远期预后。
1.2.2介绍相关生物信息学技术 (1)传统技术的限制:传统转录组测序对组织直接进行RNA提取及建库测序,这样获得的转录组结果将各种细胞类型混合,无法区分细胞类型间的差异。如,在分析免疫微环境时,无法区分T淋巴细胞、B淋巴细胞及巨噬细胞等不同类型免疫细胞;在分析肿瘤细胞时,无法精确分辨肿瘤细胞与癌旁细胞、肿瘤干细胞与高度分化细胞等,这不利于肿瘤分子分型,也不利于分析不同特征肿瘤细胞间的差异。(2)单细胞测序:单细胞转录组测序技术的出现实现了分别对每个细胞进行建库测序[9]。通过分析单个细胞转录组特征,确定其细胞类型或细胞亚型,然后分析各个细胞类型或亚型的特征、类型间的基因表达差异及不同细胞类型间的相互作用关系等[10-11]。这有助于检测并比较正常组织器官与各亚型肿瘤细胞的特征差异,发现稀有细胞类型,揭示肿瘤细胞来源,进行精确分子分型[7]。
1.2.3前沿研究实例讲解 这部分内容可以由学生分组进行文献讲解。以ZHANG等[7]发表的论文为例。该团队通过单细胞转录组测序技术检测了21例垂体瘤组织和3例健康成人垂体前叶组织(对照)中合计64 937个细胞。解析了正常垂体的单细胞转录图谱,发现在PIT1谱系内,有一群内分泌细胞(PIT1_I)不表达经典的垂体激素(生长素、催乳素、促甲状腺激素),这群细胞最接近胚胎时期PIT1祖细胞的状态,命名为成人PIT1祖细胞。
为了探索垂体瘤的起源和分化程度,研究人员将正常垂体和垂体瘤的单细胞转录组表达谱进行整合分析;对垂体瘤各细胞亚群进行打分和重新分组,将各个谱系的垂体瘤划分为高分化组和低分化组;同时探索了每个分组特征性分子标志物、转录因子活性、富集通路等特征,筛选可能有助于预测肿瘤远期复发的标志物。最后研究人员在垂体瘤患者队列中,验证了候选标志物预测肿瘤远期复发的有效性[8]。
1.3讲解与演示 首先进行示教讲解,利用GEO数据库(https://www.ncbi.nlm.nih.gov)中GSE205418单细胞转录组数据进行基本的生物信息学分析示范。该数据是10X Genomics平台测序结果,并通过官方软件Cell Ranger v3.1.0[12]将原始数据处理为表达谱矩阵(包含 barcodes.tsv.gz,features.tsv.gz和matrix.mtx.gz),采用R语言进行下游分析[13-14]。
(1)首先,打开R软件后,加载分析需要的R包:library(Seurat)library(harmony)library(dplyr)。
(2)以其中1个样本为例,读入数据:pr_name="Shili" #项目名称,可以自己定义。input_dir ="~/Desktop/Shili_1" #barcodes.tsv.gz,features.tsv.gz和matrix.mtx.gz 3个文件的位置,这里文件名不能修改。Data1<- Read10X(data.dir = input_dir) #读入Shili_1文件夹中的文件,导入数据。Or_ident= factor(c(rep("Sample1",ncol(Data1)))) #定义样本名"Sample1"。Data1= CreateSeuratObject(counts = Data1,project = pr_name) #创建Seurat对象,用于后续分析。Data1@meta.data$orig.ident< -as.factor(Or_ident) #见过定义的样本名加入Seurat对象。Data1["percent.mt"] <- PercentageFeatureSet(object = Data1,pattern = "^mt-") #计算每个细胞表达线粒体基因的比例,一般认为表达线粒体基因比例过大的细胞,其细胞状态不好,可去掉。但是其具体标准无统一要求,可结合前人相似样本的过滤标准。P1=VlnPlot(Data1,features=c("nFeature_RNA","nCount_RNA","percent.mt"),group.by="orig.ident") #对细胞表达线粒体基因比例、细胞表达基因数和捕获UMI数进行可视化,结果见图1。
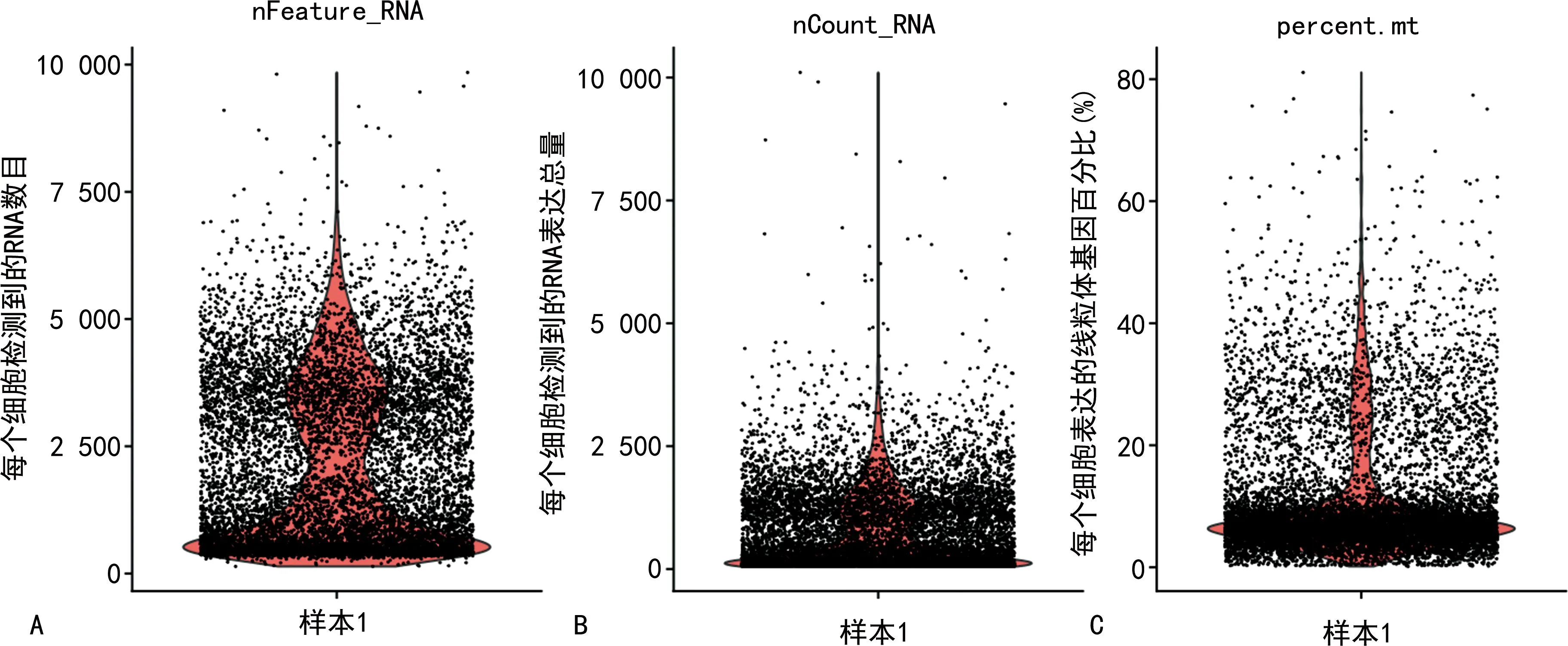
注:A为样本1中每个细胞检测到的RNA数目;B为样本1中每个细胞检测到的RNA表达总量;C为样本1中每个细胞表达的线粒体基因百分比。
Data1=subset(Data1,percent.mt <20 &nFeature_RNA >800 &nCount_RNA >1 000) #过滤低质量细胞,这里仅保留表达线粒体基因比例小于20%、表达基因数大于800且UMI数大于1 000的细胞,可以根据实际情况调整。P2=VlnPlot(Data1,features=c("nFeature_RNA","nCount_RNA","percent.mt"),group.by="orig.ident") #对过滤后细胞质量进行可视化,见图2。
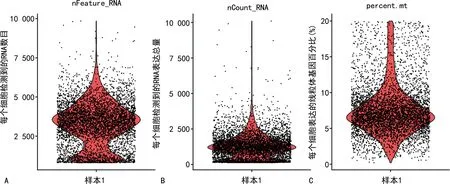
注:A为样本1中每个细胞检测到的RNA数目;B为样本1中每个细胞检测到的RNA表达总量;C为样本1中每个细胞表达的线粒体基因百分比。
对于第2个样本,读入数据,方法同上。
(3)多个样本时,对样本1(Data1)和样本2(Data2)进行数据整合:All=merge(Data1,Data2) #合并数据;All=NormalizeData(All) #数据归一化处理,减少文库测序深度的影响;All=FindVariableFeatures(All,selection.method = "vst",nfeatures = 2000) #鉴定细胞间表达量相差大的前2 000个基因,用于后续鉴定细胞类型;All=ScaleData(All) #数据标准化;All=RunPCA(All,pc.genes=All@var.genes,npcs =50,verbose = FALSE) #PCA降维并提取主成分,用于后续细胞降维;All=RunHarmony(All,group.by.vars="orig.ident",plot_convergence = TRUE) #按照"orig.ident"(样本名称)去除批次效应。
(4)对细胞进行降维、分群,见图3:All=All %>% RunUMAP(reduction = "harmony",dims= 1:50) %>% #降维算法UMAP;RunTSNE(reduction = "harmony",dims = 1:50) %>% #降维算法tSNE,UMAP和TSNE二选一即可;FindNeighbors(reduction = "harmony",dims = 1:50) %>% FindClusters(resolution = 1) %>% #resolution 参数相当于放大镜,数值越大,分群越细致。 identity(P3)=DimPlot(All,reduction = "umap",group.by= "seurat_clusters") #按照无监督聚类的细胞群进行可视化。P4=DimPlot(All,reduction = "umap",group.by = "orig.ident") #按照样本进行可视化。save(All,file="All.Rdata") #保存Seurat对象。
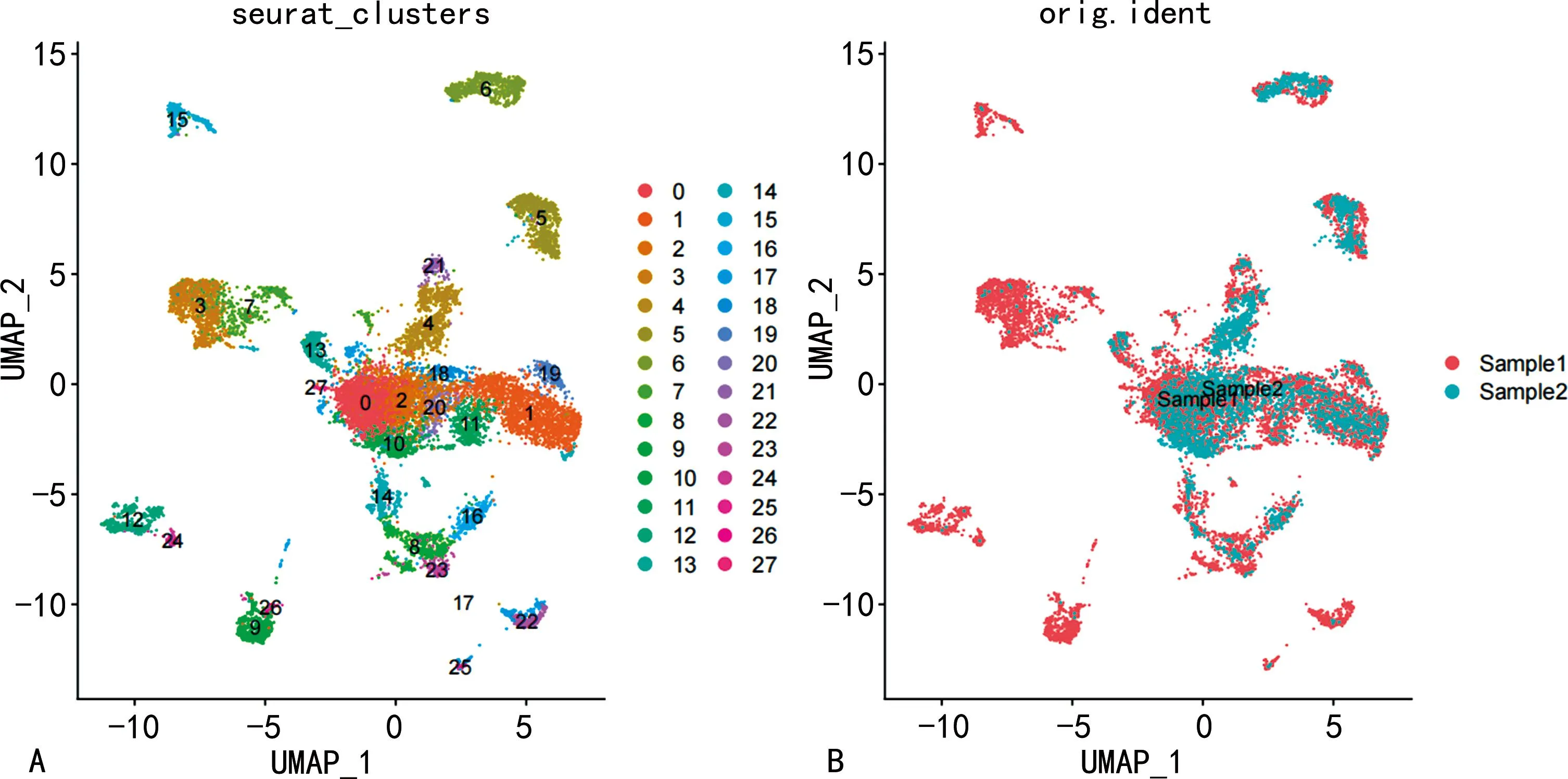
注:A为细胞降维聚类后的类群分布;B为细胞降维聚类后的样本分布;UMAP为均匀流形近似和投影,是一种非线性降维和可视化算法。
(5)计算各个细胞群的特征基因,见图4:DEG=FindAllMarkers(All,only.pos=T)Head(DEG)avg_log2FC表示该基因在组合间的差异倍数;cluster表示该基因在对应的细胞群中高表达;pct.1表示在当前细胞群中检测到的表达该基因的细胞比例;pct.2表示在其他细胞群中检测到的表达该基因的细胞比例;p_val_adj表示该基因的矫正P值;gene表示基因名称。
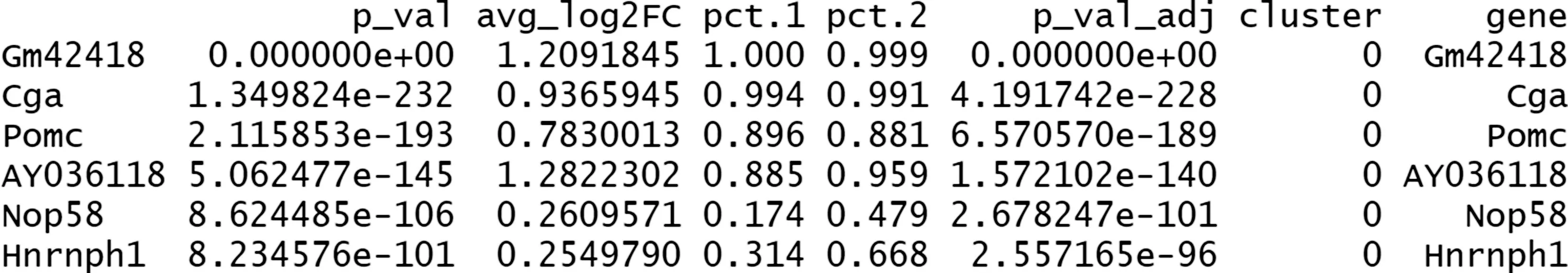
图4 特征基因计算结果示例
(6)细胞类型鉴定:根据每个细胞群的特征基因鉴定该细胞群对应的细胞类型,见表1。除了这些常见类型,其余细胞类型需要根据自己分析的样本来查阅文献和数据库,如CellMarker数据库(http://xteam.xbio.top/CellMarker/)等[15]。

表1 常见细胞类型和分子标记
(7)其他分析:后续分析可以对比不同样本类型中各个细胞类型数目的差异,寻找特有或稀有细胞类型;鉴定同一细胞类型不同样本之间的基因表达差异,如肿瘤组织和正常组织之间的差异特征基因、细胞类型之间的相关关系、同一细胞类型不同亚型之间的发育轨迹及发育过程中的重要基因等。
1.4学生分组讨论与现场实操 后续分析可让学生分组学习、探索与现场实操,将遇到的问题反馈给教师,增强教学互动,发挥学习者的能动性,利用学生多元化的学习需求推动优化教学设计。这有利于后续教学中细化需要达成的教学目标,分步骤实现,不断提升课程内容和质量,加强学生的综合能力培养。
2 结 果
在本次授课中,学生回顾了生理学课程中重要的知识点,即下丘脑-垂体系统。通过文献扩展了解了垂体瘤的现行分类标准、现行分类标准的不足之处,以及国内外相关研究前沿提出的对这些不足的解决方案。在教学内容中引入学科领域的最新成果,更新和完善讲解内容。以国内外最新研究成果为牵引,让学生具备基础医学专业知识的同时培养其国际视野,将人才培养与社会需求相结合。通过这一系列学习不仅扩展了学生在相关问题上的知识储备,而且培养了学生发现问题、分析问题和解决问题的科研能力。
此外,对文献中所用的主要生物信息学方法进行讲解和分析示范,对分析结果进行数据解读。一方面告诉学生什么是生物信息学,生物信息学有什么用处;另一方面也鼓励学生在课堂示范的基础上自主探索和学习,为学生未来从事临床或基础研究工作奠定一定基础。同时,通过课程学习,激发了学生对临床和科研工作的兴趣,让学生在未来工作中从实际出发,敬业奉献。
3 讨 论
随着生物信息学技术的发展,其与医学临床检验和基础研究的联系越来越紧密。因此,在医学生本科学习阶段开展生物信息学授课对提高学生综合能力具有重要帮助。但是,在教学设计和实施过程中应注意以下问题:(1) 在授课教师的培训方面,该课程要求教师掌握计算机语言应用和生物信息学分析方法;同时,授课教师应具有良好的医学理论基础,关注医学国际前沿研究发展,能够结合学生的医学专业背景和专业知识进行教学实践,同时紧跟国际前沿研究进展、与时俱进。(2) 在教学过程中应注意各个环节的时间分配,同时可设计翻转课堂,案例分析,小组讨论、现场实操等环节,例如,①在介绍基础课程知识点时以回顾为主,内容不宜过多;②讲解相关文献时,可由学生分组对教师指定文献进行解读。学生讲解之后,进行互动提问和教师总结;③数据分析讲解与示例部分主要由教师进行讲解和示范,与学生互动提问、现场实操和分组讨论,该部分是主要环节,可分配较长时长。(3) 在课程设置上应充分考虑临床医学和基础研究的需要,设置实用的生物信息学课程内容。例如,概括介绍高通量测序的概念、常见技术、应用领域等。在后续具体教学实例中可结合文献进行常用的高通量方法的应用示范,例如,一代测序、全外显子测序、全基因组测序、转录组测序、甲基化测序等。(4) 采用“以学生为中心”的教学模式[2-4],应关注学生在大量互动和自主学习模式下的学习效果。考查学生对知识的理解和熟悉情况,以及对教学模式的适应情况。与之配套的考核方式应将相当的比重放置在过程性考核上,关注学生在分组中的表现,注重检验学生能力提升情况。量化教学评价,培养德才兼备的高素质创新型人才。(5) 在课程设置时应注重挖掘课程的思想政治元素,结合医学基础课程的特点,利用学习思维方法引领价值观,增强职业认同感,使学生更好地肩负起维护和促进人民群众身体健康的重要使命。
通过本文中这一系列课程设置,让学生回顾本专业知识点的同时进一步扩大知识面,了解国际前沿的新发现、新理论。同时培养学生发现问题、分析问题和解决问题的能力。再通过相关生物信息学方法的讲解、分析示范和结果解读,让学生在学习过程中了解生物信息学分析的一般方法、掌握对分析结果的解读,将临床与科研相结合,提高学生综合能力。

