基于WGCNA筛选胃癌相关自然杀伤细胞基因及潜在中药预测*
郑翼欣,肖尚平,邝美霞,邝卫红
(1.广州中医药大学第一临床医学院,广东 广州 510000;2.广东医科大学药学院,广东 东莞 523000)
加权基因共表达网络分析(weighted gene co-expression network analysis,WGCNA)通过构建共表达网络确定基因模块与性状表型相关性,从而挖掘与性状表型显著相关的基因,为挖掘疾病的关键基因和机制提供了研究方法[1-2]。胃癌为世界上病死率排名第4位的恶性肿瘤。据统计,每年全世界有超100万新发诊断胃癌的患者[3]。大约70%的患者在确诊时已经是进展期或者晚期胃癌,失去了手术根治的机会。化疗是进展期胃癌患者的一线治疗方法,但是存在疗效异质性、中位生存期短、复发等问题[4-6],所以亟须研究新的治疗方法以提高晚期胃癌患者生存期。免疫治疗成为新兴的治疗手段,极大改变了胃癌的治疗格局[7]。肿瘤微环境被认为与肿瘤的预后和对免疫治疗的反应密切相关,其与免疫细胞之间的复杂相互关系在肿瘤免疫微环境中发挥重要作用并决定临床结局[8-9]。NK细胞作为免疫细胞中的重要成分,因其在肿瘤中的特异性细胞毒性和安全性,以及作为细胞疗法的潜在用途,受到人们的广泛关注[10-11]。中医药以其独特的辨证论治思路在胃癌的治疗中有巨大的发展前景。诸多研究[12-13]表明中医药能提高NK细胞的数量及活性,能够有效改善症状评分,减少化疗带来的免疫抑制。
1 材料与方法
1.1 数据获取与整理 从TCGA数据库(The Cancer Genome Atlas,https://portal.gdc.cancer.gov/)中下载全部胃癌的转录组和临床数据。利用perl语言(Strawberry perl 5.30.0.1)对数据进行解压、ID转换等整理成基因表达矩阵和提取临床数据。以“gastric cancer survival”为关键词检索GEO数据库(https://www.ncbi.nlm.nih.gov/geo/),并限定数据类型及物种进行筛选,选择GSE84433、GSE84437两个数据集,利用perl语言对数据进行整理,去除1个探针对应多个分子的探针,遇到同个分子有多个探针时,仅保留信号值最大的探针,对其进行质量控制,背景校正及补充缺失值,并提取临床数据。本研究共计纳入TCGA 375 个胃癌样本和32 个正常样本,GSE84437 和GSE84433数据集中共790个胃癌样本被纳入本研究,删除生存状态和生存时间未知的样品。
1.2 基因共表达网络的构建和模块的筛选 以免疫细胞基因集文件中的Natural.killer.cell及Natural.killer.T.cell基因集作为背景文件,使用R语言(version 4.1.2)中的“ssGSEA”方法估算TCGA-STAD及GSE84437中所有样品中上述两种免疫细胞的分数,作为WGCNA性状数据。为确定基因模块与自然杀伤细胞浸润的中枢基因,使用数据中方差前25%的基因用于构建加权基因共表达网络,先用基因两两之间相关系数的绝对值构建Person相关矩阵。选择无标度拓扑拟合指数R2超过0.90时对应的软阈值将邻接矩阵转换为拓扑重叠矩阵(topological overlap matrix,TOM)。采用动态混合剪切方法将基因分成不同的模块,每个模块至少100个基因。选择P值显著并且相关系数最高的模块,将上述两个数据集获得的相关系数最高的两个模块取交集。使用R语言“limma”包筛选出TCGA差异基因DEGs,将上述两个模块与DEGs取交集,获得胃癌相关自然杀伤细胞基因,并提取其在TCGA数据集中的表达量,采用R语言中的“pheatmap”包绘制自然杀伤细胞基因在正常组和肿瘤组的热图。(见图1)

图1 胃癌中自然杀伤细胞基因差异分析热图

图2 通过WGCNA 确定TCGA-STAD 数据集与临床特征相关的模块
1.3 GO和KEGG富集分析 为揭示胃癌相关自然杀伤细胞基因主要参与的生物学功能及信号通路,我们通过在线数据库DAVID(https://david.ncifcrf.gov/)对胃癌相关自然杀伤细胞基因进行基因本体论(gene ontology,GO)和京都基因与基因组百科全书(kyoto encylopaedia of genes and genomes,KEGG)富集分析。
1.4 获取与胃癌预后相关的基因 对TCGA-STAD和GSE84437两个基因矩阵进行归一化数据处理,使用R语言中的“sva”包中的ComBat算法校正批次效应,合并为训练集,提取胃癌相关自然杀伤细胞基因的表达量,合并生存状态及生存时间,采用R语言中的“survival”包进行单因素Cox回归分析,获取与胃癌预后相关的自然杀伤细胞基因表达矩阵,作为后续分析文件,并绘制森林图。
1.5 预后基因相关性分析 使用R语言中的“corrplot”包进行预后基因之间相关性分析,并绘制预后基因之间相关性分析图。
1.6 预后基因生存分析 采用R语言中的“survival”包对合并生存数据预后相关的基因表达矩阵文件进行临床生存分析,获取与生存显著相关的基因,并绘制生存曲线图,并使用GSE84433数据集作为验证集,对上述基因进行生存分析验证。
1.7 胃癌生存相关基因与免疫细胞浸润的相关性分析 将“1.4”获得的归一化处理的训练集,运用R语言中“CIBERSORT”算法计算22种免疫细胞在各样本中的相对含量作为免疫细胞浸润结果,根据生存相关基因在样本中的表达情况,对所有免疫细胞进行循环计算,得到基因与免疫细胞相关性结果,进一步得出基因与免疫细胞相关性,绘制生存相关基因与22种免疫细胞相关性分析热图。
1.8 潜在中药预测及统计 将生存相关自然杀伤细胞基因映射到Coremine Medical数据库中预测干预胃癌的中药,以P<0.05为截断值,并对筛选出的中药进行四气、五味、归经和功效的统计分析。
2 结果
2.1 加权基因共表达网络分析 基于加权共表达分析,TCGASTAD和GSE84437基因表达矩阵分别被分为18和9个模块,与自然杀伤细胞最相关的基因模块分别为TCGA grey60模块1541个基因和GSE magenta模块543个基因。将上述两个模块基因与STAD数据集差异表达基因DEGs进行取交集,共获得98个被认为与自然杀伤细胞最密切的基因,并通过热图将共同基因在正常组织和胃癌组织中的表达可视化。图中左边蓝色部分为正常组,右边粉色部分为胃癌组,红色为高表达基因,蓝色低表达基因,自然杀伤细胞在肿瘤组多为高表达。(见图1)
2.2 GO和KEGG富集分析 将上述98个基因输入DAVID数据库,获得GO和KEGG富集分析结果。(见图4~5)GO富集从3个方面注释了基因的生物学特性,生物学进程(biological process,BP)集中于细胞因子介导的信号通路、对病毒的反应、正向调节细胞因子的产生、细胞黏附的正向调节等;细胞成分(molecular function,CC)主要集中于质膜外侧面、内吞小泡、吞噬小泡等;分子功能(molecular function,MF)主要集中于细胞因子受体结合、细胞因子活性、免疫受体活性等免疫相关通路。KEGG主要富集于EB病毒感染、抗原处理和呈递、细胞因子-细胞因子受体相互作用、甲型流感、Th1和Th2细胞分化、吞噬体、新型冠状病毒肺炎、NOD样受体信号通路、趋化因子信号通路等通路。
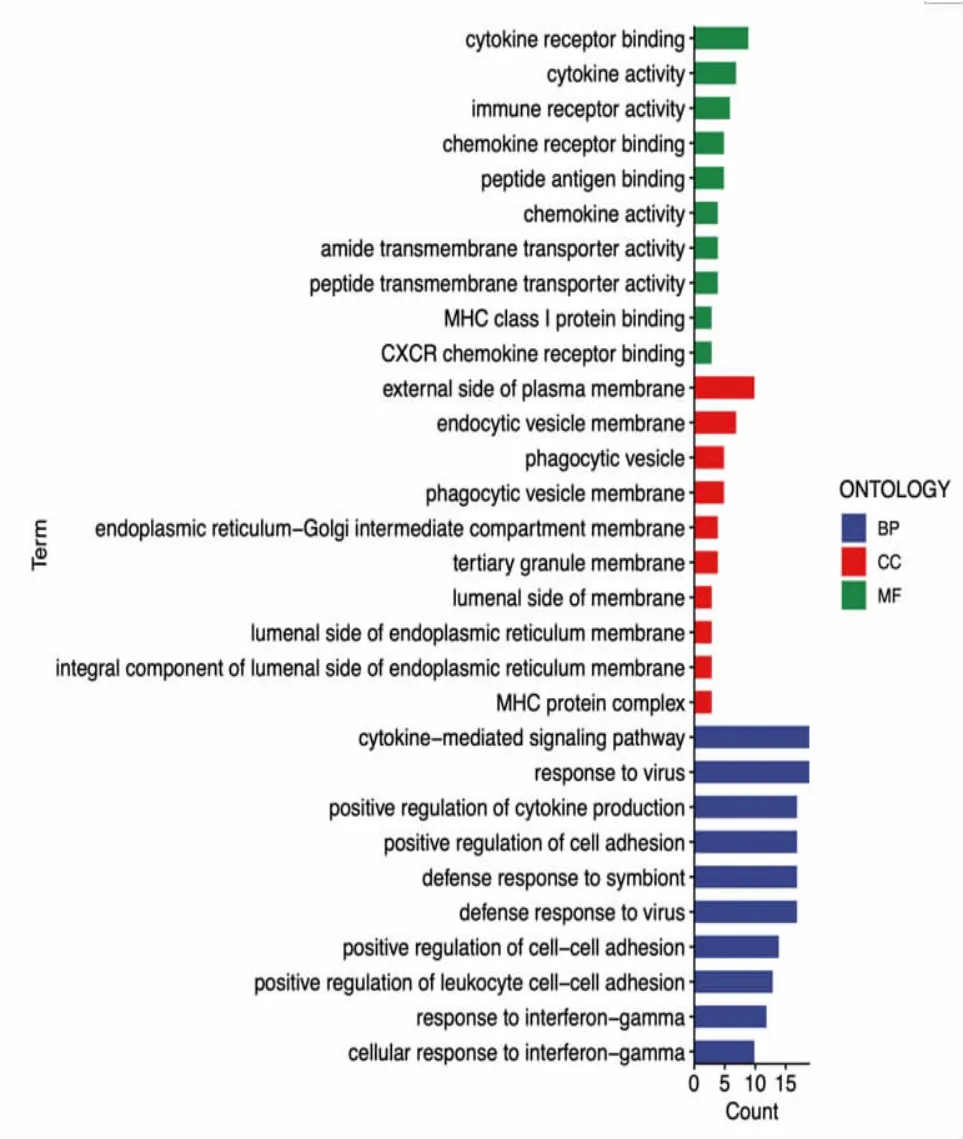
图4 GO 功能富集图
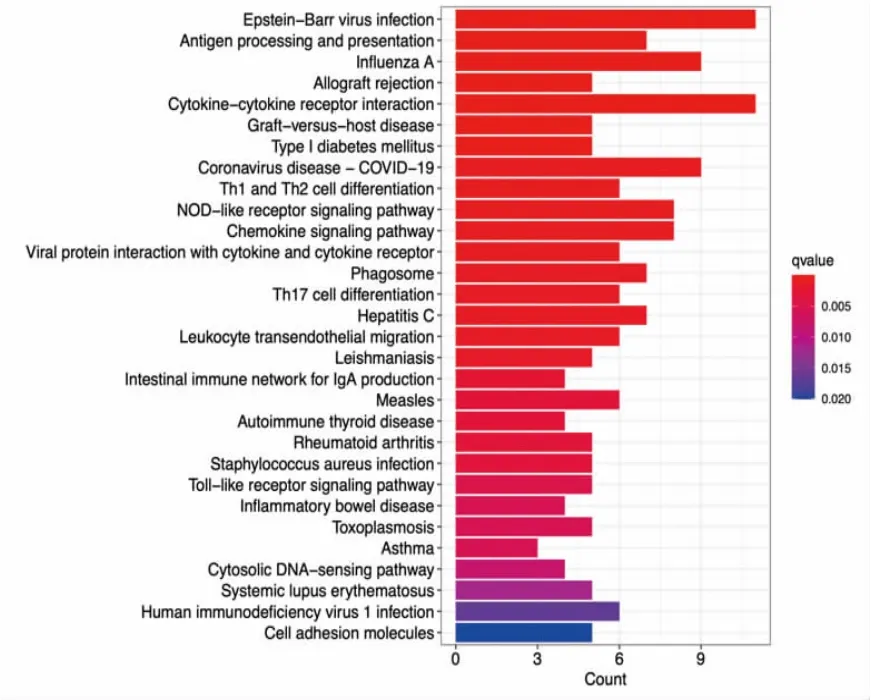
图5 KEGG 功能富集分析图
2.3 筛选与胃癌预后相关基因 通过单因素回归分析,共有22 个基因(ALOX5、BATF2、CD96、CXCL11、CXCL9、EPSTI1、GBP4、GBP5、GZMB、IFNG、IL2RB、IL32、IRF8、LAP3、MUC20、PARP14、PARP9、SNX10、STAT1、TAP1、UBE2L6、ZNF683)与胃癌预后相关。(见图6)除MUC20、ALOX5在胃癌中被视为危险基因,其余基因均为保护基因。相关性分析图显示22个基因之间存在正负相关性,其中打叉的被认为相关性不显著。正相关性最大的是CXCL9与GBP5,MUC20与大部分基因存在负相关性。其中与CXCL9和GBP5负相关性最大,相关系数为-0.34。

图6 单因素Cox 回归分析森林图及预后基因的相关性分析图
2.4 核心基因生存分析并验证 将“2.3”得到的22个预后基因进行Kaplan-Meier生存分析,结果显示5个基因(IL32、IRF8、IFNG、SNX10、BATF2)生存曲线中高、低表达组患者的预后存在显著差异。5个基因在高表达组生存时间均更长。另外,将GSE84433作为验证集,可见上述5个基因生存曲线在高、低表达组患者中的预后存在显著差异,且其在胃癌中高表达提示生存时间更长。(见图7~8)
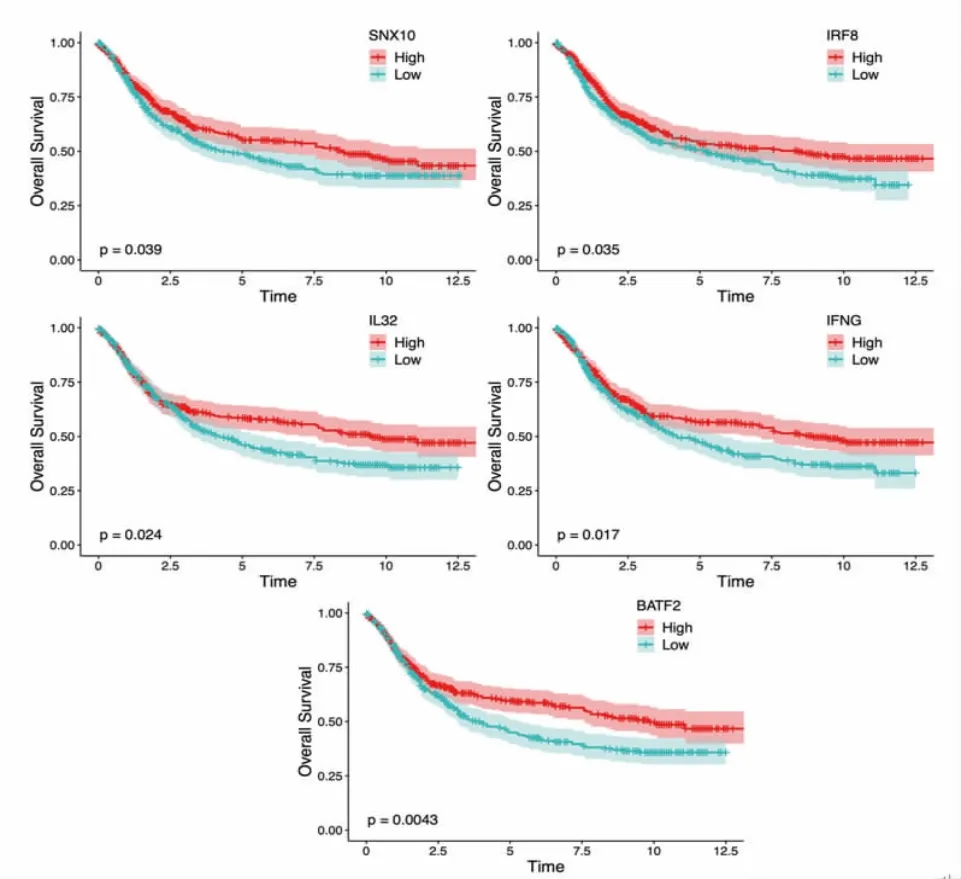
图7 IL32、SNX10、BATF2、IRF8、IFNG 在训练集中OS 患者K-M 曲线分析
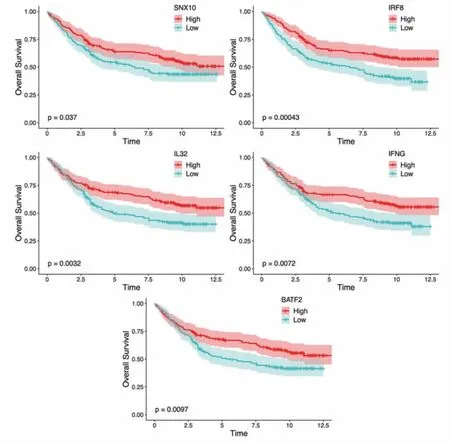
图8 IL32、SNX10、BATF2、IRF8、IFNG 在验证集GSE84433中OS 患者K-M 曲线分析
2.5 生存相关基因与免疫细胞浸润相关性分析 相关性分析可见生存相关基因与大部分免疫细胞存在相关性。如IFNG与CD8 T细胞、活化CD4记忆T细胞、浆细胞、滤泡辅助性T细胞、静息自然杀伤细胞、M1巨噬细胞存在正相关关系,与调节性T细胞、幼稚B细胞、静息CD4记忆T细胞、静息肥大细胞、活化肥大细胞、M2巨噬细胞、记忆B细胞、活化树突状细胞存在负相关关系等,与活化CD4记忆T细胞相关性最强。(见图9)
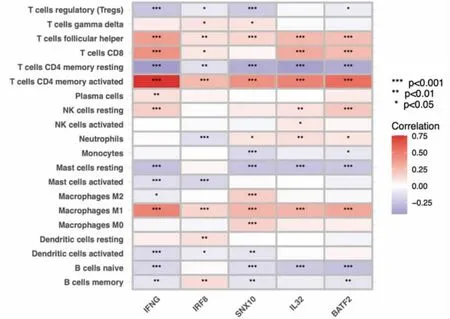
图9 生存相关基因与免疫细胞浸润的相关性分析
2.6 潜在中药预测结果及统计 使用Coremine Medical数据库(https://coremine.com/)与预后相关基因互相映射,以P<0.05为截断值,其中11个基因有较多中药被筛出,共筛选治疗胃癌的潜在中药186味,其中频数较高的中药有姜黄、莪术、人参、郁金、黄芩、山药、沙棘、高良姜、灵芝、石斛10味中药。通过翻阅《中药大辞典(2006版)》《中华本草》《中药学(第3版)》,对筛选出的中药进行四气、五味、归经及功效进行分类。四气总频数为186,四气中以温、寒为主,温>寒>平>凉>热;五味总频数为301,五味中以苦、辛、甘为主,苦>辛>甘>咸>涩>酸>淡;归经总频数为458,归经中以肝、肺、胃、脾经为主,肝经>肺经>胃经>脾经>肾经>心经>大肠经>膀胱经>胆经>小肠经>三焦经。有明确分类的药物有98味,功效分类中以补虚药、清热药为主,其中补虚药>清热药>利水渗湿药>解表药>活血祛瘀药>温里药>祛风湿药=安神药>收涩药=理气药>驱虫药=化痰药>平肝息风药=开窍药。(见表1、图10)

表1 生存相关基因及其对应中药

图10 中药四气、五味、归经及功效统计图
3 讨论
本研究发现5个与胃癌生存预后显著相关的基因,5个基因在胃癌中高表达均提示较好的生存预期。研究发现,γ干扰素(interferon γ,IFNG)能通过激活效应细胞,提高自然杀伤细胞等免疫细胞活性,发挥杀伤肿瘤细胞的作用[14]。干扰素调节因子8(interferon regulator 8,IRF8)作为IRF8/IFN-γ信号轴的关键成分,可通过介导免疫反应起到抑制肿瘤的作用[15]。SNX10是分选链接蛋白家族(sorting nexins,SNXs)相关蛋白家族中重要成员,SNX10基因突变可能与骨癌的发生有关联[16]。张伟新等[17]通过研究发现基因SNX10在结直肠癌细胞中过表达时,可以抑制结直肠癌细胞的增殖。碱性亮氨酸拉链转录因子2(basic leucine zipper transcription factor2,BATF2),是AP-1转录因子家族成员,在正常细胞中高表达。而在多个肿瘤中低表达。付晓红等[18]通过实验证实白芦藜醇可能通过上调BATF2的表达,起到抑制肿瘤细胞增殖的作用。白介素-32(interleukin-32,IL-32)是一种广泛参与炎症和肿瘤的细胞因子,IL-32过表达可以通过促进癌细胞的凋亡而抑制肿瘤细胞的增殖[19]。
胃癌属于中医学“胃脘痛”“噎膈”“积聚”等范畴。多数医家认为,胃癌的发生发展以脾虚为本[20]。有学者认为,胃癌正虚的原因责之于脾阳虚,同时因脾阳虚引起“痰、瘀、毒”等病理产物的产生[21]。“寒气生浊”理论是在阳虚基础上演变而来,有研究发现阳虚质和气虚质是胃癌常见体质类型[22-23]。这种类型体质下的机体免疫力常处于抑制状态,导致自然杀伤细胞等免疫细胞活性下降[24]。以扶助正气为基础,配合祛瘀解毒为法是中医治疗胃癌的基本法则。同时,气机郁滞是胃癌发生发展的重要病机。贾英杰在临床辨治胃癌过程尤为重视气机的调畅[25-26]。脾胃为中焦气机升降的枢纽。肝脏喜调达,其气宜升,肝气郁结则木郁克土。脾胃受克,则脾气愈虚。中医药被证实在胃癌的治疗中有效,能通过多成分、多靶点增强化疗敏感性、减轻化疗导致的不良反应,且能提高机体免疫机能以增强机体抗癌能力[27-28]。研究发现,人参、黄芩、莪术、姜黄、郁金均有抑制胃癌细胞增殖的效果[29-33]。本研究获取的中药四气以温、寒为主,五味以苦、辛、甘为主。根据中医理论,温以补阳,辛甘化阳扶助正气,以提高免疫力以抗癌。攻邪以苦寒为主。归经以肝、肺、胃、脾经为主。胃癌治疗病位在脾胃。肝、肺归经排名较前。脾胃为气机升降枢纽,而肝、肺二者以气机升降之主,可能提示胃癌治疗过程中尤应重视气机的调畅。功效以补虚药、清热药为主,补虚药占比最多。结果基本符合胃癌以正虚为本的基本病机,清热药排名靠前可能与胃病容易阳化的中医理论相契合。

