天然气库上方土壤微生物群落研究
张 凡,佘跃惠,孔淑琼,舒福昌,喻高明,侯读杰
(1.中国地质大学(北京)能源学院,北京100080;2.长江大学化学与环境工程学院,湖北荆州434023)
天然气库上方土壤微生物群落研究
张 凡1,佘跃惠2,孔淑琼2,舒福昌2,喻高明2,侯读杰1
(1.中国地质大学(北京)能源学院,北京100080;2.长江大学化学与环境工程学院,湖北荆州434023)
通过基于16S rRNA基因克隆文库的方法研究了天然气库上方土壤的微生物群落结构。针对大港油田板876气库上方同一取样点1.0 m和2.0 m的土壤样品构建了16S rDNA克隆文库DGS1和DGS2,并对其150个阳性克隆进行限制性酶切片段长度多样性分析(ARDRA)。结果表明,克隆文库DGS1有40个操作分类单元(OTU),克隆文库DGS2有39个OTU,该天然气库上方土壤中微生物较丰富;但由于土壤深度的不同两个土壤样品微生物群落结构存在着差异。每个OTU的代表克隆序列分析结果表明,克隆文库DGS1中优势菌群为芽单胞菌(Gemmatimonadetes)28%、绿弯菌(Chlorof lexi)23%和放线菌(Actinobacterium)21%;克隆文库DGS2菌群分布较平均,其中 Chlorof lexi19%、硫氧化菌(Sulfur-oxidizing)12%、酸酐菌(Acidobacteria)10%、Gemmatimonadetes10%。天然气库上方土壤微生物多样性的分子分析可为开展微生物油气勘探(MPOG)技术奠定基础。
气库;16S rRNA基因;克隆文库;微生物群落;微生物油气勘探(MPOG)
基于16S rDNA分子生物学技术已被广泛用于研究环境微生物群落多样性。克隆和16S rDNA序列分析能有效地确定研究区域中未被培养出来的微生物[1],能够使人们更准确全面地认识特定生态系统中的微生物种群多样性[2]。
基于16S rDNA的分子生物学方法已被用来研究森林、稻田、草原土壤和海湖沉积物中的微生物群落。对油田微生物群落的研究主要集中在油田产出油水样、油田地层水样中的微生物群落[3~5],而对于油气田上方土壤中的微生物群落研究报道却很少。采用微生物油气勘探(MPOG)技术检测土壤中烃氧化菌的异常,可以预测下伏油气藏的存在[6,7]。
作者在此通过构建16S rRNA基因克隆文库分子,研究了大港油田板876气库上方土壤微生物群落结构,为详细研究油藏生态系统中微生物种群组成及开展微生物油气勘探(MPOG)技术提供了可靠数据。
1 实验
1.1 取样
样品取自中石油大港油田(天津)板876气库上方土壤样品。其地层剖面图如图1所示。
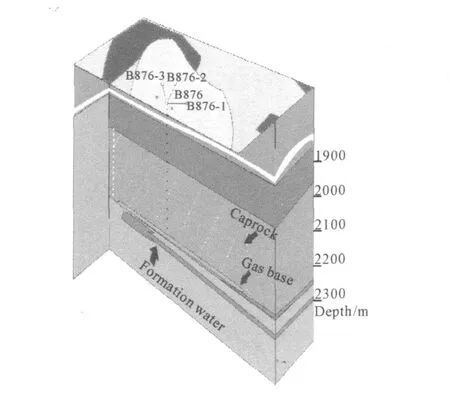
图1 板876气库地层剖面Fig.1 G eological base of Ban876 gas reservoir
取样点距注气点板876井100 m。气藏盖层上部为沙一中、上段地层,全部为暗色泥岩,总厚度200 m左右,平面上,砂体呈席状分布,气藏主体部位渗透砂岩厚度 6~9 m。孔隙度 13.0%~24.4%,平均21.1%。渗透率21.0×10-3~398.0×10-3μm2,平均164.5×10-3μm2。气层受构造控制,气水界面在2244 m处,含气高度24 m,凝析油含量72.7 g·m-3。气藏气层平面上分布连片,连通性好,气层有效厚度1.2~11.4 m,碾平厚度4.6 m,为构造气藏。天然气相对密度为0.6264(设空气的密度为1),其中甲烷含量为90%。
分别取取样点的纵向剖面0.5 m、1.0 m、1.5 m、2.0 m以及2.5 m处的5个土壤样品,立即送实验室, 20℃保存。
1.2 土壤样品处理以及DNA提取
将新鲜土壤样品碾碎,充分混匀,分别称取5个土壤样品0.5 g各3份,按 Fast DNA Spin Kit for Soil (Qbiogen,Carlsbad,CA.U.S.A)试剂盒说明提取样品总DNA。将每个样品的3份DNA混合到一起用DNA浓度测定仪(Nanodrop?ND-1000 UV-Vis Spectrophoto-meter,Nanodrop Technologies)测定所提取的DNA浓度。
1.3 16S rRNA基因全长扩增
使用 16S rDNA片段扩增通用引物 27f(5′-AGAGTTTGATCCTGGCTCA G-3′)和 1492R(5′-GGCTACCTTGTTACGACTT-3′)从基因组总DNA中扩增16S rDNA片段。PCR扩增体系及程序参见文献[8]。
1.4 克隆文库建立
用琼脂糖凝胶回收试剂盒(Agarose gel DNA purification kit,TaKaRa Biotechnology,Dalian,China)对16S rDNA扩增产物进行割胶,纯化。然后将回收产物16S rDNA片段p GEM T-easy克隆载体(Promega)转化到感受态细胞 Escherichia coliJM109 Competentcells(TaKaRa Biotechnology,Dalian, China)中。从两个文库各挑取150个阳性克隆,挑取少量菌体作为模板,扩增每个克隆的16S rRNA基因。PCR产物用限制性内切酶 HaeⅢ和 HhaⅠ进行酶切,再用2%琼脂糖凝胶电泳分析限制性片段,然后对电泳图谱进行限制性酶切片段长度多态性(ARDRA)分析。将两种酶切图谱相同的克隆归为同一种发育类型,即同一种操作分类单元(OTU)。
1.5 克隆文库库容
应用 Rarefaction分析软件(http://www.uga. edu/strata/software/Software.html)对克隆文库的库容饱和度进行分析,以检测建立的克隆文库是否已经足够全面地代表了MOB群落的多样性。
1.6 测序并构建系统发育树
挑取ARDRA分型归为一类的克隆接种于含有100μg·mL-1氨苄的液体LB培养基中,37℃培养20 h后送往测序公司(诺赛基因,北京)。所得序列通过DNAMAN软件进行处理,然后在National Center for Biotechnology(NCBI)(http://www.ncbi.nlm.nih. gov/BLAST/)数据库中比对分析,寻找亲缘关系最近的序列,用DNAMAN软件构建系统发育树。
2 结果与讨论
2.1 气库上方土壤微生物群落
在气库上方1.0 m和2.0 m土壤样品的16S rRNA基因克隆文库DGS1和DGS2中各选择了150个阳性克隆子,通过 HaeⅢ和 HhaⅠ双酶切分型分别得到了40个和39个OTU。Rarefaction分析曲线(图2)显示当阳性克隆子达到150个时曲线趋于水平,说明克隆文库能够反映样品微生物群落结构。
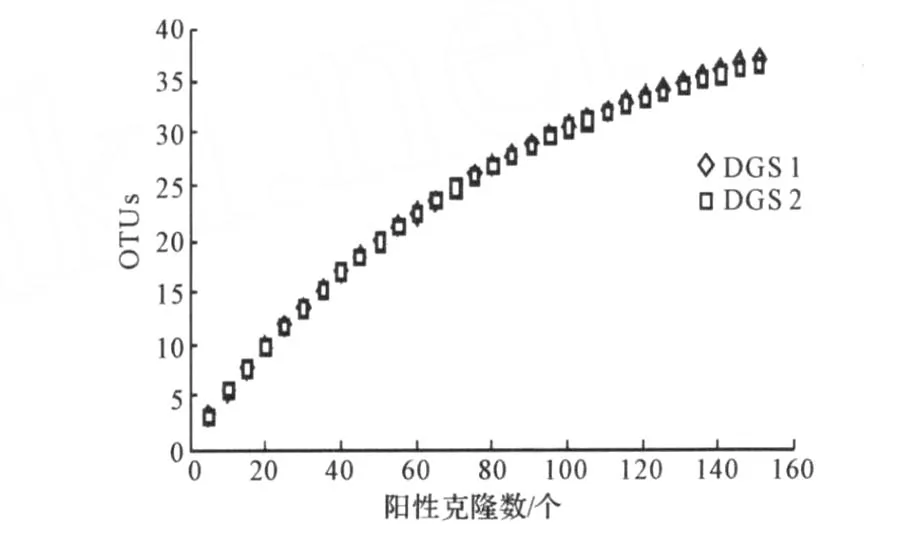
图2 克隆文库DGS1和DGS2 Rarefaction分析曲线Fig.2 Rarefaction analysis curves of clone libraries DGS1 and DGS2
根据系统发育树(图 3、图4)可知,克隆文库DGS1中优势菌群为单胞菌 (Gemmatimonadetes) 28%、绿弯菌(Chlorof lexi)23%和放线菌(Actinobacterium)21%;克隆文库DGS2菌群分布较平均,其主要菌群有 Chlorof lexi19%、硫氧化菌(Sulfur-oxidizing)12%、酸酐菌(Acidobacteria)10%、Gemmatimonadetes10%。所得序列在NCBI数据库中对比分析,结果表明,大多数序列(>80%)和GenBank中已有的16S rDNA序列同源性小于97%,说明该样品微生物系统中微生物资源比较新颖,很多属于新的种。
2.2 讨论
应用16S rRNA基因克隆建库方法对天然气库上方土壤微生物群落结构进行了初步研究。两土壤样品微生物群落结构存在差异,但微生物种类数(OTUs)较为接近。以前关于油气微生物勘探(MPOG)报道指出油气田上方土壤中的烃氧化菌中甲烷氧化菌的数量较其它非油气田上方多[9,10]。但是本研究中两个气库上方土壤样品克隆文库中没有发现烃氧化菌,证明烃氧化菌在气库上方土壤中并非以优势菌的地位存在。因此要了解油气田与非油气田上方土壤中烃氧化菌的差异,必须找到烃氧化菌的特异性引物,从而排除其它微生物的干扰。
大港油田地处渤海之滨是一片盐碱地,土壤的平均p H值为7.5。两个克隆文库中的主要微生物种群为 Chlorof lexi、Gemmatimonadetes、A cidobacteria、 Actinobacterium。海洋沉积物以及盐碱沼泽土壤微生物也包含这些微生物[11~13]。这一结果证明土壤中的微生物菌群主要是由土壤的地理位置以及土壤的理化性质决定。不同油气田由于其所属地层的化学和物理条件不同,其土壤中所含的微生物差异较大。因此, MPOG工作必须开展中前期的土壤微生物调查。
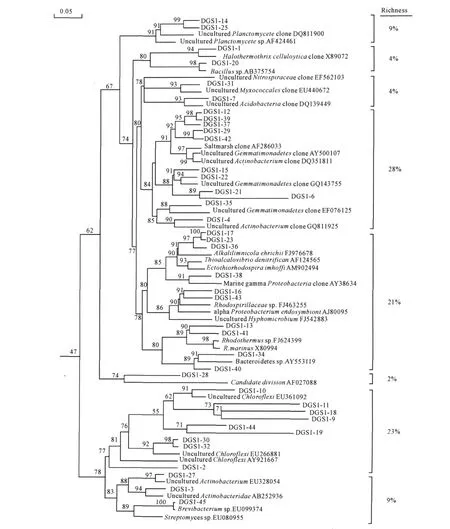
图3 16S rRNA基因克隆文库DGS1系统发育树Fig.3 Neighbor-joining phylogenetic tree of sequences of the 16S rRNA gene of clone library DGS1
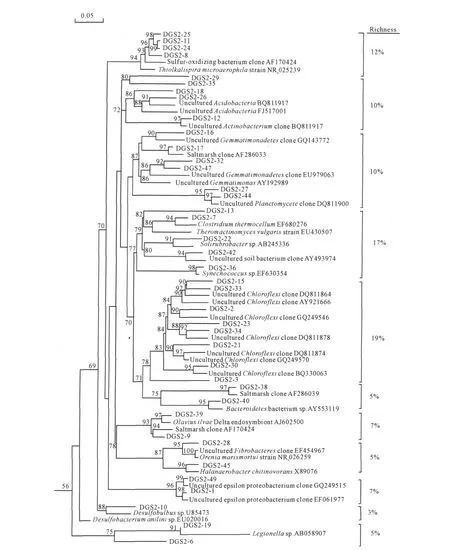
图4 16S rRNA基因克隆文库DGS2系统发育树Fig.4 Neighbor-joining phylogenetic tree of sequences of the 16S rRNA gene of clone library DGS2
3 结论
通过基于16S rRNA基因克隆文库的方法研究了天然气库上方土壤的微生物群落结构。构建了16S rDNA克隆文库DGS1和DGS2,并对其150个阳性克隆进行限制性酶切片段长度多样性分析(ARDRA)。结果表明,克隆文库DGS1有40个操作分类单元(OTU),克隆文库DGS2有39个OTU,克隆文库DGS1中优势菌群为芽单胞菌(Gemmatimonadetes) 28%、绿弯菌(Chlorof lexi)23%和放线菌(Actinobacterium)21%;克隆文库DGS2菌群分布较平均,其中有 Chlorof lexi19%、硫氧化菌 (Sulfur-oxidizing) 12%、酸酐菌(Acidobacteria)10%、Gemmatimonade-tes10%。天然气库上方土壤微生物多样性的分子分析为开展微生物油气勘探(MPOG)技术奠定了基础。
[1] Hugenholtz P,Goebel B M,Pace N R.Impact of cultured-independent studies on the emerging phylogenetic view of bacterial diversity[J].J Bacteriol,1998,180(18):4765-4774.
[2] Amann R I,Ludwig W,Schleifer K H.Phylogenetic identification and in situ detection of individual microbial cells without cultivation[J].Microbiol Rev,1995,59(1):143-169.
[3] Lysnes Kristine,BΦdtker Gunhild,Torsvik Terje,et al.Microbial response to reinjection of produced water in an oil reservoir[J]. Appl Environ Microbiol,2009,83(6):1143-1157.
[4] Kaster Krista M,Bonaunet Kristin,Berland Harala,et al.Characterization of cultured-independent and-dependent microbial communities in a high-temperature offshore chalk petroleum reservoir [J].Antonie Van Leeuwenhoek,2009,96(4):423-439.
[5] 佘跃惠,张学礼,张凡,等.大港孔店油田水驱油藏微生物群落的分子研究[J].微生物学报,2005,45(3):329-334.
[6] Wagner Manfred,Wagner Martin,Piske Joachim,et al.Case histories of microbial prospection for oil and gas[J].AAPG Studies in Geology 48 and SEG Geophysical References Series,2002,11: 453-479.
[7] 袁志华,梅博文,佘跃惠,等.二连盆地马尼特坳陷天然气微生物勘探[J].天然气地球科学,2004,15(2):162-165.
[8] Di Cello F,Bevivino A,Chiarini L,et al.Biodiversity of aBurkholderia cepaciapopulation isolated from the maize rhizosphere at different plant growth stages[J].Appl Environ Microbiol,1997, 63(11):4485-4493.
[9] Baum M G,Bleschert K H,Wagner M,et al.Application of surface prospecting methods in the Dutch North Sea[J].Petroleum Geoscience,1997,3(2):171-181.
[10] Klusman R W,Saeed M A.Comparison of light hydrocarbon microseepage mechanisms.In:Schumacher D,Abrams M A,eds. Hydrocarbon migration and its near-surface expression[J]. AAPG Memoir,1996,66:157-168.
[11] Miller S R,Strong A L,Jones K L,et al.Bar-coded pyrosequencing reveals shared bacterial community properties along the temperature gradients of two alkaline hot springs in Yellowstone National Park[J].Appl Environ Microbiol,2009,75(13):4565-4572.
[12] Li H R,Yu Y,Luo W,et al.Bacterial diversity in surface sediments from the Pacific Arctic Ocean[J].Extremophiles,2009,13 (2):233-246.
[13] Pearson A,Rusch D B.Distribution of microbial terpenoid lipid cyclases in the global ocean metagenome[J].ISME J,2009,3 (3):352-363.
Study on Microbial Community in Soil Above A G as Reservoir
ZHANG Fan1,SHE Yue-hui2,KONGShu-qiong2,SHU Fu-chang2,YU G ao-ming2,HOU Du-jie1
(1.School ofEnergy Resources,China University of Geosciences(Beijing),Beijing100083,China; 2.College of Chemistry and Environmental Engineering,Yangtze University,J ingzhou434023,China)
16S rRNA gene clone library construction was used to analyze the microbial communities of soil sampled from above a known Ban876 gas reservoir.Two clone libraries DGS1 and DGS2 were constructed. There were 150 clones from the two libraries respectively for amplified ribosomal DNA restriction analysis (ARDRA).40 Taxanomic operational units(OTUs)were found in library DGS1,and 39 OTUs were found in library DGS2.Sequence analysis of the representative clone of each OTU showed that dominant bacteria of the DGS1 were affiliated withGemmatimonadetes28%,Chlorof lexi23%,Actinobacterium21%;andChlorof lexi 19%,Sulfur-oxidizing 12%,Acidobacteria10%,Gemmatimonadetes10%for DGS2,which provides foundation for application of MPOG(Microbial prospecting of oil and gas).
gas reservoir;16S rRNA gene;clone library;microbial community;microbial prospecting of oil and gas(MPOG)
Q 939 Q 785
A
1672-5425(2010)06-0054-05
中石油集团公司中青年创新基金资助项目(07E1025)
2010-03-19
张凡(1980-),女,湖北咸宁人,博士研究生,研究方向:石油微生物学;
侯读杰,教授,博士生导师。E-mail: bjbios001@126.com。

