植物几丁质酶纯化测定及应用研究进展
杨海霞,邓建军,2,张 建,3,赵广华,*
(1.中国农业大学食品科学与营养工程学院,北京100083;2.西北大学食品科学与工程系,陕西西安710069;3.石河子大学食品学院,新疆石河子832000)
植物几丁质酶纯化测定及应用研究进展
杨海霞1,邓建军1,2,张 建1,3,赵广华1,*
(1.中国农业大学食品科学与营养工程学院,北京100083;2.西北大学食品科学与工程系,陕西西安710069;3.石河子大学食品学院,新疆石河子832000)
植物几丁质酶是植物体中能够水解几丁质多聚体的一种致病性相关蛋白(Pathogenesis-related proteins)。近年来对于几丁质酶的研究报道中,大量新型的植物几丁质酶被分离纯化,并建立了不同的酶活测定方法,在几丁质酶的结构及分类方面也逐步有了系统的研究。从几丁质酶的结构及分类,分离纯化以及已建立的酶活测定方法等方面取得的新进展进行了综述,并展望了几丁质酶在农业、食品生产及药用领域的应用前景。
植物,几丁质酶,分类,纯化,酶活测定
植物在受到致病性微生物侵害时,会诱导表达大量不同基因编码的蛋白质——致病性相关蛋白(Pathogenesis-related proteins)来保护植株免受病原菌的侵害[1-4]。而在这些诱导的致病性相关蛋白质中,几丁质酶(Chitinase,E.C.3.2.1.14)被证明参与了寄主植物与真菌病原菌的相互作用[5-7]。几丁质酶又称壳多糖酶,能够水解几丁质内部的β-1,4-糖苷键,生产N-乙酰葡萄糖胺寡聚体。它广泛存在于植物体中,人们已从植物的根、茎、叶、果实以及种子中分离纯化得到多种几丁质酶[8-10],并且发现几丁质酶可以通过破坏菌丝顶端及其侧壁抑制真菌生长,从而达到抗真菌感染的目的;还可以通过释放寡糖诱导植物抗霉素的产生。抑菌实验结果表明当几丁质酶与β-1,3-葡聚糖酶共同作用时,抑菌效果会大大提高[7]。由于几丁质酶具有很强的抗真菌活性,近年来,几丁质酶已经成为工业、农业、生物技术以及食品等领域研究的热点。本文就植物来源的几丁质酶在结构、分类、分离纯化方法、酶活测定方法以及应用前景等方面进行了综述,为今后植物源几丁质酶的开发和应用提供技术参考。
1 几丁质酶的结构及分类
1.1 几丁质酶的结构
近年来,对多种植物的几丁质酶基因进行了克隆,研究发现植物几丁质酶的核苷酸序列与所编码的氨基酸序列在结构上存在一些规律性的变化,植物几丁质酶前体一般都含有N-端信号区(Signal peptide)、催化区(Catalytic domain)和 C-端延伸区(C-terminal extension),有的在N-端信号区之后有富含半胱氨酸的几丁质结合区域(Chitin binding domain,CBD),它与主结构区之间有一段可变交联区
(Flexible hinge region)[11](如图 1)。N-端信号区一般由20多个氨基酸组成,为疏水性,作用是调节酶的分泌作用,引导几丁质酶通过细胞膜运输到内质网后被信号肽酶裂解掉。有的胞内几丁质酶是没有N-端信号区的[12],不同几丁质酶的N-端信号区也是不同的。CBD是一段由40个氨基酸组成的富含半胱氨酸的高度保守结构[3],6个半胱氨酸残基位置基本固定,之间形成二硫键,可能是维持三维结构和蛋白之间的相互作用。可变交联区富含甘氨酸和脯氨酸,不同的几丁质酶的交联区长度差异很大。催化区的作用是水解底物,序列对比显示催化区有两段高度保守的区域,其中SxGG是结合底物部位,DxxDxDxE是催化水解底物区域[13],需要说明的是催化区的底物结合部位和CBD是两个独立的区域,研究表明CBD不能够增加几丁质水解速率,只能够增加几丁质酶的结合能力[14-15]。C-端延伸区的作用是用来引导几丁质酶进入植物液泡[16]。
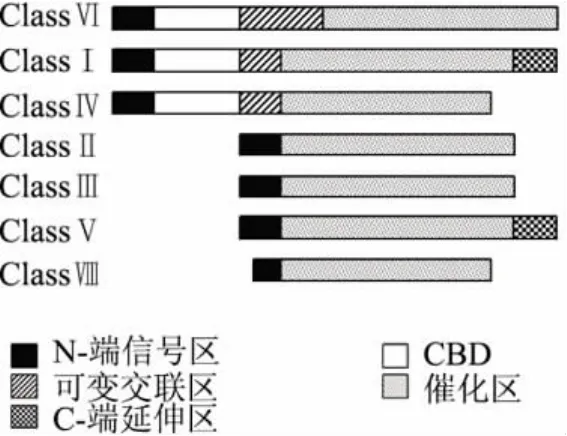
图1 植物几丁质酶的结构分类图
1.2 几丁质酶的分类
几丁质酶依据结构特点和氨基酸序列被分为以下几类[17],如图1所示:ClassⅠ在 N-端信号区后有几丁质交联区(CBD)以及可变交联区,连接着催化区,一般含有约300个氨基酸,此类几丁质酶呈碱性,大部分存在于液泡内;ClassⅡ的主要氨基酸序列和ClassⅠ有很强的同源性,只是缺少CBD和可变交联区,此类几丁质酶多数呈酸性,存在于胞质外;ClassⅢ的结构和前两类植物几丁质酶的结构很不相同,但是和细菌、真菌几丁质酶具有较高的同源性,同样不存在CBD区[18],它是胞外水解酶,在酶活性方面和溶菌酶有较大的相似性;ClassⅣ和ClassⅠ具有序列同源性,也同样含有CBD和保守的主要结构,ClassⅣ和ClassⅠ可用血清学方法来区分,ClassⅣ的成熟蛋白质基本含有241~255个氨基酸,分子量比ClassⅠ略小是由于有部分片段缺失[3]。Melchers el从烟草分离到一种新的几丁质酶,此酶的氨基酸序列与几种细菌的几丁质外切酶相似,但与已报道的植物几丁质酶不同,故将其定义为ClassesⅤ[10]。目前在烟草、荨麻、甜菜、豆科植物以及裸子植物中均已发现 ClassesⅤ几丁质酶[19-20]。ClassⅥ几丁质酶的N端序列含有两个重复排列的可变交联区域,而ClassⅦ则缺失CBD区域,与ClassⅣ有较高的同源性,已从小麦、棉花中提取得到此类酶[21-22]。
如果依据糖基水解酶系统分类,植物几丁质酶可以被分为两个家族,即Family 18和Family 19。所有的真菌、动物、细菌几丁质酶以及植物ClassⅢ、Ⅳ几丁质酶均为Family 18,植物 ClassⅠ、Ⅱ、Ⅴ、Ⅵ、Ⅶ为Family 19[23]。二者在氨基酸序列、三维结构以及分子机制等方面的差异表明它们分别起源于不同的祖先[21]。
2 植物几丁质酶的分离纯化方法
植物几丁质酶的分子量介于25~35kD之间。在以几丁质为底物时,最适反应pH一般低于7,其等电点介于3~10之间,植物几丁质酶有着较好的热稳定性(有的在60℃仍很稳定),且抗蛋白酶降解[24]。
植物几丁质酶可以通过不同的方法分离纯化,主要有硫酸铵盐析、离子交换层析、疏水作用层析、凝胶过滤层析、几丁质-亲和层析以及电泳等方法。分离纯化过程最好在低温(4℃)下进行。几丁质酶的酶活会随着纯化步骤的增加有所降低。提取植物几丁质酶一般按如下方法进行:将植物原料用适当pH的缓冲液匀浆,搅拌后过滤,得到的滤液离心去除不溶性淀粉及杂质,上清液中加入适当浓度的硫酸铵,使几丁质酶沉淀,静置一段时间,离心取沉淀,用缓冲液重新溶解成溶液状态,透析去除盐分等杂质,即得到几丁质酶的粗提液。纯化植物几丁质酶的方法有很多,依据不同几丁质酶的生物化学性质选择合适的纯化方法。Bloch and Burger利用几丁质与几丁质酶结合的原理,将酶粗提液过几丁质亲和柱,收集洗脱液即为纯化的几丁质酶[25]。Takeshi Watanabe等人通过离子交换层析和凝胶过滤层析对几丁质酶进行纯化[26],纯化过程中要选择适当的pH、缓冲液、洗脱盐浓度、流速等条件,也可利用电泳(Polyacrylamide Gel Electrophoresis)的方法制备得到纯化的几丁质酶[27]。
3 植物几丁质酶的酶活测定方法
植物几丁质酶主要是水解几丁质多聚体中的β-1,4-糖苷键,几丁质酶的水解活性主要有三种方式,即内切几丁质酶(Endochitinase)、外切几丁质酶(Exochitinase)以及 N-乙酰氨基己糖苷酶(GlcNAcase)[13,27]。内切几丁质酶水解位点为同聚物任意中间位点的β-1,4-糖苷键,生成可溶的、低分子量的 N-乙酰氨基葡糖多聚体,如:chitotetraose、chitotriose以及diacetylchitobiose,其作用底物大多为四糖以上的低聚糖,而且酶与底物亲和性也随着底物聚合度提高而增大[28];外切几丁质酶则作用于非还原端口依次切下单糖,它生成的产物只有diacetylchitobiose,没有单糖或低聚糖;N-乙酰氨基己糖苷酶利用外切形式将diacetylchitobiose和几丁质多聚体裂解成N-乙酰氨基葡糖单体[13]。离体实验表明,几丁质酶的活性底物还有其它物质;有的几丁质酶具有溶菌酶活性可作用于细菌肽聚糖、N-乙酰胞壁酸和N-乙酰葡萄糖之间的β-1,4-糖苷键;有的还能催化转糖基反应[29]。一般几丁质酶的内切作用和外切作用是同时进行的[12]。从许多植物包括大麦、玉米、烟草、马铃薯、大豆和大米中提取的几丁质酶都属于内切几丁质酶,主要产物为低聚糖[30]。
3.1 还原糖测定方法
目前,关于检测几丁质酶酶活的方法报道很多。最常用的方法是用胶体几丁质(Colloidal chitin)做底物,通过检测酶解后的产物还原糖进行酶活测定。胶体几丁质制备方法如Imanaka T等人所述[31],选择合适的酶解条件进行酶解反应后,生成产物还原糖利用Imato和Yagishita的方法测定,1U等于释放1μg 还原糖所需的酶量[32]。Nitoda T.K.等人发现用胶状几丁质的颗粒均一大小的粉末作底物,灵敏度比用胶状几丁质上清液高4倍[33]。几丁质的可溶性衍生物乙二醇几丁质(Glycolchitin)以及脱乙酰壳聚糖(Chitosan)被用作十分重要的测定酶活底物,酶解后通过紫外吸光值的测定反应酶活[34]。还有报道认为用胶状几丁质做底物测定还原糖的含量只测出外切几丁质酶的活性,如要测内切几丁质酶活性还需加入蜗牛肠酶酶解,再测定还原糖含量[35]。
3.2 生色底物测定方法
为了更好地检测溶液中几丁质酶的酶活,生色测定方法被建立并逐渐成熟起来。底物N-acetyl-D- glucosaminide(GlcNAc)、β - D - N,N′-diacetylchitobiose[(GlcNAc)2]以 及 β-D-N,N′,N″-triacetylchitotrios[(GlcNAc)3]可用生色基团p-nitrophenyl(pNP)分别标记为 pNP-GlcNAc,pNP-(GlcNAc)2以及 pNP-(GlcNAc)3。内切几丁质酶、外切几丁质酶以及N-乙酰氨基己糖苷酶分别酶解pNP-(GlcNAc)3、pNP-(GlcNAc)2以及 pNP-GlcNAc后释放pNP,通过荧光测定反应酶活。Nagel R.等人提出利用pNP-GlcNAc作底物比固体几丁质灵敏,几丁质酶对pNP-GlcNAc的水解速率是固体几丁质的三倍,1U等于释放1nmol pNP所用的酶量[36]。同样,底物被4-methylumbelliferyl(4-MU)修饰的测定酶活的方法也被建立起来。4-MU-(GlcNAc)3、4-MU-(GlcNAc)2和4-MU-GlcNAc酶解后可释放4-MU,通过荧光(激发波355nm,发射波460nm)分别测定反应内切几丁质酶、外切几丁质酶以及N-乙酰氨基己糖苷酶的酶活,1U即释放1nmol 4-MU所用的酶量。
3.3 染料底物测定方法
有些学者利用染料修饰胶状几丁质,利用酶解释放的染料具有特异吸光值来测定几丁质酶的活性。早在1964年Hackman和Goldberg就提出用有色几丁质衍生物Chitin Azure作底物,酶解后通过550nm处测定吸光值反映几丁质酶酶活,1U酶活被定义为吸光值增加0.001所需的酶量[37]。M.Go′mez Ramı′rez提出了一种快速灵敏的方法,利用Remazol Brilliant Blue RR(RBB)染色的胶状几丁质作底物,培养酶解后通过在595nm下测定吸光值反映几丁质酶的活性[38]。Ostazin Brilliant Red用作标记几丁质底物,酶解后释放的被染料标记的GlcNAc在530nm测 定 吸 光 值[39]。Mojtaba Mamarabadi将 底 物 用Remazol Brilliant Violet(RBV)修饰,酶解后在550nm下测定吸光值反映酶活[40]。
4 植物几丁质酶在食品中的应用前景
近些年来,人们从几丁质酶的酶学特性、抗菌机理以及分子克隆方面已经有了系统的研究,但是对于几丁质酶防御反应的机理还有待阐明;此外,目前学者的兴趣主要集中于寻找或重组有更好动力学特性的新型几丁质酶,以及对几丁质酶活性位点残基的识别[27]。由于植物几丁质酶的重要作用,其在食品及药物方面有着广泛的应用。
4.1 食品添加剂
由于几丁质酶具有良好的抑菌活性[7],所以在食品生产中,几丁质酶与其它抗微生物酶(葡萄糖氧化酶和乳过氧化物酶)或与传统的防腐措施(如山梨酸盐、乙醇、温度和低pH)联合使用,可以提高食品微生物的安全性,在糕点或饮料等食品中适当添加,可防止微生物的繁殖,起到防腐作用。
另外,几丁质对动植物而言是一种较难吸收利用的氮素来源,几丁质酶可将食品中的几丁质酶解,作用后所产生的N-乙酰几丁寡糖和壳聚几丁寡糖都具有清爽的甜味,吸湿性和保湿性好,而且在水中溶解度比单糖低,有助于调整食品的水活性,增进保水性,兼具调味和改良食品质构的功能。
4.2 降解产物的利用
几丁质酶的降解产物寡聚糖、葡糖胺以及N-乙酰氨基葡糖在医药及食品方面有着广阔的应用前景,我国渔业食品工业生产中的废弃物中含有大量几丁质,而传统工艺用酸水解虾壳等生产几丁寡糖成本昂贵且对容器腐蚀性大,利用几丁质酶水解法制备其降解产物,不仅能彻底有效的处理废壳,减少对环境的污染,还可以变废为宝,使降解产物得以利用,这种环保、经济、快速的方法无疑为企业开拓一条新的途径。
4.3 食品基因工程中的应用
由于几丁质酶具有显著的抗菌特性[7],近年来,通过转基因技术,将水稻、大麦、烟草等几丁质酶基因转入水稻、番茄等植物中用于病虫害防治,效果十分显著,不仅获得了高效而广谱的抗性,还克服了转基因植物的表达水平低的问题,应用前景十分广阔。
5 小结
总之,无论从几丁质资源的开发利用、减轻污染和保护环境的角度来看,还是几丁质原料及几丁质酶在农业、工业、食品及药物中的重要作用,充分研究几丁质酶,利用几丁质酶的特性开发新的应用前景将会给社会以及人类带来巨大帮助。
[1]Kitajima S,Sato F.Plant pathogenesis- related proteins:molecular mechanisms of gene expression and protein function[J].The Journal of Biochemistry,1999,125(1):1-8.
[2]Tyler B M.Entering and breaking:virulence effector proteins of oomycete plant pathogens[J].Cell Microbiology,2009,11(1):13-20.
[3]Collinge D B,Kragh K M,Mikkelsen J D,et al.Plant chitinases[J].Plant Journal,1993,3(1):31-40.
[4]Ellis J G,Rafiqi M,Gan P et al.Recent progress in discovery and functional analysis of effector proteins of fungal and oomycete plant pathogens[J].Current Opinion in Plant Biology,2009,12(4):399-405.
[5]Saito A,Fujii T,Shinya T,et al.The msiK gene,encoding the ATP- hydrolysing component of N,N′- diacetylchitobiose ABC transporters,is essential for induction of chitinase production in Streptomyces coelicolor A3(2) [J].Microbiology,2008,154(Pt 11):3358-3365.
[6]Shinya T,Hanai K,Galis I,et al.Characterization of NtChitIV,a class IV chitinase induced by beta-1,3-,1,6-glucan elicitor from Alternaria alternata 102:Antagonistic effect of salicylic acid and methyl jasmonate on the induction of NtChitIV[J].Biochemical and Biophysical Research Communication,2007,353(2):311-317.
[7]El G A,Wilson C L,Callahan A M.Induction of Chitinase,beta-1,3- Glucanase,and Phenylalanine Ammonia Lyase in Peach Fruit by UV- C Treatment[J].Phytopathology,2003,93(3):349-355.
[8]Graham L S,Sticklen M B.Plant chitinases[J].Canadian Journal of Botany,1994,72:1057-1083.
[9]Mauch F,Mauch-Mani B,Boller T.Antifungal hydrolases in pea tissue:II.Inhibition of fungal growth by combinations of chitinase and beta-1,3-glucanase[J].Plant Physiology,1988,88(3):936-942.
[10]Melchers L S,Apotheker-De G M,van der Knaap J A,et al.A new class of tobacco chitinases homologous to bacterial exochitinases displays antifungal activity[J].Plant Journal,1994,5(4):469-480.
[11]欧阳石文,赵开军,冯兰香,等.植物几丁质酶的研究进展[J].生物工程进展,2001,21:30-34.
[12]Seidl V,Huemer B,Seiboth B,et al.A complete survey of Trichoderma chitinases reveals three distinct subgroups of family 18 chitinases[J].Federation of European Biochemical Societies Journal,2005,272(22):5923-5939.
[13]Duo- Chuan L.Review offungalchitinases[J].Mycopathologia,2006,161(6):345-360.
[14]Kuranda M J,Robbins P W.Chitinase is required for cell separation during growth of Saccharomyces cerevisiae[J].Journal of Biological Chemistry,1991,266(29):19758-19767.
[15]Limon M C,Margolles-Clark E,Benitez T,et al.Addition of substrate-binding domains increases substrate-binding capacity and specific activity of a chitinase from Trichoderma harzianum[J].FEMS Microbiology Letters,2001,198(1):57-63.
[16]Neuhaus J M,Sticher L,Meins F J,et al.A short C-terminal sequence is necessary and sufficient for the targeting of chitinases to the plant vacuole[J].Proceedings of the National Academy of Sciences of the United States of America,1991,88(22):10362 -10366.
[17]Shinshi H,Neuhas J M,Ryals J,et al.Structure of a tobacco endochitinase gene:evidence that different chitinase genes can arise by transposition of sequences encoding a cysteine-rich domain[J].Plant Molecular Biology,1990,14(3):357-368.
[18]Kuo C J,Liao Y C,Yang J H,et al.Cloning and characterization ofan antifungalclass III chitinase from suspension- cultured bamboo(Bambusa oldhamii)cells[J].Journal of Agricultural and Food Chemistry,2008,56(23):11507-11514.
[19]Van Damme E J,Culerrier R,Barre A,et al.A novel family of lectins evolutionarily related to class V chitinases:an example of neofunctionalization in legumes[J].Plant Physiology,2007,144(2):662-672.
[20]Taira T,Hayashi H,Tajiri Y,et al.A plant class V chitinase from a cycad(Cycas revoluta):biochemical characterization,cDNA isolation,and posttranslational modification [J].Glycobiology,2009,19(12):1452-1461.
[21]Henrissat B,Bairoch A.New families in the classification of glycosyl hydrolases based on amino acid sequence similarities[J].Biochemical Journal,1993,293:781-788.
[22]Suzuki K,Taiyoji M,Sugawara N,et al.The third chitinase gene(chiC)of Serratia marcescens 2170 and the relationship of its product to other bacterial chitinases[J].Biochemical Journal,1999,343:587-596.
[23]Powning R F,Irzykiewicz H.Studies on the chitinase system in bean and other seeds[J].Comparative Biochemistry and Physiology,1965,14:127-133.
[24]Bloch R,Burger M M.Purification of wheat germ agglutinin using affinity chromatography on chitin[J].Biochemical and Biophysical Research Communications,1974,58(1):13-19.
[25]Watanabe T,Kobori K,Miyashita K et al.Identification of glutamic acid 204 and aspartic acid 200 in chitinase A1 of Bacillus circulans WL-12 as essential residues for chitinase activity[J].Journal of Biological Chemistry,1993,268(25):18567-18572.
[26]Dahiya N,Tewari R,Hoondal G S.Biotechnological aspects of chitinolytic enzymes:a review[J].Applied Microbiology and Biotechnology,2006,71(6):382-773.
[27]Molano J,Polacheck I,Duran A,et al.An endochitinase from wheat germ[J].Journal of Biological Chemistry,1979,254(11):4901-4907.
[28]Song H K,Suh S W.Refined structure of the chitinase from barley seeds at 2.0 a resolution[J].Acta Crystallographica Section D-biological Crystallography,1996,52(Pt 2):289-298.
[29]Imanaka T,FukuiT,Fujiwara S.Chitinase from Thermococcus kodakaraensis KOD1[J].Methods in Enzymology,2001,330:319-329.
[30]Imoto T,Yagishita K.A simple activity measurement of lysozyme[J].Agricultural and Biological Chemistry,1971,35:1154-1156.
[31]Nitoda T,Kurumatani H,Kanzaki H,et al.Improved bioassay method for Spodoptera litura chitinase inhibitors using a colloidal chitin powder with a uniform particle size as substrate[J].Pesticide Science,1999,55(5):563-565.
[32]Liau C Y,Lin C S.A modified coomassie brilliant blue G 250 staining method for the detection of chitinase activity and molecular weight after polyacrylamide gel electrophoresis[J].Journal of Bioscience and Bioengineering,2008,106(1):111-113.
[33]Boller T,Vogeli U.Vacuolar localization of ethylene-induced chitinase in bean leaves[J].Plant Physiology,1984,74(2):442-444.
[34]Kovacs K,Szakacs G,Pusztahelyi T,et al.Production of chitinolytic enzymes with Trichoderma longibrachiatum IMI 92027 in solid substrate fermentation[J].Applied Biochemistry and Biotechnology,2004,118(1-3):189-204.
[35]Nagel R,Birch R G,Manners J M.Detection of bacterial chitinase activity in transformed plant tumour cells using a specific exochitinase substrate[J].Plant Cell Reports,1990,8(12):729-732.
[36]Hackman R H,Goldberg M.New substrates for use with chitinases[J].Analytical Biochemistry,1964,8:397-401.
[37]Gomez R M,Rojas A L,Rojas A N,et al.Colloidal chitin stained with Remazol Brilliant Blue R,a useful substrate to select chitinolytic microorganisms and to evaluate chitinases[J].Journal of Microbiological Methods,2004,56(2):213-219.
[38]Shirazi F,Kulkarni M,Deshpande M V.A rapid and sensitive method for screening of chitinase inhibitors using Ostazin Brilliant Red labelled chitin as a substrate for chitinase assay[J].Letters in Applied Microbiology,2007,44(6):660-665.
[39]Mamarabadi M,Jensen B,Lubeck M.Three endochitinaseencoding genes identified in the biocontrol fungus Clonostachys rosea are differentially expressed[J].Current Genetics,2008,54(2):57-70.
Review on purification,enzyme assay and application of plant chitinases
YANG Hai-xia1,DENG Jian-jun1,2,ZHANG Jian1,3,ZHAO Guang-hua1,*
(1.College of Food Science and Nutritional Engineering,China Agricultural University,Beijing 100083,China;2.Department of Food Science and Engineering,Northwest University,Xi’an 710069,China;3.Food College,Shihezi University,Shihezi 832003,China)
Plant chitinases which hydrolyze the chitin is one of pathogenesis-related proteins and can be induced in resistance of plants to fungal pathogens.Thus,plant chitinases has a wide range of application as a antisepticise material.Recently,chitinases from different plants have been purified and their enzymatic activities have been assayed with varied methods.The structures and classifications,different methods on enzymatic activity assay and purification were summarized.Moreover,applications of chitinases such as in food and medicine field were prospected.
plant;chitinases;classification;purification;enzymatic activity assay
TS201.2+5
A
1002-0306(2011)06-0431-05
2010-05-13 *通讯联系人
杨海霞(1984-),女,博士研究生,研究方向:蛋白质化学与营养。

