猪瘟病毒RT-nPCR检测方法的建立及陕西部分地区猪瘟病毒E0基因分子特征分析
吴旭锦,朱小甫
(咸阳职业技术学院 畜牧兽医研究所,动物疫病分子生物学诊断实验室,陕西 咸阳 712000)
猪瘟(Classical swine fever,CSF)是由猪瘟病毒(Classical swine fever virus,CSFV)引起的一种高度接触传染性疫病,发病率和死亡率高,广泛分布于世界各国,严重威胁着养猪业的发展[1]。CSFV是黄病毒科、瘟病毒属成员,为单股正链RNA病毒。CSFV基因组长度约为12.3 kb,编码1个独立的多聚蛋白,自氨基端到羧基端排列顺序为Npro-C-E0-E1-E2-P7-NS2.3-NS4A-NS4B-NS5A-NS5B[2]。多聚蛋白被宿主蛋白酶修饰后可转变成不同的成熟蛋白,在这些蛋白中,E0蛋白能诱导机体产生中和性抗体,也是CSFV感染细胞后惟一可以分泌到细胞培养上清液中的糖蛋白[3]。研究发现,E0蛋白具有RNA酶活性,且E0可能是导致病毒持续感染宿主的重要原因[4]。由于猪瘟对养猪业有重要影响,多年来发展出了多种猪瘟诊断技术,在病原诊断上主要有免疫荧光技术、酶联免疫吸附试验、免疫胶体金技术和RT-PCR技术,其中以RT-PCR技术最为敏感,是现代诊断重点技术之一[5-6]。CSFVE0基因相对保守,是CSFV分子诊断的重要候选基因。长期以来,我国在猪瘟防控上一直坚持疫苗免疫为主的策略,但随着时间的推移,许多学者发现猪瘟病毒已经发生了变异,并怀疑猪瘟流行形势的变化与基因变异有关[7-8]。因此,本研究拟建立一种RT-nPCR扩增E0基因的检测方法,同时进行E0序列分析,以期为临床提供一种快速、灵敏、特异的诊断手段,并通过序列分析了解CSFV分子衍化情况,为猪瘟防控提供参考。
1 材料与方法
1.1 材 料
1.1.1 病 毒 猪瘟病毒参考株为疫苗毒株,参照毒株牛病毒性腹泻黏膜病病毒(BVDV)XY08株、蓝耳病病毒(PRRSV)CH-1R株、圆环病毒(PCV-2)SD06株、伪狂犬病毒(PRV) Bartha-K61株、细小病毒(PPV)ZJ05株为疫苗毒或分离毒株,均由咸阳职业技术学院畜牧兽医研究所动物疫病分子生物学诊断实验室保存。
1.1.2 组织病料 疑似猪瘟组织病料由动物疫病分子生物学诊断实验室采集,共计32份,分别来自咸阳市(11份)、西安市(5份)、宝鸡市(9份)、渭南市(5份)和汉中市(2份)猪场。组织病料包括脾脏、肝脏、肾脏、淋巴结和扁桃体,研磨处理后,12 000 r/min 高速离心10 min,取上清液于-70 ℃保存备用。
1.1.3 主要试剂 TRIzol Reagent为Invitrogen公司生产;反转录酶(AMV)、RNA酶抑制剂、DEPC处理水、rTaq酶、dNTP、EcoRⅠ、UNIQ-10柱式DNA胶回收试剂盒均购自生工生物工程(上海)有限公司;pMD18-T载体克隆试剂盒、BamHⅠ及Hind Ⅲ限制性内切酶均为TaKaRa公司产品;DH5α大肠杆菌由动物疫病分子生物学诊断实验室保存。
1.1.4 引物设计与合成 根据GenBank上发表的猪瘟病毒HCLV株(GenBank登录号:AF531433)、Shimen株(GenBank登录号:AF092448)全基因序列,设计2对引物,采用RT-nPCR方法扩增E0基因,引物序列如下,E0-1F:5′-AACCACCAGAATCTAGGAAG-3′,E0-1R:5′-GTGTTTTTGGGGAGGCAAGC-3′;E0-2F:5′-AAAGCCCTATTGGCATGGG-3′,E0-2R:5′-GGTGCAGTTGTTAGTGTACC-3′。预期扩增片段801 bp,引物由生工生物工程(上海)有限公司合成,用DEPC处理水溶解,终浓度为25 μmol/L,-20 ℃保存备用。
1.2 方 法
1.2.1 CSFV总RNA的提取及反转录 取CSFV疫苗毒250.0 μL,用TRIzol Reagent提取总RNA,空气中自然干燥。RNA干燥过程中,配制反转录反应液,组成为:DEPC处理水12.5 μL,5×AMV Buffer 4.0 μL,dNTP 2.0 μL,下游引物E0-1R(10 μmol/L)1.0 μL, AMV 0.25 μL,RNA酶抑制剂 0.25 μL,总体积20.0 μL。待RNA干燥后用配制好的反转录反应液充分溶解,4 000 r/min瞬时离心5 s,置42 ℃水浴反转录90 min,取出后立即冰浴,用微量紫外分光光度计测定cDNA浓度。
1.2.2 CSFV RT-nPCR检测方法的建立 以已知浓度的cDNA溶液作为模板,摸索扩增条件。第1次扩增反应体系中,cDNA 2.0 μL,10×PCR Buffer 2.5 μL,dNTP 1.0 μL,E0-1F、E0-1R(10 μmol/L)各0.5 μL,改变rTaqDNA聚合酶用量(0.25~1.0 μL)和Mg2+用量(0.5~3.0 μL),用超纯水补足25.0 μL。条件设定为95 ℃预变性5 min;进入循环后94 ℃ 50 s变性,退火温度由52 ℃至58 ℃退火1 min,72 ℃延伸 1 min,共30个循环;最后72 ℃延伸10 min。第2次扩增反应体系中,取2.0 μL第1次扩增产物作为模板,体系及条件同第1次扩增,引物为E0-2F、E0-2R。摸索出反应体系和反应条件的最佳组合。
1.2.3 RT-nPCR方法的灵敏度试验 将已知浓度的cDNA溶液做10倍梯度稀释,按照摸索出的最优体系与条件进行RT-nPCR反应,取5.0 μL第2次扩增产物,进行10 g/L琼脂糖凝胶电泳,于凝胶成像系统中照相观察。
1.2.4 RT-nPCR方法的特异性试验 提取CSFV、PRRSV、BVDV等RNA病毒的核酸并反转录获得cDNA;按照DNAzol Reagent试剂说明提取PCV-2、PRV和PPV等DNA病毒的DNA模板,用E0-1F/E0-1R、E0-2F/E0-2R引物对分别进行第1次和第2次扩增,用10 g/L琼脂糖凝胶电泳检测PCR产物。
1.2.5 病料中E0基因的PCR扩增 用以上建立的检测方法对收集的32份组织病料进行检测。选取不同地区E0基因阳性PCR产物,用UNIQ-10柱式DNA胶回收试剂盒回收PCR扩增条带,与pMD18-T载体进行连接,然后转化DH5α感受态细胞,经Amp+平板筛选,挑取单个菌落,摇动培养后进行菌液PCR鉴定。提取阳性菌质粒,进行BamH Ⅰ及Hind Ⅲ双酶切鉴定,将阳性质粒送生工生物工程(上海)有限公司进行测序。
1.2.6E0基因序列及推导氨基酸序列的比对分析 用DNAstar软件,将获得的8株猪瘟病毒E0基因序列与GenBank中发表的ALD(登录号D49532)、Alfort187(X87939)、Brescia(AF091661)、C HVRI(AY805221)、GXWZ02(AY367767)、HCLV(AF531433)、Paderborn (AY072924)、Rimes(AY259122)和Shimen(AF092448)等国内外猪瘟代表毒株,进行核苷酸和氨基酸同源性比对分析,绘制系统发生树。
2 结果与分析
2.1 CSFV RT-nPCR检测方法的建立
通过改变反应体系中rTaqDNA聚合酶用量和Mg2+用量,改变反应条件中的退火温度,优化反应体系和反应条件,最终确定最优体系和条件为:第1次扩增cDNA 2.0 μL,超纯水16.25 μL,10×PCR Buffer 2.5 μL,Mg2+2.0 μL,dNTP 1.0 μL,E0-1F、E0-1R(10 μmol/L)各0.5 μL,rTaqDNA聚合酶 0.25 μL,总体积25.0 μL。条件为:95 ℃预变性5 min;94 ℃ 50 s,54 ℃ 1 min,72 ℃ 1 min,共进行30个循环;最后72 ℃延伸10 min。第2次扩增时,取2.0 μL第1次扩增产物作为模板,体系同第1次扩增,引物为E0-2F/E0-2R。条件为:95 ℃预变性5 min;94 ℃ 50 s,58 ℃ 1 min,72 ℃ 1 min,共30个循环;最后72 ℃延伸10 min。
2.2 CSFV RT-nPCR检测方法的灵敏度
测定反转录第一链cDNA含量为670 ng/L,10倍梯度稀释后进行套式扩增。从图1可见,用所建立的方法能够扩增出可见目的条带的最大稀释度为107,即检测的cDNA含量极限为6.7×10-5ng/L,表明此方法灵敏度高,完全满足CSFV临床检测需要。
2.3 CSFV RT-nPCR检测方法的特异性
用所建立的方法对CSFV、PRRSV、BVDV、PCV-2、PRV和PPV阳性毒进行扩增,结果只有CSFV扩增出了801 bp的目的条带(图2),与预期条带长度一致,其他5种毒均未扩增出条带,提示所建立的方法特异性较好。
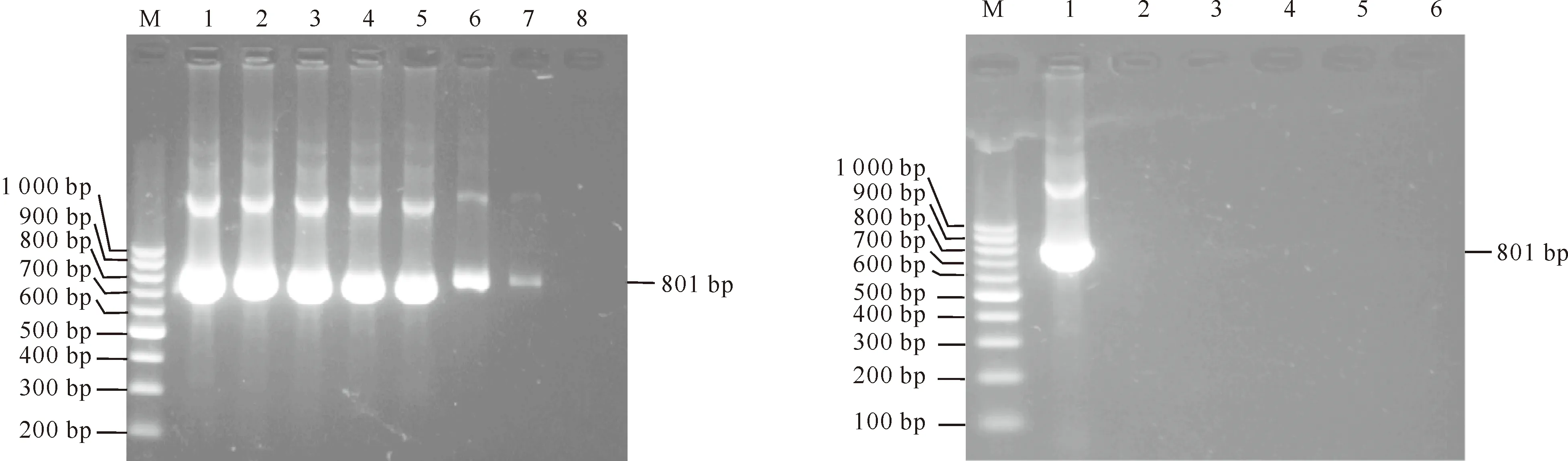
图1 CSFV E0基因 RT-nPCR检测方法的灵敏度
2.4 疑似病料的CSFV检测
用建立的RT-nPCR方法,检测采集的陕西省部分地区32份疑似猪瘟的淋巴结、脾脏、肝脏、肾脏和扁桃体等组织病料,结果有12份病料呈现阳性,阳性率为37.5%,部分病料检测结果见图3。
2.5 E0基因序列的测定
选取陕西不同地区8株CSFV毒株的E0克隆基因,回收产物连接pMD18-T载体,转化感受态细胞后提取阳性质粒,经BamH Ⅰ及Hind Ⅲ双酶切鉴定,获得一个完整的801 bp片段或538 bp和263 bp 2个小片段,原因是部分E0基因中存在Hind Ⅲ酶切位点(图4)。测序后获得8株CSFV流行毒株E0基因序列,分别命名为SXXY01-E0、SXXY02-E0、SXBJ01-E0、SXWN01-E0、SXWN02-E0、SXXA01-E0、SXXA02-E0和SXHZ01-E0。

图3 部分疑似CSF病料中CSFV E0基因的检测
2.6 E0基因及推导氨基酸序列的比对分析
将获得的8株E0基因核苷酸序列、推导氨基酸序列与9株参考毒株进行比对分析,同源性见表1,系统发生树见图5。
从表1可以看出,8株CSFV流行毒株E0核苷酸及其编码氨基酸同源性最高的是SXXA02-E0与SXHZ01-E0,分别为99.3%和98.5%,最低的是SXXY01-E0与SXWN02-E0,分别为97.0%和94.8%,其他各株间的同源性介于此范围内;这8个流行毒株之间的同源性较高。与参考毒株相比,核苷酸同源性最高的是SXXA02-E0与GXWZ02,为95.4%, 最低的是SXWN02-E0与HCLV,为83.4%;氨基酸同源性最高的仍为SXXA02-E0与GXWZ02,为98.1%,最低的是SXXA01-E0与Rimes,仅为85.8%。
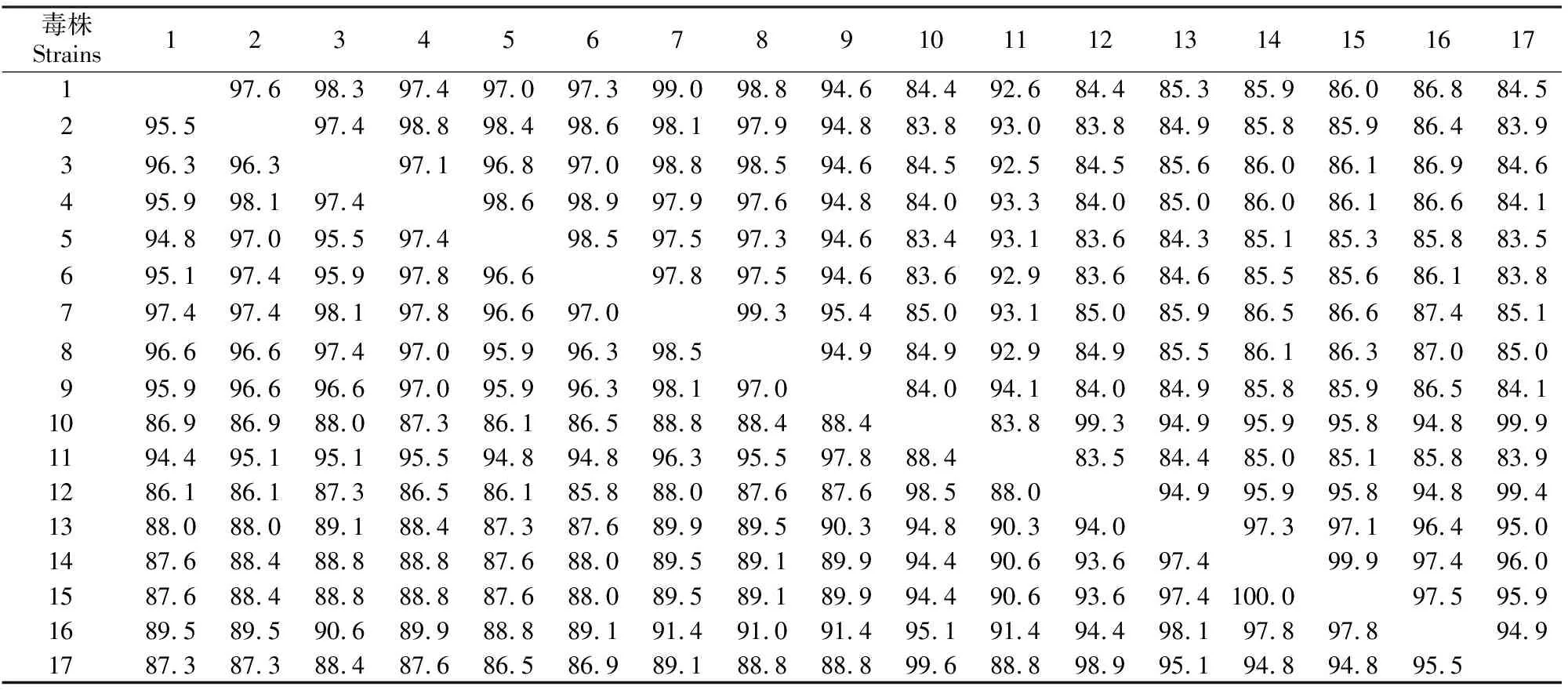
表1 不同CSFV毒株E0核苷酸、氨基酸序列同源性比对
从进化树(图5)可以看出,所有比对毒株分为2大基因群, HCLV、Rimes、Shimen、ALD、Alfort187、Brescia和C HVRI形成一个大的分支,为基因Ⅰ群;而本研究获得的8个(CSFV)流行毒株与GXWZ02、Paborn形成另一个大的分支,为基因Ⅱ群。
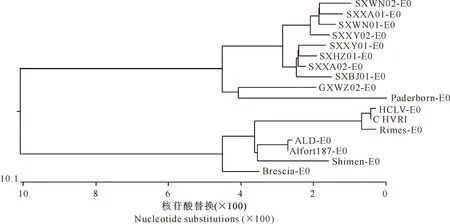
图5 CSFV流行毒株与参考毒株E0基因的系统发生树
E0蛋白中,氨基酸基序SLHGIWPE和EWNKHGWC具有Rnase活性,分别定位于第47~54位和第94~101位(图6)。比较发现,8个CSFV流行毒株E0蛋白的SLHGIWPE基序相同,与参考毒株相比,第54位的氨基酸大多数为E,但C HVRI、HCLV和Rimes 3个疫苗株均为G(图6左)。EWNKHGWC基序比较发现,除SXWN02第94位由E变异为K外,其他毒株均未发生任何变异(图6右)。

图6 CSFV E0蛋白2个Rnase活性区域的氨基酸序列比较
3 讨 论
猪瘟是猪群重大疫病之一,其快速诊断方法的建立对于防控猪瘟具有重要作用。目前诊断猪瘟的主要方法有荧光抗体染色、兔体中和试验、酶联免疫吸附试验、免疫胶体金技术和RT-PCR等技术。在这些方法中,RT-PCR技术具有快速、灵敏、特异的优点,适合大批量检测。许多学者也针对CSFV的不同基因建立了RT-PCR诊断方法,主要针对E2、NS5B、3′-UTR等区域[9]。本研究选取E0作为靶基因,建立了CSFV的RT-nPCR检测方法,原因是E0基因相对保守,便于扩增,并且扩增产物可直接进行回收测序,对研究E0基因的变异有重要意义。试验表明,所建立方法检测cDNA含量的极限为6.7×10-5ng/L,在常见的猪群疫病病毒中仅有CSFV可扩增出特异性条带,提示建立的方法灵敏度高、特异性好。
应用所建立的方法对采集的陕西省部分地区32份疑似猪瘟病料进行检测发现,有12份病料呈现阳性,阳性率为37.5%,表明所建立的方法能够很好地检测组织病料。此外,本试验结果显示,在陕西部分猪场猪瘟感染强度仍然较高,猪瘟仍是猪场防控的重点疫病,这一结果与其他学者近年的研究观点[10]相似。
近年来,猪瘟免疫失败的报道屡见不鲜,有许多研究认为,CSFV在长期免疫压力下基因变异是导致这一现象的原因之一[11-12]。E0蛋白具有Rnase活性,与病毒持续感染机体有直接关系,因而研究E0基因的变异趋势有重要意义[13-14]。本研究发现,测定的8株流行毒株E0基因与我国疫苗毒株HCLV、C HVRI的同源性仅为83.4%~85.1%,氨基酸同源性仅为86.1%~89.1%,呈现出较明显的远离疫苗株的变异趋势。E0蛋白中具有Rnase活性的氨基酸基序SLHGIWPE和EWNKHGWC,且8个流行毒株E0蛋白的SLHGIWPE基序相同。不同的是,3个疫苗株C HVRI、HCLV和Rimes第54位的氨基酸均为G,其他毒株均为E,这一变异是否与毒株的毒力有关,尚需进一步研究。比较发现,除SXWN02株EWNKHGWC基序第94位由E变异为K外,其他毒株均未发生任何变异。SXWN02株的这一变异为首次发现,尚未见到相似的公开报道,这一变异对毒株毒力、致病性有何影响,值得深入研究。
总之,本研究建立了一种特异性好、灵敏度高的猪瘟病毒RT-nPCR检测方法,并对克隆的E0基因进行了序列分析。以前研究认为,E0蛋白中具有Rnase活性的氨基酸基序高度保守,未发现变异[4,9-10,14],但本研究首次发现了Rnase活性氨基酸基序的变异现象,值得关注。综合多位学者的研究成果,CSFV基因变异并逐渐远离疫苗株已成为普遍现象,且在一些关键位点上呈现变异增多的趋势。这一情况需要引起高度重视,虽然目前疫苗尚能保护猪群[15],但随着病毒变异的积累,其后果难以预料。因此,我国需要做好针对流行毒株的新疫苗开发工作。
[参考文献]
[1] Barbara E S,Jeffrey J Z,Sylvie D A,等.猪病学 [M].9版.赵德明,张仲秋,沈建忠,译.北京:中国农业大学出版社,2008:325-335.
Barbara E S,Jeffrey J Z,Sylvie D A,et al.Diseases of swine [M].9th ed.Zhao D M,Zhang Z Q,Shen J Z,translation.Beijing:China Agricultural University Press,2008:325-335.(in Chinese)
[2] Meyers G,Thiel H J.Molecular characterization of pestiviruses [J].Advance Virus Research,1996,47:53-118.
[3] Rumenapf T,Unger G,Strauss J H,et al.Processing of the envelope glycoprotein of pestivirus [J].Journal of Virology,1993,67(5):3288-3294.
[4] Hulst M M,Panoto F E,Hoekman A,et al.Inactivation of the Rnase activity of glycoprotein Erns of classical swine fever virus in a cytopathogenic virus [J].Journal of Virology,1998,72(1):151-157.
[5] 朱小甫,张 志,李晓成,等.猪瘟病毒RT-nested PCR检测方法的优化和应用 [J].西北农林科技大学学报:自然科学版,2007,35(6):11-14.
Zhu X F,Zhang Z,Li X C,et al.Optimization and application of RT-nested PCR detection for classical swine fever virus [J].Journal of Northwest A&F University:Nat Sci Ed,2007,35(6):11-14.(in Chinese)
[6] 朱小甫,李晓成,陈德坤,等.猪瘟诊断技术研究进展 [J].中国动物检疫,2007,24(2):45-47.
Zhu X F,Li X C,Chen D K,et al.Progress of swine fever diagnostic techniques [J].China Animal Quarantine,2007,24(2):45-47.(in Chinese)
[7] Wirz B,Trascbin J D,Muler H.Detection hog cholera virus by polymerase chain reaction [J].Journal of Clinical Microbiology,1993,31:1148-1154.
[8] 王 波,张 鹏,杨增岐,等.陕西省PRRSV与CSFV,PCV2,PRV混合感染的检测 [J].西北农业学报,2009,18(5):27-30.
Wang B,Zhang P,Yang Z Q,et al.Detections of the co-infection of PRRSV,CSFV,PCV2 and PRV in Shaanxi Province [J].Acta Agriculturae Boreali-occiaentalis Sinica,2009,18(5):27-30.(in Chinese)
[9] 吴旭锦,朱小甫,陈德坤.2006-2007年陕西省古典猪瘟流行毒株E0基因的克隆及序列分析 [J].浙江大学学报:农业与生命科学版,2010,36(1):16-22.
Wu X J,Zhu X F,Chen D K.Cloning and sequence analysis of E0 gene of virulent classical swine fever virus isolates in Shaanxi Province in 2006-2007 [J].Journal of Zhejiang University:Agric & Life Sci,2010,36(1):16-22.(in Chinese)
[10] 王 琴,宁宜宝,王在时,等.猪瘟病毒流行株与疫苗株Erns基因的序列分析 [J].中国农业科学,2004,37(3):446-452.
Wang Q,Ning Y B,Wang Z S,et al.Sequence analysis of the Ernsgene of field isolates and vaccine strains of hog cholera virus [J].Scientia Agricultura Sinica,2004,37(3):446-452.(in Chinese)
[11] 朱小甫,吴旭锦.陕西省部分地区猪瘟流行毒株与疫苗毒株E2基因主要抗原区序列变异分析 [J].上海交通大学学报:农业科学版,2011,29(6):24-28.
Zhu X F,Wu X J.Sequence analysis of E2 gene of virulent classical swine fever virus isolates in Shaanxi province and vaccine strains [J].Journal of Shanghai Jiaotong University:Agricultural Science,2011,29(6):24-28.(in Chinese)
[12] 万 婧,陈 宁,童 超,等.猪瘟病毒浙江分离株体外生长特性及其囊膜糖蛋白E2分子的变异分析 [J].中国兽医科学,2011,41(8):794-798.
Wan J,Chen N,Tong C,et al.Growth characteristics of classical swine fever virus strains isolated in Zhejiang and genetic variations of their envelope glycoprotein E2 [J].Chinese Veterinary Science,2011,41(8):794-798.(in Chinese)
[13] Fernandez-Sainz I,Holinka L G,Gavrilov B K,et al.Altcration of the N-linked glycosylation condition in E1 glycoprotcin of classical swine fever virus strain brescia alters virulence in swine [J].Virology,2009,386(1):210-216.
[14] Meyers G,Saalmuller A,Buttner M.Mutations abrogating the Rnase activity in glycoprotein Ernsof the pestivirus classical swine fever virus lead to virus attenuation [J].Journal of Virology,1999,73(12):10224-10235.
[15] 王在时,丘惠深,郎洪武,等.猪瘟病毒流行株与疫苗株主要抗原编码基因差异研究 [J].中国兽药杂志,2001,35(1):1-3.
Wang Z S,Qiu H S,Lang H W,et al.Studies on the essential antigen coding gene difference of the field isolate strains and vaccine strain of hog cholera virus [J].Chinese Journal of Veterinary Drug,2001,35(1):1-3.(in Chinese)

