植原体基因组学研究进展
杨 毅, 车海彦, 曹学仁, 罗大全
(中国热带农业科学院环境与植物保护研究所,农业部热带作物有害生物综合治理重点实验室,海南省热带农业有害生物监测与控制重点实验室,海口 571101)
专论与综述
植原体基因组学研究进展
杨 毅, 车海彦, 曹学仁, 罗大全*
(中国热带农业科学院环境与植物保护研究所,农业部热带作物有害生物综合治理重点实验室,海南省热带农业有害生物监测与控制重点实验室,海口 571101)
植原体(phytoplasma)是一类重要的植物病原细菌,可经叶蝉、飞虱等昆虫介体传播,感染1 000多种植物,产生丛枝、黄化、韧皮部黑斑坏死等症状,给农业、林业生产带来巨大的损失。本文对植原体基因组学研究的历史、现状及当前植原体效应蛋白等方面的研究进展进行了综述。
植原体; 基因组; 效应蛋白; 病原-寄主互作
植原体(phytoplasma)是一类植物病原原核生物,分类上隶属于软壁菌门(Tenericutes,又称无壁菌门)柔膜菌纲(Mollicutes)植原体暂定属[Candidatus(Ca.) genus Phytoplasma]。它可通过具刺吸式口器的叶蝉、飞虱、木虱、蝽等半翅目昆虫感染一千多种植物。在被感染的植物中,植原体主要定殖于韧皮部的筛管细胞中,引起植物的病症有丛枝、黄化、植株退化矮缩、花变叶、花绿化以及韧皮部黑斑坏死等(图1)。在昆虫介体中,植原体随植物汁液被昆虫取食后进入昆虫肠道,再穿过肠壁细胞进入昆虫的体液循环,最后主要在唾腺细胞中增殖。当昆虫介体再次在植物的韧皮部取食时,植原体又随着昆虫回吐的唾液进入植物中,完成侵染循环[1]。植原体病害在世界各地广泛分布,对粮食作物、经济作物、林业及果树生产带来巨大的危害,我国发生的泡桐丛枝病、枣疯病、小麦蓝矮病、槟榔黄化病以及国外发生的葡萄黄化病、苹果簇生病、椰子致死黄化病等都是由植原体引起。
植原体只能寄生在植物或昆虫的活细胞内,迄今为止无法在人工培养基上进行培养,很难进行生理生化特性、代谢需求、致病机理以及与寄主互作等方面的研究。获得植原体的全基因组序列,是研究植原体这种难培养细菌的重要基础,也一直是植原体研究的热点与难点。
1 植原体的物理图谱研究
早期的植原体基因组研究,测定了植原体基因组的G+C含量和染色体的大小,并构建了植原体染色体的物理图谱。
G+C含量是基因组的一个重要特征,具有种属特异性,也是柔膜菌纲细菌鉴定和分类的重要参数。柔膜菌纲细菌的G+C含量大多在24%~35%之间[2]。1989年,Kollar等用氯化铯密度梯度离心结合高效液相色谱(HPLC)方法研究发现,苹果簇生植原体、长春花小叶植原体等基因组的G+C含量在23.0%~26.2%之间,同时在植原体的基因组中只检测到了很少量的甲基化碱基[3]。同年,Sears等用相似的技术方法对晚樱草(OenotherahookeriT. & G. strain Johansen)变叶病植原体基因组进行了研究,发现G+C含量小于30%,DNA复杂度相对较低,质粒有较高的拷贝数[4]。

图1 植物感染植原体后的症状Fig.1 Typical symptoms associated with phytoplasma presence
测定技术和手段的进步对促进自然科学的发展有重要作用,1983年发明的脉冲场电泳(pulsed-field gel electrophoresis, PFGE)是一种重要的分离大分子量DNA的电泳技术,远远突破了传统琼脂糖电泳只能分离不超过50 kb DNA片段的限制,使得分离的DNA片段大小上限达到了Mb级[5]。脉冲场电泳技术的发明,使得可以从整体上研究植原体的基因组。
1993年,Neimark等用脉冲场电泳方法,第一次从植物寄主中分离出完整的植原体染色体,研究发现植原体具有环形的染色体,大小为640~1 185 kb[6]。此后,Marcone等测定了12个主要植原体组中的共71个植原体的染色体,全面地对植原体的染色体大小进行了准确测定[7]。Marcone等的结果表明,翠菊黄化植原体组的植原体染色体大小在660~1 130 kb之间;僵化植原体组的植原体染色体在860~1 350 kb之间,其中STOLF 株系有已知最大的植原体染色体(1 350 kb);西方X病植原体组的染色体大小在670~1 075 kb之间;百慕大草白叶植原体(Bermuda grass white leaf phytoplasma)有最小的染色体(约为530 kb),不仅是柔膜菌纲中最小的染色体,而且是当时已知的细胞生物中最小的染色体;其余测定的植原体染色体都小于1 000 kb;试验中还发现有的样品中有不止一条植原体染色体带存在的现象[7]。
用脉冲场电泳,同时结合内切酶酶切和Southern杂交技术,迄今为止共构建出5个植原体株系染色体的物理图谱,分别为西方X病植原体(Western X-disease phytoplasma)、苹果簇生植原体AT株系(apple proliferation phytoplasma strain AT)、甜薯小叶植原体V4株系(sweet potato little leaf phytoplasma strain V4)、欧洲核果黄化植原体GSFY1株系(European stone fruit yellows phytoplasma strain GSFY1)和葡萄金黄化植原体FD92株系(flavescence dorée phytoplasma strain FD92),研究发现植原体都含有2个不连锁的rRNA操纵子[8-12]。植原体染色体物理图谱的获得,进一步明确了植原体染色体的大小、结构和rRNA操纵子、tuf、fus、gid等基因在植原体染色体上的排列和位置,以及植原体基因组之间的差异和遗传结构的多样性。
2 植原体基因组研究
2.1 植原体基因组测序
由于植原体全基因组序列对于植原体研究的重要性,许多实验室用不同的方法对植原体进行全基因组测序。
Liefting等用PFGE的方法纯化西方X病植原体的染色体,构建了染色体片段的粘粒文库(cosmid libraries),测得了西方X病植原体基因组的部分序列[13]。Melamed等试图从植原体的昆虫介体中分离无寄主DNA污染的植原体基因组DNA[14]。Garcia-Chapa等用氯化铯密度梯度离心结合PCR的方法对梨衰退植原体的基因组进行了研究[15]。Cimerman等用抑制消减杂交技术(suppression subtractive hybridization,SSH)研究了僵化植原体基因组[16]。Saccardo等用CTAB结合差速离心的方法提取植原体基因组,并用测序技术Illumina构建了植原体16SrIII组4个株系的基因组草图[17]。Chung等用鸟枪法对花生丛枝植原体基因组进行了研究,构建的基因组草图有13个染色体片段的重叠群,共562 kb,覆盖了90%的染色体[18]。我国也启动了小麦蓝矮植原体和泡桐丛枝植原体全基因组测序研究,并取得了前期结果[19-20]。
2004年,日本Namba实验室发表了首个植原体全基因组序列—洋葱黄化植原体全基因组序列。这是过去十年中,植原体研究最重要的进展。迄今,共有4个植原体的全基因组序列被报道,分别为日本Namba实验室报道的洋葱黄化植原体M株系(CandidatusPhytoplasma asteris line onion yellows-mild,OY-M)[21]、美国Hogenhout实验室报道的翠菊黄化植原体丛枝株系(aster yellows phytoplasma strain witches′ broom,AY-WB)[22]、澳大利亚Tran-Nguyen实验室报道的澳大利亚植原体暂定种tuf-Australia I;rp-A(CandidatusPhytoplasma australiense subgrouptuf-Australia I;rp-A)[23]和德国Kube实验室报道的苹果簇生植原体(CandidatusPhytoplasma mali strain AT)[24]。
尽管现在测序技术十分成熟,获得植原体的全基因组序列还是要克服很多困难,主要有以下原因。一、植原体是活细胞内专性寄生,不能体外培养,从寄主植物或昆虫介体中提取的植原体基因组中总是有大量寄主DNA污染,很难获得无污染的植原体DNA。二、某些植原体,尤其是寄生在木本植物中的植原体,通常含量较低,富集困难。测定苹果簇生植原体基因组序列时,先把感染苹果树、梨树和桃树的苹果簇生植原体通过菟丝子传染到长春花和烟草中,使植原体在长春花和烟草中大量富集,再从长春花和烟草中提取植原体DNA[24]。三、虽然用PFGE方法可以较好地去除植原体DNA中寄主DNA的污染,但此方法操作复杂,且由于植原体没有细胞壁,差速离心以及PFGE过程中很容易使菌体破裂DNA降解。用PFGE纯化植原体DNA,需要此植原体株系在自然寄主中含量丰富或可以转到其他植物中大量富集。Chung等进行花生丛枝植原体测序时,由于花生丛枝植原体在寄主长春花中含量很低,无法用PFGE进行纯化[18]。四、植原体基因组富含AT碱基,在构建文库时,寄主DNA比植原体DNA更容易克隆到载体里,很少的寄主DNA污染就会在文库中产生大量的非植原体克隆[25]。五、大多植原体基因组中富含重复序列。重复序列更容易克隆到文库中,这导致构建很大的文库才能覆盖全部植原体基因组[25]。大量的重复序列也给测序片段的拼接带来困难。六、某些植原体基因组的G+C含量非常低,这就伴随有大量的长同聚体和低复杂性的序列,导致用PCR方法填补拼接留下的空缺(gap)难以实现[18]。
2.2 植原体基因组的基本特征
比较分析已知的4个植原体基因组,有以下主要特征(表1)。植原体的染色体主要为环形,偶有线形染色体存在;G+C含量在21%~28%之间;600~800 kb的染色体上,约有70%的区域编码蛋白,基因组共有500~800个基因;有31~35个tRNA;有2个rRNA操纵子;植原体与无胆甾原体属的细菌一样用UGA作为终止密码子,而与植原体同属柔膜菌纲的支原体等大多数细菌用UGA编码色氨酸[21-24]。
植原体一般含有0~4个质粒(表1),不同株系中质粒的拷贝数从1至2个到几百个不等[26]。质粒上一般都有与质粒滚环复制相关的rep基因或danG基因以及ssb基因[22, 26-29]。质粒上的大多数基因都被预测为编码分泌蛋白和膜蛋白[22, 27],质粒上编码的膜蛋白可能与昆虫介体的传播以及寄主特异性有关[30-31],且质粒上的基因比染色体上的基因有更快的进化速度[32]。
2.3 植原体的生理生化及代谢特征
植原体有独特的生理生化及代谢特征。通过对基因组序列的分析发现,植原体缺少参与氨基酸合成途径、脂肪酸合成途径、氧化磷酸化、三羧酸循环的许多基因,植原体很可能是依赖糖酵解途径产生能量[21, 24]。柔膜菌纲的大多数细菌以及大肠杆菌(Escherichiacoli)、枯草芽胞杆菌(Bacillussubtilis)等细菌,都有保守的磷酸戊糖途径。细胞通过磷酸戊糖途径合成的NADPH来获得还原能力。磷酸戊糖途径还提供核苷酸合成所必需的5-磷酸核糖。而在植原体的基因组中,找不到参与磷酸戊糖途径的基因,植原体也缺少大多数合成核苷酸必需的基因[21, 24]。与植原体进化关系较远的立克次氏体,也具有不完整的核苷酸合成途径。立克次氏体与植原体都是寄生在细胞内,这些相同的基因丢失可能反映了趋同进化[21]。大多数细菌,都可以合成ATP,编码ATP合成酶,且所有的ATP合成酶亚基基因都被认为是独立复制的具细胞结构的生物必不可少的基因。而在植原体基因组中,8个ATP合成酶亚基基因都没有发现[21, 23]。植原体有较多的编码转运蛋白的基因,一些还是多拷贝基因,可能植原体主要从寄主中吸收所需的代谢物质[21, 24]。以上代谢途径和基因的缺失,可能是植原体长期寄生在营养丰富的细胞内,植原体的基因组产生了简化进化[21]。
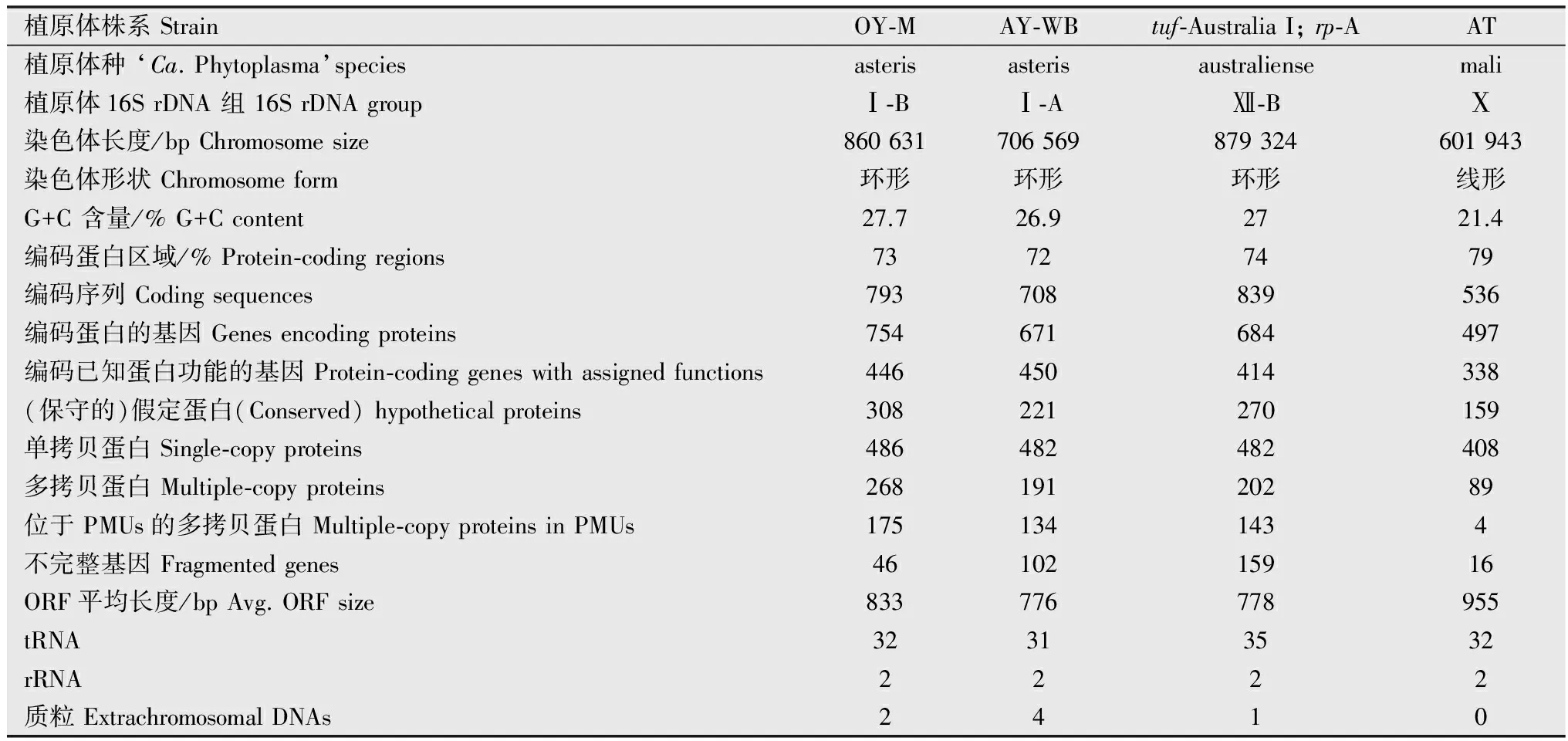
表1 植原体基因组的主要特征1)Table 1 General feature of phytoplasma genomes
1) 本表修改自Hogenhout等[25]。 This table is compiled based on Hogenhout et al., 2009[25].
2.4 PMU
尽管植原体基因组很小,但却富含重复序列。重复序列中,多个特定基因组成的基因簇,称为潜在移动元件(potential mobile unit, PMU)[21-24, 33]。植原体AY-WB株系的基因组中有7个PMU,其中1个PMU位于染色体195 949 bp至216 042 bp之间、包含21个ORF,称为PMU1[22]。PMU1的一端是328 bp的插入序列(insertion sequences,ISs)和1个完整的转座酶基因(tra5),另一端是反向重复的327 bp ISs和1个不完整的tra5,被tra5 IS包在中间的序列有DNA引发酶基因(dnaG)、DNA解旋酶基因(dnaB)、胸苷激酶基因(tmk)、Zn依赖蛋白酶基因(hflB)、DNA结合蛋白HU基因(himA)、单链DNA结合蛋白基因(ssb)和专化sigma因子(sigF)以及一些未知功能的基因[22]。在植原体AT株系中,预测有两个PMU,其中一个与AY-WB株系的PMU1相似,但是缺少himA,且一侧的tra5缺失,两侧的反向重复序列都缺失[24]。
PMU的序列特征和包含的基因表明,PMU可能以复制形式进行转座,且参与基因组的重组和修复。与支原体相比,植原体缺少一些DNA重组和修复功能。植原体很可能通过PMU参与的重组增加变异,使植原体适应不同的植物和昆虫寄主细胞环境[22]。
3 植原体的效应蛋白
由于植原体不能在体外培养,很难鉴定植原体的效应蛋白,植原体与植物寄主和昆虫寄主的互作研究一直以来进展缓慢。获得植原体全基因组序列信息以后,运用生物信息学方法,植原体的效应蛋白鉴定与分析研究进入了高速发展阶段。对植原体效应蛋白的深入研究将有助于揭示植原体与寄主互作的分子机制,为植原体病害新的防控策略提供理论依据。
3.1 SAP11
Hogenhout实验室用生物信息学软件SignalP、TMHMM2.0、PSORT、PredictNLS、BPROM、Neural Network Promoter Prediction等对植原体AY-WB株系的全基因组进行了分析,在693个假定蛋白中鉴定出76个具有N端信号肽(N-terminal signal peptide, SP)的蛋白,这76个蛋白中有56个分泌到寄主细胞质中,命名为AY-WB分泌蛋白(secreted AY-WB proteins, SAP),其中SAP11有1个N端信号肽和1个真核细胞核定位信号(nuclear localization signal, NLS)[34]。
Hogenhout实验室研究发现,植原体AY-WB感染的植物中,可检测到sap11基因转录,且SAP11蛋白定位于植物细胞核中并在细胞核中积累,突变了核定位信号的SAP11不在细胞核中积累[34]。这些结果表明,SAP11是植原体AY-WB编码的一个定位于寄主植物细胞核的蛋白,可能是植原体与寄主植物互作的一个效应蛋白。进一步的研究发现,SAP11通过操纵植物发育和植物防御素的合成,有利于其昆虫介体的繁殖[35]。
3.2 TENGU
Namba实验室对植原体OY基因组的分泌蛋白进行了研究,在鉴定了30多个分泌蛋白的功能后发现了1个效应蛋白,将其命名为TENGU[36]。TENGU是一个只有4.5 ku、38个氨基酸的分泌蛋白,N端的11个氨基酸是TENGU的功能域[37]。
Namba等研究发现,转tengu基因的烟草和拟南芥都表现出与感染植原体后相似的丛枝和矮化症状[36]。TENGU蛋白可从植物的韧皮部运输到顶端分生组织,并通过抑制植物的生长素合成及信号调控途径来影响植物发育[36]。对泡桐丛枝植原体的研究发现,泡桐丛枝植原体可显著影响泡桐体内细胞分裂素和ABA的合成[38]。植原体可能通过扰乱植物体内的激素平衡,以此影响植物发育,产生丛枝、矮化等症状[36]。
3.3 SAP54
花变叶和绿化症状(图1c),是植原体病害的典型症状之一。已有研究发现,在植原体感染的植物中,参与花器官形成的多种基因调控失常,有关花器官发育关键基因的表达发生改变[39-40]。
MacLean等研究发现,植原体AY-WB的效应蛋白SAP54在拟南芥中超表达后,可改变拟南芥的花发育,使拟南芥产生花变叶症状,且只在韧皮部特异表达SAP54也产生同样的症状[41]。
4 展望
全基因组序列是分子水平研究植物病原菌致病机理的重要基础,对于植原体这类不能体外培养的病原菌来说,获得全基因组序列尤为重要。由于植原体全基因组测序十分困难,迄今只有4种植原体株系的全基因组序列被测定。这4种植原体全基因组序列的发表,极大地推动了植原体致病机理的研究。在了解全基因组序列的基础上,已进一步明确了植原体独特的生理生化及代谢特征,鉴定出引起植物寄主丛枝、花变叶等植原体感染典型症状以及植原体与寄主互作的一些效应蛋白,但是这些效应蛋白与寄主互作的机理还有待进一步研究。
随着更多种植原体株系全基因组序列的测定, 并在此基础上进行的比较基因组学、转录组学以及功能基因组学研究,将会鉴定出更多的植原体效应蛋白,使我们深入了解植原体的致病机制、介体传毒和病原寄主互作的机理,最终掌握植原体病害的发生流行规律,发展出新的植原体病害防控对策。
[1]Weintraub P G, Beanland L. Insect vectors of phytoplasmas[J].Annual Review of Entomology, 2006,51:91-111.
[2]Razin S. Molecular biology and genetics of mycoplasmas(Mollicutes)[J]. Microbiological Reviews, 1985, 49(4):419-455.
[3]Kollar A, Seemuller E.Base composition of the DNA of mycoplasma-like organisms associated with various plant-diseases[J].Journal of Phytopathology-Phytopathologische Zeitschrift, 1989, 127(3):177-186.
[4]Sears B B, Lim P O, Holland N, et al. Isolation and characterization of DNA from a mycoplasma-like organism[J]. Molecular Plant-Microbe Interactions,1989,2(4):175-180.
[5]Schwartz D C, Saffran W, Welsh J, et al. New techniques for purifying large DNAs and studying their properties and packaging[J]. Cold Spring Harbor Symposia on Quantitative Biology, 1983, 47:189-195.
[6]Neimark H, Kirkpatrick B C. Isolation and characterization of full-length chromosomes from non-culturable plant-pathogenic mycoplasma-like organisms[J]. Molecular Microbiology, 1993, 7(1):21-28.
[7]Marcone C, Neimark H, Ragozzino A, et al. Chromosome sizes of phytoplasmas composing major phylogenetic groups and subgroups[J]. Phytopathology, 1999, 89(9):805-810.
[8]Firrao G, Smart C D, Kirkpatrick B C. Physical map of the western X-disease phytoplasma chromosome[J]. Journal of Bacteriology, 1996, 178(13):3985-3988.
[9]Lauer U, Seemuller E.Physical map of the chromosome of the apple proliferation phytoplasma[J]. Journal of Bacteriology, 2000, 182(5):1415-1418.
[10]Padovan A C, Firrao G, Schneider B, et al. Chromosome mapping of the sweet potato little leaf phytoplasma reveals genome heterogeneity within the phytoplasmas[J]. Microbiology, 2000, 146:893-902.
[11]Marcone C, Seemuller E.A chromosome map of the European stone fruit yellows phytoplasma[J]. Microbiology, 2001, 147:1213-1221.
[12]Malembic-Maher S, Constable F, Cimerman A, et al. A chromosome map of the flavescence doree phytoplasma[J]. Microbiology, 2008, 154:1454-1463.
[13]Liefting L W, Kirkpatrick B C. Cosmid cloning and sample sequencing of the genome of the uncultivable mollicute, Western X-disease phytoplasma, using DNA purified by pulsed-field gel electrophoresis[J]. FEMS Microbiology Letters, 2003, 221(2):203-211.
[14]Melamed S, Tanne E, Ben-Haim R, et al. Identification and characterization of phytoplasmal genes, employing a novel method of isolating phytoplasmal genomic DNA[J]. Journal of Bacteriology, 2003, 185(22):6513-6521.
[15]Garcia-Chapa M, Batlle A, Rekab D, et al. PCR-mediated whole genome amplification of phytoplasmas[J]. Journal of Microbiological Methods, 2004, 56(2):231-242.
[16]Cimerman A, Arnaud G, Foissac X. Stolbur phytoplasma genome survey achieved using a suppression subtractive hybridization approach with high specificity[J]. Applied and Environmental Microbiology, 2006, 72(5):3274-3283.
[17]Saccardo F, Martini M, Palmano S, et al. Genome drafts of four phytoplasma strains of the ribosomal group 16SrⅢ[J]. Microbiology, 2012,158:2805-2814.
[18]Chung W C, Chen L L, Lo W S, et al. Comparative analysis of the peanut witches′-broom phytoplasma genome reveals horizontal transfer of potential mobile units and effectors[J]. PLoS ONE,2013,8(4):e62770.
[19]陈旺, 李艳, 吴云锋. 小麦蓝矮植原体染色体DNA的分离[J]. 微生物学通报, 2013, 40(4):706-710.
[20]杨毅, 杨旭光, 林彩丽, 等. 泡桐丛枝植原体染色体全长及两个rRNA操纵子定位研究[J]. 植物检疫, 2011, 25(4):5-9.
[21]Oshima K, Kakizawa S, Nishigawa H, et al. Reductive evolution suggested from the complete genome sequence of a plant-pathogenic phytoplasma[J].Nature Genetics,2004,36(1):27-29.
[22]Bai X D, Zhang J H, Ewing A, et al. Living with genome instability:the adaptation of phytoplasmas to diverse environments of their insect and plant hosts[J]. Journal of Bacteriology, 2006, 188(10):3682-3696.
[23]Tran-Nguyen L T T, Kube M, Schneider B, et al. Comparative genome analysis of “CandidatusPhytoplasma australiense”(subgrouptuf-Australia I;rp-A) and “Ca. Phytoplasma asteris” strains OY-M and AY-WB [J]. Journal of Bacteriology, 2008, 190(11):3979-3991.
[24]Kube M, Schneider B, Kuhl H, et al. The linear chromosome of the plant-pathogenic mycoplasma ‘CandidatusPhytoplasma mali’[J]. BMC Genomics, 2008, 9:306.
[25]Hogenhout S A, Music M S. Phytoplasma genomics, from sequencing to comparative and functional genomics-what have we learnt?[C]∥Weintraub P G, Jones P. Phytoplasmas:genomes, plant hosts and vectors. Wallingford, UK:CABI, 2009:19-36.
[26]Petrzik K, Sarkisova T, Curnova L. Universal primers for plasmid detection and method for their relative quantification in phytoplasma-infected plants[J]. Bulletin of Insectology, 2011, 64:25-26.
[27]Petrzik K, Krawczyk K, Zwolinska A.Two high-copy plasmids found in plants associated with strains of “CandidatusPhytoplasma asteris”[J]. Plasmid, 2011, 66(2):122-127.
[28]Lin C L, Zhou T, Li H F, et al. Molecular characterisation of two plasmids from paulownia witches′-broom phytoplasma and detection of a plasmid-encoded protein in infected plants[J]. European Journal of Plant Pathology, 2009,123(3):321-330.
[29]郑文虎, 车海彦, 符瑞益, 等. 长春花小叶病植原体质粒DNA克隆及其分子特征[J]. 热带作物学报, 2012,33(1):94-98.
[30]Ishii Y, Kakizawa S, Hoshi A, et al. In the non-insect-transmissible line of onion yellows phytoplasma(OY-NIM), the plasmid-encoded transmembrane protein ORF3 lacks the major promoter region[J]. Microbiology,2009,155:2058-2067.
[31]Nishigawa H, Oshima K, Kakizawa S, et al. A plasmid from a non-insect-transmissible line of a phytoplasma lacks two open reading frames that exist in the plasmid from the wild-type line[J]. Gene, 2002,298(2):195-201.
[32]Ishii Y, Oshima K, Kakizawa S, et al. Process of reductive evolution during 10 years in plasmids of a non-insect-transmissible phytoplasma[J]. Gene, 2009,446(2):51-57.
[33]Arashida R, Kakizawa S, Hoshi A, et al. Heterogeneic dynamics of the structures of multiple gene clusters in two pathogenetically different lines originating from the same phytoplasma[J]. DNA and Cell Biology, 2008, 27(4):209-217.
[34]Bai X D, Correa V R, Toruno T Y, et al. AY-WB phytoplasma secretes a protein that targets plant cell nuclei[J]. Molecular Plant-Microbe Interactions, 2009, 22(1):18-30.
[35]Sugio A, Kingdom H N, MacLean A M, et al. Phytoplasma protein effector SAP11 enhances insect vector reproduction by manipulating plant development and defense hormone biosynthesis[J]. Proceedings of the National Academy of Sciences of the United States of America, 2011, 108(48):1254-1263.
[36]Hoshi A, Oshima K, Kakizawa S, et al. A unique virulence factor for proliferation and dwarfism in plants identified from a phytopathogenic bacterium[J]. Proceedings of the National Academy of Sciences of the United States of America, 2009, 106(15):6416-6421.
[37]Sugawara K, Honma Y, Komatsu K, et al. The alteration of plant morphology by small peptides released from the proteolytic processing of the bacterial peptide TENGU[J]. Plant Physiology, 2013, 162(4):2005-2014.
[38]Mou H Q, Lu J, Zhu S F, et al. Transcriptomic analysis of paulownia infected by paulownia witches′-broom phytoplasma[J]. PloS ONE, 2013, 8(10).
[39]Pracros P, Renaudin J, Eveillard S, et al. Tomato flower abnormalities induced by stolbur phytoplasma infection are associated with changes of expression of floral development genes[J].Molecular Plant-Microbe Interactions,2006,19(1):62-68.
[40]Himeno M, Neriya Y, Minato N, et al. Unique morphological changes in plant pathogenic phytoplasma-infected petunia flowers are related to transcriptional regulation of floral homeotic genes in an organ-specific manner[J]. Plant Journal, 2011, 67(6):971-979.
[41]MacLean A M, Sugio A, Makarova O V, et al. Phytoplasma effector SAP54 induces Indeterminate leaf-like flower development in Arabidopsis plants[J]. Plant Physiology, 2011, 157(2):831-841.
Researchprogressinphytoplasmagenomes
Yang Yi, Che Haiyan, Cao Xueren, Luo Daquan
(InstituteofEnvironmentalandPlantProtection,ChineseAcademyofTropicalAgriculturalSciences;KeyLaboratoryofIntegratedPestManagementonTropicalCrops,MinistryofAgriculture,P.R.China;HainanKeyLaboratoryforMonitoringandControlofTropicalAgriculturalPests,Haikou571101,China)
Phytoplasmas are important bacterial plant pathogens transmitted by insects, such as leafhoppers and plant hoppers. Phytoplasmas can infect thousands of plants and also frequently induce witches’ broom(proliferation of stems, branches, and leaves), generalized yellowing and phloem necrosis, which can cause devastating yield losses. This review summarized the research history of phytoplasma genomes, current opinions and phytoplasma effectors.
phytoplasma; genome; effector; pathogen-host interaction
2014-01-18
:2014-05-13
国家自然科学基金(31201492);中央级公益性科研院所基本科研业务费专项(2012hzs1J001)
S 432.1
:ADOI:10.3969/j.issn.0529-1542.2014.06.001
* 通信作者 Tel: 0898-66969235; E-mail: luodaquan@163.com

