激活沉默基因簇发掘微生物次级代谢产物的研究进展
杨康敏,高向东,顾觉奋
·综述·
激活沉默基因簇发掘微生物次级代谢产物的研究进展
杨康敏,高向东,顾觉奋
天然产物是指动物、植物、昆虫、海洋生物和微生物体内的组成成分或其代谢产物以及人和动物体内许许多多内源性的化学成分的总称。在医药产业,天然产物已是新药的重要来源之一,新的化学单体中,40%以上都由天然产物衍生而来,而天然产物多数是由微生物的次级代谢过程产生[1]。自20世纪70年代以来,尽管人们对治疗传染性疾病和癌症的药物需求越来越多,但是,发现新的活性天然产物的速度却在不断下降,传统方法已很难获得新的次级代谢产物。如今的DNA测序技术已经能够快速、方便地检测大量基因组序列。其结果提示我们:作为天然活性产物的重要来源,尽管细菌和真菌已被深入研究,但其次级代谢产物合成基因簇的数目远远超出我们的预期,仍有大量的次级代谢产物等待发掘。有两个原因限制了新化合物的发现:①能够进行人工培养的微生物种类比例极低,目前为止仅有0.1%~1%的微生物家族成员被成功地在实验室培养[2];②大部分次级代谢产物合成基因簇在常规实验条件下处于沉默状态。若这些潜在的生物合成途径被激活,则有可能发现许多新活性产物,打破现有新药研发的瓶颈[3]。
目前,为激活沉默基因簇,已报道了多种方法,其中包括改变微生物的培养条件,例如培养基成分、通气速率、pH、温度、光照等,使它们产生更多类型或更大数量的次级代谢产物,即one strain many compounds(OSMAC)法。此方法被成功应用于aspoquinolone A~D的发现,研究人员发现在构巢曲菌基因组中存在可编码多种邻氨基苯甲酸合酶的基因,后者可催化喹啉或喹唑啉生物碱的合成,通过在不同条件下培养构巢曲菌,利用紫外光谱法和质谱法筛选后,获得了新化合物aspoquinoloneA~D(图1)。但OSMAC法在许多情况下(例如改变培养条件)并不能使微生物生产新的次级代谢产物,应用范围受到极大限制。近年来,研究人员通过采用不同手段、不同途径探索激活沉默基因簇,并取得一定成果,本文就这一领域的最新进展进行简要综述。
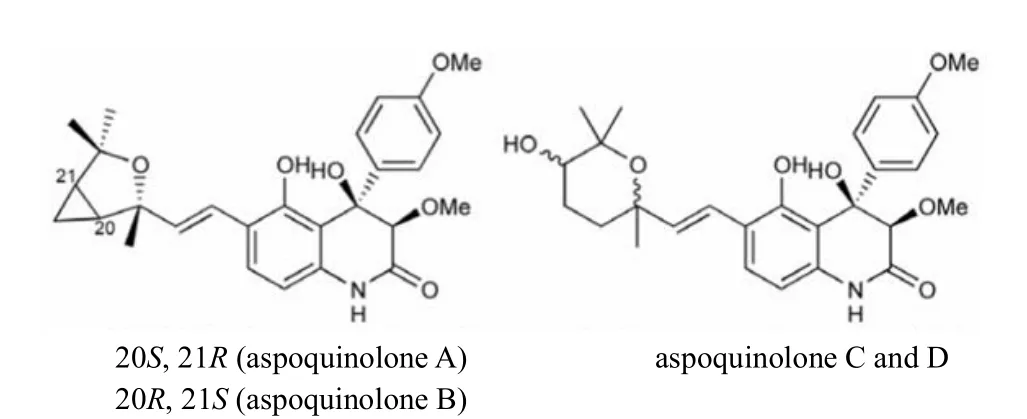
图1 利用OSMAC法在构巢曲菌发酵产物中分离得到的aspoquinoloneA~D
1 聚酮合酶和非核糖体肽合酶系统
绝大多数次级代谢产物都由非核糖体多肽或多聚酮衍生而来,其表达过程都有多功能酶——聚酮合酶(PKSs)和非核糖体肽合酶(NRPSs)的参与。PKSs和NRPSs分别利用简单的丙二酰或氨基酸来构建分子片段,并用非常相似的方法进行组装[4]。完整的PKS系统由许多模块组成,每个模块负责一个独立的链延伸步骤,并且可被细分为不同的域(domain)。一个典型的PKS模块至少包含一个负责选择和转移延伸单元的酰基转移酶域AT,一个负责荷载延伸单元的酰基载体蛋白域ACP和负责延伸单元脱羧化缩合的酮脂酰合成酶域KS。同样地,NRPS模块至少包含一个负责氨基酸活化的腺苷酰化域A,一个硫醇化域T和一个催化肽键形成的缩合域C;此外,有些模块还包含许多非必需域(图2)。在细菌中,有许多证据证明PKS和NRPS系统还可以共同作用,产生杂合产物[5]。近年来,根据PKS和NRPS系统,并利用基因工程技术,多种激活沉默基因的有效方法被发展起来。
2 过表达通路特异性转录因子
过表达某种通路特异性转录因子是激活沉默基因簇的有效方法之一,它可同时提高整个基因簇的表达,产生最终的次级代谢产物,只需操控一个很小的基因。此技术首次被应用于过表达构巢曲菌中的转录因子基因apdR。apdR是沉默基因簇apd的一部分,而后者中部包含一段PKS-NRPS杂合基因。Bergmann等[6]发现,过表达apdR可以激活整段基因簇,最终导致了一类新细胞毒性化合物——aspyridoneA、B的发现(图3)。但此技术也存在一定缺陷,其操作的成功性目前还不可预测。此外,某些通路特异性转录因子需要进行翻译后修饰,这可能会降低待表达次级代谢产物的产量。例如,在构巢曲菌中,调控柄曲霉素基因簇表达的特异性转录因子AflR在被翻译后必须进行磷酸化才能成功进入核内[7]。
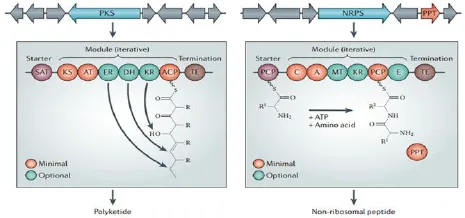
图2 PKS和NRPS模块结构
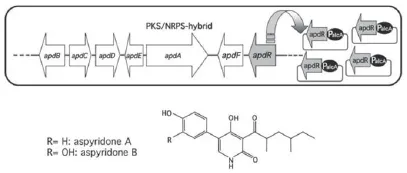
图3 在构巢曲菌中过表达apdR基因以产生aspyridoneA、B
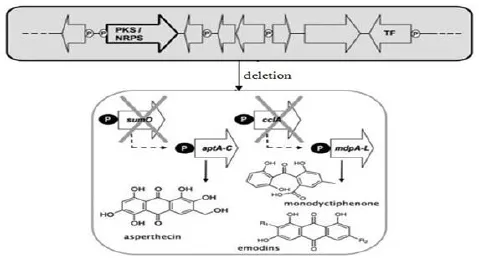
图4 在构巢曲菌中敲除sumO基因以提高asperthecin产量;敲除cclA基因以获得monodyctiphenone、emodins
3 过表达通用转录因子
除了通路特异性转录因子,微生物基因组中还存在一些通用转录因子,例如LaeA,它是一种核内蛋白,具有调控曲霉属真菌次级代谢产物表达的功能[8]。敲除laeA基因会降低构巢曲菌中柄曲霉素、青霉素以及烟曲霉菌中胶霉毒素的表达。相反,过表达laeA能够增加这些次级代谢产物产量。利用此方法,研究人员在构巢曲菌中成功表达了terrequinoneA。Terrequinone A具有抗癌活性,有着广阔的应用前景和市场潜力[9]。
4 表观遗传调控/染色质水平的重构
组蛋白修饰可以通过改变染色质的乙酰化、甲基化、磷酸化、泛素化和类泛素化等来调节基因转录。Shwab等[10]证实了组氨酸乙酰化在调节曲霉菌天然产物合成过程中的重要作用。曲霉菌基因组中的hdaA基因可编码组蛋白脱乙酰酶,将其敲除可提高杂色曲霉素和青霉素的产量。此外,COMPASS复合物能够促进组蛋白H3第四位赖氨酸(H3K4)的甲基化,而后者被认为是染色质变松散、转录因子被活化的标志[11]。Bok等[12]将构巢曲菌中编码COMPASS复合物的cclA基因敲除,发现其可诱导大黄素及其衍生物以及组织蛋白酶K抑制剂F-9775A和F-9775B等合成基因的表达。类似地,Szewczyk等[13]将构巢曲菌中的类泛素化基因sumO敲除后,可激活某些次级代谢途径,使asperthecin产量增加(图4)。
5 次级代谢生物合成基因簇的异源表达
过表达转录因子以及染色质水平的调节只能应用于拥有较高级分子遗传系统的微生物中,若要将这些技术普遍应用于其他微生物,必须引入一套完善的分子遗传系统,或者将待表达的次级代谢生物合成基因簇引入到合适的异源宿主微生物中。例如,唐德链霉菌S.tendae Tu1028含有吩嗪化合物生物合成基因簇,但在常规条件下处于沉默状态,Saleh等[14]将该基因簇引入到宿主菌天蓝链霉菌S.coelicolor M512基因组中进行表达,通过在关键基因前加入红霉素抗性基因强启动子,成功分离到了具有抗肿瘤和抗细菌等多种活性的吩嗪化合物。目前,放线菌沉默基因簇异源表达的常用宿主菌有白色链霉菌、天蓝链霉菌和大肠杆菌等。异源表达存在的问题是,由于包含PKSs和NRPSs基因的基因簇可长达100 kb,将其引入异源宿主中相对困难[15]。此外,异源表达时必须将宿主微生物基因簇中原有启动子替换为强启动子或可诱导启动子,当次级代谢产物合成通路中含有较多基因时,此方法便会受到限制。
6 核糖体工程
野生型变铅青链霉菌本身由于相应合成基因的休眠通常不会生产抗生素,但是当其核糖体蛋白S12突变,引入链霉素抗性后,便能生产大量蓝色的放线紫红素。另一方面,细菌的信号素ppGpp(高度磷酸化的鸟苷酸分子,包括鸟苷酸五磷酸及四磷酸,简称ppGpp)在核糖体中产生,它可以和RNA聚合酶(RNAP)结合,导致抗生素的产生[16]。以上研究说明,引入某种抗性的RNAP修饰可模拟ppGpp结合方式,从而激活合成基因簇的表达。据此,Chai等[17]建立了“核糖体工程”方法,以核糖体S12蛋白、转录因子以及RNAP等为靶,通过改造,调控转录(翻译)途径,激活或提高沉默(或低表达)的基因簇的表达。他们将海洋真菌产紫青霉菌G59经50%DMSO介导2 mg/ml庆大霉素处理5 d,使其核糖体蛋白S12发生突变,筛选得到庆大霉素抗性突变株,在其发酵产物中分离纯化得到了麦角甾醇(ergosterol)、fructigenine A、rugulosuvineA等具有抗肿瘤活性的新次级代谢产物。
7 利用物种内部通讯机制
次级代谢产物的功能之一是为产生它的微生物应对周围环境提供便利。某些情况下次级代谢产物被用以对抗一些紫外线辐射等非生物介质,但更多情况下微生物利用其次级代谢产物来对抗其他微生物,这提示我们,可能存在某些感应机制来控制次级代谢产物的产生。如果可以利用微生物与环境之间的相互作用,便有可能激活沉默基因簇产生新次级代谢产物。Schroeckh等[18]通过细菌-真菌相互作用,利用基因芯片技术监测了真菌沉默基因簇的选择性激活。将构巢曲菌和采集的58种不同的放线菌进行共培养,发现其中一种放线菌——S.rapamycinicus,在与构巢曲菌接触后,可诱导两个次级代谢基因簇的表达,其中一个可以产生芳香多酮类化合物苔色酸(orsellinic acid)、红粉苔酸(lecanoric acid)以及组织蛋白酶K抑制剂F-9775A和F-9775B。透析实验和电镜成像显示,构巢曲菌与S.rapamycinicus之间的物理接触是激活沉默PKSs基因的必要条件。此方法也证明了将不同微生物共培养以获得新次级代谢产物的可行性。
8 展望
目前,先进的基因组测序技术证明了大量未知的次级代谢生物合成基因簇的存在,还有大量的次级代谢产物等待被发现。各种卓有成效的激活沉默基因簇的手段使得对新天然产物的发掘已进入了一个新的阶段。据统计,通过上述方法,已有多达50种新次级代谢产物被发现[19]。研究人员还在不断探索新的方法,生物信息学在此领域正扮演越来越重要的角色,其最终目标是利用基因组的测序信息,在未知的基因簇中预测新的天然产物。此外,近年来,蛋白质组学也扩充了鉴定沉默基因簇的方法。今后寻找新次级代谢产物的趋势便是筛选方法的自动化以及基因组学、分子遗传学、生物化学等方法的协同组合。随着越来越多的方法被发明以及改善,沉默的生物合成途径将会成为新药开发的宝贵资源。
此外,寻找这些基因簇在常规条件下处于沉默的原因也同样十分重要,它们是否会在我们还未知的环境下自发表达?这些特殊的环境是怎样的?在这种环境下,微生物所产生的次级代谢产物的固有功能是什么?可以预见,当了解隐藏在这些沉默基因簇背后的机制后,人们对微生物次级代谢途径的利用将会更加充分。
[1]Scharf DH,Brakhage AA.Engineering fungal secondary metabolism:a roadmap to novel compounds.J Biotechnol,2013,163(2):179-183.
[2]Yin J,Straight PD,Hrvatin S,et al.Genome-wide high-throughput mining of natural-product biosynthetic gene clusters by phage display. Chem Biol,2007,14(3):303-312.
[3]Ochi K,Hosaka T.New strategies for drug discovery:activation of silent or weakly expressed microbial gene clusters.Appl Microbiol Biotech,2013,97(1):87-98.
[4]Hertweck C.The biosynthetic logic of polyketide diversity.Angew Chem Int Ed Engl,2009,48(26):4688-4716.
[5]Wenzel SC,Kunze B,Höfle G,et al.Structure and biosynthesis of myxochromides S1-3 in Stigmatella aurantiaca:evidence for an iterative bacterial type I polyketide synthase and for module skipping in nonribosomal peptide biosynthesis.Chem Bio Chem,2005,6(2):375-385.
[6]Bergmann S,Schümann J,Scherlach K,et al.Genomics-driven discovery of PKS-NRPS hybrid metabolites from Aspergillus nidulans. Nat Chem Biol,2007,3(4):213-217.
[7]Chiang YM,Chang SL,Oakley BR,et al.Recent advances in awakening silent biosynthetic gene clusters and linking orphan clusters to natural products in microorganisms.Curr Opin Chem Biol,2011,15(1):137-143.
[8]Chiang YM,Lee KH,Sanchez JF,et al.Unlocking fungal cryptic natural products.Nat Prod Commun,2009,4(11):1505-1510.
[9]Bok JW,Hoffmeister D,Maggio-Hall LA,et al.Genomic mining foraspergillus natural products.Chem Biol,2006,13(1):31-37.
[10]Shwab EK,Bok JW,Tribus M,et al.Histone deacetylase activity regulates chemical diversity in Aspergillus.Eukaryotic Cell,2007,6(9):1656-1664.
[11]Song BY,Zhu WG.Advances in effector protein of histone methylation.Hereditas,2011,33(4):285-292.(in Chinese)
宋博研,朱卫国.组蛋白甲基化修饰效应分子的研究进展.遗传,2011,33(4):285-292.
[12]Bok JW,Chiang YM,Szewczyk E,et al.Chromatin-level regulation of biosynthetic gene clusters.Nat Chem Biol,2009,5(7):462-464.
[13]Szewczyk E,Chiang YM,Oakley CE,et al.Identification and characterization of the asperthecin gene cluster ofAspergillus nidulans. Appl Environ Microb,2008,74(24):7607-7612.
[14]Saleh O,Bonitz T,Flinspach K,et al.Activation of a silent phenazine biosynthetic gene cluster reveals a novel natural product and a new resistance mechanism against phenazines.Med Chem Commun,2012,3(8):1009-1019.
[15]Chen LY,Wang YM,Zhao XQ.Nature product discovery of marine actinobacteria by genome mining:strategies and prospects.Microbiol China,2013,40(10):1896-1908.(in Chinese)
陈亮宇,王玉梅,赵心清.基因组挖掘技术在海洋放线菌天然产物研究开发中的应用及展望.微生物学通报,2013,40(10):1896-1908.
[16]Ochi K.From microbial differentiation to ribosome engineering. Biosci Biotechnol Biochem,2007,71(6):1373-1386.
[17]Chai YJ,Cui CB,Li CW,et al.Activation of the dormant secondary metabolite production by introducing gentamicin-resistance in a marine-derived Penicillium purpurogenum G59.Mar Drugs,2012,10(3):559-582.
[18]SchroeckhV,ScherlachK,NützmannHW,etal.Intimate bacterial-fungalinteractiontriggersbiosynthesisofarchetypal polyketides in Aspergillus nidulans.Proc Natl Acad Sci U S A,2009,106(34):14558-14563.
[19]Brakhage AA,Schroeckh V.Fungal secondary metabolites-strategies to activate silent gene clusters.Fungal Genet Biol,2011,48(1):15-22.
10.3969/cmba.j.issn.1673-713X.2015.01.015
210009南京,中国药科大学生命科学与技术学院
顾觉奋,Email:yqyan1@126.com,高向东,Email:xdgao@ cpu.edu.cn
2014-06-03

