葡萄糖基氟虫腈处理下蓖麻内参基因的稳定性分析
毛根林, 解 云, 赵俊龙, 陈 炎, 徐汉虹, 林 菲
(华南农业大学 农学院/天然农药与化学生物学教育部重点实验室, 广东 广州 510642)
葡萄糖基氟虫腈处理下蓖麻内参基因的稳定性分析
毛根林, 解云, 赵俊龙, 陈炎, 徐汉虹, 林菲
(华南农业大学 农学院/天然农药与化学生物学教育部重点实验室, 广东 广州 510642)
摘要:【目的】筛选葡萄糖基氟虫腈(GTF)及溶剂二甲基亚砜(DMSO)处理下蓖麻Ricinuscommunis稳定的内参基因,为研究GTF的韧皮部装载机制提供参考。【方法】选取Actin,ARC,ef1a,SamDC,TUA6为内参基因,通过实时荧光定量PCR分析基因表达量并利用geNorm,NormFinder,BestKeeper,Delta CT软件及RefFinder在线分析工具综合比较不同时间和不同浓度的GTF与DMSO处理后,5个候选内参基因在蓖麻幼苗子叶中表达的稳定性。【结果】各软件分析得出的内参基因稳定性排名依次为geNorm:Actin=ef1a>SamDC>ARC>TUA6;NormFinder:SamDC>ARC>Actin>ef1a>TUA6;BestKeeper:Actin>ef1a>SamDC>ARC>TUA6;Delta CT:SamDC>Actin>ARC>ef1a>TUA6; RefFinder:Actin>SamDC>ef1a>ARC>TUA6,而单独分析DMSO处理时,稳定性排名则为:ef1a>SamDC>Actin>TUA6>ARC。【结论】综合分析GTF和DMSO处理,Actin的表达最稳定;在DMSO处理下,则ef1a最为稳定。
关键词:蓖麻; 葡萄糖基氟虫腈; 二甲基亚砜; 实时荧光定量PCR; 内参基因; 稳定性
农药施用减量增效要求在减少农药施用量的同时提高农药的使用效率,降低农药残留,保证食品安全,这也是当前农药研究者共同努力的方向。徐汉虹等[1]提出的“导向农药”理念完美契合农药减量增效的目的,对保障食品安全具有非常重要的意义。导向农药能够在植物内源转运蛋白的作用下定向积累至病虫为害部位,它的作用方式可以分为2种:一是当植物受到危害时,导向农药分子能够被植物应激反应产生的酶消解并释放出农药母体分子作用于靶标;二是具有农药活性的导向分子直接作用于靶标从而达到防治病虫害的目的。植物内源糖从源叶合成,在糖转运蛋白介导下源源不断地通过韧皮部转运至植物库组织[2];基于导向农药的理念,Yang等[3]合成了葡萄糖基氟虫腈耦合物,并发现其有可能是通过蓖麻糖转运蛋白介导进入蓖麻韧皮部[4]。充分利用植物对内外源化合物的吸收与转运机制对于提高农作物产量,探索病虫害防治新策略具有非常重要的意义。
利用植物对内外源糖物质的吸收转运规律,对关键基因进行修饰从而提高作物产量;合成能够被植物转运载体介导的新型农药分子,实现农药分子在植物体内的主动运输和定向积累,对于病虫害防治能起到事半功倍的效果。研究发现草甘膦、百草枯、葡萄糖基氟虫腈、部分农药分子与氨基酸的耦合物L-lysine-2,4-D等虽然能被磷酸转运蛋白、聚胺转运蛋白、糖转运蛋白、氨基酸转运蛋白所介导[4-7],但却无法明确具体的转运子,无法明确化合物与转运载体之间的构效关系,大大降低了这类农药的开发效率。
蓖麻Ricinuscommunis是用来研究内外源化合物韧皮部输导性的模式植物,随着其基因组数据库的释放,蓖麻中的各类转运蛋白家族将会被鉴定,利用RT-qPCR技术分析并克隆能被载体介导的化合物处理前后差异表达的基因,是鉴定参与化合物韧皮部装载的相关载体蛋白的重要手段。RT-qPCR自1995年由美国PE公司成功开发以来,便以其操作简便、检测灵敏度高、重复性好、检测速度快等优点,被广泛应用于生命科学、分子诊断学、农学与医学、食品微生物学等领域[8]。在基因表达分析过程中,RNA的质量和产量、不同组织或细胞间反转录效率的差异等因素均有可能影响到目的基因表达分析结果的准确性[9]。为了获取准确的结果,除了提高RNA质量,选择合适的内参基因来校正和标准化mRNA是最首要的方式[10]。理想的内参基因的表达不受试验因素的影响而恒定表达在各种类型的组织或细胞中[11]。常用的内参基因是构成细胞器骨架的基本组分或是参与生物体的基本生化代谢过程的基因[12-14]。同一个内参基因在不同的试验条件下表达的稳定性是不一样的,如玉米根萤叶甲DiabroticavirgiferavirgiferaLeconte在不同的试验条件下β-Actin在其整个生命周期内表达稳定,RPS9则在dsRNA处理后的玉米根萤叶甲中表达稳定性最好,而BT处理后,EF-1在不同的组织中表达最为稳定[15];在荔枝果实不同发育阶段用外源生长调节剂处理后β-Actin比GAPDH、18SrRNA、UBQ、eEF和25S rRNA的稳定性更好[16-17],由此可见筛选出化合物处理前后都能稳定表达的内参基因是RT-qPCR技术分析并克隆能被载体介导的化合物处理后差异表达的基因的基础。
本研究以蓖麻幼苗子叶为材料,选取常用的内参基因Actin、ARC、SamDC、ef1a、TUA6作为候选基因,用RT-qPCR方法检测上述常用内参基因在不同浓度及不同时间的GTF及其溶剂DMSO处理下的表达量,分别采用常用的内参基因稳定性评价软件geNorm[10]、NormFinder[18]、BestKeeper[19]、Delta CT[20]进行数据分析,最后用RefFinder[21]在线工具对上述4种软件的计算结果进行综合评价,为研究GTF相关的转运载体基因的表达分析研究筛选出最为稳定的内参基因。
1材料与方法
1.1材料
蓖麻种子由淄博市农业科学院提供。成熟蓖麻种子在湿脱脂棉上于27 ℃下催芽48 h后,选出发芽的种子,播入蛭石中,置于人工气候箱内进行培养。培养条件为:温度为27 ℃,相对湿度为75%~85%,光照度260~350 lx,每天光照14 h,连续培养6 d的蓖麻幼苗作为供试材料。
1.2方法
1.2.1蓖麻幼苗的药物处理将蓖麻幼苗小心剥去胚乳,用手轻轻擦洗子叶表面,切勿折叠或弄破子叶。用子叶培养缓冲液(20 mmol·L-1MES, 0.25 mmol·L-1MgCl2, 0.5 mmol·L-1CaCl2, 2 mmol·L-1EDTA, pH 5.0)配制相应浓度的GTF或者DMSO后,将蓖麻子叶浸泡于相应含药缓冲液中,同时将蓖麻根部置于根部培养缓冲液(0.5 mmol·L-1CaCl2, 1 mmol·L-1MES)中。浓度梯度试验设定7个GTF和DMSO浓度梯度,分别为10、25、50、100、200、500、1 000 μmol·L-1,于人工气候箱内培养6 h后收集样品;时间梯度试验设置为以100 μmol·L-1的GTF或DMSO处理蓖麻幼苗,分别于0、3、6、9、12 h后收集子叶。每个处理设3个重复,以不含GTF及DMSO的缓冲液为对照组,完成相应的处理后在蓖麻幼苗胚柄处剪断,将子叶装入锡纸袋,用液氮速冻后置于-80℃冰箱保存备用。
1.2.2蓖麻子叶总RNA的提取及第一链cDNA的合成使用Omega植物RNA提取试剂盒提取经过处理的蓖麻子叶总RNA,并用核酸蛋白定量仪(ND1000,Eppendorf)检测RNA样品的D260 nm/D280 nm和D260 nm/D230 nm值,确定RNA的浓度和纯度,同时用含甲醛(w=12.5%)的琼脂糖凝胶电泳检测RNA的完整性。随后RNA用RQ1 RNase-free DNase处理,并用反转录试剂盒(Bio-Rad)进行第一链cDNA的反转录。转录完成后以合成的cDNA为模板克隆内参基因片段以进行RT-qPCR试验。
1.2.3候选内参基因的处理根据水稻内参基因的氨基酸序列[20], 在NCBI上获得对应的蓖麻内参基因登录号和基因序列,并通过Primer Premier 5.0软件设计特异性引物,由北京六合华大基因科技有限公司合成。选取的内参基因引物序列见表1。

表1 RT-qPCR候选内参基因的引物序列
以含候选基因片段的质粒为模板构建其对应的标准曲线。利用cDNA为模板获得候选内参基因片段。通过20 g·L-1凝胶电泳检测并用 DNA凝胶纯化试剂盒回收目的片段,将回收产物连接pMD20-T载体上,蓝白斑筛选阳性克隆并测序。经验证正确的阳性克隆扩大培养后,用质粒小提试剂盒(Omega)提取质粒。取500 ng质粒用Dilution Buffer(Takara)稀释, 每个梯度稀释10倍,依次稀释5~8个梯度,则浓度分别为50、 5、 1/2、1/20、 1/200和1/2 000、1/20 000、1/200 000 ng·μL-1。以稀释后的质粒为模板进行RT-qPCR反应,PCR仪自带的计算软件CFX Manager TM Software自动计算获得熔解曲线,并根据所得Ct值绘制标准曲线,得到斜率(k)和线性相关系数(R)。根据公式E= 5-1/k-1,计算得出扩增效率(E)。候选内参基因引物特异性则通过RT-qPCR的熔解曲线的峰形图来评价。
1.2.4RT-qPCR 分析RT-qPCR反应用Bio-Rad CFX-96 Real-Time PCR System完成。反应体系按照IQTMSYBR® Green Supermix说明书进行配制,反应总体积为20 μL;反应条件如下:95℃ 30 s,95 ℃ 5 s,62 ℃ 40 s,40个循环后进行65~95 ℃熔解曲线分析。每个样品设定3个重复。
1.3数据分析
根据Bio-Rad CFX-96 Real-Time PCR System获得的各样品的Ct值,采用geNorm,NormFinder,BestKeeper,Delta CT 4种算法对各候选内参基因的稳定性进行比较,同时参考RefFinder的计算结果,筛选出在GTF及DMSO处理下稳定性最好的内参基因。
2结果与分析
2.1候选内参基因引物特异性及扩增效率
以反转获得的cDNA为模板扩增候选内参基因片段,电泳检测结果显示所得目的条带大小与理论值一致, 且没有发现其他条带,扩增特异性好(图1)。
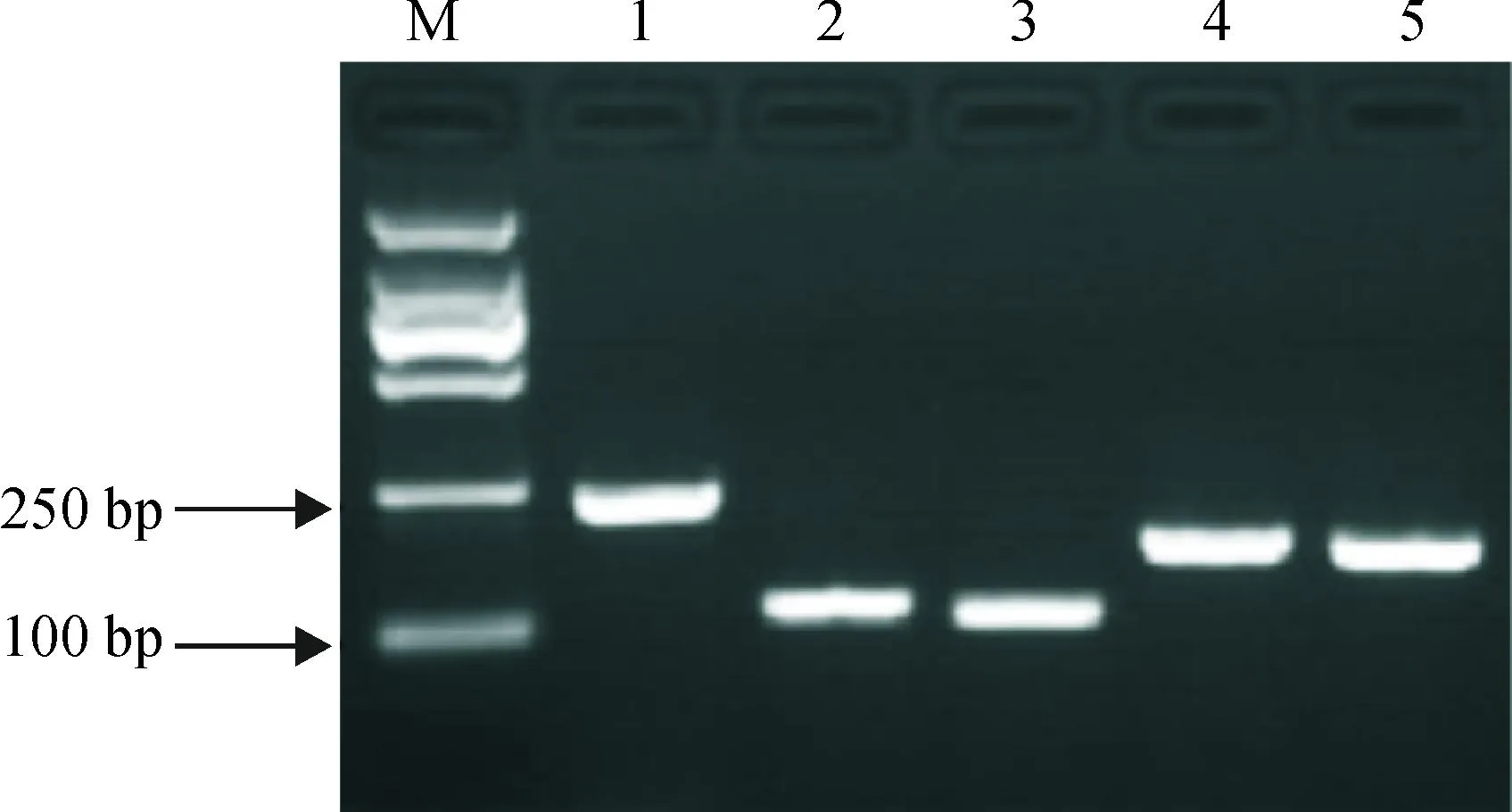
M: DNA Marker DL 2 000;1:Actin;2:ARC;3:ef1a; 4:SamDC; 5:TUA6。
图1候选内参基因PCR扩增产物检测
Fig.1Detection of PCR amplification products of five candidate reference genes
RT-PCR的结果表明,各候选内参基因的相关系数(R)≥ 0.996, 引物的扩增效率为95.0%~107.7%,均符合RT-qPCR对扩增效率的要求(表2);进一步分析熔解曲线,发现各候选内参基因都只产生单一的熔解峰。上述结果说明所设计的引物特异性良好,RT-qPCR反应的专一性高,准确性好。

表2 5个候选内参基因的标准曲线参数
2.2候选内参基因的表达谱分析
在RT-qPCR试验中,Ct值与基因的表达量成反比。本研究结果表明同一个扩增反应的3个重复的标准差很小,Ct值非常接近,说明扩增反应重复性好,能准确反映基因的转录水平;5个候选内参基因的Ct值均位于17~24,同时都处于相应候选内参基因标准曲线的Ct跨距之内;同一个基因,浓度处理对其表达量影响较大;其中对ef1a的表达量最大(17~21),SamDC及ARC的表达量相对较小(18~24);在所有的样品中,ef1a的Ct值变化范围最小,△Ct为2.77,其次是Actin,SamDC的Ct值变化最大,△Ct为4.23。同一个基因在不同的样品中表达量的差异说明有必要对这些常用内参基因进行筛选,以选择合适的内参基因或组合来进行接下来的定量试验。
2.3不同处理候选内参基因稳定性分析
2.3.14种软件分析内参基因稳定性将RT-qPCR试验所获得的Ct值处理后导入geNorm(V3.5)计算出各候选内参基因的稳定值(M值),M值越大,表示稳定性越差。计算结果(图2a)表明,5个候选内参基因均相对较稳定(M<1.5),Actin和ef1a的M值最低,稳定性最好;ARC的M值最高,稳定性最差;这5个候选内参基因的稳定性从高到低依次为:Actin=ef1a>SamDC>ARC>TUA6,即在geNorm的算法中,Actin与ef1a的稳定性最好。
按NormFinder软件要求,将处理好的数据导入软件进行计算,得出各候选内参基因的稳定值M,M值越大,说明基因的稳定性越差;反之则说明基因的表达稳定性越好。经计算获得5个候选内参基因的M值,按稳定性由高到低依次为:SamDC>ARC>Actin>ef1a>TUA6(图2b),即在NormFinder算法中,SamDC的稳定性最好。
将RT-qPCR反应获得的Ct值导入BestKeeper软件,根据各候选基因标准偏差值(SD)来确定候选基因的相对稳定性,SD值越小,稳定性越好。在本次试验中,所有基因的SD值均小于1,其SD大小顺序依次为:Actin>ef1a>SamDC>ARC>TUA6(图2c),即在BestKeeper软件的算法中Actin的稳定性最好。
利用Delta CT软件计算5个候选内参基因的Ct值平均标准偏差(SD),SD越小,表明基因稳定性越高。将计算结果按稳定性由高到低进行排序,依次为:SamDC>Actin>ARC>ef1a>TUA6(图2d),即在Delta CT软件的算法中SamDC的稳定性最好。
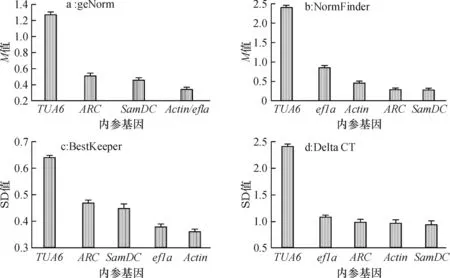
图2 不同软件分析5种候选内参基因的稳定性
Fig.2Expression stability of five candidate reference genes calculated by geNorm,NormFinder software,BestKeeper software and Delta CT software
2.3.2RefFinder综合分析由以上分析可知,geNorm,NormFinder,Delta CT和BestKeeper 4种软件由于不同的算法分析出来的结果也存在差异。此时可以利用RefFinder在线分析工具来进行综合分析从而选出表达相对最为稳定的内参基因。RefFinder可以根据各种算法所得的候选内参基因稳定表型排名的几何平均数作为综合排名,从而筛选出最稳定的内参基因。基因综合稳定参数与基因表达稳定性成反比。根据计算结果可知GTF及DMSO处理下,候选内参基因表达稳定性由高到低依次为:Actin>SamDC>ef1a>ARC>TUA6(图4)。

图3 RefFinder综合分析比较候选内参基因稳定性
Fig.3Expression stability of five candidate reference genes analyzed by RefFinder software
2.3.3DMSO处理下内参基因的稳定性分析DMSO在蓖麻体系中的韧皮部输导性研究中常被用作溶剂,有必要对其处理的样品单独进行内参基因稳定性分析,以作参考。RefFinder分析结果(图4)表明,在10~1 000 μmol·L-1的浓度梯度及0~12 h的时间梯度的DMSO处理下ef1a是最稳定的内参基因。
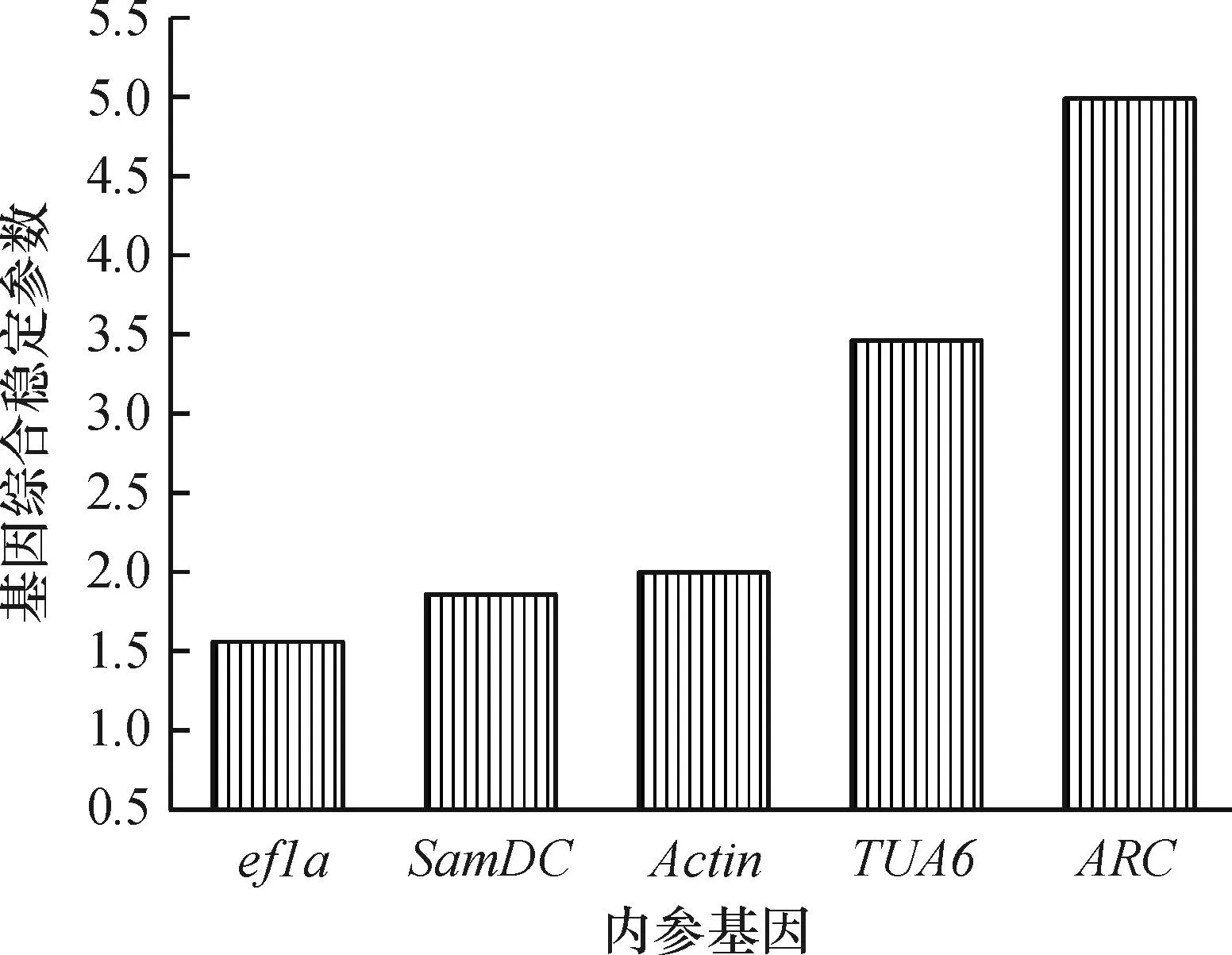
图4DMSO在不同处理浓度及不同处理时间下的内参基因稳定性分析
Fig.4Expression stability of five candidate reference genes analyzed by RefFinder in all tested samples treated with different concentrations of DMSO and different times
3结论
本研究比较了Actin、ARC、SamDC、ef1a、TUA6 基因在不同浓度及不同时间的GTF及其溶剂DMSO处理前后的表达量,并分别采用geNorm、NormFinder、BestKeeper、Delta CT 4个软件及RefFinder在线分析工具对各候选内参基因的稳定性进行计算分析,以期获得相对稳定的用于校正和标准化RT-qPCR试验的一个或是一组内参基因。geNorm软件的计算结果中,Actin与ef1a为稳定性最好的组合;在NormFinder与Delta CT的算法中,SamDC是稳定性最好、最适合的内参基因;而通过BestKeeper获得的结果表明,Actin是最稳定的内参基因。由于各软件算法的不同导致计算结果不尽相同,难以取舍。而RefFinder在线工具可以根据以上4种算法所得的候选内参基因稳定性的几何平均数综合排名,筛选出表达最为稳定的基因,结果表明GTF、DMSO共同处理综合分析时Actin的稳定性最好。单独对DMSO处理后的内参基因稳定性分析时发现ef1a是最稳定的内参基因。本文分析了5个常用内参基因在GTF和 DMSO共同处理及DMSO单独处理下内参基因的稳定性,可为相关研究提供参考。
参考文献:
[1]徐汉虹,张志祥,程东美,等. 导向农药[J].世界农药,2004(5): 3-9.
[2]DOIDY J, GRACE E, KUHN C, et al. Sugar transporters in plants and in their interactions with fungi[J]. Trends Plant Sci, 2012, 17(7):413-422.
[3]YANG W, WU H, XU H, et al. Synthesis of glucose-fipronil conjugate and its phloem mobility[J]. J Agr Food Chem, 2011, 59(23):12534-12542.
[4]WU H, YANG W, ZHANG Z, et al. Uptake and phloem transport of glucose-fipronil conjugate inRicinuscommunisinvolve a carrier-mediated mechanism[J]. J Agr Food Chem, 2012, 60(24): 6088-6094.
[5]DENIS M, DELROT S. Carrier-mediated uptake of glyphosate in broad bean (Viciafaba) via a phosphate transporter[J]. Physiol Plantarum, 1993, 87(4):569-575.
[6]FUJITA M, SHINOZAKI K. Identification of polyamine transporters in plants: Paraquat transport provides crucial clues[J]. Plant Cell Physiol, 2014, 55(5): 855-861.
[7]CHOLLET J, DELETAGE C, FAUCHER M, et al. Synthesis and structure-activity relationships of some pesticides with an alpha-amino acid function[J]. Biochim Biophys Acta, 1997, 1336(2): 331-341.
[8]POSTOLLEC F, FALENTIN H, PAVAN S, et al. Recent advances in quantitative PCR (qPCR) applications in food microbiol[J]. Food Microbiol, 2011, 28(5): 848-861.
[9]袁伟,万红建,杨悦俭. 植物实时荧光定量PCR内参基因的特点及选择[J]. 植物学报, 2012(4): 427-436.
[10]VANDESOMPELE J, DE PRETER K, PATTYN F, et al. Accurate normalization of real-time quantitative RT-PCR data by geometric averaging of multiple internal control genes[J]. Genome Biol, 2002, 3(7): H34.
[11]TONG Z, GAO Z, WANG F, et al. Selection of reliable reference genes for gene expression studies in peach using real-time PCR[J]. BMC Mol Biol, 2009, 10(1): 71.
[12]HUGGETT J, DHEDA K, BUSTIN S, et al. Real-time RT-PCR normalisation, strategies and considerations[J]. Genes Immun, 2005, 6(4): 279-284.
[13]HONG S, SEO P J, YANG M, et al. Exploring valid reference genes for gene expression studies in Brachypodium distachyon by real-time PCR[J]. BMC Plant Biol, 2008, 8(1):112.
[14]SUZUKI T, HIGGINS P J, CRAWFORD D R. Control selection for RNA quantitation[J]. Biotechniques, 2000, 29(2): 332-337.
[15]BARROS R T, KHAJURIA C, WANG H, et al. Validation of reference housekeeping genes for gene expression studies in western corn rootworm (Diabroticavirgiferavirgifera)[J/OL]. PLoS One, 2014, 9(10): e109825.http://www.plosoneorg.sci-hub.org/article/fetchobject.action?uri=info:doi/10.1371/journal.pone.0109825&rep resentation=PDF.
[16]魏永赞,赖彪,胡福初,等. 用于荔枝qPCR分析的内参基因克隆及稳定性分析[J]. 华南农业大学学报, 2012,33(3): 301-306.
[17]GUTIERREZ L, MAURIAT M, GUENIN S, et al. The lack of a systematic validation of reference genes: A serious pitfall undervalued in reverse transcription-polymerase chain reaction (RT-PCR) analysis in plants[J]. Plant Biotechnol J, 2008, 6(6): 609-618.
[18]ANDERSEN C L, JENSEN J L, ORNTOFT T F. Normalization of real-time quantitative reverse transcription-PCR data: A model-based variance estimation approach to identify genes suited for normalization, applied to bladder and colon cancer data sets[J]. Cancer Res, 2004, 64(15): 5245-5250.
[19]PFAFFL M W, TICHOPAD A, PRGOMET C, et al. Determination of stable housekeeping genes, differentially regulated target genes and sample integrity: BestKeeper:Excel-based tool using pair-wise correlations[J]. Biotechnol Lett, 2004, 26(6): 509-515.
[20]SILVER N, BEST S, JIANG J, et al. Selection of housekeeping genes for gene expression studies in human reticulocytes using real-time PCR[J]. BMC Mol Biol, 2006, 7(1):33.
[21]XIE F, SUN G, STILLER J W, et al. Genome-wide functional analysis of the cotton transcriptome by creating an integrated EST database[J/OL]. PLoS One, 2011, 6(11): e26980.http://www.plosone.org.sci-hub.org/article/fetchobject.action?uri=info:doi/10.1371/journal.pone.0026980&representation=PDF.
[22]JAIN M, NIJHAWAN A, TYAGI A K, et al. Validation of housekeeping genes as internal control for studying gene expression in rice by quantitative real-time PCR[J]. Biochem Biophys Res Commun, 2006, 345(2): 646-651.
【责任编辑霍欢】
Stability analysis of internal reference gene of Ricinus communis
treated by glucose-fipronil
MAO Genlin, XIE Yun, ZHAO Junlong, CHEN Yan, XU Hanhong, LIN Fei
(College of Agriculture, South China Agricultural University/Key Laboratory of Natural Pesticide and
Chemical Biology, Ministry of Education, Guangzhou 510642, China)
Abstract:【Objective】 To screen reliable internal reference genes of castor,Ricinuscommunis,treated by glucose-fipronil (GTF) and the solvent dimethyl sulfoxide (DMSO),and to provide a basis for studying the uptake mechanism of GTF. 【Method】Actin,ARC,ef1a,SamDCandTUA6 genes were selected as candidate reference genes. Specific primers for each gene were designed and real-time quantitative PCR was conducted. Five softwares,including geNorm, NormFinder, BestKeeper, Delta CT and RefFinder,were employed to analyze the gene expression stabilities in cotyledon of castor seedlings treated by GTF and DMSO with different concentration and time. 【Result】 The stabilities were variant according to different softwares. The rank orders of stability were geNorm:Actin=ef1a>SamDC>ARC>TUA6;NormFinder:SamDC>ARC>Actin>ef1a>TUA6; BestKeeper:Actin>ef1a>SamDC>ARC>TUA6;Delta CT:Actin>ef1a>SamDC>ARC>TUA6;RefFinder:Actin>SamDC>ef1a>ARC>TUA6. With a single analysis of DMSO treatment by RefFinder showed thatef1a>SamDC>Actin>TUA6>ARC.【Conclusion】Actinis the stablest reference gene under GTF and DMSO treatments, whileef1ais the stablest one under DMSO treatment.
Key words:Ricinuscommunis; GTF; DMSO; RT-qPCR; internal reference gene; stability
中图分类号:S511;S502
文献标志码:A
文章编号:1001-411X(2016)01-0052-06
基金项目:国家自然科学基金(31171889);高等学校博士学科点专项科研基金(20114404110020,20134404130003)
作者简介:毛根林(1989—),男,硕士研究生,E-mail:maogenlin@163.com;通信作者:徐汉虹(1961—),男,教授,博士,E-mail:hhxu@scau.edu.cn;林菲(1977—),女,副研究员,博士,E-mail:resistanc@scau.edu.cn
收稿日期:2015-04-02优先出版时间:2015-12-07
优先出版网址:http://www.cnki.net/kcms/detail/44.1110.s.20151207.1132.020.html
毛根林, 解云, 赵俊龙,等.葡萄糖基氟虫腈处理下蓖麻内参基因的稳定性分析[J].华南农业大学学报,2016,37(1):52-57.

