BraSDG2 RNA干扰载体的构建和拟南芥的遗传转化
周雅智 姜玲 黄勇

摘要:多倍化是植物进化过程中的自然现象,也是植物进化的重要源泉。在前期的研究中,通过生物信息学分析发现二倍体的白菜型油菜中存在两个拟南芥SDG2的同源基因BraSDG2a(Bra037086)和BraSDG2b(Bra039562)。经序列比对,选取258 bp的保守序列构建RNA干扰载体pFGC5941-BraSDG2-RNAi。通过浸花法转化野生型拟南芥(Col-0),经抗性筛选及PCR检测,获得1株表型稳定的转基因植株。通过对T4代转基因拟南芥(纯合体)的表型观察发现,BraSDG2 RNA干扰转基因植株具有拟南芥sdg2突变体相似的生物学表型,植株弱小、种子稀少。本试验为进一步研究BraSDG2的生物学功能奠定基础。
关键词:白菜型油菜;BraSDG2;RNA干扰;拟南芥;遗传转化
中图分类号:S634.301文献标识号:A文章编号:1001-4942(2016)10-0007-05
据估计,50%~70%的被子植物在其进化过程中至少经历过1次多倍化过程[1],基因表达的变化在多倍体化过程中起着重要作用[2]。组蛋白甲基化修饰作为一种重要的表观遗传修饰,在参与重塑染色质结构与基因表达方面起到重要作用,其主要包括赖氨酸和精氨酸的甲基化[3]。组蛋白赖氨酸(K)甲基化修饰包括组蛋白H3的K4、K9、K27、K36和组蛋白H4的K20的单、双和三甲基化的修饰状态。组蛋白H3K4及H3K36的甲基化修饰被认为参与基因表达的激活,而H3K9、H3K27和H4K20的甲基化修饰则被认为与基因沉默及异染色质结构的维持相关[4]。
拟南芥SDG2编码一个包含2 335个氨基酸的蛋白质,其含有SET、Post-SET、GYF保守结构域,具有特异的H3K4me3甲基转移酶活性。SDG2突变后,拟南芥的育性完全丧失,50%的四分体包含小孢子的数量少于四个,且BT3、SPL、MS1、EDA31、MEE65等11个与雄配子发育相关基因的表达发生明显下调[5]。因此,SDG2介导的H3K4me3甲基转移酶活性影响着雄配子体的发育及植物育性。
RNA干扰(RNA interference,RNAi)是由双链RNA(double-stranded RNA,dsRNA)所介导的基因沉默现象,它在转录、转录后以及翻译水平上干扰基因的表达。RNAi技术是一种研究功能未知基因的有效方法。我们前期的研究表明,在二倍体的白菜型油菜中,BraSDG2具有2个拷贝,并具有不同的组织表达模式,这些结果暗示在基因多倍体化过程中组蛋白甲基转移酶基因的命运可能发生了明显分化[6]。本研究拟通过构建BraSDG2的RNA干扰载体,经浸花法转化哥伦比亚野生型拟南芥(Col-0),筛选BraSDG2基因沉默的稳定遗传的转基因拟南芥植株并观察其表型特征,为进一步研究白菜型油菜BraSDG2的生物学功能奠定基础。
1材料与方法
1.1试验材料
白菜型油菜(Brassica rapa)B2;大肠杆菌(Escherichia coli)DH5α;根癌农杆菌GV3101;载体pMD19-T vector和pFGC5941(含PPT抗性)。以上试验材料均来自湖南农业大学植物发育与表观遗传调控实验室。
1.2试验方法
1.2.1白菜型油菜BraSDG2-RNAi片段的克隆从Tair与白菜基因组数据库BRAD收集拟南芥SDG2、白菜BraSDG2a和BraSDG2b CDS序列,经比对分析后选取一段258 bp的保守序列,命名为BraSDG2-RNAi。设计引物序列BraSDG2-RNAi F:5′-CCATGGTCTAGATTGGTGGGCTGCCAGATTG-3′(下划线分别表示NcoⅠ、XbaⅠ酶切位点);BraSDG2-RNAi R:5′ -GGCGCGCCGGATCCGATCCCCAAACACACGTCTCATAAC-3′(下划线分别表示Asc Ⅰ、BamHⅠ酶切位点)。
高保真酶PCR扩增后获得RNA干扰目的片段,切胶回收后连接到pMD19-T载体上,命名为pMD19-T-BraSDG2-RNAi,将连接好的载体通过热激法转化至感受态大肠杆菌DH5α,挑选PCR检测为阳性的单菌落提取质粒测序。
1.2.2RNAi载体的构建使用BamH Ⅰ和Xba Ⅰ双酶切载体pMD19-T-BraSDG2-RNAi和pFGC5941。将反向BraSDG2-RNAi片段导入终止子与CHSA intron之间,获得中间载体p-BraSDG2-RNAi;再通过AscⅠ和NcoⅠ对载体pMD19-T-BraSDG2-RNAi和p-BraSDG2-RNAi进行双酶切,将正向BraSDG2-RNAi片段导入35S启动子与CHSA intron之间,最终获得RNA干扰载体pFGC5941-BraSDG2-RNAi。对获得的RNA干扰载体进行酶切验证。
1.2.3拟南芥的遗传转化及筛选将pFGC5941-BraSDG2-RNAi导入到工程菌GV3101后,挑选阳性菌落用于拟南芥转化。通过浸花法将该重组质粒转化至野生型拟南芥(Col-0)中,采用浓度为100 mg/mL的PPT筛选T0代拟南芥转化子。设计检测引物35S825 F:5′-ATCCCACTATCCTTCGCAAGACCCTT-3′,Intron825 R:5′-TGAC-TCCATCTTATTCCCTCCGTTTC-3′,对获得的抗性苗进行PCR检测筛选转基因植株。
1.2.4转化子的表型观察将野生型、T3代和T4代(纯合体)转基因拟南芥种子经4℃春化2~3 d后,置于22℃长日照(16 h/8 h)条件下培养37 d,对其进行表型观察。
2结果与分析
2.1BraSDG2-RNAi片段的克隆
经序列比对,选取一段长度为258 bp的保守序列作为RNAi片段,命名为BraSDG2-RNAi,并成功克隆到该基因片段,见图1。将克隆的目的片段与pMD19-T载体连接并导入感受态工程菌DH5α中,选取编号为13和16的两个阳性单菌落(图2)送至公司测序。结果如图3所示,目的
片段与预期片段比对,序列基本一致(5个位点的差异是由于本实验室所提供的白菜型油菜品种与白菜基因组数据库品种有所差异导致)。
2.2BraSDG2干扰载体的构建
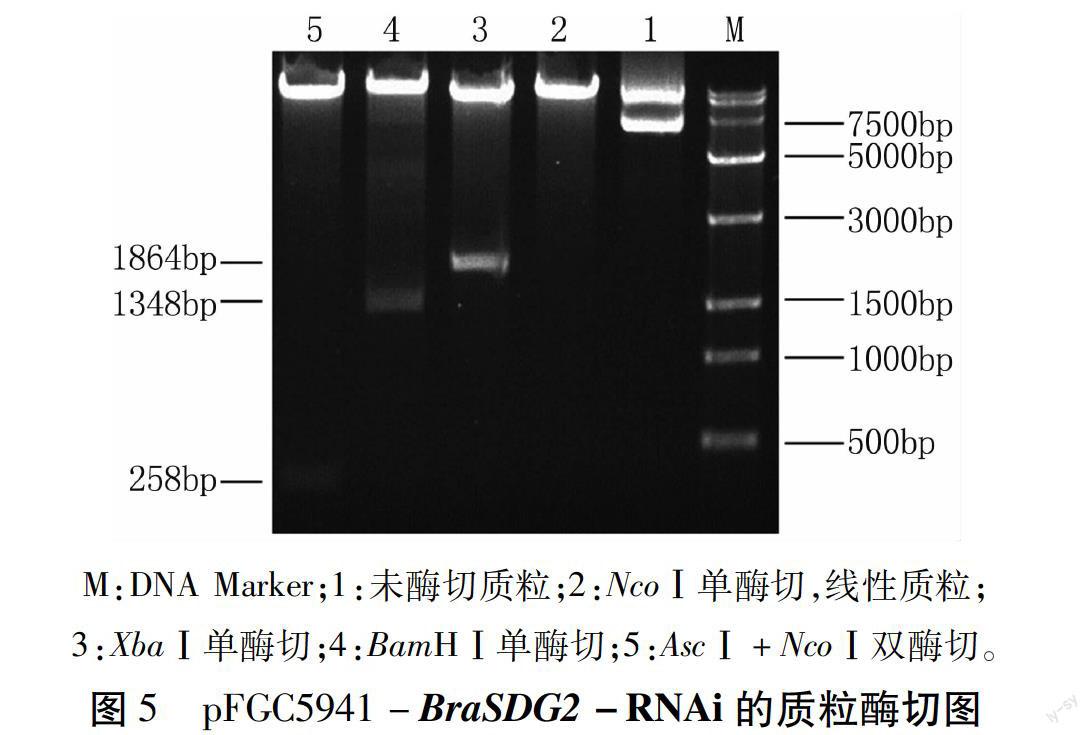
将BraSDG2-RNAi片段正反向插至目的载体pFGC5941上,结构如图4所示。通过酶切验证,确认BraSDG2 RNA干扰载体pFGC5941-BraSDG2-RNAi构建成功,酶切后的片段大小与预期一致,如图5所示。
2.3拟南芥的遗传转化
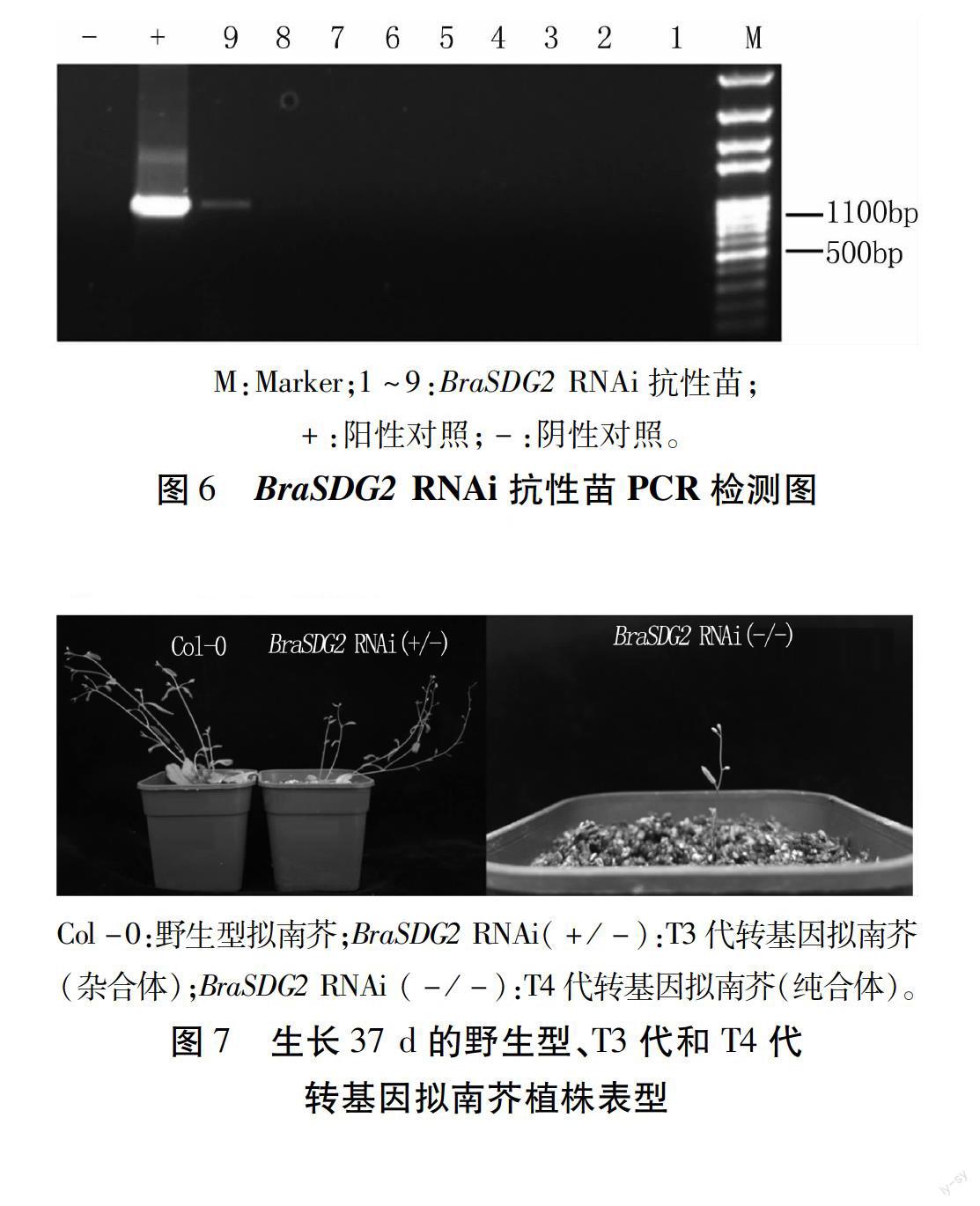
经浸花法将构建的重组质粒转化野生型拟南芥(Col-0),通过100 mg/mL PPT筛选得到抗性苗并进行PCR检测,扩增结果如图6所示。结果显示,获得了1株转基因植株。
2.4BraSDG2 RNAi转基因植株表型观察
在相同长日照条件下(16 h/8 h),对生长37 d的T3、T4代 BraSDG2 RNAi转基因拟南芥植株与Col-0进行观察。相比Col-0,T4代BraSDG2 RNAi转基因植株(纯合体)形态特征表现为植株矮小且出现败育现象(图7),其表型与拟南芥sdg2缺失突变体相近。
3讨论与结论
组蛋白赖氨酸甲基化修饰是一种重要的表观遗传学标记,其作用机理是通过特异的组蛋白赖氨酸甲基转移酶催化改变染色质结构,间接影响基因表达,在植物生长发育中起到重要的生物学作用。拟南芥核组蛋白的赖氨酸残基被甲基化常见的有4类, histoneH3lysine4(H3K4)、H3K9、H3K27、H3K36。现有研究的基因组规模分析的结果显示不同位点不同程度的甲基化会导致转录异常,从而影响基因的表达[7-15]。
研究表明,拟南芥的大多数基因都存在组蛋白甲基化现象,其中发挥重要作用的就是组蛋白赖氨酸甲基转移酶(histone lysine methyltransferase,HLMT),这是一类包含130~150个氨基酸保守 SET结构域的蛋白[16],该保守结构域也是其发挥催化作用的活动中心。SDG蛋白家族都含有SET结构域,拟南芥SDG2功能表现为特异性地催化组蛋白,对H3K4位点造成双甲基化与三甲基化,从而调控相关基因表达[17]。
已有试验表明,对拟南芥SDG2进行RNAi,会导致植株弱小、叶片变小、根长变短、莲座叶数目变少。本研究通过序列比对获得白菜型油菜中与拟南芥SDG2同源的基因片段BraSDG2-RNAi,并构建BraSDG2 RNA干扰载体转化拟南芥,结果表明构建的载体可以有效干扰拟南芥SDG2基因的表达,BraSDG2 RNAi拟南芥转基因纯合植株表型与sdg2拟南芥突变体相似,都表现为植株矮小且育性极低,从而佐证了组蛋白甲基化转移酶SDG2同系物在植物中是高度保守的。有研究显示,拟南芥SDG2的缺失导致H3K4me3甲基转移酶在体内剧烈下降,突变体具有强烈的多向性的表型以及许多基因的错误调控,植株矮小和败育就是其中重要的表型[18]。本试验证明了白菜型油菜中SDG2的同系物BraSDG2可能对H3K4me3甲基转移酶具有相似的功能,该结果为进一步研究组蛋白甲基转移酶SDG家族的作用机制与白菜中BraSDG2的生物学功能奠定基础。
参考文献:
[1]Masterson J. Stomatal size in fossil plants: evidence for polyploidy in majority of angiosperms[J]. Science, 1994,264(5157):421-423.
[2]Wendel J F. Genome evolution in polyploids[J]. Plant Mol. Biol., 2000,42(1):225-249.
[3]Martin C, Zhang Y. The diverse functions of histone lysine methylation[J]. Nat. Rev.Mol.Cell Biol., 2005,6(11):838-849.
[4]Liu C, Lu F, Cui X, et al. Histone methylation in higher plants[J]. Annu. Rev. Plant Biol., 2010,61:395-420.
[5]Berr A, McCallum E J, Ménard R, et al. Arabidopsis SET DOMAIN GROUP2 is required for H3K4 trimethylation and is crucial for both sporrophyte and gametophyte development[J]. Plant Cell, 2010,22(10):3232-3248.
[6]Huang Y, Liu C, Shen W H, et al. Phylogenetic analysis and classification of the Brassica rapa SET-domain protein family[J]. BMC Plant Biol., 2011,11:175.
[7]Lippman Z, Gendrel A V, Black M, et al. Role of transposable elements in heterochromatin and epigenetic control[J]. Nature, 2004,430 (6998):471-476.
[8]Turck F, Roudier F, Farrona S, et al. Arabidopsis TFL2/LHP1 specifically associates with genes marked by trimethylation of histone H3 lysine 27[J]. PLoS Genet.,2007,3(6):e86.
[9]Zhang X, Clarenz O, Cokus S, et al. Whole-genome analysis of histone H3 lysine 27 trimethylation in Arabidopsis[J]. PLoS Biol.,2007,5(5):e129.
[10]Bernatavichute Y V, Zhang X, Cokus S, et al. Genome-wide association of histone H3 lysine nine methylation with CHG DNA methylation in Arabidopsis thaliana[J]. PLoS ONE, 2008,3(9):e3156.
[11]Oh S, Park S, van Nocker S. Genic and global functions for Paf1C in chromatin modification and gene expression in Arabidopsis[J]. PLoS Genet., 2008,4(8):e1000077.
[12]Zhang X. The epigenetic landscape of plants[J]. Science, 2008,320(5875):489-492.
[13]Zhang X Y, Bernatavichute Y V, Cokus S, et al. Genome-wide analysis of mono-, di- and trimethylation of histone H3 lysine 4 in Arabidopsis thaliana[J]. Genome Biol., 2009, 10:R62.
[14]Charron J B, He H, Elling A A, et al. Dynamic landscapes of four histone modifications during deetiolation in Arabidopsis[J]. Plant Cell, 2009, 21(12):3732-3748.
[15]Jacob Y, Stroud H, LeBlanc C, et al. Regulation of heterochromatic DNA replication by histone H3 lysine 27 methyltransferases[J]. Nature, 2010, 466:987-991.
[16]宋江华,曹家树. 植物SET蛋白[J]. 细胞生物学杂志, 2007, 29(3):384-388.
[17]Xu L, Zhao Z, Dong A, et al. Di- and tri- but not monomethylation on histone H3 lysine 36 marks active transcription of genes involved in flowering time regulation and other processes in Arabidopsis thaliana[J].Molecular and Cell Biology, 2008, 28(4):1348-1360.
[18]Guo L, Yu Y, Law J A, et al. SET DOMAIN GROUP2 is the major histone H3 lysine 4 trimethyltransferase in Arabidopsis[J]. PNAS, 2010, 107(43):18557-18562.

