miR-210在马氏珠母贝外套膜矿化机制中的结构和功能预测
潘晓艳,郑 哲,田荣荣,焦 钰,杜晓东
(广东海洋大学水产学院,广东 湛江 524025)
miR-210在马氏珠母贝外套膜矿化机制中的结构和功能预测
潘晓艳,郑 哲,田荣荣,焦 钰,杜晓东
(广东海洋大学水产学院,广东 湛江 524025)
为进一步探究Pm-miR-210在马氏珠母贝贝壳形成的作用,利用miR-RACE技术验证Pm-miR-210的成熟体序列并进行序列分析,通过生物信息学分析来获得Pm-miR-210的前体序列,并进行组织定量和靶位点预测。结果表明,Pm-miR-210的miR-RACE结果与高通量测序结果一致,利用转录组数据库与Pm-miR-210成熟体序列比对,获得的前体序列长度为80 bp并具有典型的颈环结构。多序列比对结果显示,Pm-miR-210成熟体序列的种子区域在不同物种间具有极高的保守性。荧光定量结果得出,Pm-miR-210在外套膜边缘膜和中央膜中均有表达,但表达量差异不具统计学意义(P > 0.05)。靶向预测分析结果显示,钙调蛋白(calmodulin)、钙调磷酸酶-A亚基(calcineurin A subunit)、N19以及Shematrin-3为Pm-miR-210的候选靶基因。
马氏珠母贝;Pm-miR-210;靶向预测;贝壳形成
microRNA(miRNA)是一类内生性、在序列结构上具有很大保守性、长度为21~25 nt的非编码单链小分子RNA[1-2]。miRNA 主要是通过其种子序列(5' 端第2—8 nt)与靶mRNA 的3'UTR进行完全或不完全互补配对,从而抑制mRNA的翻译或直接使其降解或沉默,行使转录后调控功能[3-6]。如果miRNA与其靶mRNA完全互补,作用方式和功能与siRNA非常类似,则最后切割靶mRNA[7]。目前,发现的数以千计的 miRNA通过对靶基因的调控[8-10],参与了包括生长发育、细胞分化、细胞调亡等几乎所有己知细胞生理过程[11-14],并与生物体的感染、肿瘤、代谢性疾病等多种病理过程有关[15]。
miRNA-210是miRNA中的一种类型,作为一种靶基因的沉默子,主要与癌症及癌细胞转移的研究有关[16-17]。有研究报道,miRNA-210位于人体染色体的11p15.5位点上[18],通过参与机体的上皮间质转化过程(epithelial-mesenchymal transition,EMT)[19],使得癌细胞和干细胞一样具有了浸染和转移的能力。但最新研究发现,miRNA-210还可能与骨髓间充质干细胞分化有关[20]。
马氏珠母贝(Pinctada martensii),又称合浦珠母贝(Pinctada fucata),属软体动物门珠母贝属,是我国南方沿海地区人工培育海水珍珠的最常用贝类[21]。在前期的实验中本课题组发现马氏珠母贝中也存在miR-210[22],鉴于miR-210在调节成骨细胞矿化以及调控人骨髓间充质干细胞分化中发挥作用,推测miR-210在贝壳形成过程中行使了相应的功能。为了进一步探究miR-210在马氏珠母贝矿化中的作用机制,本研究利用miR-RACE、生物信息学及实时荧光定量PCR技术对Pm-miR-210进行序列验证和分析以及组织定量检测,并进行Pm-miR-210靶位点基因的筛选,为贝类矿化机制研究提供理论依据。
1 材料与方法
1.1 材料与试剂
实验用马氏珠母贝取自湛江市徐闻县广东岸华集团珍珠养殖基地,选取活力旺盛、生长状况良好的贝体进行实验。马氏珠母贝外套膜边缘膜和中央膜材料离体后立即放入液氮速冻,之后于-80℃冰箱保存备用。RNAiso for Small RNA购自Takara,Trizol购自Invitrogen公司,SYBR® Select Master Mix 购自Applied Biosystems公司,GeneJET PCR Purification Kit 购自Thermo公司、DNA Marker、Reverse Transcriptase M-MLV反转录酶、Taq 聚合酶、pMD19-T vector购自大连宝生物工程有限公司,大肠杆菌DH5α为本实验室自备。
1.2 方法
1.2.1 Small RNA提取和模板 cDNA制备 采用RNAiso for Small RNA试剂盒及说明书提取马氏珠母贝外套膜的small RNA,得到的small RNA需经过0.01 g/mL 琼脂糖凝胶电泳检测其完整性,并使用紫外分光光度计来检测其浓度。选取质量较好的small RNA,参照TaKaRa Poly(A) Polymerase说明书进行后续的3' 端加Poly A尾和5' 端加接头实验,然后通过M-MLV反转录酶以及通用引物制备cDNA模板。整个实验过程所用耗材均要求RNase-free。
1.2.2 miR-RACE的扩增 分析已获得的马氏珠母贝转录组数据库中 Pm-miR-210,采用上述步骤合成的cDNA模板,通过Primer Premier 5.0软件分别对Pm-miR-210设计3' miR-RACE、5' miR-RACE特异性引物GSP1和GSP2,结合3' miR-RACE和5' miR-RACE所对应的通用引物进行PCR扩增,引物序列如表1。采用茎环法[23],设计茎环引物。PCR扩增体系及反应程序均参照TaKaRa公司提供的说明书进行。依据不同引物的退火温度Tm值调整。PCR扩增产物通过0.01 g/mL琼脂糖凝胶电泳检测后,将目的基因片段连接到pMD19-T载体上,16℃放置16 h。之后转化到感受态细胞中,菌落PCR后挑取阳性单克隆送往上海生工测序[24]。

表1 引物序列及用途Table 1 Primer sequences and application
1.2.3 Pm-miR-210序列分析 经测序验证 PmmiR-210的成熟体序列后,通过Clustal W2在线软件进行序列同源性分析,然后从马氏珠母贝基因组数据库找到包含Pm-miR-210在内的scaffold序列,利用 M-fold 软件在线预测其二级结构,得到Pm-miR-210前体序列及颈环结构。
1.2.4 荧光定量PCR检测Pm-miR-210的表达
利用 Trizol法提取马氏珠母贝外套膜边缘膜和中央膜的总RNA,经0.01g/mL琼脂糖凝胶电泳检测其完整性,并使用紫外分光光度计测定A260/A280值,之后对总RNA进行筛选,使用质量较好且条带完整无损的总 RNA进行荧光定量PCR模板的制备。在此过程中使用茎环引物对Pm-miR-210进行特异性反转录。把反转录生成的cDNA模板稀释100倍,以U6作为内参[22],按照如下反应体系进行荧光定量 PCR:SYBR Mix 5 μL,cDNA 0.5 μL,上下游引物各0.4 μL,ddH2O 3.7 μL。反应条件为:95℃ 7 min ,95℃15 s,Tm 10 s,72℃35 s,40个循环。采用 2-△△Ct法计算Pm-miR-210在马氏珠母贝外套膜边缘膜和中央膜中的相对表达量,并且根据SPSS 20.0进行两组间的差异性统计分析[25]。
1.2.5 生物信息学进行Pm-miR-210的靶位点基因预测 从NCBI数据库中下载具有完整3' UTR序列的马氏珠母贝矿化相关基因,利用靶位点预测软件RNA-hybrid预测Pm-miR-210的靶位点与矿化相关基因的3'UTR作用位点,以自由能小于-20 kcal/mol 为筛选标准,获得Pm-miR-210的靶基因。
2 结 果
2.1 Pm-miR-210的序列克隆及分析
利用miR-RACE技术扩增得到Pm-miR-210的3'端序列和5'端序列,测序结果显示,测序结果与预测序列完全一致,说明高通量测序的准确度很高。通过Clustal W2在线软件,将人类(Homo sapiens)、家鼠( Mus musculus)、果蝇( Drosophila pseudoobscura)和海蠕虫(Capitella teleta)的miR-210成熟体序列同马氏珠母贝Pm-miR-210序列进行同源性比较,结果显示物种间miR-210在种子区域(5'端第2-8nt)完全一致,Pm-miR-210与果蝇的成熟体同源性达到82%,只有4个碱基的差异。与人类和家鼠的成熟体同源性也高达73%,虽与海蠕虫相比较,同源性较低,但也达到了64%。说明Pm-miR-210与其他物种的miR-210序列具有较高的保守性(图1)。并且,采用M-fold在线软件获得了Pm-miR-210前体序列的二级结构,发现其与典型的miRNA一样,具有发夹结构(图2)。
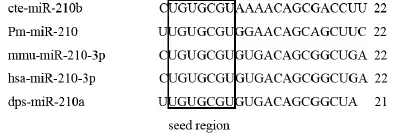
图1 序列比对结果Fig.1 Sequence alignment results
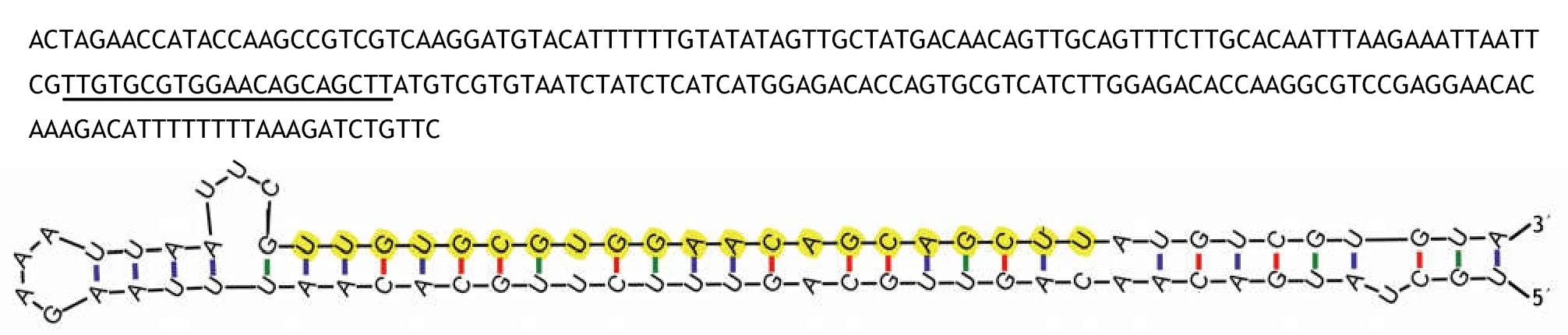
图2 前体序列及二级结构Fig.2 Precursor sequence and two-stage structure
2.2 Pm-miR-210的外套膜表达量分析
提取马氏珠母贝外套膜边缘膜和中央膜的RNA后,使用茎环引物特异性反转录成cDNA模板进行实时荧光定量 PCR。荧光定量结果显示,Pm-miR-210在外套膜边缘膜和中央膜都有较高表达,CT值分别为11.9和11.1,而且在边缘膜的含量比在中央膜的含量高出0.442631,但差异不具有统计学意义(P>0.05)。
2.3 马氏珠母贝Pm-miR-210靶基因的筛选
将马氏珠母贝转录组数据库中矿化相关的unigene序列靶点筛选后得出Pm-miR-210对多个靶位点都具有不同程度的调控作用,结合miR-210的文献报道及在线预测软件 RNAhybrid显示的最小自由能,最终确定钙调蛋白(calmodulin)、钙调磷酸酶-A亚基(calcineurin A subunit)、N19以及shematrin-3为Pm-miR-210的候选靶基因(图3)。

图3 候选靶基因Fig.3 Candidate target genes
3 讨 论
本实验以课题组前期研究中构建的马氏珠母贝miRNA转录组数据库中miR-210的序列为基础,通过高通量测序后获得Pm-miR-210序列,通过与其他物种的成熟体序列比对得出,Pm-miR-210成熟体的序列的保守性较高,在种子区域(miRNA 5'端第2-8个核苷酸)完全一致,说明miR-210在进化过程中比较保守。研究表明,“种子”序列[26]和靶基因的碱基互补配对对于miRNA发挥调控功能至关重要。从荧光定量结果来看,Pm-miR-210在外套膜中央膜和边缘膜中均有表达,证实了Pm-miR-210在外套膜中的存在,初步推测它参与了棱柱层和珍珠层的生物矿化。
马氏珠母贝的贝壳及珍珠的形成是一个十分复杂的生物矿化过程,外套膜是矿化作用中最关键的组织。外套膜细胞分泌的有机基质(基质蛋白)和钙离子等前驱物经过沉淀最终形成贝类的贝壳和珍珠[27,28]。一般来说,外套膜边缘区外表皮细胞主要分泌形成棱柱层,外套膜套膜区及中央区外表皮细胞主要分泌形成珍珠层[29]。从荧光定量结果得出,Pm-miR-210在外套膜的边缘膜和中央膜有较高的表达量,CT值分别为11.9和11.1。因此推测miR-210可能参与了马氏珠母贝的矿化过程,即参与了珍珠层和棱柱层形成的表达调控。
与贝类贝壳形成的矿化机制相似,生物体骨骼的形成也是一个矿化作用的过程。骨髓间充质干细胞(Bone Mesenehymal Stem Cells,BMSCs)具有强大的增殖能力和多向分化潜能,在适宜的体内或体外环境下可分化为成骨细胞[30],成骨细胞分泌的骨基质与钙离子经过一系列的矿化作用,最终形成新骨。在这一过程中,一些miRNA的调控会影响成骨细胞的分化,进而干扰或抑制矿化生理过程。Mizuno等[31]利用实时定量PCR法得出,在间充质干细胞ST2自然增殖过程中,miR-125b表达上调,且随着表达量的增加,细胞的增殖作用逐渐减弱,由此推断,miR-125b可能起到抑制ST2增殖的作用。Wang等[32]发现在hFOB1.19(人SV40转染成骨细胞)成骨分化的过程中,miR-27表达水平上升,随后将miRNA-27或其模拟物转染hFOB1.19,实时定量PCR结果显示ALP和骨钙素mRNA表达水平显著升高,提示 miR-27对成骨分化有正调控作用。同样,在贝类珍珠层形成中,miRNA也参与了表达调控。郑哲[33]等研究表明,miR-2305通过靶向调控马氏珠母贝外套膜pif177和pearlin的表达而对珍珠质的形成产生影响。
本研究中的Pm-miR-210是否参与贝类的矿化作用?许啸等[20]研究表明,miR-210基因在成骨细胞分化过程中表达显著性上调,且具有诱导骨髓间充质干细胞分化为成骨的作用,分化形成的成骨细胞会分泌骨基质,骨基质经过矿化作用最终形成骨骼[34]。Mizuno等[35]用基因芯片分析BMP-4诱导骨髓基质细胞ST2成骨分化过程中miRNA表达的差异性,发现miR-210表达显著上调。另外,贝壳的形成受有机基质控制比较复杂,由大约95%的文石碳酸钙和5%的有机基质交替叠加,层层复合而成,有机基质的含量虽少,却直接决定了整个贝壳的生物矿化过程[36]。鉴于miR-210参与了骨骼形成的相关靶基因的调控和有机基质在生物矿化中的重要调控机制,因此,我们推测miR-210可能也是通过调控马氏珠母贝外套膜分泌的有机基质的靶基因,来影响其矿化作用的。本研究中预测的Pm-miR-210的候选靶基因为calmodulin、calcineurin A subunit、N19和shematrin-3。其中,calmodulin和calcineurin属于钙离子结合蛋白,通过调控钙的摄取、转运和吸收等新陈代谢过程,对贝壳的形成起到关键作用[37-38];N19和shematrin-3属于基质蛋白,主要参与珍珠层和棱柱层形成。Shematrin家族在贝壳矿化过程中起到框架蛋白的作用,N19参与马氏珠母贝的矿化结晶并负调控珍珠层矿化的形成过程[39-40]。此4种候选靶基因均在贝壳的生物矿化中发挥了有机基质的调控作用,因此可以断定Pm-miR-210参与了马氏珠母贝贝壳形成的过程。
RNAhybrid是用来筛选靶位点基因快速有效的方法,但这种方法只是对miRNA靶基因的一个预测,要彻底研究miR-210对靶基因的调控机制,还需要进一步做基因的过表达实验和基因功能验证。对于筛选出来的miR-210的靶基因是如何参与马氏珠母贝外套膜的矿化机制的,更需从激活靶蛋白的信号通路方面进行深入的研究。
[1]BUSHATI N,COHEN S M.microRNA functions [J].Annual review of cell and developmental biology,2007,23:175-205.
[2]史孟婧,赵远鹏,邹仲敏,等.MiR-210在缺氧中调控机制的研究进展[J].现代生物医学进展,2013,13(16):3173-3175.
[3]EULALIO A,HUNTZINGER E,IZAURRALDE E.Getting to the root of miRNA-mediated gene silencing [J].Cell,2008,132(1):9-14.
[4]FILIPOWICZ W,BHATTACHARYYA S N,SONENBERG N.Mechanisms of post-transcriptional regulation by microRNAs:are the answers in sight[J].Nature reviews Genetics,2008,9(2):102-114.
[5]TANG G.siRNA and miRNA:an insight into RISCs[J].Trends Biochem Sci,2005,30:106-114.
[6]方福德.microRNA的研究方法与应用[M].北京:中国协和医科大学出版社,2008.
[7]KHVOROVA A,REYNOLDS A,JAYASENA S D.Functional siRNAs and miRNAs exhibit strand bias[J].Cell,2003,115(2):209-216.
[8]FARH K,GRIMSON A,JAN C,et al.The widespread impact of mammalian MicroRNAs on mRNA repression and evolution[J].Science,2005,310:1817-1821.
[9]BARTEL D.Micro RNAs:genomics,biogenesis,mechanismand function[J].Cell,2004,116:281-297.
[10]KLOOSTERMAN W,PLASTERK R.The diverse functions of microRNAs in animal development and disease[J].Dev Cell,2006,11:441-450.
[11]SEILA A,SHARP P.Small RNAs tell big stories in Whistler[J].Nat Cell Biol,2008,10:630-633.
[12]KARP X,AMBROS V.Developmental biology.Encountering microRNAs in cell fate signaling[J].Science,2005,310:1288-1289.
[13]LEUNG A,SHARP P.microRNAs:a safe guard against turmoil[J].Cell,2007,130:581-585.
[14]GUARNIERI D,DILEONE R,MicroRNAs:a new class of gene regulators[J].Ann Med,2008,40:197-208.
[15]CROCE C,CALIN G.miRNAs,cancer,and stem cell division[J].Cell,2005,122:6-7.
[16]YU P F,FAN S F,HUANG L,et al.MIR210 as a potential molecular target to block invasion and metastasis of gastric cancer[J].Medical Hypotheses,2015,84:209-212.
[17]IVAN M,HUANG X.miR-210:fine-tuning the hypoxic response[J].Adv Exp Med Biol,2014,772:205-227.
[18]XIONG L,WANG F,HUANG X,et al.DNA demethylation regulates the expression of miR-210 in neural progenitor cells subjected to hypoxia[J].FEBS Journal,2012,279:4318-4326.
[19]TAKIKAWA T,MASAMUNE A,HAMADA S,et al.miR210 regulates the interaction between pancreatic cancer cells and stellate cells[J].Biochem Biophys Res Commun,2013,437:433-439.
[20]许啸.miRNA-210在人骨髓间充质干细胞诱导成骨分化中的作用及意义[D].西安:第四军医大学,2010.
[21]田荣荣,田群莉,杜晓东,等.马氏珠母贝SRF基因的分子特征及组织特异性表达[J].基因组学与应用生物学,2015,34(2):272-278.
[22]焦钰,田群莉,杜晓东,等.马氏珠母贝 LST8基因cDNA的分子特征及表达分析[J].基因组学与应用生物学,2014,33(1):10-15.
[23]石琰.miR-210及其靶基因转录因子E2F3与喉鳞状细胞癌的相关研究[D].太原:山西医科大学第一临床医学院,2012.
[24]潘晓艳,郑哲,田荣荣,等.马氏珠母贝组蛋白H3基因的克隆与表达特性分析[J].基因组学与应用生物学,2015,34(7):1421-1428.
[25]JIAO Yu,ZHENG Zhe,DU Xiaodong,et al.Identification and characterization of MicroRNAs in pearl oyster Pinctada martensii by Solexa deep sequencing [J].Marine Biotechnology,2014,16(1):54-62.
[26]颜宇琦,王梅梅,熊亚南,等.miR-210过表达载体的构建及 Northern鉴定[J].河北联合大学学报(医学版),2012,14(3):306-308.
[27]张学骜,王建方,吴文健,等.贝壳珍珠层生物矿化及其对仿生材料的启示[J].无机材料学报,2006,21(2):257-265.
[28] 徐在宽.三角帆蚌不同部位外套膜的比较矶究[J].海洋湖沼通报,1991(1):71-76.
[29]石安静,张矛,吴中文,等.三角帆蚌珍珠囊形成的研究[J].水产学报,1985,9(3):248-252.
[30]MAJUMDAR M K,THIEDE M A,MOSCA J D,et al.Phenotypic and functional comparison of cultures of marrow- derived mesenchymal stem cells(MSCs)and stromal cells[J].J Cell Physiol,1998,176:57- 66.
[31]MIZUNO Y,YAGI K,TOKUZAWA Y.miR-215b inhibits osteoblastic differentiation by down-regulation of cell proliferation[J].Biochem Biophys Res Commun,2008,368:267-272.
[32]WANG T,XU Z.miR-27 promotes osteoblast differentiation by modulating Wnt signaling[J].Biochem Biophys Res Commun,2010,402:186-189.
[33]郑哲.马氏珠母贝珍珠层形成microRNA的筛选和功能的研究[D].湛江:广东海洋大学,2013.
[34] 刘晓丹,李春风,谷大海,等.成骨细胞的研究进展[J].中国畜牧兽医,2011,38(7):53-56.
[35]MIZUNO Y,TOKUZAWA Y,NINOMIYA Y,et al.miR-210 promotes osteoblastic differentiation through inhibition of AcvR1b[J].FEBS Lett,2009,13:2263-2268.
[36]DAUPHIN Y.Comparison of the soluble matrices of the calcitic prismatic layer of Pinna nobilis(Mollusca,Bivalvia,Pteriomorpha).Comparative Biochemistry And Physiology a-Molecular And Integrative Physiology.2002,132(3):577-590.
[37]张君诚,孟玉环,等.植物Ca2+-caM信号系统及其调控研究进展[J].重庆师范大学学报,2005,22(4):49-52.
[38]符民桂,唐朝枢.钙调神经磷酸酶的研究进展[J].生物化学与生物物理进展,2000,27(2):157-159.
[39]YANO M,NAGAI K,MORIMOTO K,et al.Shematrin:A family of glycine-rich structural proteins in the shell of the pearl oyster Pinctada fucata [J].Comparative Biochemistry and Physiology B-Biochemistry & Molecular Biology,2006,144:254-262.
[40]YANO M,NAGAI K,MORIMOTO K,et al.A novel nacre protein N19 in the pearl oyster Pinctada fucata[J].Biochemical and Biophysical Research Communications,2007,362(1):158-163.
(责任编辑:陈庄)
The Structure and Function Prediction of miR-210 in the Mineralization Mechanism of Mantle in Pinctada martensii
PAN Xiao-yan,ZHENG Zhe,TIAN Rong-rong,JIAO Yu,DU Xiao-dong
(Fisheries College,Guangdong Ocean University,Zhanjiang 524025,China)
To explore the structure and function of miR-210 and the regulation mechanism in Pinctada martensii,miR-RACE technology was used to verify and analyze the mature sequence of the Pm-miR-210.Bioinformatics analysis was performed to obtain the precursor of the sequence of Pm-miR-210 and predict target genes.The miR-RACE result was consistent with the result of high-throughput sequencing.We got the precursor sequence of Pm-miR-210 with 80 bp neck ring structure by the transcriptome data analysis and the mature sequence of the Pm-miR-210.Multiple sequence alignment revealed that the seeds of miR-210 mature sequence area among species are highly conservative.Pm-miR-210 is highly and similarly expressed in the mantle central and mantle edge(P >0.05).Targeted forecast analysis indicated that calmodulin,calcineurin A subunit,N19 and Shematrin-3 are candidate target genes of Pm-miR-210.
Pinctada martensii;Pm-miR-210;Target prediction;mineralization
S917.4
A
1673-9159(2016)03-0009-06
10.3969/j.issn.1673-9159.2016.03.002
2016-03-21
国家自然科学基金(31272635);国家自然科学基金(41206141);国家自然科学基金(31372526)
潘晓艳(1990—),女,硕士研究生,主要从事珍珠贝分子生物学研究。
焦钰(1981—),女,博士,主要从海洋生物分子生物学研究。Tel.:0759-2383346,Email:jiaoyu1981@hotmail.com

