小麦类钙调素TaCML79基因的克隆和表达分析
周 硕,刘永伟,董福双,杨 帆,赵 和,柴建芳,吕孟雨,孙果忠,王海波
(河北省农林科学院 遗传生理研究所,河北省植物转基因中心,河北 石家庄 050051)
小麦类钙调素TaCML79基因的克隆和表达分析
周 硕,刘永伟,董福双,杨 帆,赵 和,柴建芳,吕孟雨,孙果忠,王海波
(河北省农林科学院 遗传生理研究所,河北省植物转基因中心,河北 石家庄 050051)
为了研究小麦类钙调素基因的功能和在植物逆境响应中的分子机制,以电子克隆结合RT-PCR的方法,在小麦中克隆到一个类钙调素基因TaCML79。该基因的开放阅读框长度为588 bp,编码的蛋白长度为195个氨基酸,推测的分子量为20.45 kDa,等电点为4.6,属于酸性蛋白,具有2个典型的和一个不完整的EF-hand结构域。多序列比对和系统进化树分析表明,TaCML79与野生稻的ObCML35和水稻的OsCML10亲缘关系较近,相似性分别为84.6%和86.8%。该基因的表达在根和地上部分受到热激、NaCl、渗透和冷胁迫的诱导或抑制,初步推断该基因可能与植物抗逆有密切的关系。
小麦;类钙调素;EF-hand结构域;基因克隆;基因表达
钙信号通路在植物的生长发育和耐受逆境胁迫中起着至关重要的作用。大多数的外界刺激都会引起植物细胞内钙离子(Calcium,Ca2+)水平发生改变,其变化的频率、幅度和胞内的位置等的不同,会形成复杂的Ca2+信号,由下游的钙离子结合蛋白感知并传导,激活下游的信号转导通路[1-5]。钙调素(Calmodulin,CaM)和类钙调素(Calmodulin-like protein,CML)是植物中重要的Ca2+感受蛋白,其本身没有酶的活性,可通过与Ca2+结合,改变自身构象,从而激活下游的靶蛋白发挥功能[6-7]。典型的CaM非常保守,一般由149个氨基酸组成,具有4个EF-hand结构域,参与了植物的热激(TaCaM1-2和AtCaM3)[8-10]、盐胁迫(GmCaM4)[11]、氧化胁迫(CaCaM1)[12]、生物胁迫(GmCaM4)[11]、光形态建成(AtCaM7)[13]等信号通路。CML是与CaM相似性较高的钙离子结合蛋白大家族,拟南芥中有50个成员,水稻中有32个成员,蛋白长度一般为83~315个氨基酸不等,具有2~6个EF-hand结构域[6-7,14]。CML参与了植物的盐胁迫(AtCML9和OsMSR2)[15-17]、ABA信号(AtCML37和AtCML24)[18-20]、抗坏血酸合成(AtCML10)[21]、过氧化物酶体代谢(AtCML3)[22]、生物胁迫(AtCML42)[23]等抗逆相关的信号转导过程。然而,对CaM/CML的研究多集中在拟南芥和水稻中,作为世界上最重要的作物小麦,CaM/CML基因的克隆和功能研究则相对滞后。目前,仅克隆了10个小麦CaM基因[24],尚未见关于CML基因的报道,小麦CML基因的表达特性和生物学功能基本上还处于未知的状态。由于CaM/CML基因在结构和功能上具有高度的保守性,推测小麦的CaM/CML基因可能参与植物的抗逆信号通路。本研究在对小麦CaM/CML基因电子克隆的基础上,从小麦cDNA中分离克隆了一个新的CML基因TaCML79,对其进行了生物信息学分析,并研究了其基本特性及逆境胁迫下的表达特征,为深入研究和利用该基因奠定了基础。
1 材料和方法
1.1 材料及逆境胁迫处理
试验于2015-2016年在河北省农林科学院遗传生理研究所植物转基因中心进行。以河北省农林科学院遗传生理研究所育成的国审小麦品种金禾9123为研究材料。取籽粒饱满、大小均匀一致的小麦种子,经表面消毒后置于含有双层湿润滤纸的培养皿中。待种子萌发后,选取发芽状态较一致的种子播于蛭石盒中,在光照培养箱(光周期:16 h/8 h;温度:24~26 ℃)中培养14 d后进行热激、NaCl、渗透和冷胁迫处理。具体处理方法为:热激处理组将幼苗置于42 ℃光照培养箱,于0,10,20,30,60,120,180 min分别取样;NaCl胁迫组用200 mmol/L NaCl处理,渗透胁迫组用16.1%的PEG6000处理,冷处理组将幼苗置于4 ℃光照培养箱,后3种处理分别于0,1,3,6,12,24 h取样。4种胁迫处理时间点取样,皆为根和地上部分2组样本,且均为10株幼苗的混合样,取样后置于液氮速冻,于-80 ℃保存备用。
1.2 植物总RNA的提取及cDNA第1链的合成
利用RNAiso Plus(TaKaRa)提取植物总RNA。cDNA第1链的合成使用PrimeScript RT reagent Kit with gDNA Eraser(TaKaRa)。
1.3 小麦TaCML79基因的克隆和序列分析
以小麦TaCaM1-1蛋白序列为探针,在NCBI的小麦EST数据库中进行tBlastN比对(http://blast.ncbi.nlm.nih.gov/Blast.cgi),下载一致性较高的ESTs序列,然后利用DNAStar软件对ESTs序列进行拼接,得到多个可能的小麦CaM/CML基因。根据其中一个小麦CML基因TaCML79的拼接序列,设计基因的开放阅读框(Open reading frame,ORF)扩增引物,上游引物TaCML79-F:5′-GCATTCATCTC CTTTGAGCAG-3′,下游引物TaCML79-R:5′-GGAGG AGGAGCCTGGCGTAG-3′,由上海捷瑞生物工程有限公司合成。PCR反应使用TransStart FastPfu Fly DNA Polymerase(北京全式金生物技术有限公司),体系为50 μL:FastPfu Fly DNA Polymerase 1 μL,5×PCR Buffer 10 μL,PCR Stimulant 10 μL,10 μmol/L的上下游引物各2 μL,小麦cDNA 2 μL,去离子水23 μL。PCR反应条件为:98 ℃ 2 min;98 ℃ 20 s,56 ℃ 20 s,72 ℃ 1 min,32个循环;72 ℃ 5 min。回收的PCR产物目的片段连接至Blunt vector(北京全式金生物技术有限公司),转化E.coliDH5α,PCR、酶切筛选阳性克隆,并对阳性克隆进行测序验证。序列的ORF分析、蛋白质翻译、分子量和等电点预测使用DNAStar软件包;利用ProSite(http://prosite.expasy.org/)在线分析蛋白的保守结构;利用DNAman 8.0软件对蛋白序列进行比对;利用MEGA 6.0软件Neighbor-Joining方法构建系统进化树(Bootstrap:1 000;Mode/Method:Poisson model;Gaps/Missing Data Treatment:Pairwise deletion)。
1.4 实时荧光定量PCR
参照TaKaRa SYBR Green qPCR 试剂盒的实时荧光定量PCR 引物原则,设计目标基因qRT-PCR引物。上游引物TaCML79-QF:5′-GGACTTCGCGCG CATGATGA-3′,下游引物TaCML79-QR:5′-GGAGG AGGAGCCTGGCGTAG-3′。以小麦18SrRNA为内参,上游引物18S-QF:5′-GATCCATTGGAGGGCAA GTC-3′,下游引物18S-QR:5′-GATGCTTGCTTTGAG CACTC-3′。实时荧光定量PCR反应体系为20 μL:qPCR Master Mix (2×) 10 μL、 ROX Dye 0.4 μL、cDNA模板2.5 μL、10 μmol/L的上下游引物各0.5 μL和去离子水6.1 μL。每个样品设置3个重复。反应在ABI 7500 Real-time System上进行。反应条件为:95 ℃ 30 s;95 ℃ 5 s,58 ℃ 30 s,72 ℃ 34 s,40个循环;在72 ℃ 反应步骤时收集荧光信号。根据扩增曲线确定每个基因和样本相应的Ct值,以18S为内参基因,将“0”时间点样本的表达量设为“1”,相对表达量采用2-ΔΔCt方法计算。
2 结果与分析
2.1TaCML79基因克隆及序列分析
根据拼接的TaCML79基因的序列,设计了上下游引物,从小麦幼苗的cDNA中扩增获得一条长度为683 bp的序列(图1)。测序结果表明,其核苷酸序列与电子克隆序列一致。TaCML79基因的ORF长度为588 bp,编码的蛋白长度为195个氨基酸,推测的蛋白分子量为20.45 kDa,等电点为4.6。TaCML79蛋白具有2个典型的EF-hand结构域,位于第108~143,148~183个氨基酸,2个典型的钙离子结合区位于第121~133,161~173个氨基酸;还具有一个不完整的EF-hand结构域,位于第73~88个氨基酸,一个不完整的钙离子结合区位于第72~77个氨基酸。将TaCML79基因序列提交到NCBI的GenBank数据库,登录号为KX094887。
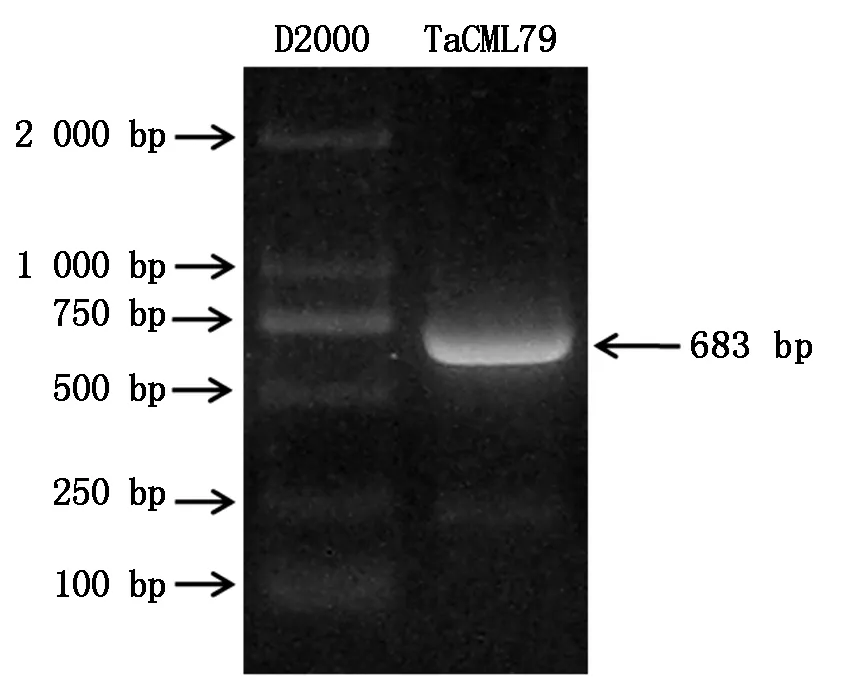
D2000.D2000 DNA分子量标准;TaCML79.TaCML79基因的扩增结果。D2000.D2000 DNA Marker;TaCML79.Gene amplification of TaCML79.
2.2 TaCML79蛋白的同源序列比对和系统进化树分析
将TaCML79预测的氨基酸序列与来自野生稻(Oryzabrachyantha)(ObCML35:XP_006645392.1)、水稻(Oryzasativa)(OsCML10:XP_015647311.1)、短柄草(Brachypodiumdistachyon)(BdCML35:XP_003565061.3)、拟南芥(Arabidopsisthaliana)(AtCML7:NP_172089.1)、小麦(Triticumaestivum)(TaCaM1-1:AAC49578)的蛋白序列进行同源比对(图2),结果显示,TaCML79与野生稻的ObCML35和水稻的OsCML10序列相近,相似性分别为84.6%和86.8%;而与短柄草BdCML35、拟南芥AtCML7和小麦TaCaM1-1的蛋白序列存在较大差异,相似性分别为30.1%,27.9% 和25.0%。表明TaCML79是一个新的小麦CML基因。
将TaCML79与上述蛋白序列,以及来自玉米(Zeamays)(ZmCML1:ACG37375)、水稻(Oryzasativa)(OsCML36:XP_015619632.1)、黄瓜(Cucumissativus)(CsCML35:XP_004136671.2)、烟草(Nicotianatomentosiformis)(NtCML3:XP_009607745.1)、拟南芥(Arabidopsisthaliana)(AtCaM1:NP_198594.1)的蛋白序列进行系统进化树分析(图3),结果显示,TaCML79与ObCML35、OsCML10、ZmCML1、OsCML36和CsCML35归为一组,属于CML家族,且TaCML79与ObCML35和OsCML10的亲缘关系较近。系统进化分析还表明,BdCML35、NtCML3和AtCML7归为一组,同属CML家族,但是与CaM家族的亲缘关系较近;AtCaM1与TaCaM1-1归为一组,属于CaM家族。

图2 TaCML79推测的氨基酸序列与其他CaM/CML蛋白序列的比对Fig.2 Alignments of deduced amino acid sequence of TaCML79 and other CaM/CML proteins
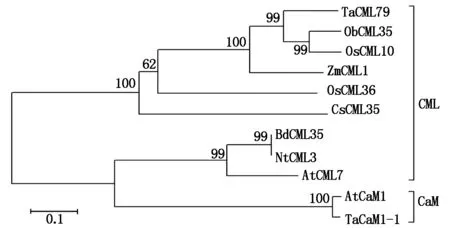
图3 小麦TaCML79蛋白与其他CaM/CML蛋白的系统进化树分析Fig.3 Phylogenetic tree of TaCML79 and other CaM/CML proteins
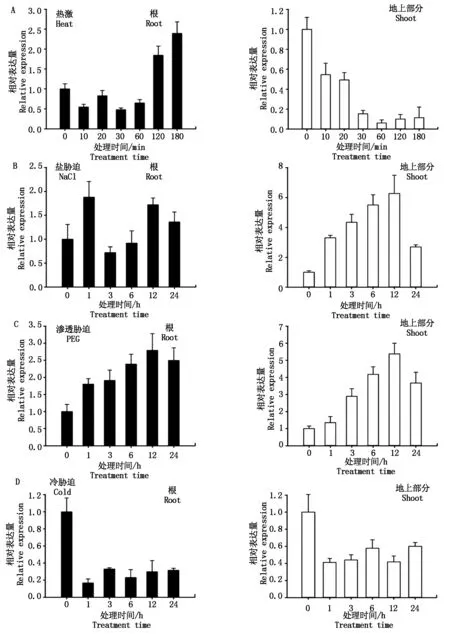
图4 TaCML79基因在各种处理下的相对表达量Fig.4 Relative expression of TaCML79 under various treatments
2.3TaCML79基因的表达特性
为了解TaCML79基因在各种胁迫下的表达特性,设计特异引物进行了荧光定量PCR的检测。结果显示,热激处理时,TaCML79基因在根中的表达在60 min前没有明显的变化,2 h时明显上升;在地上部分的表达受到热激的抑制,60 min时达到最低值,之后没有明显变化(图4-A)。
NaCl处理时,TaCML79基因在根中的表达量有明显的波动,1 h时表达量上升,随后下降到初始水平,12 h时又上升;在地上部分的表达持续上升,12 h上升到最高值,之后表达量下降(图4-B)。
渗透处理时,TaCML79基因在根和地上部分的表达均持续上升,到12 h达到最高值,之后略有下降(图4-C)。
冷处理时,TaCML79基因在根和地上部分的表达均受到明显的抑制,但在处理的不同时间点变化不大(图4-D)。
总之,TaCML79基因的表达受到热激、盐、渗透和冷胁迫的诱导或抑制,可能与小麦抵御非生物胁迫有密切的关系。
3 讨论与结论
为了克隆小麦CML基因,并研究其表达特性,本研究通过电子克隆结合RT-PCR的方法,得到一个小麦CML基因TaCML79。生物信息学分析显示,该基因的ORF长588 bp,编码的蛋白长度为195个氨基酸;蛋白序列的同源比对和系统进化树分析显示,TaCML79与来自野生稻的ObCML35和水稻的OsCML10序列相近,表明TaCML79是一个新的小麦CML基因。并且TaCML79不仅具有2个完整的EF-hand结构域,还具有一个不完整的EF-hand结构域,其是否具有结合Ca2+的特性还需更深入的研究。
CML基因广泛参与到植物的多种信号通路中,并且已证明多个CML基因与植物耐受逆境胁迫有关。AtCML9的表达量受到NaCl、冷、干旱和ABA处理的诱导,其功能缺失突变体对NaCl和干旱胁迫更加敏感[15];水稻中的一个CML基因,OsMSR2,其表达量不仅受到热激、盐、干旱和冷胁迫的诱导,过表达该基因还可以提高拟南芥和水稻对盐胁迫的抗性[16-17];而AtCML24却负调节了植物对ABA和盐离子胁迫的抗性[20]。本研究中的TaCML79基因,热激可以诱导该基因在根中的表达,抑制其在地上组织中的表达,表明TaCML79基因在热激时可能在根和地上部分发挥不同的功能;在NaCl和渗透胁迫中,TaCML79在根和地上组织中的表达均上升,24 h后略有下降,表明TaCML79可能在植物耐受盐和渗透胁迫中发挥重要的功能,植物通过提高TaCML79的表达来应对NaCl和渗透胁迫,并且随着胁迫时间的延长,植物逐渐适应逆境,TaCML79的表达随之回落;TaCML79的表达受到冷胁迫的抑制,说明TaCML79在植物抗冷反应中参与了与上述3种逆境胁迫不同的信号转导通路。
CML基因只在植物和某些原生生物中发现,所以一般认为CML基因是植物为了适应各种逆境胁迫由CaM基因进化而来[25-26]。植物在生长过程中,需要感受热激、盐、干旱、冷等各种不同的刺激,使胞内产生复杂的钙信号,这就需要由多种不同的钙结合蛋白感知、解码并传导至下游不同的信号通路。本研究中的TaCML79基因,其表达量在根和地上部分受到多种胁迫处理的诱导或抑制,不仅暗示了该基因在植物耐受逆境胁迫中发挥着重要的作用,也说明植物钙信号转导通路在不同的逆境胁迫和不同组织中的复杂性。不过,本研究仅探索了TaCML79基因的生物信息学特性和表达模式,认识其生物学功能还需要更多分子遗传学的证据。
本研究克隆到一个新的小麦CML基因TaCML79,该基因的ORF长588 bp,编码的蛋白长度为195个氨基酸,具有2个典型的和一个不完整的EF-hand结构域,与来自野生稻的ObCML35和水稻的OsCML10序列相近。TaCML79的表达受到热激、NaCl、渗透和冷胁迫的诱导和抑制,说明该基因可能与植物对非生物胁迫的抗性相关。本研究为探讨小麦CML在植物抗逆中的功能提供了新的候选基因,为小麦抗逆分子生物学及转基因研究提供了新的线索。
[1] Reddy A S,Ali G S,Celesnik H,et al.Coping with stresses:roles of calcium-and calcium/calmodulin-regulated gene expression[J].Plant Cell,2011,23(6):2010-2032.
[2] Nomura H,Shiina T.Calcium signaling in plant endosymbiotic organelles:mechanism and role in physiology[J].Molecular Plant,2014,7(7):1094-1104.
[3] Marti M C,Stancombe M A,Webb A A.Cell-and stimulus type-specific intracellular free Ca2+signals inArabidopsis[J].Plant Physiology,2013,163(2):625-634.
[4] 郑仲仲,沈金秋,潘伟槐,等.植物钙感受器及其介导的逆境信号途径[J].遗传,2013,35(7):875-884.
[5] 张晶晶,李金金,年洪娟.钙/钙调素信号途径在胁迫中的作用研究进展[J].中国微生态学杂志,2013,25(7):858-860.
[6] Mccormack E,Braam J.Calmodulins and related potential calcium sensors ofArabidopsis[J].New Phytologist,2003,159(3):585-598.
[7] Mccormack E,Tsai Y C,Braam J.Handling calcium signaling:ArabidopsisCaMs and CMLs[J].Trends in Plant Science,2005,10(8):383-389.
[8] Liu H T,Li B,Shang Z L,et al.Calmodulin is involved in heat shock signal transduction in wheat[J].Plant Physiology,2003,132(3):1186-1195.
[9] Zhang W,Zhou R G,Gao Y J,et al.Molecular and genetic evidence for the key role of AtCaM3 in Heat-shock signal transduction inArabidopsis[J].Plant Physiology,2009,149(4):1773-1784.
[10] 李 冰,周人纲.钙-钙调素信号系统参与热激信号转导的研究[J].西北植物学报,2004,24(7):1322-1328.
[11] Rao S S,El-Habbak M H,Havens W M,et al.Overexpression of GmCaM4 in soybean enhances resistance to pathogens and tolerance to salt stress[J].Molecular Plant Pathology,2014,15(2):145-160.
[12] Choi H W,Lee D H,Hwang B K.The pepper calmodulin gene CaCaM1 is involved in reactive Oxygen species and nitric oxide generation required for cell death and the defense response[J].Molecular Plant-Microbe Interactions,2009,22(11):1389-1400.
[13] Abbas N,Maurya J P,Senapati D,et al.ArabidopsisCAM7 and HY5 physically interact and directly bind to the HY5 promoter to regulate its expression and thereby promote photomorphogenesis[J].Plant Cell,2014,26(3):1036-1052.
[14] Boonburapong B,Buaboocha T.Genome-wide identification and analyses of the rice calmodulin and related potential Calcium sensor proteins[J].BMC Plant Biology,2007,7(1):4.
[15] Magnan F,Ranty B,Charpenteau M,et al.Mutations in AtCML9,a calmodulin-like protein fromArabidopsisthaliana,alter plant responses to abiotic stress and abscisic acid[J].Plant Journal,2008,56(4):575-589.
[16] Xu G Y,Rocha P,Wang M L,et al.A novel rice calmodulin-like gene,OsMSR2,enhances drought and salt tolerance and increases ABA sensitivity inArabidopsis[J].Planta,2011,234(1):47-59.
[17] Xu G Y,Cui Y C,Li M J,et al.OsMSR2,a novel rice calmodulin-like gene,confers enhanced salt tolerance in rice(OryzasativaL.)[J].Australian J Crop Sci,2013,7(3):368.
[18] Scholz S S,Vadassery J,Heyer M A,et al.Mutation of the arabidopsis Calmodulin-like protein CML37 deregulates the jasmonate pathway and enhances susceptibility to herbivory[J].Molecular Plant,2014,7(12):1712-1726.
[19] Scholz S S,Reichelt M,Vadassery J,et al.Calmodulin-like protein CML37 is a positive regulator of ABA during drought stress inArabidopsis[J].Plant Signal Behavior,2015,10(6):e1011951.
[20] Delk N A,Johnson K A,Chowdhury N I,et al.CML24,regulated in expression by diverse stimuli,encodes a potential Ca2+sensor that functions in responses to abscisic acid,daylength,and ion stress[J].Plant Physiology,2005,139(1):240-253.
[21] Cho K M,Kim Ha Thi Nguyen,Kim S Y,et al.CML10,a variant of calmodulin,modulates ascorbic acid synthesis[J].New Phytologist,2016,209(2):664-678.
[22] Dolze E,Chigri F,Höwing T,et al.Calmodulin-like protein AtCML3 mediates dimerization of peroxisomal processing protease AtDEG15 and contributes to normal peroxisome metabolism[J].Plant Molecular Biology,2013,83(6):607-624.
[23] Vadassery J,Reichelt M,Hause B A,et al.CML42-Mediated Calcium signaling coordinates responses to spodoptera herbivory and abiotic stresses inArabidopsis[J].Plant Physiology,2012,159(3):1159-1175.
[24] Yang T,Segal G,Abbo S,et al.Characterization of the calmodulin gene family in wheat:structure,chromosomal location,and evolutionary aspects[J].Molecular & General Genetics,1996,252(6):684-694.
[25] Zeng H Q,Xu L Q,Singh A,et al.Involvement of calmodulin and calmodulin-like proteins in plant responses to abiotic stresses[J].Frontiers in Plant Science,2015,6:600.
[26] Zhu X Y,Dunand C,Snedden W,et al.CaM and CML emergence in the green lineage[J].Trends in Plant Science,2015,20(8):483-489.
Cloning and Expression Analysis of TaCML 79 in Wheat
ZHOU Shuo,LIU Yongwei,DONG Fushuang,YANG Fan,ZHAO He,CHAI Jianfang,LÜ Mengyu,SUN Guozhong,WANG Haibo
(Institute of Genetics and Physiology,Hebei Academy of Agriculture and Forestry Sciences,Plant Genetic Engineering Center of Hebei Province,Shijiazhuang 050051,China)
In order to study the function and molecular mechanism of wheat calmodulin-like protein gene(CML) in plant stress response,oneCMLgeneTaCML79 was cloned in wheat based on electron cloning and RT-PCR.Sequence analysis showed that the open reading frame (ORF) ofTaCML79 was 588 bp,encoding 195 amino acids of protein containing two typical and one incomplete EF-hand domains,with predicted molecular weight of 20.45 kDa and isoelectric point of 4.6.Multiple sequence alignment and phylogenetic tree analysis showed that TaCML79 was closely related to the ObCML35 and OsCML10,with 84.6% and 86.8% identity respectively.The expression ofTaCML79 was induced or inhibited by heat shock,NaCl,osmotic and cold stress in root and shoot tissue,suggesting thatTaCML79 might be closely related to plant resistance to abiotic stress.
Triticumaestivum;Calmodulin-like proteins;EF-hand domain;Gene cloning;Gene expression
2016-08-26
河北省农林科学院博士基金项目(F15E0008);北京全式金生物技术有限公司Trans助研梦想基金(Trans-RasDF-003);国家自然科学基金项目(31600216);河北省农林科学院科学技术研究与发展计划项目(A2015110102)
周 硕(1985-),男,河北邢台人,助理研究员,博士,主要从事作物分子生物学研究。周硕、刘永伟为同等贡献作者。
王海波(1958-),男,河北保定人,研究员,博士,主要从事作物遗传、生理、育种及转基因研究。
Q78;S512.1
A
1000-7091(2016)06-0001-06
10.7668/hbnxb.2016.06.001

