玉米ZmCPK9基因在非生物胁迫下的表达分析
足木热木·吐尔逊,陈勋基,陈 果,李建平,郝晓燕,黄全生
(新疆农业科学院核技术生物技术研究所,乌鲁木齐 830091)
玉米ZmCPK9基因在非生物胁迫下的表达分析
足木热木·吐尔逊,陈勋基,陈 果,李建平,郝晓燕,黄全生
(新疆农业科学院核技术生物技术研究所,乌鲁木齐 830091)
目的钙依赖蛋白激酶(CPKs)是一类依赖于Ca2+的丝氨酸/苏氨酸蛋白激酶,仅在植物和部分低等生物中存在。Ca2+作为第二信使,在植物生长发育和抗逆应答过程中起着重要作用。研究玉米ZmCPK9基因在幼苗期不同逆境胁迫下的动态表达,为分析该基因在玉米逆境应答中的功能和机制奠定基础。方法利用生物信息学方法,分析ZmCPK9基因序列特征,采用Real-time PCR技术分析ZmCPK9在干旱、低温、盐和ABA诱导下的表达特征。结果利用生物信息学方法从玉米基因组中鉴定出CPK基因,命名为ZmCPK9。序列分析表明ZmCPK9 cDNA序列长1 548 bp,编码515个氨基酸。生物信息学分析显示,在蛋白质序列和结构上该基因与其他物种CPK基因一样非常保守。Real-time PCR的结果表明,在干旱、低温、盐和ABA诱导下,ZmCPK9表达量均有所上调。结论ZmCPK9是玉米CPKs基因家族中一个新的成员,对干旱、低温、盐和ABA等非生物胁迫具有应答反应,推测ZmCPK9对植物防御非生物胁迫具有一定作用。
玉米;CPK9基因;非生物逆境
0 引 言
【研究意义】玉米是重要的粮食作物和经济作物之一。生产上,玉米常常受到干旱、低温、盐等非生物因素胁迫,可导致减产甚至绝收,对农业生产造成不可限量的损失。而Ca2+是转导途径中的第二信使,在调节植物生长发育和抵抗逆境胁迫中发挥着重要作用。钙依赖的蛋白激酶(Calcium dependent protein kinases),全称是钙依赖的钙调素不依赖的蛋白激酶(Calcium-dependent calmodulin-independent protein kinases),简称CPKs[1]。生物信息学分析发现玉米ZmCPK9基因与其他物种中的CPKs基因有相似或部分相似的功能,推测可能参与了非生物胁迫的应答。因此,研究ZmCPK9基因在幼苗期对干旱、低温、盐和ABA等不同逆境胁迫下的动态表达,为对搞清该基因在玉米逆境应答中的功能和机制具有实际意义。【前人研究进展】CPKs在大豆中[2]存在以来,相继在多种植物中发现蛋白激酶。研究表明CPKs广泛存在于植物体内,是植物体内最大的Ca2+调控蛋白激酶家族,在拟南芥[3]、水稻[4]、小麦[5]和玉米[6]等植物中分别发现有34、31、20和40个CPKs基因家族成员。数目众多的CPKs基因家族和广泛的细胞内定位都暗示了CPKs基因在植物信号转导途径中具有广泛的功能。目前,对来自不同物种的CPKs基因的生物学功能仍了解甚少,其表达和代谢过程的调节机制等方面还有待进一步研究。【本研究切入点】研究报道CPKs基因与植物的抗逆作用息息相关[7],对玉米ZmCPK1、ZmCPK17、ZmCPK22、ZmCPK28和ZmCPK34基因克隆并进行了功能鉴定[8],而对玉米ZmCPK9基因尚待研究。研究玉米ZmCPK9基因在幼苗期不同逆境胁迫下的动态表达。【拟解决的关键问题】以玉米自交系B73为材料,用生物信息学和Real-time PCR对ZmCPK9基因进行分析。
1 材料与方法
1.1 材 料
玉米瑞德黄马牙(Reid yellow Dent)系列B73,属坚秆综合种,具有早熟和配合力高等优点,是美国当代商业育种中重要玉米自交系的祖先种,在生产上已得到广泛应用[9]。因此,选择玉米自交系B73为实验材料(实验室保存)。当幼苗生长至三叶期,分别进行干旱(土壤含水量达到60%开始处理)、低温(4 ℃)、NaCl(200 mmol/L水培)和ABA(100 μmol/L水培)4种胁迫处理,在处理后0(CK)、1、3、6、12和24 h取整株幼苗,置于液氮中速冻,-80℃冰箱保存备用。实验设置3次重复。
所用Trolzol试剂、Real-timePCR试剂盒、反转录酶、TaqDNA聚合酶和RNA酶抑制剂均购自宝生物工程有限公司;引物及其他试剂均购自生工生物工程(上海)有限公司。
1.2.1 ZmCPK9的生物信息学
利用NCBI中Blastp[10]、CCD[11]和Cobalt[12]工具分别对ZmCPK9进行氨基酸同源序列分析、功能域分析、基因序列比对分析和不同物种CPKs基因进化树分析,并采用neighber-joning方法构建系统进化树。利用ExPASy中ProtParampI/Mw[13]在线工具对ZmCPK9基因的蛋白序列的理化性质、氨基酸组成等一级结构进行预测,利用SignalP 4.1 Server[14]进行信号肽分析,利用ProtScale[13]进行疏水性/亲水性预测,Profun2.2 Server[15, 16]进行功能预测,利用Psort[17]在线工具对编码蛋白亚细胞定位进行预测,GORIV[18]对蛋白进行二级结构预测,通过SWISS-MODEL[19]在线软件同源建模。除特殊说明外,软件均设置为默认参数。
1.2.2 总RNA的提取和引物设计
参照试剂盒使用说明,采用TROLZOL试剂分别提取材料总RNA。根据Maize Sequence数据库中(http://www.maizegdb.org)ZmGAPDH内参基因和ZmCPK9基因序列,用软件Primer 5.0设计Real-time PCR特异性引物:
ZmGAPDH-F5'-CAACGACCCCTTCATCACCAGG-3',
ZmGAPDH-R5'-ATACTCAGCGCCAGCCTCACCC-3';
ZmCPK9-FCCACGTTTTGCCGAAGAAA,
ZmCPK9-RCCGCTTTTTTCGTGGCACT。
涤尘居卧室。灯下、床上,赤裸的柳含烟就像掉进冰窖里脸色苍白。她吃力地睁开眼睛看到床前萧飞羽的身影。“我不是有意的,我是想,我是想……”她没有说下去,因为她竟然没有听到自己的声音。
1.2.3 Real-time PCR
按Trolzol法提取总RNA样品反转录合成cDNA,均一化后作为Real-time PCR模板,进行Real-time PCR扩增,检测不同胁迫处理后ZmCPK9基因表达量变化。
Real-time PCR按SYBRPremixExTaqTMII试剂盒操作说明进行,20 μL反应体系:SYBR Green PCRMix 10 μL,上下游引物各0.5 μL,cDNA模板1 μL,ddH2O 8 μL。反应条件:95℃,1 min;95℃,10 s,60℃,20 s,72℃,30 s,40次循环。设置3次重复。以玉米ZmGAPDH为内参基因,相对表达量计算按照2-△△Ct法[20]。
1.3 数据处理
用GraphPad prism 6.0软件进行统计分析,数值为平均值±标准差(means±SD),n=3,差异显著性分析采用Dunnett’st检验。
2 结果与分析
2.1 ZmCPK9基因序列的获得与克隆
以拟南芥和水稻CPK基因序列和蛋白序列,经Blastx比对,结合Maize GDB数据库中(http://www.maizegdb.org)分析,获得一致性较高的ZmCPK基因家族序列,根据这些家族序列在Maize GDB数据库中查找ZmCPK核酸序列,依据Blastx蛋白比对结果构建进化树进行分析,可知玉米ZmCPK基因家族与水稻OsCPK和拟南芥的AtCPK基因家族的进化关系较近,可能在功能上具有相关性或相似性,根据Real-time PCR的实验分析结果,对干旱、低温、NaCl和ABA胁迫有应答的基因进行了基因的全长cDNA克隆[21]。
2.2 ZmCPK9基因的生物信息学
2.2.1 ZmCPK9基因特征
通过PT-PCR获得ZmCPK9基因cDNA全长,在NCBI数据库中,cDNA序列比对结果显示,基因序列长1 548 bp,编码515个氨基酸。Blastp发现,ZmCPK9与AtCPK9(Q42396.1)具有同源性,一致性为100%,因此,命名为ZmCPK9。CCD功能域分析显示具有典型的CPK保守结构域,N末端均有丝氨酸/苏氨酸蛋白激酶域、Ca2+结合位点,在CPK特有的C端调控区(即是钙结合区),有4个EF-hands结构域。用Cobalt工具比较不同物种中CPKs蛋白序列,与玉米ZmCPK11、水稻OSCPK11、OSCPK9一致性分别为88%、90%和88%,这基因与玉米与水稻亲缘关系相对较近。系统发育进化树中ZmCPK9和水稻相应基因聚在一起也验证了这一结果。图1
2.2.2 ZmCPK9蛋白特征
ZmCPK9蛋白分子式为C2 508H3 955N693O759S25,分子量为56 761.68 kD,等电点(pI)9.14,平均疏水性(GRAVY)-0.295,脂肪系数(AI)83.75。GORIV软件预测蛋白无规则卷曲所占的比例最高,占47.38%,其次是α-螺旋41.55%和延伸链11.07%,不含β-螺旋。通过在线SWISS-MODEL软件对ZmCPK9和AtCPK9蛋白的三维结构进行预测,研究表明,两者的三维结构预测结果十分相似。根据SignalP4.1Server软件预测结果显示ZmCPK9基因所编码的蛋白不存在信号肽,为非分泌蛋白,它们在细胞质中合成后可能不被运转。ProtScale软件预测蛋白为弱亲水性蛋白。Psort软件预测ZmCPK9蛋白据其概率大小依次可能存在于内质网(膜)、质膜、微体(过氧物酶体)和细胞核。Profun2.2 Server软件预测显示ZmCPK9可能参与调控转录调控,属裂解酶类。图2
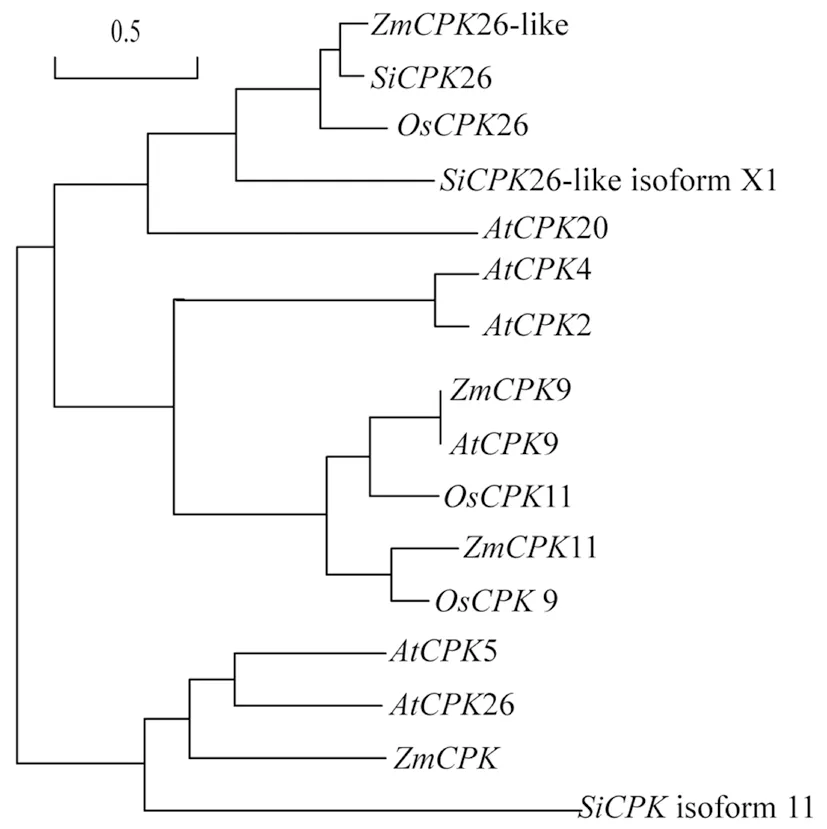
图1 ZmCPK9和其他物种基于氨基酸序列的进化树
Fig.1 Phylogenetic tree of ZmCPK9 and other plant species based on acid sequences
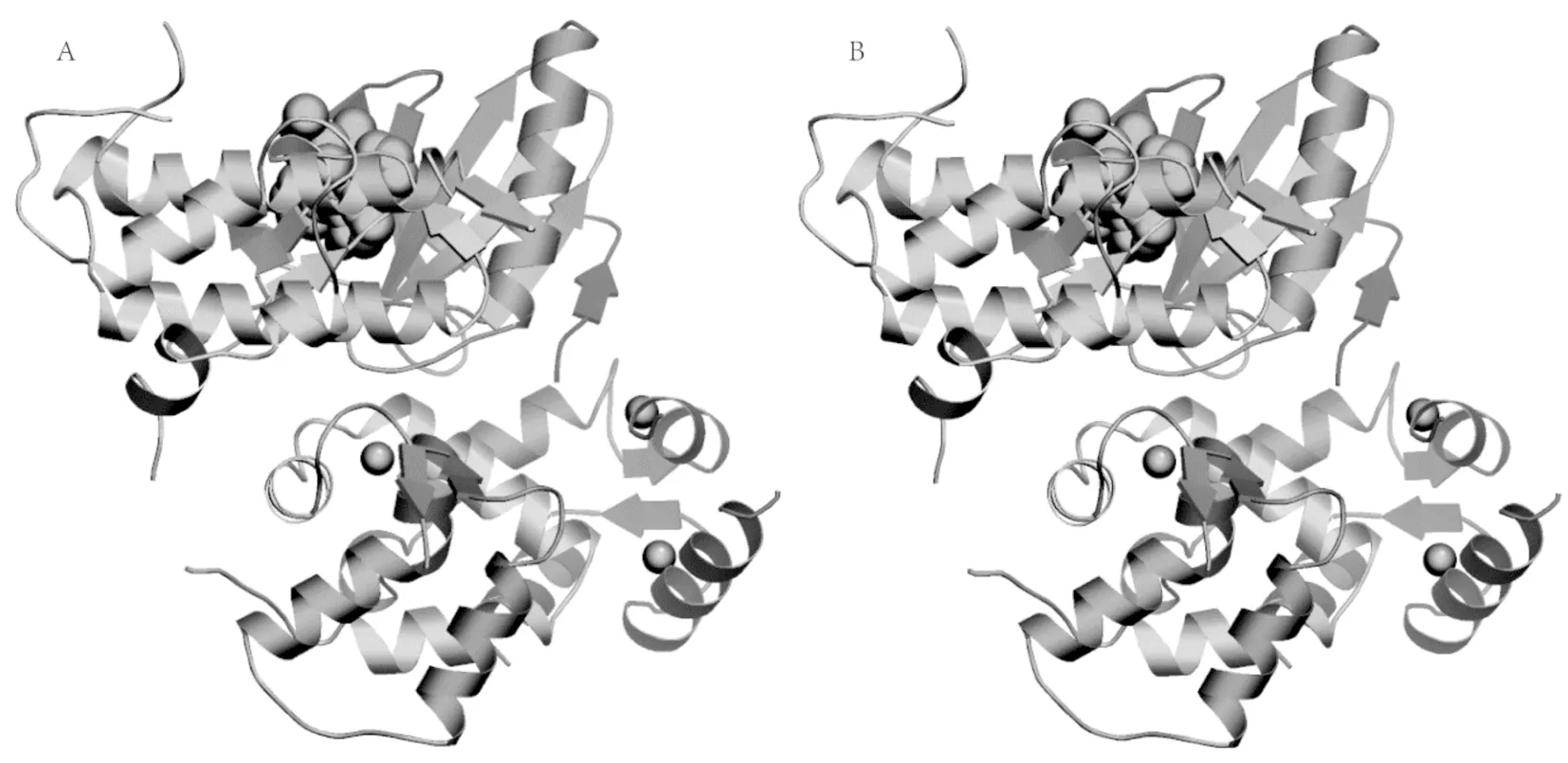
A:ZmCPK9 蛋白质三维结构预测;B:AtCPK9 蛋白质三维结构预测
A: Prediction ofZmCPK9 protein 3D structure; B: Prediction ofAtCPK9 protein 3D structure
图2 ZmCPK9与AtCPK9蛋白质三维结构比较
Fig.2 Comparison Prediction of ZmCPK9with AtCPK9 protein 3D structure
2.3不同非生物胁迫下ZmCPK9基因表达
用热酚法提取幼苗总 RNA,经反转录后得到4种不同胁迫条件下的 cDNA 模板,进行PCR扩增后,获得1.5 kb 左右的片段(图3A),与cDNA序列长度一致。
利用Real-time PCR方法,分析ZmCPK9基因在在干旱、低温、NaCl和ABA 4种胁迫条件下的表达模式。研究表明,4种胁迫处理下,在不同处理时间下,ZmCPK9基因表达量均有所上调,但不尽相同。在干旱、低温、NaCl和ABA处理时,ZmCPK9基因相对表达量上调幅度较大,分别在24、12、12和1 h时其相对表达量达到最大值,分别是对照的2.25(P<0.05)、235.11(P<0.05)、1.22(P<0.05)和3.28(P<0.05)倍,而在高温42℃处理时相对表达量下调幅度较大,在3 h时其相对表达量达到最小值,分别是对照的0.08倍(P<0.001)。在干旱胁迫条件下,随着处理时间延长而有所升高,至24 h时表达量达到最大值,为2.24±0.22,是对照的2.24倍(P<0.05)(图3B)。在250 mmol/L NaCl胁迫条件下,随着处理时间延长而其相对表达量有所上升,12 h时其相对表达量达最大值,为230.10±23.51,是对照的235倍(P<0.05),但随后开始下降(图3C)。在100 mol/L ABA胁迫条件下,随着处理时间延长而其相对表达量有所上升,12 h时其相对表达量达到最大值,为1.22±0.12,是对照的1.2倍(P<0.05),但随后其相对表达量下调至0.12±0.10,是对照的0.12倍(P<0.05)(图3D)。在4℃冷激胁迫条件下,随着处理时间延长而其相对表达量有所上升,在1 h时,其相对表达量达到最大值3.28±0.26,是对照的3.3倍(P<0.05),随时间延长后,其相对表达量逐渐下调(图3E)。ZmCPK9参与了干旱、盐、ABA和低温等信号传导调控,对植物防御非生物胁迫具有一定作用。图3
3 讨 论
3.1 ZmCPK9是玉米CPKs家族中的新成员
ZmCPK9与植物中的CPKs基因具有同源性,同时含有CPKs基因的结构域。功能域分析显示ZmCPK9含有丝氨酸/苏氨酸激酶催化域等,CPKs蛋白具有的保守功能域。用Cobalt工具比较不同物种中CPKs蛋白序列,发现ZmCPK9与水稻等中CPKs基因具有同源性,而且有非常保守的序列和结构,与玉米ZmCPK11、水稻OSCPK11、OSCPK9一致性分别为88%、90%和88%,这与玉米与水稻亲缘关系相对较近的现象是一致的(玉米和水稻同属禾本科植物)。系统发育进化树中ZmCPK9和水稻相应基因聚在一起也验证了这一结果,玉米和水稻具有较近的亲缘关系。另一方面,也暗示在逆境应答或其他信号通路中可能有相似的功能。说明ZmCPK9是植物CPKs家族中的1个新成员。
3.2ZmCPK9与参与了干旱、盐、ABA和低温的应答
对ZmCPK9基因的生物信息学特征进行了预测,结果显示ZmCPK9很多特征和参数与CPKs基因非常类似,这些结论不仅验证了它们同属CPKs基因家族,而且预示ZmCPK9可能共同参与或调控某些信号途径。功能预测结果显示ZmCPK9基因可能会参与ABA或其他激素介导的生理生化过程,预测的部分功能和已证实的不同物种中CPKs基因功能是一致的,已有研究结果也表明CPK基因在逆境(干旱、盐、激素和低温等)应答中起重要作用[22, 3, 23]。因此,分别进行干旱、低温、NaCl和ABA 4种胁迫处理玉米自交系B73三叶期幼苗,用Real-timePCR分析其在不同胁迫处理下的相对表达量情况。玉米ZmCPK9基因在4种胁迫处理下不同时间点其表达量均有所上调,但具体动态表达模式不尽相同。在玉米ZmCPK11表达量受机械伤害、H2O2和ABA诱导,而ZmCPK5参与了H2O2和ABA应答过程[23, 24]。这些试验结果与其在拟南芥、水稻和玉米中的同源基因有着相似的结论,这些结论也暗示ZmCPK9与其他物种中的同源基因有相似或部分相似的功能,参与了干旱、盐、ABA和低温的应答,但在不同的逆境应答过程中具体参与调控模式有所不同。
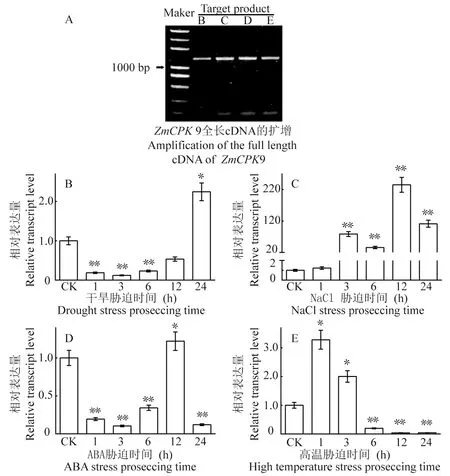
Dunnett’s t检验,*P<0.05,**P<0.01
Dunnett’s test,*P<0.05,**P< 0.01
图3 玉米ZmCPK9在不同处理条件下的转录水平和全长cDNA扩增
Fig.3 Transcriptional level of ZmCPK9 under different treated conditions and its amplification of the full length cDNA
4 结 论
该文报道的ZmCPK9是ZmCPKs基因家族中新的一员,对干旱、低温、盐和ABA等非生物胁迫具有应答反应,推测该基因可能对植物防御非生物胁迫具有一定作用。
References)
[1] 万丙良, 查中萍, 戚华雄. 钙依赖的蛋白激酶与植物抗逆性[J]. 生物技术通报, 2009, (1): 7-10.
WAN Bing-liang, ZHA Zhong-ping, QI Hua-xiong. (2009). Calcium-dependent Protein Kinases(CDPKs)and Plant Tolerance to Environmental Stresses [J].BiotechnologyBulletin, (1): 7-10.( in Chinese)
[2]Harmon, A. C., Putnam-Evans, C., & Cormier, M. J. (1987). A calcium-dependent but calmodulin-independent protein kinase from soybean.PlantPhysiology, 83(4): 830-837.
[3]Boudsocq, M., & Sheen, J. (2013). Cdpks in immune and stress signaling.TrendsinPlantScience, 18(1): 30-40.
[4]Asano, T., Tanaka, N., Yang, G., Hayashi, N., & Komatsu, S. (2005). Genome-wide identification of the rice calcium-dependent protein kinase and its closely related kinase gene families: comprehensive analysis of the cdpks gene family in rice.Plant&CellPhysiology, 46(2): 356-366.
[5] Li AL, Zhu YF, Tan XM, et al. (2008). Evolutionary and functional study of the CDPK gene family in wheat (TriticumaestivumL.)[J].PlantMolecularBiology, 66(4): 429-443.
[6]Kong, X., Wei, L., Jiang, S., et al. (2013). Genome-wide identification and expression analysis of calcium-dependent protein kinase in maize.BmcGenomics, 14(1): 1-15.
[7]Asano, T., Hayashi, N., Kobayashi, M., et al. (2012). A rice calcium-dependent protein kinase oscpk12 oppositely modulates salt-stress tolerance and blast disease resistance.PlantJournalforCell&MolecularBiology, 69(1): 26-36.
[8]Weckwerth, P., Ehlert, B., & Romeis, T. (2015). Zmcpk1, a calcium-independent kinase member of the zea mays cdpk gene family, functions as a negative regulator in cold stress signalling.PlantCell&Environment, 38(3): 544-558.
[9] 吴权明. 美国玉米种质中BSSS与Reid的区别与联系[J]. 玉米科学, 2014, 22(3): 19-23.
WU Quan-ming. (2014). Distinction and Relationship of BSSS and Reid in American Maize Germplasm [J].JounalofMaizeSciences, 22(3): 19-23. ( in Chinese)
[10]Altschul, S. F., Wootton, J. C., Gertz, E. M., Agarwala, R., Morgulis, A., & Schffer, A. A., et al. (2010). Protein database searches using compositionally adjusted substitution matrices.FebsJournal, 272(20): 5,101-5,109.
[11]Marchler-Bauer A; Derbyshire MK; Gonzales NR; Lu S; Chitsaz F; Geer LY; Geer RC; He J; Gwadz M; Hurwitz DI; Lanczycki CJ; Lu F; Marchler GH; Song JS; Thanki N; Wang Z; Yamashita RA; Zhang D; Zheng C; Bryant SH. (2015). Cdd: ncbi's conserved domain database.NucleicAcidsResearch, 43 (Database issue): 222-226.
[12]Papadopoulos, J. S., & Agarwala, R. (2007). Cobalt: constraint-based alignment tool for multiple protein sequences.Bioinformatics, 23(9):1,073-1,079.
[13]Gasteiger, E., Hoogland, C., Gattiker, A., Duvaud, S., Wilkins, M. R., & Appel, R. D., et al. (2005).ProteinIdentificationandAnalysisToolsontheExPASyServer.TheProteomicsProtocolsHandbook. Humana Press.
[14]Brunak, S., Nielsen, H. Heijne, G.,. Nordahl, P. & Heijine, G. Von. (2010). SignalP 4.0: discriminating signal peptides from transmembrane regions.Naturemethods, 8(10):785-786.
[15] Jensen, L. J., Gupta, R., Blom, N., et al. (2002). Prediction of human protein function from post-translational modifications and localization features.JournalofMolecularBiology, 319(5): 1,257-1,265.
[16]Jensen, L. J., Gupta, R., Staerfeldt, H. H., & Brunak, S. (2003). Prediction of human protein function according to gene ontology categories.Bioinformatics, 19(5): 635-642.
[17]Yu, N. Y., Wagner, J. R., Laird, M. R., Melli, G., Rey, S., & Lo, R., et al. (2010). Psortb 3.0: improved protein subcellular localization prediction with refined localization subcategories and predictive capabilities for all prokaryotes.Bioinformatics, 26(13): 1,608-1,615.
[18]Garnier, J., Gibrat, J. F., & Robson, B. (1996). GOR secondary structure prediction method version IV.MethodsinEnzymology, (266): 540-543.
[19]Biasini, M., Bienert, S., Waterhouse, A., Arnold, K., Studer, G., & Schmidt, T., et al. (2014). Swiss-model: modelling protein tertiary and quaternary structure using evolutionary information.NucleicAcidsResearch, 42(Web Server issue), W252-258.
[20]Livak, K. J., & Schmittgen, T. D. (2001). Analysis of relative gene expression data using real-time quantitative pcr and the 2(-delta delta c(t)) method.Methods, 25(4): 402-408.
[21]张毓露. 玉米CDPK基因家族的克隆、表达分析及ZmCPK12功能初探[D]. 乌鲁木齐: 新疆农业大学硕士学位论文, 2012.
ZHANG Yu-lu. (2012).ExpressionAnalysisoftheCDPKGeneFamilyandFunctionResearchofZmCPK12inMaize[D]. Master Thesis. Xinjiang Agricultural University, Urumqi. (in Chinese)
[22]Asano, T., Hayashi, N., Kikuchi, S., & Ohsugi, R. (2012). Cdpk-mediated abiotic stress signaling.PlantSignaling&Behavior, 7(7): 817-821.
[23]Ding, Y., Cao, J., Lan, N., Yuan, Z., Zhang, A., & Tan, M., et al. (2013). Zmcpk11 is involved in abscisic acid-induced antioxidant defence and functions upstream of zmmpk5 in abscisic acid signalling in maize.JournalofExperimentalBotany, 64(4): 871-884.
[24]Szczegielniak, J., Borkiewicz, L., Szurmak, B., Lewandowskagnatowska, E., Statkiewicz, M., & Klimecka, M., et al. (2012). Maize calcium-dependent protein kinase (zmcpk11): local and systemic response to wounding, regulation by touch and components of jasmonate signaling.PhysiologiaPlantarum, 146(1): 1-14.
Abstract:【Objective】 Calcium dependent proteinkinases (CPKs), a kind of serine/threonine protein kinases, are only found in plants and lower animals. Ca2+, as a second messenger, plays important roles in various aspects of plant physiology and is involved in many cellular processes. The purpose of this paper is to study the dynamic expression ofZmCPK9 gene in seedling stage under different stress conditions and to lay the foundation for further analysis of the function and mechanism of this gene in maize adverse stress response.【Method】ZmCPK9 gene sequence features were analyzed by using bioinformatics methods, and the expression characteristics ofZmCPK9 under drought, low temperature, salt and ABA stress were analyzed by real-time PCR technique. 【Result】The sequence analysis showed thatZmCPK9 had a length of 1,548 bp in the genome, encoding 515 aminoacids residues. Bioinformatics analysis revealed that the protein sequence and structure of theZmCPK9 gene were as conservative as those of other species, theCPKgene. Real-time PCR analysis discovered that the expression ofZmCPK9 was up-regulated by ABA, cold, drought and salt stress, respectively. 【Conclusion】ZmCPK9 is a new member in cornCPKsgene family in maize. The gene is involved in signal transduction regulation under the condition of drought, low temperature, salt, and ABA stress. We speculateZmCPK9 has a certain role in plant defense under abiotic stress.
Keywords: maize; calcium-dependent protein kinase 9; abiotic stress
ExpressionAnalysisofZmCDPK9GeneinMaizeunderAbioticStress
Zumuremu Turxun,CHENG Xun-ji, CHEN Guo, LI Jian-ping, HAO Xiao-yan, HUANG Quan-sheng
(ResearchInstituteofNuclearandBiotechnologies,XinjiangAcademyofAgriculturalSciences,Urumqi830091,China)
10.6048/j.issn.1001-4330.2017.09.005
S513;S188
A
1001-4330(2017)09-1606-07
2017-06-19
国家自然科学基金项目(31260331);新疆农业科学院优秀青年科技人才基金项目(2014026)
足木热木·吐尔逊 (1981-),女,新疆人 ,助理研究员,研究方向为玉米分子生物学,(E-mail) azze128@163.com
黄全生(1964-),男,新疆乌鲁木齐人,研究员,博士,研究方向为作物耐逆基因资源及分子生物学,(E-mail) hquansheng@126.com
Supported by: National Natural Science Foundation of China (31260331) and Outstanding Young Scientific Talent Foundation of Xinjaing Academy of Agricultural Sciences.(2014026)
Corresponding author:HUANG Quan-sheng(1964-),male, Urumqi, Xinjiang, Professor, supervisor, the main research directions for crop adversity molecular biology,(E-mail) hquansheng@126.com

