Wnt6、Wnt3A、LEF1和FZD1基因启动子甲基化在结直肠癌筛查及早期诊断中的临床应用
杨齐华 叶国良 徐拔群 洪晓波
结直肠癌(colorectal cancer,CRC)是常见的恶性肿瘤。最新统计显示全球有超过140万例CRC患者,每年约有69.39万例死亡,占所有癌症死亡人数的8%[1],居癌症死亡率的第4位[2]。生活环境、饮食习惯、遗传和表观遗传调控间的多步骤、多基因的复杂过程参与了肿瘤的发生和发展[3]。其中表观遗传调控的一个主要机制是DNA甲基化,主要是启动子区域CpG岛的甲基化,同时在启动子区外也有内含子及外显子区域CpG岛的甲基化,还有非CpG岛的甲基化[4-5]。DNA甲基化在细胞分化、细胞增殖、胚胎发育、遗传印记及人类肿瘤的发生中具有重要作用[6-7],是当前研究的热点。相关研究发现在CRC中,不同病理分型患者的部分甲基化基因差异较为明显[8]。另有研究发现,与其基因本身的调控比较,DNA甲基化可能对基因表达的调控更有优势[9-18]。人Wnt基因定位于12q13,在胚胎发育中,Wnt通路是Wnt基因调控的重要信号传导系统[19]。在CRC研究中,Wnt信号转导通路在调控结直肠肿瘤发生、发展、侵袭、转移等方面具有重要作用,可能成为分子靶向抗结直肠肿瘤药物[20-22]。基于当前主要芯片平台的研究和目前CRC诊断手段的优缺点,本研究主要探讨Wnt6、Wnt3A、LEF1和FZD1基因在CRC组织及正常结直肠组织中的甲基化状态,同时探讨以上基因启动子高甲基化后是否会导致基因表达下调,进而推断表观遗传是否起了主导作用。
1 对象和方法
1.1 对象 选择本院2015年12月至2016年12月收治的CRC患者55例为观察组,其中男35例,女20例;年龄≤60岁30例,>60岁25例;肿瘤位置:近端37例,远端18例;TNM分期Ⅰ~Ⅱ期21例,Ⅲ~Ⅳ期34例;淋巴结转移24例;远处转移5例;所有患者术中病理检查确诊为CRC,术前均未接受过放化疗或其他联合治疗。选择本院同期正常健康体检者55例为对照组,其中男33例,女22例;年龄≤60岁29例,>60岁26例。两组受检者性别、年龄比较,差异均无统计学意义(均P>0.05)。本研究经医院伦理委员会审核通过,所有受检者签署知情同意书。
1.2 方法
1.2.1 主要试剂及仪器 β-actin引物(上海生工生物技术公司合成)、QIAGEN试剂盒(Qiagen公司)、PCR仪(Eppendorf公司)、Trizol试剂盒(上海闪晶分子生物科技有限公司)、DNA提取试剂盒(QIAGEN试剂盒)、离心机(长沙英泰仪器有限公司)、微量紫外分光光度计(上海让奇仪器科技有限公司)、CT转换试剂盒(上海晶抗生物工程有限公司)、DNA甲基化试剂盒(北京天漠科技开发有限公司)。
1.2.2 标本收集 (1)CRC组织及对照者结直肠组织标本收集(对照者自愿接受肠镜检查及组织标本采集):收集组织标本后立即放于标本管中,同时加入RNA保存液,放于冰块中,随后迅速放于-80℃冰箱内保存。(2)CRC患者术前血标本及对照者外周血标本收集:采集血标本3~5ml,放于紫色管中,EDTA抗凝;随后迅速放于-80℃冰箱内保存。(3)CRC患者术前粪便标本及对照者粪便标本收集:收集粪便标本,分装后迅速放于-80℃冰箱内保存。
1.2.3 基因组DNA提取 提取结直肠组织基因组DNA以及血液、粪便中脱落细胞的DNA。取样本,溶解,处理。加20μl蛋白酶K,涡旋混匀,简单离心。加200μl缓冲液裂解液,涡旋15s,70℃水浴10min,简单离心。加200μl无水乙醇,涡旋混合。转移混合物至离心柱中,该柱子放在2ml收集管中,8 000r/min离心1min。把离心柱放在干净的2ml收集管中,收取管子,弃滤液。小心打开离心柱,加500μl缓冲液洗涤液1,8 000r/min离心1min。将离心柱放在干净的2ml收集管中,弃滤液。小心打开离心柱,加500μl缓冲液洗涤液2,25 000r/min离心3min。把离心柱放在新的2ml收集管中,弃滤液。30 000r/min离心1min。把离心柱放在干净的1.5ml离心管,弃滤液。小心打开离心柱,加200μl蒸馏水,清洗滤柱膜。室温静置1min,8 000r/min离心1min。最后使用微量紫外分光光度计测定所提取的基因组DNA浓度,要求>30ng/μl,A260/A280范围为 1.8~2.0。测好浓度后置于-20℃冰箱存储。血液及粪便样本的操作步骤与组织标本类似。随后进行亚硫酸氢钠修饰及CT转换试剂盒、DNA甲基化试剂盒处理作甲基化转化,置于-20℃冰箱存储。
1.2.4 甲基化特异性PCR(MSP) 通过在线 Meth-Primer软件分别设计针对甲基化和非甲基化序列的引物,并进行PCR扩增;然后进行琼脂糖凝胶电泳,确定与引物互补的DNA序列的甲基化状态。MSP法显示Wnt6、Wnt3A、LEF1和FZD1基因分别大于相应的截断值,即甲基化阳性。分别计算粪便脱落细胞DNA中Wnt6、Wnt3A、LEF1和FZD1基因启动子甲基化阳性率(甲基化阳性例数/每组例数)和粪便脱落细胞DNA中相关基因启动子甲基化对Ⅰ~Ⅱ期CRC的发现率(甲基化阳性例数/Ⅰ~Ⅱ期CRC例数)。引物信息见表1。

表1 MSP引物信息
1.2.5 RT-PCR 采用Trizol法抽提组织中总RNA。取50~100mg组织置于1.5ml离心管中,加1ml Trizol充分匀浆,室温静置5min。加0.2ml氯仿,振荡15s,静置2min。4℃、15 000r/min离心15min,取上清液。加0.5ml异丙醇,将管中液体轻轻混匀,室温静置10min。4℃、15 000r/min离心10min,弃上清液。加入1ml 75%乙醇,轻轻洗涤沉淀。4℃、8 000r/min离心5min,弃上清液。晾干,加入适量的DEPC H2O溶解(5℃促溶10~15min)。随后使用nanodrop2000微量紫外分光光度计检测RNA浓度及纯度,取A260/A280为1.8~2.0的RNA进行反转录,以两步法进行RT-PCR,然后进行2%琼脂糖凝胶电泳,利用软件分析电泳条带灰度值,相对含量=目的基因/内参β-actin。
1.2.6 Western-blot 采用Western-blot法提取组织中总蛋白。将少量组织置于1~2ml匀浆器中球状部位,尽量碾碎。加400μl单去污剂裂解液(含L-苯甲基磺酰氟)在匀浆器中进行匀浆,冰置数分钟后,再研磨组织,重复数次,使组织尽量碾碎。裂解30min后取裂解液,4℃、12 000r/min离心5min。取上清液分装并存于-20℃冰箱内。使用BCA蛋白浓度测定试剂盒检测上清液蛋白浓度。提取的组织蛋白进行聚丙烯酰氨凝胶(SDSPAGE)电泳,使用12%的分离胶分离 Wnt6、Wnt3A、LEF1和FZD1蛋白,电泳后采用半干式转移法转移到聚偏二氟乙烯膜上,用5%的脱脂牛奶封闭1h(37℃),加Wnt6、Wnt3A、LEF1 和 FZD1 抗体(1∶1 000),37℃孵育1h;HRP 标记的兔抗鼠二抗(1∶1 000),37℃孵育 1h;βactin 单克隆抗体(1∶100)作为内参照,孵育 3h。使用ECL化学发光试剂盒检测杂交信号显示条带。
1.3 统计学处理 应用SPSS 17.0统计软件。计量资料用表示,组间比较采用两独立样本t检验;计数资料用率表示,组间比较采用χ2检验。P<0.05为差异有统计学意义。
2 结果
2.1 DNA提取结果及人基因组DNA的确认 取A260/A280为1.8~2.0的110例粪便脱落细胞DNA样本进行PCR反应扩增,均可以扩增出人β-actin基因片段,部分扩增产物结果见图1。

图1 人基因β基actin扩增片段电泳图
2.2 两组受检者粪便脱落细胞DNA中相关基因启动子甲基化阳性结果 MSP显示Wnt6、Wnt3A、LEF1和FZD1基因存在甲基化或非甲基化,见图2。观察组粪便脱落细胞DNA中Wnt6、Wnt3A、LEF1和FZD1基因启动子甲基化阳性率分别为 69.1%(38/55)、63.6%(35/55)、54.5%(30/55)和 56.4%(31/55),4 种基因总甲基化阳性率为90.9%(50/55);对照组粪便脱落细胞DNA中Wnt6、Wnt3A、LEF1和FZD1基因启动子甲基化阳性率分别为 0.0%、1.8%(1/55)、0.0%和 1.8%(1/55),4 种基因总甲基化阳性率为3.6%(2/55)。

图2 粪便脱落细胞DNA中Wnt6、Wnt3A、LEF1和FZD1基因启动子甲基化的电泳图(P1-P7为平行PCR;U为非甲基化;M为甲基化,dH2O为单蒸水)
2.3 粪便脱落细胞DNA中相关基因启动子甲基化对Ⅰ~Ⅱ期CRC的发现率 粪便脱落细胞DNA中Wnt6、Wnt3A、LEF1和FZD1基因启动子甲基化对Ⅰ~Ⅱ期CRC 的发现率分别为 80.9%(17/21)、71.4%(15/21)、57.1%(12/21)和 57.1%(12/21),联合检测 4 种基因启动子甲基化对Ⅰ~Ⅱ期CRC的发现率为90.5%(19/21)。
2.4 CRC患者粪便脱落细胞DNA中相关基因启动子甲基化与临床病理特征的关系 CRC患者粪便脱落细胞DNA中Wnt6、Wnt3A、LEF1和FZD1基因启动子甲基化与患者性别、年龄、远处转移、肿瘤部位、TNM分期及淋巴结转移均未见相关(均P>0.05),见表2。
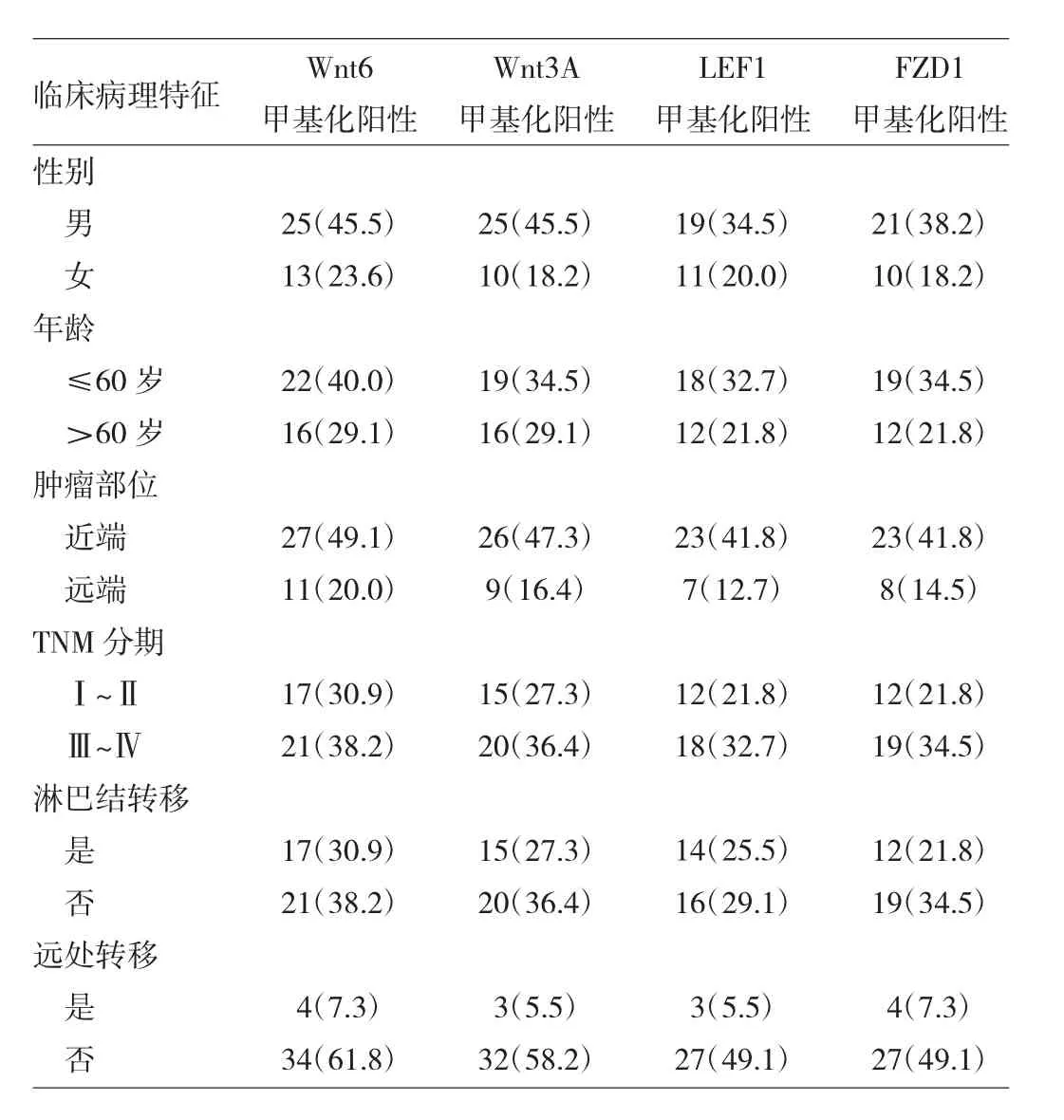
表2 55例CRC患者粪便脱落细胞DNA中相关基因启动子甲基化与临床病理特征的关系[例(%)]
2.5 不同标本类型相关基因启动子甲基化阳性率比较观察组与对照组结直肠组织、血标本、粪便标本中Wnt6、Wnt3A、LEF1和FZD1基因启动子甲基化阳性率比较,差异均有统计学意义(均P<0.01),见表3。

表3 不同标本类型相关基因启动子甲基化阳性率[例(%)]
2.6 两组受检者结直肠组织中相关基因启动子甲基化后mRNA水平比较 两组受检者结直肠组织中Wnt6、Wnt3A、LEF1和FZD1基因启动子甲基化后mRNA水平均有所下调,观察组与对照组比较差异均有统计学意义(均 P<0.01),见图 3。

图 3 两组受检者结肠组织中相关基因启动子甲基化后 mRNA 水平比较(a:WNT6;b:WNT3A;c:LEF1;d:FZD1;*与对照组比较,P<0.05)
2.7 两组受检者结直肠组织中相关基因启动子甲基化后蛋白表达水平比较 两组受检者结直肠组织中Wnt6、Wnt3A、LEF1和FZD1基因启动子甲基化后蛋白水平均有不同程度的下调,观察组与对照组的电泳图差异明显,见图4。

图4 两组受检者结直肠组织中相关基因启动子甲基化后蛋白表达的电泳图
3 讨论
表观遗传调控的一个主要机制是DNA甲基化。大多数脊椎动物基因组DNA都有少量的甲基化胞嘧啶,DNA的CG两个核苷酸的胞嘧啶在甲基转移酶的催化下被选择性地甲基化,形成5-甲基胞嘧啶。这常见于基因的5′-CG-3′序列,DNA甲基化的主要位点是CpG位点,常在基因上游调控区的启动子区域高度聚集,以CpG岛的形式存在。被甲基化的基因位点可随DNA的复制而复制,从而遗传下去。DNA甲基化能改变染色质结构、DNA稳定性、DNA构象以及DNA与蛋白质相互作用的方式,从而起到调控基因表达的作用。甲基化模式包括特异基因的高甲基化或低甲基化,与异常基因转录相关[14-15],DNA甲基化通常针对转座因子或改变沉默相邻基因的转录(DNA甲基化直接干扰特异转录因子与各自启动子的识别区结合)。如AP-2、CAREB、E2F转录因子能识别含CpG残基的序列,当CpG残基上的C被甲基化后,结合作用即被抑制;序列特异性的甲基化DNA结合蛋白与启动子区甲基化CpG岛结合,募集组蛋白去乙酰化酶(HDAC),形成转录抑制复合物,阻止转录因子与启动子区靶序列的结合,从而影响基因的转录[16-18]。因此,与其基因本身的调控比较,DNA甲基化可能对基因表达的调控具有明显优势。
目前临床上诊断CRC的方法很多。(1)螺旋CT对CRC的诊断被广泛使用,但它对晚期疾病的灵敏度、特异度较好,早期诊断的准确性较差,且为有创检查;(2)肿瘤标志物如CA19-9、CA153等为无创手段,但在诊断CRC的阳性率较低,准确性较差;(3)内镜检查目前主要用于CRC的早期诊断,是一种有创检查,部分患者可能会拒绝此项检查[23-25]。目前相关研究发现,DNA甲基化可能成为诊断癌症的新分子标志物,特别在无创、快捷取得血液、尿液、痰等标本以及诊断价值高等方面具有优势,在提前发现癌症方面具有很大潜力[26-28]。本研究结果发现,血液及粪便脱落细胞中DNA均有高甲基化,可见血液或粪便基因启动子甲基化的检测有望成为CRC无创诊断或高风险人群筛查的一个新工具。
甲基化检测根据研究位点不同,可分为2种,即基因组整体水平的甲基化检测和特异位点甲基化的检测。其中基因组整体水平的甲基化检测方法主要有免疫化学法、高效液相色谱柱(HPLC)法、CpG甲基转移酶法等;而特异位点甲基化的检测方法包括直接测序法、MSP 法、Pyrosequencing(PSQ)测序、荧光法等,这些方法都是基于亚硫酸氢盐处理的基因启动子甲基化检测方法。其中MSP法是一种快速检测方法,它的优点归纳如下:(1)不涉及使用限制性内切酶,不会出现因使用内切酶而引发的相关实验问题;(2)灵敏度高,可检出比例为1∶1 000的甲基化等位基因片段,不需要要求太高的DNA质量即可达到基因高甲基化检出率;(3)对DNA标本来源的要求不高,石蜡包埋组织DNA或少量DNA即可进行甲基化分析;(4)实验操作简便,费用低。本研究采用MSP法检测 Wnt6、Wnt3A、LEF1和 FZD1基因启动子甲基化情况,结果显示观察组与对照组结直肠组织、血标本、粪便标本中Wnt6、Wnt3A、LEF1和FZD1基因启动子甲基化阳性率比较,差异均有统计学意义;联合检测粪便标本中4种基因启动子甲基化对Ⅰ~Ⅱ期CRC的发现率为90.5%。以上结果提示MSP法检测粪便脱落细胞DNA中Wnt6、Wnt3A、LEF1和FZD1基因启动子甲基化是一种CRC早期筛查的有效方法。
综上所述,MSP法检测结直肠组织、血液及粪便脱落细胞DNA中Wnt6、Wnt3A、LEF1和FZD1基因启动子甲基化是一种CRC早期筛查的有效方法。
[1]Torre LA,Bray F,Siegel RL,et al.Global cancer statistics[J].CA Cancer J Clin,2015,65(12):87-108.
[2]Global Burden of Disease Cancer Collaboration.The Global Burden of Cancer 2013[J].JAMA oncology,2015,1(4):505-527.doi:10.1001/jamaoncol.2015.0735.
[3]Dawson MA,Kouzarides T.Cancer epigenetics:from mechanism to therapy[J].Cell,2015,150(66):12-27.
[4]Chiang NJ,Shan YS,Hung WC,et al.Epigenetic regulation in the carcinogenesis of cholangiocarcinoma[J].Int J Biochem Cell Biol,2015,67(43):110-114.
[5]Monk D.Germline-derived DNA methylation and early embryo epigenetic reprogramming:The selected survival of imprints[J].Int J Biochem CellBiol,2015,67(2):128-138.
[6]Ouadid-Ahidouch H,Rodat-DespoixL,MatifatF,etal.DNAmethylation of channel-related genes in cancers[J].Biochim Biophys Acta,2015,1848(10):2621-2628.
[7]Robinson MD,Kahraman A,Law CW,et al.Statisticalmethods for detecting differentially methylated lociand regions[J].Front Genet,2014,5(11):324.
[8]Ji M,Guan H,Gao C,et al.Highly frequent promoter methylation and PIK3CA amplification in non-small cell lung cancer(NSCLC)[J].BMC Cancer,2011,11(21):147.
[9]Pilozzi E,Maresca C,Duranti E,et al.Left-sided early-onset vs late-onset colorectal carcinoma:histologic,clinical,and molecular differences[J].Am J Clin Pathol,2015,143:374-384.
[10]Meng H,Cao Y,Qin J,et al.DNA methylation,its mediators and genome integrity[J].Int J BiolSci,2015,11(42):604-617.
[11]Okada Y.Regulation of endothelial cell-specific Robo4 gene expression by DNA methylation and non-lineage specific transcription factors[J].Yakugaku Zasshi,2014,134(20):817-821.
[12]Wang YJ,He L,Yuan M,et al.Epigenetic changes of TIMP-3,GSTP-1 and 14-3-3 sigma genes as indication of status of chronic inflammation and cancer[J].Int J Biol Markers,1900,29(3):e208-214.
[13]Esteller M.Epigenetics in cancer[J].N Engl J Med,2018,358:1148-1159.
[14]Orta ML,Dominguez I,Pastor N,et al.The role of the DNA hypermethylating agent Budesonide in the decatenating activity of DNAtopoisomeraseⅡ[J].Mutat Res,2010,694(32):45-52.
[15]Gronniger E,Weber B,Heil O,et al.Aging and chronic sun exposure cause distinct epigenetic changes in human skin[J].PLoS Genet,2010,6(14):e1000971.
[16]Migheli F,Migliore L.Epigenetics of colorectal cancer[J].Clin Genet,2012,81(33):312-318.
[17]Taberlay PC,Jones PA.DNA methylation and cancer[J].Prog Drug Res,2011,67(27):1-23.
[18]Suzuki MM,Bird A.DNA methylation landscapes:provocative insights fromepigenomics[J].NatRevGenet,2008,9(34):465-476.
[19]Bienz M.Beta-Catenin:A pivot between cell adhesion and Wnt signalling[J].Curr Biol,2005,15(23):R64-67.
[20]李悠然,侯毅,谷云飞,等.Wnt/β-catenin信号通路在结直肠癌中的研究进展[J].世界华人消化杂志,2015,21(15):1930-1936.
[21]Malcomson FC,Willis ND,Mathers JC.Is resistant starch protective against colorectal cancer via modulation of the WNT signalling pathway?[J].Proc Nutr Soc,2015,43(12):1-10.
[22]Sebio A,Kahn M,Lenz HJ.The potential of targeting Wnt/beta-catenin in colon cancer[J].Expert Opin Ther Targets,2014,18(34):611-615.
[23]高剑波.胃肠道肿瘤螺旋CT诊断的新进展[J].中华放射学杂志,2001,35(23):312-314.
[24]李岩,王孟春.消化道早期肿瘤的内镜诊断[J].中国实用内科杂志,2005,25(29):208-209.
[25]左强,李爱民,缪景霞,等.多种肿瘤标志物蛋白芯片检测系统对消化道肿瘤的诊断价值[J].中国肿瘤临床,2004,31(6):337-339.
[26]Mikeska T,Craig JM.DNA methylation biomarkers:cancer and beyond[J].Genes(Basel),2014,5(29):821-864.
[27]Mansour H.Cell-free nucleic acids as noninvasive biomarkers for colorectalcancer detection[J].Front Genet,2014,5(17):182.
[28]Brambilla C,Fievet F,Jeanmart M,et al.Early detection of lung cancer:role of biomarkers[J].Eur Respir J Suppl,2003,39(24):36s-44s.

