黄秋葵花和果荚转录组测序及类黄酮代谢差异表达分析
姚运法,张少平,练冬梅,赖正锋,黄慧明,洪建基
(福建省农业科学院 亚热带农业研究所,福建漳州 363005)
黄秋葵,学名咖啡黄葵[Abelmoschusesculentus(Linn.)Moench],为锦葵科(Malvaceae)秋葵属(Abelmoschus)一年或多年生草本植物。原产于非洲,自20世纪90年代初引入中国,现国内各地均有栽培。黄秋葵花和果荚作为重要开发价值的器官,富含蛋白质、果胶多糖、总黄酮等营养成分[1-4],其花主要开发黄秋葵花茶,经济附加值高[5];果荚具有降血压、血脂和提高机体抗疲劳等功效,在黄秋葵保健品开发方面具有巨大潜力[6]。当前,国内对黄秋葵研究主要从栽培技术、营养成分提取、功效分析和产品加工[7-10]等方面,但其基础理论研究方面还很薄弱, 例如花、果荚中类黄酮物质等主要功能成分及相关代谢途径等均属研究空白。
类黄酮(flavonoids)属于植物重要次生代谢产物之一,是指2个苯环(A-与B-)通过3个碳原子连接形成具有C6-C3-C6基础结构的一类化合物[11],由于其纯净状态呈现黄色,故称黄酮。研究表明,类黄酮物质对人体具有抗癌、抗氧化、抗动脉硬化等功能[12-13],据其结构的差异将类黄酮主要分为黄烷酮(flavanols)、异黄酮(isoflavones)、黄酮(flavones)、黄酮醇(flavonols)、二氢黄酮(2H-flavanones)与花色素(anthoyanidins)等6大类[14]。
转录组测序技术是连接基因组与代谢组的重要纽带,本团队前期对紫色黄秋葵叶片[15]、果荚[16]转录组测序和分析,初步探讨了黄秋葵次生代谢物质合成的遗传基础。本研究利用Illumina Hi-seq 2500高通量测序技术,根据黄秋葵花、果荚的转录组数据及其类黄酮代谢途径关键基因分析,探讨黄秋葵花、果荚类黄酮合成机理和关键差异表达基因,为后续黄秋葵关键基因克隆和功能验证、遗传改良和加工利用等提供研究基础。
1 材料和方法
1.1 试验材料
试验材料为黄秋葵品种‘闽秋葵3号’,种植于福建省农业科学院亚热带农业研究所试验农场,2017年6月10日种植,8月15日开始采集样品(花/果荚)。采集同一株黄秋葵植株花朵(上午9:00)和果荚(开花后8 d),花朵只保留完整花瓣,果荚去除果柄部位。花和果荚均取3重复,后立即液氮速冻,置于-70 ℃超低温冰箱备用。
1.2 试验方法
1.2.1高通量测序及数据组装提取黄秋葵花、果荚总RNA,分别采用Nanodrop、Qubit2.0、Aglient 2100技术检测RNA样品的纯度、浓度和完整性等,构建cDNA文库,再分别使用Qubit2.0、Agilent 2100和Q-PCR对文库的质量进行检测。合格后,用Illumina HiseqTM2500进行测序。测序读长为PE125。获得原生数据,进行数据过滤,去除Reads中测序引物、接头等人工序列,用Trinity对有效数据(Clean Data)进行组装。
将测序Reads构建K-mer库,去除错误的K-mer;将高频率的K-mer作为种子向两端进行扩展,不断循环直到耗光K-mer库;再对Contig进行聚簇,得到片段集合(Component);对每个Component中的Contig构建De Bruijn图;De Bruijn图进行简化;解开De Bruijn图,获得转录本序列。
1.2.2基因FPKM值估计与差异表达分析采用Bowtie[17]将测序得到的Reads与单基因数据(Unigene)库进行比对,根据比对结果,结合RSEM[18]进行表达量水平估计。利用FPKM值(fragments Per Kilobase of transcript per Million mapped reads)[19]表示对应Unigene的表达丰度。使用EBSeq进行差异表达分析[20]。采用Benjamini-Hochberg方法对原假设试验得到的显著性P值进行校正,校正后P值,即伪发现率(false discovery rate)小于0.01且差异倍数(fold change, FC)≥2作为筛选标准[21]。其中,FC表示两样品(组)间FPKM的比值。
1.2.3差异基因的功能注释与代谢途径富集分析使用Blast软件将Unigene序列与NR、Swiss-Pro、GO、COG、KOG、KEGG数据库比对,再用KOBAS2.0得到Unigene在KEGG中的Orthology,预测氨基酸序列,使用HMMER软件与Pfam数据库分析,获得Unigene的注释信息。系统分析基因产物代谢途径及功能,并将差异表达基因(DEGs)对比到KEGG数据库,得到DEGs的代谢途径。
1.2.4类黄酮代谢关键差异基因分析利用注释信息检索法,对黄秋葵花、果荚类黄酮(黄烷酮、黄酮、异黄酮、黄酮醇、黄烷酮和花青素)关键词进行数据库检索,分析类黄酮物质关键基因在KEGG数据库功能注释及代谢路径。
1.2.5类黄酮代谢关键差异基因验证取 1 μg黄秋葵花或果荚的总 RNA,利用反转录试剂盒反转录成 cDNA,设计合成引物(表1),采用qRT-PCR检测黄秋葵花、果荚组织部位与类黄酮代谢相关差异表达基因,设置3个重复;统计 8个基因 (含 1 个内参) 在待测样品中的 Ct 值,计算相对表达量。
2 结果与分析
2.1 数据组装及分析
经高通量测序和质量控制,共获得15.26 Gb有效数据,其中黄秋葵花为7.12 Gb,果荚为8.14 Gb,碱基百分比均达到91.0%以上(表2)。数据质量良好,适宜后续分析。对组装结果进行统计(表3),转录本序列组装出154 721条转录本序列,平均长度为937.5 bp,N50长1 308 bp;由单基因序列(Unigene)组装出65 436条Unigene,平均长度为782.32 bp,N50长1 215 bp。
2.2 Unigene功能注释
经与NR、Swiss-Prot、KEGG、COG、KOG、GO和Pfam数据库的比对,对Unigene功能注释进行统计(表4),在65 436条单基因序列中,共获得39 245条Unigene的注释结果,占单基因序列总数59.97%。其中与COG数据库比对,获得9 366条同源序列,占单基因序列总数14.31%;与KOG数据库比对,获得20 535条同源序列,占单基因序列总数31.38%;与GO数据库比对,获得15 931条同源序列,占单基因序列总数24.35%;与KEGG数据库比对,获得14 110条同源序列,占单基因序列总数21.56%;与Pfam数据库比对,获得22 580条同源序列,占单基因序列总数34.51%;与Swissprot数据库比对,获得24 494条同源序列,占单基因序列总数37.43%;与NR数据库比对,获得38 905条同源序列,占单基因序列总数59.46%。
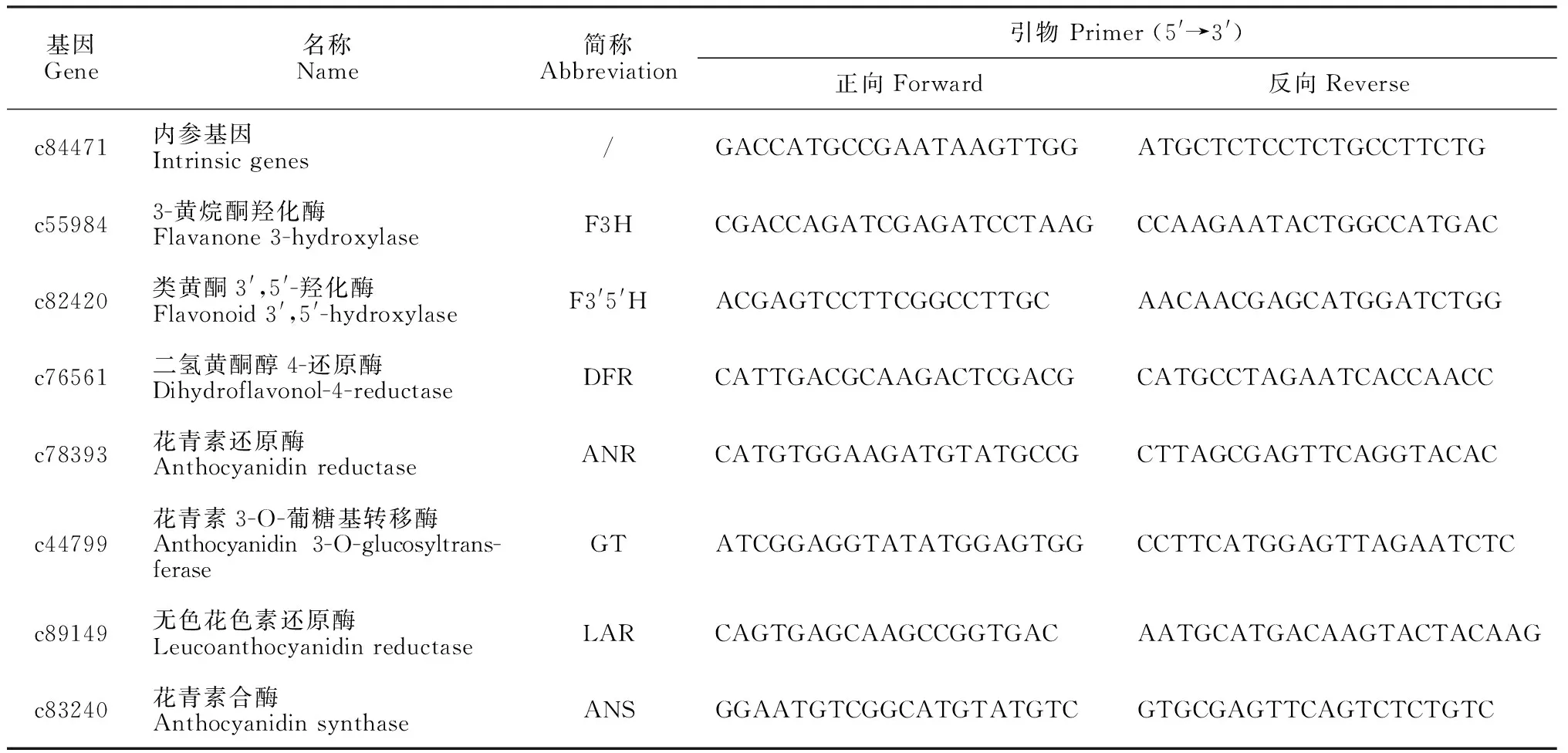
表1 实时荧光定量PCR引物

表2 有效数据评估统计
2.3 DEGs的筛选与功能注释
2.3.1DEGs筛选通过DEGs的筛选,获得黄秋葵花与果荚DEGs 1 336个,其中表达量上调的基因319个,下调基因1 017个;将DEGs单基因序列分别注释到COG、GO、KEGG、KOG、Pfam、Swiss-Prot、NR7大数据库,共有1 131个基因获得功能注释,其中GO数据库455个,COG数据库281个,KOG数据库472个,KEGG数据库372个,Pfam数据库844个,Swiss-Prot数据库807个,NR数据库1 123个,其中NR数据库注释比高最高,达99.29%。
2.3.2GO功能注释GO数据库分为3大类,分别为B生物学过程(biological process),C细胞组成(cellular component)和M分子功能(molecular function),分别用来描述基因编码产物所参加的生物过程、所具有的分子功能和所处的细胞环境等[22]。对黄秋葵花、果荚进行GO分类统计显示,455个DEGs被归到41个功能小类(表5)。生物学过程中DEGs“代谢过程”、“单一生物过程”和“细胞过程”3个功能小类占比最高;细胞组分过程中DEGs在“细胞组分”、“细胞”和“细胞器”3个功能小类占比最高;分子功能过程中DEGs在“催化活性”和“结合活性”2个功能小类占比最高。
2.3.3KOG功能注释将注释到KOG数据库的472个DEGs进行直系同源分类,获得23个功能分类,其中功能类别为R(一般功能预测),获得96个注释结果,占比20.33%;O(次生代谢产物生物合成、转运和代谢),获得67个注释结果,占比14.20%;T(信号转导机制)获得52个注释结果,占比11.01%;Q(次生代谢产物的合成、转运和代谢)获得44个注释结果,占比9.32%。另外在G(碳水化合物转运与代谢)、K(转录)也分别获得44和39个注释结果,分别占比9.32%和8.26%(表6)。
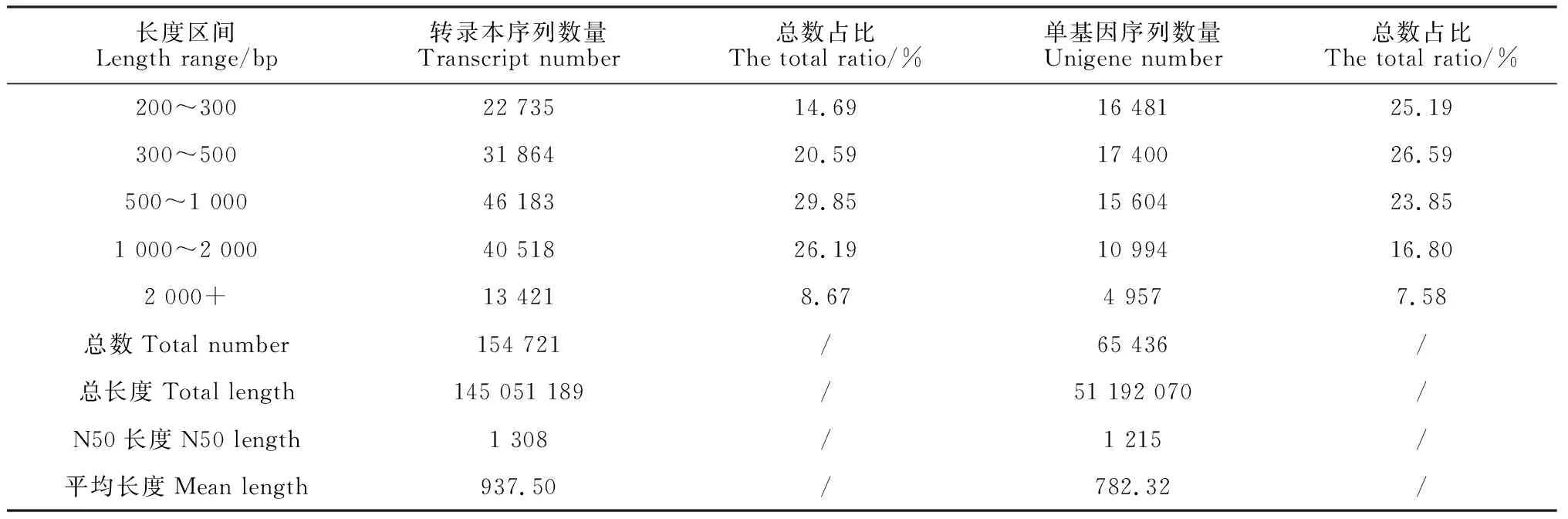
表3 组装结果统计分析


表5 差异表达基因GO功能注释
2.3.4KEGG功能注释将DEGs通过KEGG数据库比对(表7),有372条基因得到注释,分别富集在80条代谢通路,包括植物激素信号转导(39个)、淀粉和蔗糖代谢(33个)、戊糖、葡萄糖醛酸转换(25个)、氨基酸的生物合成(16个)、类苯基丙烷生物合成(14个)和碳代谢(14个)等22条代谢通路。本研究着重选择类苯基丙烷生物合成代谢通路,寻找类黄酮代谢差异发生的关键基因。
2.4 类黄酮代谢关键差异基因分析
据KEGG类黄酮代谢途径和表8数据分析,黄秋葵类黄酮合成代谢途径中,查尔酮合酶(chalcone synthase, CHS)和查尔酮-黄烷酮异构酶(chalcone isomaerase, CHI)基因在花、果荚中均无差异表达;3-黄烷酮羟化酶(flavanone 3-hydroxylase, F3H)、二氢黄酮醇4-还原酶(Dihydroflavonol-4-reductase, DFR)在黄秋葵花中具有显著表达优势;类黄酮3′,5′羧化酶(flavanone 3′,5′-hydroxylase, F3′5′H)、花青素还原酶(anthocyanidin reductase, ANR)、无色花色素还原酶基因(leucoanthocyanidin reductase, LAR)则在黄秋葵果荚中具有显著表达优势;而花青素苷合成酶(anthocyanidin, ANS)、葡萄糖基转移酶(glycosyl transferases, GT)基因则分别在花、果荚中具有显著表达。黄秋葵花、果荚中类黄酮代谢表现为:P-香豆素-CoA(P-coumaroyl-CoA)和3分子丙二醛-CoA(malnoyl-CoA),在CHS催化下,生成查尔酮(chalcone),查尔酮又在CHI作用下形成柚皮素(naringenin, NAR),此过程花与果荚均无显著差异。黄秋葵花中NAR在F3H催化下生产二氢山奈酚(dihydokaempferol, DHK),DHK在DFR作用下,生成无色天竺葵苷元,后在ANS(c83240)作用下生成花青素苷元,花青素苷元分别在GT(c44799/c82004)等转移酶的作用下,生成稳定的花青素苷;黄秋葵果荚则在F3′5′H作用下将NAR生成二氢杨梅素(dihydromyricetin, DHM),后在FLS催化下,进入黄酮醇代谢途径,部分DHM在DFR、ANS (c94125)、GT(c44799)作用下生成飞燕草素苷元(delphinidin),飞燕草素苷元在ANR作用下,进入原花青素代谢途径,无色飞燕草素苷元在LAR催化下也进入原花青素代谢途径。另外,GT、ANS在黄秋葵花和果荚中分别有3个和2个差异表达拷贝,且表达量在不同组织中具有显著互补性。

表6 差异表达基因KOG功能注释

表7 差异表达基因KEGG功能注释
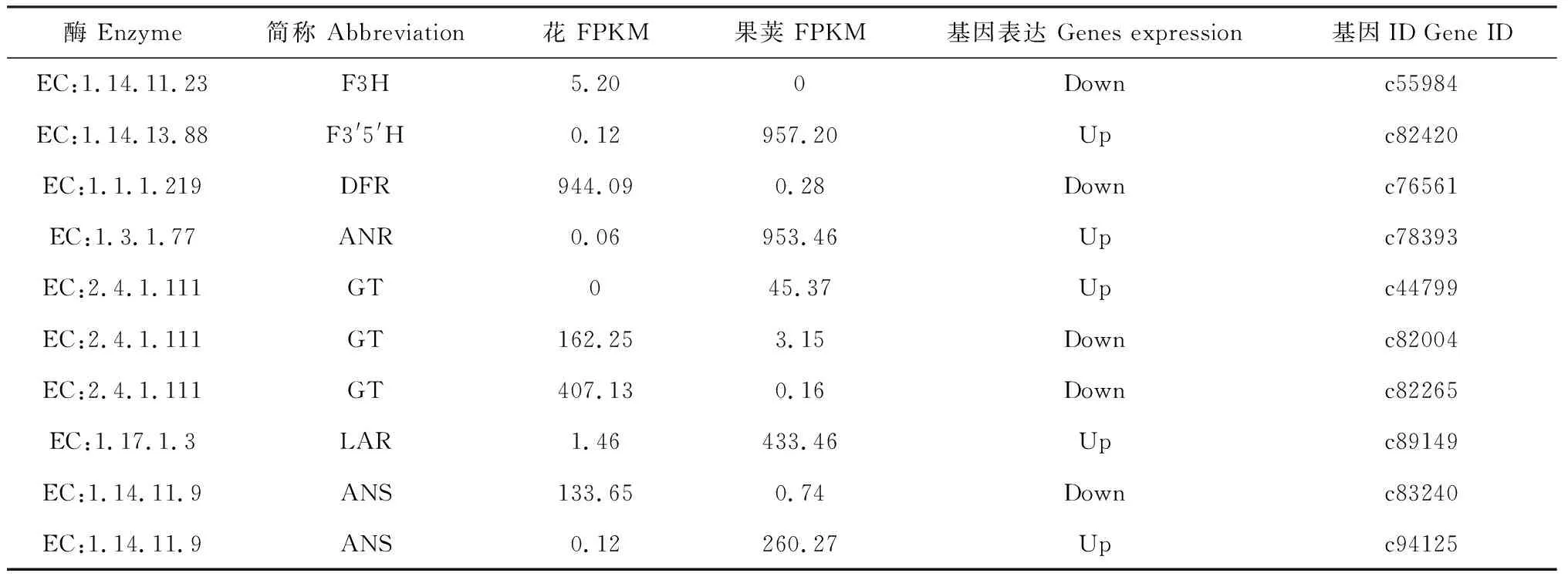
表8 黄秋葵花、果荚类黄酮代谢关键基因差异表达情况
2.5 类黄酮代谢关键差异基因验证分析
将表10中部分DEGs进行荧光实时定量(RT-PCR),其中GT和ANS基因随机选取多拷贝中1个,共7个。差异表达基因中c82420、c78393和c44799 3个基因表达量上调;c55984、c76561、c89149和c83240 4个基因表达量下调,以黄秋葵c84471基因为内参基因, 进行qRT-PCR验证。由图1可知,7个基因qRT-PCR分析得到的相对表达量与转录组表达谱分析趋势完全一致, 但表达的变化大小存在一定差异,说明基因表达谱的分析结果基本可靠,其中DFR和ANR基因分别特异性在黄秋葵花、果荚中表达,且表达量差异极大。

1.c55984;2.c82420;3.c76561;4.c78393;5.c44799;6.c89149;7.c94125图1 差异表达基因qRT-PCR相对表达Fig.1 The relative expression levels of DEGs by qRT-PCR
3 讨 论
通过Illumina HiSeqTM2500测序技术构建黄秋葵花、果荚转录组数据库,花和果荚分别获得7.12 Gb和8.14 Gb 有效数据,碱基百分比(Q30)均达到91.0%以上。对Unigene进行功能注释,在65 436条单基因序列中,共获得39 245条Unigene的注释结果,占单基因序列总数59.97%。通过DEGs分析,获得差异基因1 336个,其中上调基因319个,下调基因1 017个。获得功能注释基因有1 131个,GO数据库将455个DEGs归到41个功能小类,代谢过程、催化活性、单一生物过程和细胞过程等4个功能小类占比最高;KOG数据库将472个DEGs进行直系同源分类,获得23个功能分类,其中与次生代谢直接相关过程O和Q类别获得111个注释结果,合计占比23.52%;通过KEGG数据库比对,将372个DEGs注释到80条代谢通路上,其中富集在类苯基丙烷生物合成途径有14个DEGs,再通过类黄酮关键基因分析,共获得10个关键差异基因。
植物类黄酮合成代谢是目前研究最清楚的此生代谢路径之一[23],该通路上存在2个重要基因群,即上游基因群(early biosynthetic genes, EBGs)与下游基因群(late biosynthetic genes, LBGs)。EBGs主要包括 CHS、 CHI、F3H、F3′H、F3′5′H等基因[24-25];LBGs主要包括 DFR、 FLS、 ANS、ANR、 GT、酰基转移酶(Acyl transferase, AT)和甲基转移酶(Aethyl transferase, MT)等[26]。本研究发现,前体合成阶段关键酶(CHS、CHI)均无显著差异表达,这与CHS 和CHI基因编码区和结构都十分保守,在不同科植物间、不同组织部位上均具有较高的保守性相一致[27]。黄秋葵花、果荚类黄酮代谢路径中发现F3H、F3′5′H、DFR、ANR、ANS、LAR和GT存在差异表达,其中F3H、DFR在黄秋葵花中表现上调效应,F3′5′H、ANR、LAR在黄秋葵果荚中表现显著上调效应,ANS、GT则分别在花和果荚中均有上调或下调效应。NAR作为类黄酮合成的关键分支点,黄秋葵花通过F3H、DFR、ANS(c83240)、GT(c82265/c82004)途径,生成紫红色花葵素-3-葡萄糖苷(pelagonidin-3-glucoside)[28],这与黄秋葵花喉深红色表现可能有关。黄秋葵果荚则通过F3′5′H、FLS催化下,将NAR生成杨梅素(Myricetin)[29]等;部分NAR在F3H、DFR、ANS、LAR作用下,进入花青素苷元(表焙儿茶素)和原花青素(没食子儿茶素)合成途径。初步推断,黄秋葵花中富含花青素苷(葵素-3-葡萄糖苷)成分,果荚富含黄酮醇(杨梅素等)、花青素苷元(表焙儿茶素)原花青素(没食子儿茶素等)等类黄酮物质。另外ANS和GT基因在黄秋葵花、果荚中均存在多拷贝和底物特异性现象[30],推断GT(c44799)、GT(c82004)在黄秋葵花中特异性的催化无色天竺葵素苷元,GT(c44799)在黄秋葵果荚中特异性的催化无色飞燕草苷元;ANS分别在花和果荚里高表达,ANS(c83240)可能与黄秋葵花瓣颜色(黄色)有关,ANS (c94125)可能与果荚颜色(浓绿色)有关,具体GT、ANS特异性底物种类、产物及最终呈色关系还需要进一步功能验证。
利用高通量转录组测序技术,深入挖掘黄秋葵不同组织部位DEGs,从宏观上理清黄秋葵花、果荚差异基因及调控机理等。近年来,黄秋葵营养成分研究在国内越来越受到重视,特别是黄酮提取[31]和功能分析[32]逐渐成为研究热点,备受食品、医药等行业青睐。本研究丰富了黄秋葵花、果荚转录组水平生物数据信息,为类黄酮等关键基因克隆和功能验证等提供遗传基础。

